转Cry1Ab/Cry1Ac基因大米粉饲喂印度谷螟后的比较转录组分析
唐培安,陶冶心,王康旭,吴学友,陈二虎
(南京财经大学 食品科学与工程学院,江苏高校粮油质量安全控制及深加工重点实验室,江苏 南京 210023)
印度谷螟Plodia interpunctella(Hübener)是一种全球范围内广泛发生的储粮害虫,不仅能够通过直接取食对粮食造成损失,其生命活动过程还可以提高储粮温度,从而引起粮堆霉变和品质劣变。苏云金芽孢杆菌Bacillus thuringiensis(Bt)是一种革兰氏阳性细菌,能够产生具有杀虫效果的晶状蛋白(cry),对鳞翅目和鞘翅目害虫具有明显的防治效果[1]。随着转基因技术的不断发展,利用转基因进行 cry蛋白异源表达的农作物越来越多,目前广泛用于田间生产的有转基因棉花,主要用于防治棉铃虫。另外,转基因水稻也可以用于二化螟、大螟和稻纵卷叶螟等害虫的防治,同时,研究人员发现转基因稻谷对印度谷螟也有不错的防效[2]。在Bt抗虫研究领域还存在几个亟需回答的问题,一是 cry蛋白的毒性机理还未有定论,目前存在孔道假说和信号传导假说两个潜在的致死机制。另外,研究表明对印度谷螟进行室内筛选可以使试虫种群获得Bt抗性,但是相关的抗性发生机理尚未得到阐明[3]。
高通量测序技术,是核酸测序中最常用的工具,其标志就是能一次并行对几十万到几百万条基因序列进行测定。在高通量测序技术的迅猛发展下,生物学问题可以通过基因表达差异分析、突变分析等手段进行解决。在昆虫学研究领域,高通量测序可以用于进化发育、生理生化以及毒理学等方面的研究。但是,印度谷螟应用高通量测序技术的报道很少,相关基因数据有限。本研究通过对长期饲喂转基因大米粉和常规大米粉的印度谷螟种群进行转录组高通量测序(Illumina HiSeqTM 2000),组装后分别得到了41 597和42 306条潜在基因序列,注释其中的关键基因,最后比较分析差异表达基因。本研究一方面大大扩充了该物种的基因信息,另一方面也为转基因稻谷对印度谷螟乃至其他害虫的抗虫机理提供新的见解。
1 材料与方法
1.1 供试印度谷螟
本试验所用的印度谷螟源来源于江苏省吴江市,后经实验室多代饲养。其中,非胁迫品系(SS)在实验室条件下饲养20代以上((30±0.5) ℃、相对湿度70%~80%,无光照),饲料为正常米粉(不含Bt毒素)。从非胁迫品系中随机选择部分试虫用含有2%转基因米粉的混合饲料饲养,筛选培养5个月后,视该种群为胁迫种群(RS)。
试虫非胁迫品系(SS)饲料为:明恢63(普通稻谷)大米粉;试虫胁迫品系(RS)饲料为:明恢63(普通稻谷)和华恢1号(Cry1Ab/Cry1Ac转基因稻谷)大米粉混合物。上述两种稻谷均来自华中农业大学作物遗传改良国家重点实验室。
1.2 RNA制备以及cDNA文库构建
以3龄幼虫作为供试对象,提取RNA后送测。总RNA提取按照RNeasy Plus Universal Mini Kit(Qiagen)试剂盒说明书进行,用DNase I (Takara)去除基因组杂质,测定RNA浓度,并通过1.5%琼脂糖凝胶电泳检测RNA完整性。然后,使用华大基因HiSeq TM 2000 (Illumina)构建cDNA文库并进行测序。
1.3 生物信息学分析
筛选剔除低质量序列后以高质量的 reads进行生物信息学分析。首先,Trinity组合某一重叠长度的reads以形成更长的contigs片段[4];然后,将reads进行末端读数配对,确定contigs间的差异情况;最后,利用 Trinity将 contigs连接成没有Ns的unigene序列。将组装的unigenes与NR、Swiss-Prot、KEGG和COG数据库进行比对(设置临界 E值<10–5)[5]。Unigene序列结果全部提交至 NCBI SRA(登录号分别为 SRP060836,SRP060680)。
1.4 差异Unigenes分析
利用RPKM方法(每kb每百万读数)来鉴定两个样品(RS和SS)之间的基因表达差异[6],同时使用FDR方法来确定P值的阈值[7]。基因显著差异表达的阈值为“FDR≤0.001和 log2比≥1的绝对值”。最后以GO富集和KEGG代谢通路深入分析转录组数据[8]。
1.5 显著差异基因的GO分析
将DEG对应GO数据库,计算GO项基因数,分析GO功能差异,计算公式如下[10]:
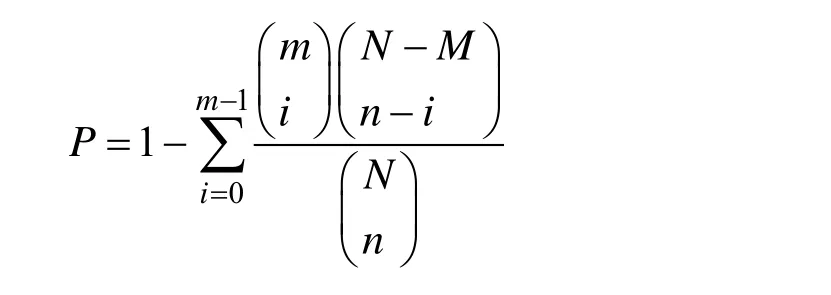
N:KEGG基因数;n:N中DEG数;M:特定途径基因数;m:M中的DEG数。
1.6 显著差异基因的代谢通路分析
通过公共代谢路径相关数据库KEGG本体富集分析进一步了解显著差异基因的生物学功能[9],计算公式同上[10]。
2 结果与分析
2.1 转录组数据De novo组装
我们从胁迫种群(RS)和非胁迫种群(SS)中均获得超过 51(百万)的 reads数,且Q20>98%,测序质量较好,深度和覆盖率结果如图 1所示。之后,通过 Trinity软件组装 reads,共分别获得54 648和54 647个contigs,平均长度分别均在600 bp以上。之后再进行unigenes组装,RS-Unigenes和SS-unigenes个数分别为41 597和42 306个,平均长度均超过1 000 bp,另外还获得 37 246个All-unigenes(表 1)。

表1 测序产量统计

图1 不同种群Unigene的分布情况
2.2 Unigene功能注释与COG分类
在37 246个unigenes中有23 310个unigenes可以被注释,其中有22 211个unigenes在NR数据库中可以被注释。通过NR数据库中的E值分析,20.5%的注释序列E值小于1.0E–100,15.5%注释序列E值范围在1.0E–100到1.0E–60之间,表明二者之间具有强同源性(表2)。序列相似性分布表明,26.1%的序列之间有较高的同源性,而另外73.9%的序列同源性介于17%~80%。另外,与印度谷螟相似度最高的物种是帝王蝶(American monarch, 67.60%),其次是家蚕(Bombyx mori, 6.5%)和赤拟谷盗(Tribolium castaneum, 3.4%)(图 2)。

表2 注释结果统计

图2 Nr注释的物种分布
2.3 注释基因功能研究
10 694个序列可以注释到 GO的 58个功能组,主要包括:生物过程,细胞组分和分子功能(图3)。同时,细胞加工(cellular process)(6 665,62.32%)、细胞组分(cell)(5 063, 47.34%)和催化活性(catalytic activity)(5 101, 47.7%)为主。

图3 Unigene的GO分类图
共9 348条unigenes注释到COG并分为25个功能类别(图 4)。其中,最大的聚类组是“一般功能基因”(3 662, 39.17%),其次是“复制、重组和修复基因”(1 575, 16.85%)以及“翻译、核糖体结构和生物发生”(1 519, 16.25%)。而其它类别基因较少,如“细胞外结构”(92, 0.98%),“RNA加工和修饰”(88, 9.41%)和“核结构”(5,0.05%)。

图4 COG功能注释
KEGG注释结果显示,15 750条unigenes被注释到258个KEGG通路。其中,以新陈代谢通路、肌动蛋白细胞骨架调节通路为主,另外有少数的 unigene被注释到了生物素代谢和赖氨酸生物合成通路。
2.4 不同品系印度谷螟基因表达分析
对印度谷螟胁迫品系与非胁迫品系的差异基因进行比较分析发现,有15 741条基因上调,18 725条基因下调(图5)。进一步利用阈值(FDR≤0.01)和log2Ratio (≥1)分析发现,两个种群中差异极显著的基因共有10 224条,其中4 520条基因上调,5 704条基因下调;除此之外,有1 245条基因在胁迫品系中表达,同时,有503条基因只在非胁迫种群中表达(图6)。

图5 印度谷螟不同品系差异表达的基因

图6 印度谷螟不同品系显著差异表达基因
2.5 印度谷螟解毒和Bt毒素受体相关基因分析
结果表明,有 87条羧酸酯酶和谷胱甘肽-S-转移酶的相关基因在胁迫和非胁迫种群中具有表达差异,其中60.91%的基因在胁迫品系中表达量上调(表3)。
另外,我们还对不同种群试虫的Bt毒素受体相关的基因进行了比较分析。结果表明,有78条注释为氨肽酶–N,碱性磷酸酶,钙黏蛋白,糖脂类的显著差异基因在两个品系中被发现,其中有55.13%的基因在胁迫品系中上调。值得注意的是,所有注释为 APN的基因在胁迫品系中都显著上调。之后,我们对筛选出来的APN基因进行了系统发育树构建,6个APN基因能够划到4个相关家族,与家蚕、小菜蛾、烟草天蛾等鳞翅目昆虫的APN蛋白序列相似度较高(图7)。

表3 印度谷螟胁迫和敏感品系中差异显著的羧酸酯酶和谷胱甘肽-S-转移酶基因

图7 印度谷螟和其他鳞翅目昆虫中APN基因系统发育分析
3 讨论
印度谷螟危害严重且全球性分布,是转基因水稻的主要靶标害虫之一。过去几年,和Bt抗性机制相关的解毒代谢机制以及毒素受体被很多人研究[11-12]。然而印度谷螟对Bt毒素的抗性机理尚不清晰。本研究中,我们用二代测序技术对印度谷螟3龄敏感及经转基因大米粉饲喂过的胁迫品系进行转录组比较分析,以研究试虫对Bt的相关毒理学机制。通过测序,总计产出超过100万nt数据,这些转录数据经过过滤被组装为Unigenes,并注释到NR,NT,KEGG,COG和GO数据库,其中,注释到NR中的Unigenes最多,有22 211个,同时发现和印度谷螟最相似的物种是帝王蝶(67.6%),其次是家蚕(6.5%)。差异表达基因分析结果发现,约有46%的差异表达基因在胁迫品系中显著上调。大量研究表明,氨肽酶-N、碱性磷酸酶、钙黏蛋白、糖脂类可能是Bt毒素的识别受体[13-14]。在本研究,我们发现9个编码APN的基因,这些基因在胁迫品系中的表达量明显升高,另外还发现 55个编码 Cadherin的基因,10个编码ALP和4条编码Glycolipid的基因,但这些受体基因在不同品系中的表达量无显著差异。由此可以推测,印度谷螟对转基因大米粉的毒理学机制和APN受体密切相关。另外,我们的另一项针对取食转基因大米粉后的印度谷螟体内抗氧化酶的活力动态进行了深入探索,这将与本研究一起为后续的相关研究提供了坚实的理论依据[15]。
本研究通过二代测序技术对印度谷螟转录组进行了解析,同时利用生物信息分析方法对不同转基因大米粉饲喂种群的表达差异基因进行了比较研究,研究结果将有利于后续基因功能验证和印度谷螟的生物学相关研究,为储粮害虫防治提供了重要的理论依据和参考资料。
备注:本文的彩色图表可从本刊官网(http://lyspkj.ijournal.cn/ch/index.axpx)、中国知网、万方、维普、超星等数据库下载获取。

