全基因组数据分析工具TB Profiler v2.8.0、Mykrobe v0.7.0和PhyResSE v1.0在耐药结核病检测中的价值
李冰莹 郑旭彬 胡屹 徐飚
耐多药结核病(MDR-TB)流行给全球结核病控制带来了严峻挑战。中国是结核病高负担国家,耐药率也高于全球平均水平[1]。传统药敏试验(drug-susceptibility testing, DST)通常被视为耐药结核病诊断的金标准,但该方法耗时较长[2]。GeneXpert、线性探针等分子诊断技术虽能快速诊断耐药性,但仅能检测少数药品最常见的耐药突变位点,无法提供全面的抗结核药品耐药信息[3-4]。随着高通量测序技术的发展和测序成本的下降,全基因组测序(whole-genome sequencing, WGS)技术为结核病耐药检测提供了新的手段。WGS可以在全基因组水平上同时检测耐药基因的变化信息,受到了世界卫生组织的肯定,有广阔的应用前景[5-6]。然而WGS产生的数据量庞大,需要运用复杂的生物信息学才能得到耐药结果,给临床实际应用带来挑战[7]。因此,WGS的临床应用必须要借助高效、自动化的信息平台,从而使非生物信息学专业的临床人员可以常规使用。
最近,研究人员开发了几款针对结核分枝杆菌的自动化分析工具,只需导入原始测序数据即可得到药敏试验结果,克服了WGS在临床使用上的限制[8-10]。为了指导临床诊断和治疗,这些分析工具必须要有高度的准确性。但是一方面,已有的研究结果并不一致,芬兰的研究表明,TB Profiler v2.8.0、Mykrobe v0.7.0和PhyResSE v1.0(以下简称“TB Profiler、Mykrobe和PhyResSE”)诊断MDR-TB的敏感度从74%~80%不等,而一项2019年的研究则显示诊断MDR-TB的敏感度在90%左右[11-12]。另一方面,不同地区主要流行菌株不同,耐药相关基因突变频率和类型也不同,会导致耐药检测的准确性有差异。因此,本研究使用TB Profiler、Mykrobe和PhyResSE这3种工具对534株中国临床分离菌株进行耐药检测,以更好地评估这些工具的诊断性能。
资料和方法
一、数据来源
在PubMed中利用“whole genome sequencing”“Mycobacteriumtuberculosis”“drug resistance”和“China”等关键词进行组合检索,筛选2019年5月1日前符合以下纳入标准的研究:(1)对4种一线抗结核药品和至少2种二线抗结核药品进行了DST;(2)至少对10株结核分枝杆菌菌株进行了测序;(3)均为来自中国的结核分枝杆菌临床分离菌株;(4)能批量下载测序数据和DST结果。最终筛选得到符合标准的2项研究,并从美国国立生物技术信息中心核酸数据库(National Center for Biotechnology Information Sequence Read Archive, NCBI SRA)中下载了这2项研究所上传的534株结核分枝杆菌的全基因组序列[13-14]。534株结核分枝杆菌菌株均为来自中国的临床菌株,表型药敏试验显示,457株为耐药菌株,77株为敏感菌株。
二、软件工具
评估的3种结核分枝杆菌全基因组数据分析工具分别是TB Profiler v2.8.0(http://tbdr.lshtm.ac.uk/)、Mykrobe v0.7.0(https://www.mykrobe.com/)和PhyResSE v1.0(www.phyresse.org)。下文中所涉及的TB Profiler、Mykrobe和PhyResSE均与此处版本相同。这3种工具都不需要使用命令行界面和编程语言,只需把原始测序数据上传到网页或导入软件即可检测所有一线抗结核药品和大多数二线抗结核药品的耐药性,具体药品目录见表1。
TB Profiler、Mykrobe和PhyResSE操作简单,给出的耐药检测结果简单易懂,并且给出了菌株家系信息(表2)。在处理时间方面,一个100倍测序深度的样本(约450兆)使用网页版TB Profiler和 PhyResSE从上传完成到得到耐药结果需要约90 min,而使用软件版Mykrobe则速度较快,只需要约30 min。在批量操作方面,PhyResSE网页版提供批量上传,TB Profiler和Mykrobe只能在命令行版本批量分析数据。此外,TB Profiler和Mykrobe可以根据用户个性化需求修改使用的突变数据库,从而纳入新发现的耐药突变,不断提高检测的准确性。TB Profiler在命令行版本中通过编辑源代码来修改突变数据库,Mykrobe在命令行版本中通过使用Python代码生成器进行调整。
三、评价指标
以DST结果为金标准,使用统计软件R(版本3.1.0)计算敏感度、特异度,包括相应的95%置信区间。
结 果
一、TB Profiler、Mykrobe和PhyResSE对一线抗结核药品耐药的检测效能
以DST结果为参照标准,Mykrobe和PhyResSE检测异烟肼耐药的敏感度分别为76.42%(350/458)和76.20%(349/458),略高于TB Profiler(69.43%,318/458),三者特异度几乎相同(表3)。TB Profiler、Mykrobe和PhyResSE检测利福平耐药的敏感度和特异度均较高,敏感度分别为90.81%(415/457)、87.75%(401/457)和90.81%(415/457),特异度分别为97.40%(75/77)、96.10%(74/77)和96.10%(74/77)。对于乙胺丁醇,TB Profiler、Mykrobe和PhyResSE的敏感度相近,分别为81.61%(213/261)、79.31%(207/261)和79.69%(208/261),三者特异度也相近,在83%左右。对于吡嗪酰胺,TB Profiler的敏感度(72.82%,150/206)高于Mykrobe(61.65%,127/206)和PhyResSE(50.97%,105/206),三者特异度相近,均高于92%。
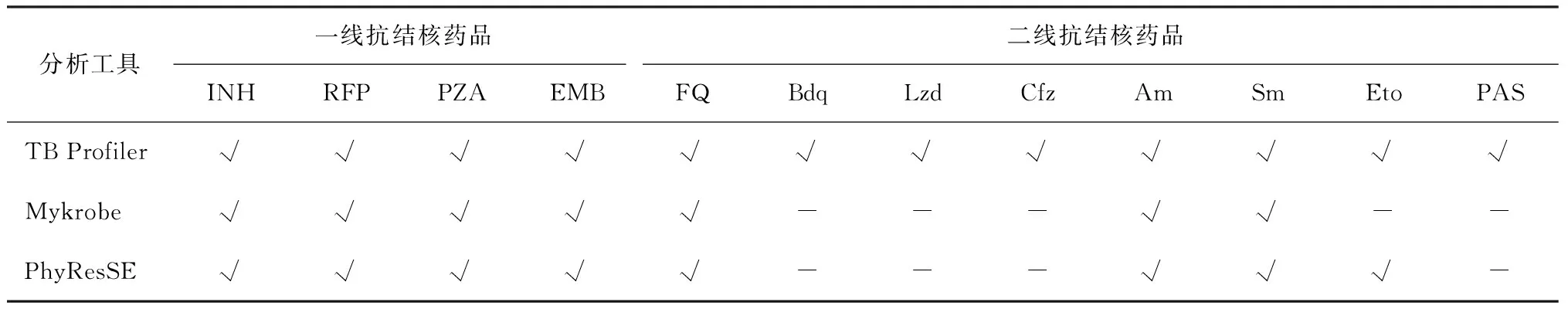
表1 3种全基因组数据分析工具检测药品种类情况
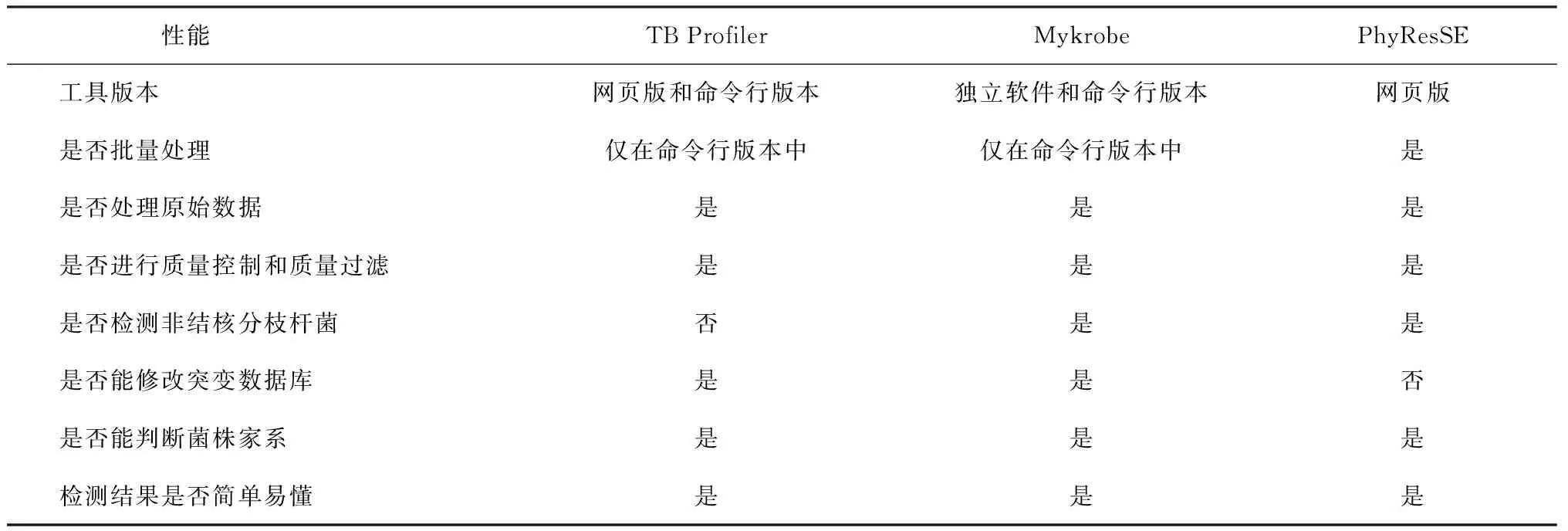
表2 不同性能在3种全基因组数据分析工具间的比较
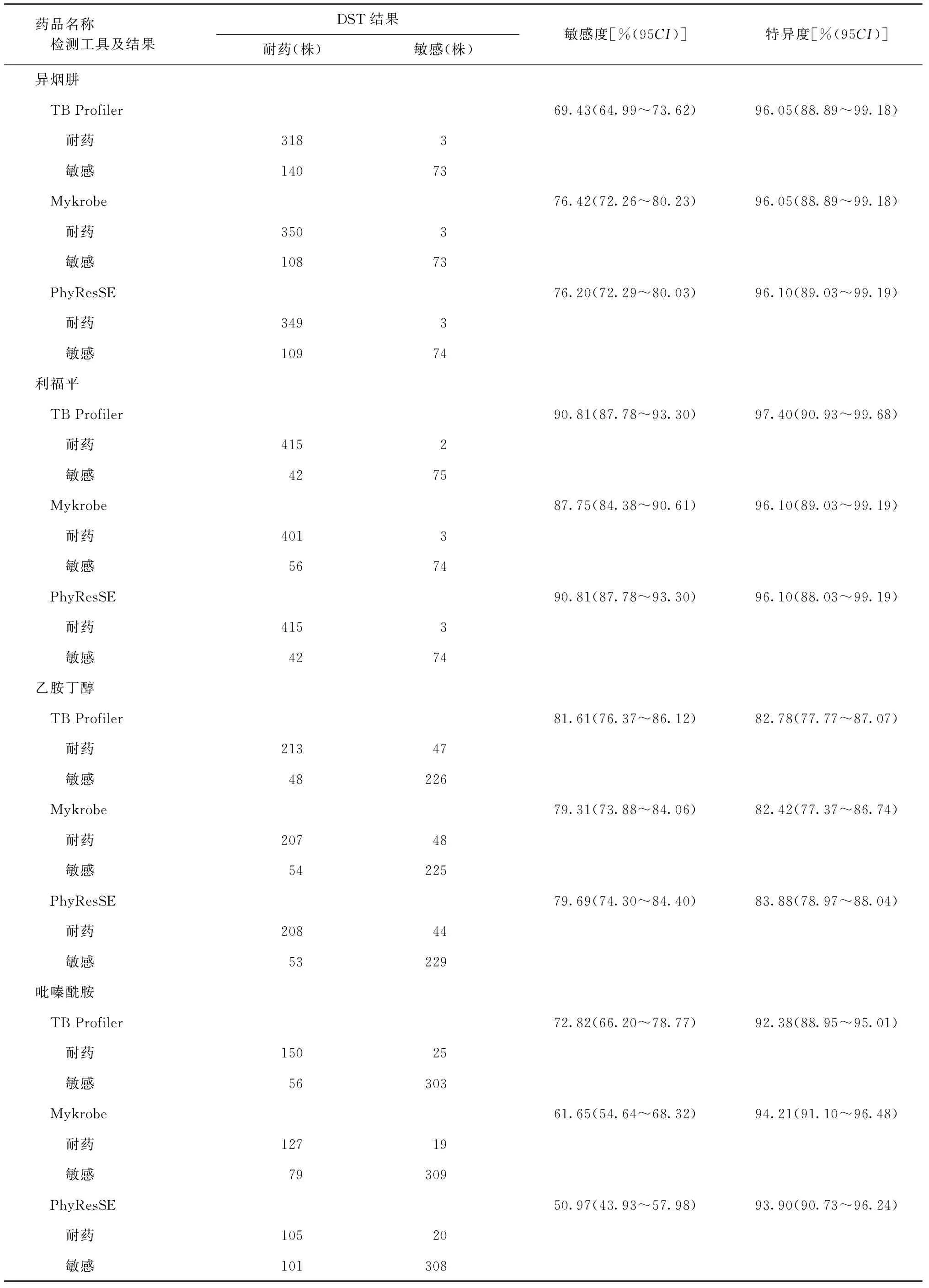
表3 以DST为金标准判断TB Profiler、Mykrobe和PhyResSE对一线抗结核药品耐药的检测效能
二、TB Profiler、Mykrobe和PhyResSE对二线抗结核药品耐药的检测效能
TB Profiler、Mykrobe和PhyResSE检测二线抗结核药品耐药的特异度均较高,而敏感度在不同药品和工具之间差异较大(表4)。PhyResSE检测氟喹诺酮类耐药的敏感度为88.27%(143/162),略高于TB Profiler(81.48%,132/162)和Mykrobe(82.10%,133/162)。对于阿米卡星,PhyResSE的敏感度为60.00%(27/45),略高于TB Profiler(48.89%,22/45)和Mykrobe(55.56%,25/45)。TB Profiler、Mykrobe和PhyResSE检测耐链霉素的敏感度相近,分别为78.77%(256/325)、76.00%(247/325)和79.38%(258/325)。TB Profiler、Mykrobe和PhyResSE检测氟喹诺酮类和阿米卡星耐药的特异度均高于95%,检测耐链霉素的特异度略低,分别为88.89%(184/207)、89.37%(185/207)和90.34%(187/207)。根据DST结果,本研究纳入的菌株中有3株耐贝达喹啉、12株耐利奈唑胺和35株耐氯法齐明,TB Profiler对这3种药品进行了耐药检测,但只检测了Rv0678和rrl基因上少数突变位点,没有检出耐药菌株。
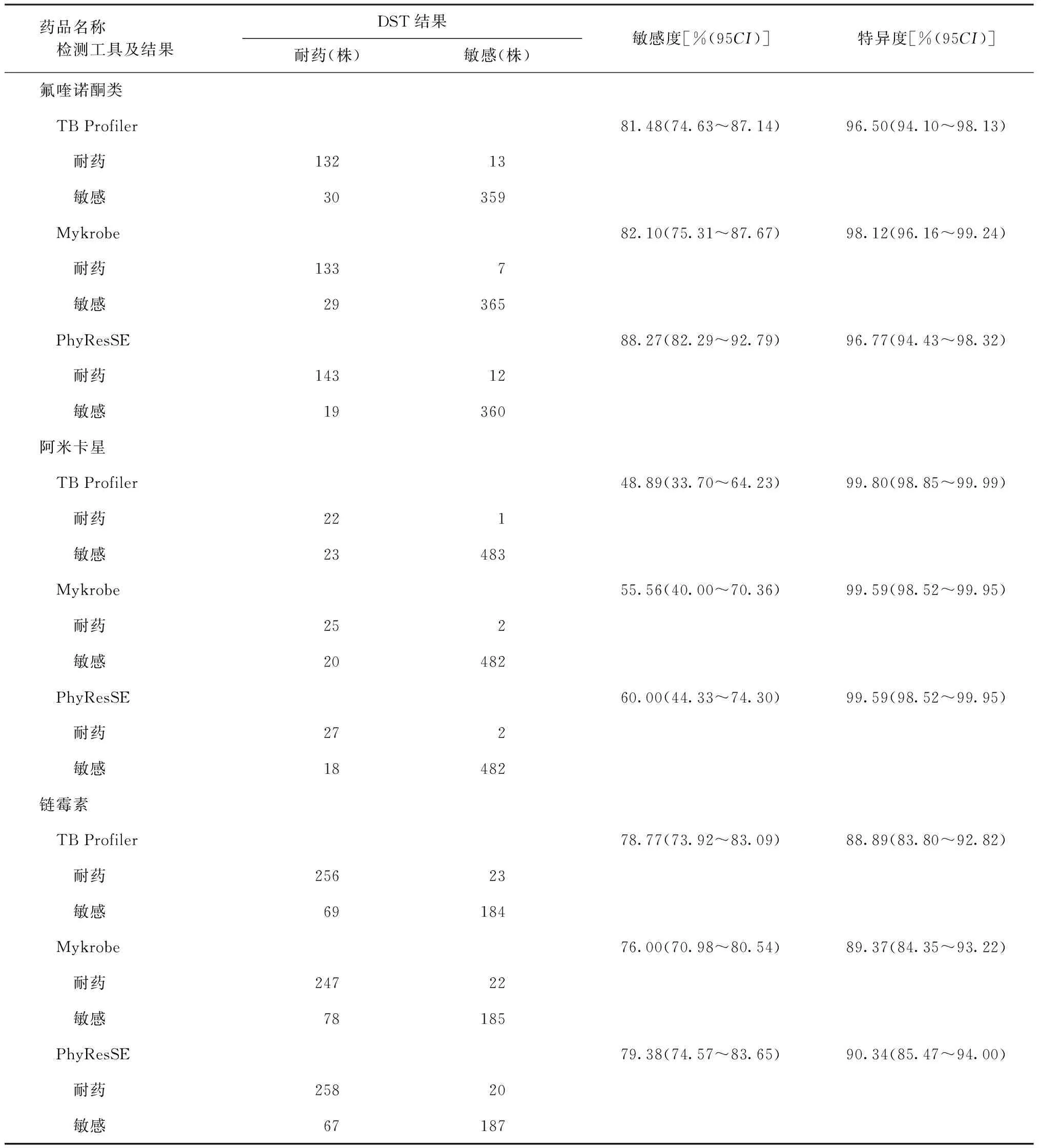
表4 以DST为金标准判断TB Profiler、Mykrobe和PhyResSE对二线抗结核药品耐药的检测效能
讨 论
快速检测结核分枝杆菌耐药性可以指导临床治疗、改善患者预后、降低传播风险,对于控制结核病至关重要。使用WGS可以在5 d左右获得耐药结果,显著减少了耐药诊断所需的时间,具有良好的发展潜力[11]。但是WGS数据处理复杂,不利于临床人员使用,WGS的临床应用必须要借助高效、自动化的数据分析工具。本研究评估了TB Profiler、Mykrobe和PhyResSE这3种针对结核分枝杆菌开发的全基因组数据分析工具的检测性能,以利于WGS技术在结核病中的应用。结果显示,TB Profiler、Mykrobe 和PhyResSE总体性能良好,检测各种药品耐药的特异度均较高,但敏感度在不同的药品和工具之间差异较大,这与先前的研究结果相一致[11,15-16]。总体而言,一线抗结核药品(PZA除外)、氟喹诺酮类药品和链霉素的耐药检测敏感度较好,其余药品敏感度较差。
TB Profiler、Mykrobe和PhyResSE检测吡嗪酰胺耐药的敏感度较低的原因不是工具算法无法检测到菌株的遗传变异,而是由于分析时只纳入了早期研究发现的耐药有关的点突变,但是近期研究发现插入与缺失也是吡嗪酰胺耐药的重要机制[17]。一项日本的研究表明,通过手动修改TB Profiler等工具的突变数据库,增加吡嗪酰胺耐药有关的插入和缺失,可以显著提高检测的准确性[18]。手动修改突变数据库不适用于临床环境,因此需要开发者加快更新速度,及时调整默认的突变数据库,不断提高耐药检测的准确性。但是由于缺少持续的资金支持,这些工具的维护和完善面临挑战。例如,KvarQ是一款2014年开发耐药检测软件,但自从开发之后未曾进行过更新维护,影响用户使用[19]。因此,可考虑采取可持续的商业模式,以确保这些工具的临床实用性[16]。
TB Profiler、Mykrobe和PhyResSE中只有TB Profiler对贝达喹啉、利奈唑胺和氯法齐明进行了耐药检测,但是却没有检测出耐药菌株。这主要是由于贝哒喹啉等药品的耐药机制尚不清楚,TB Profiler检测时只纳入了Rv0678和rrl基因上少数突变位点,检测效果差。这提示对于贝达喹啉等敏感度表现不佳的药品首先要加强耐药机制研究,获得高质量的耐药有关突变,才能提高耐药检测的准确性。此外,值得注意的是WGS测序深度也会影响耐药检测的敏感度。当菌群中存在敏感菌和耐药菌混合感染时,表型药敏试验能检测出1%菌群比例的耐药,而WGS检测限值与测序深度有关,测序深度较浅时可能会将混合感染的样本诊断为敏感[20]。
本研究首次针对性地纳入中国结核病患者的临床分离菌株以更好地评估TB Profiler、Mykrobe和PhyResSE的诊断性能。但本研究也存在一定的局限性。首先,贝达喹啉、利奈唑胺和氯法齐明耐药菌株数量较少,可能会影响敏感度评估的准确性。其次,本研究中耐药菌株占比高,不能反映耐药率水平,因此,无法估计阳性预测值和阴性预测值。
本研究证实了TB Profiler、Mykrobe和PhyResSE这3种针对结核分枝杆菌开发的全基因组数据分析工具总体性能良好,对于指导临床用药有较高价值。但目前还面临吡嗪酰胺和二线药品检测敏感度低等限制,离大规模临床应用尚有一段距离,有望在将来彻底改变耐药结核病的快速临床诊断。为了确保这些工具的临床实用性,还需要加强抗结核药品耐药机制研究(尤其是吡嗪酰胺和二线药品),提高耐药突变数据库的质量,从而提高检测的准确性。

