基于改进的残差神经网络的白细胞分类
马 力,帅仁俊+,刘文佳,李文煜
(1.南京工业大学 计算机科学与技术学院,江苏 南京 211816;2.南京医科大学附属医院南京鼓楼医院 消化内科,江苏 南京 211166)
0 引 言
在临床医学上,人体内血液主要由红细胞、白细胞和血小板组成[1]。其中白细胞在人体免疫系统中发挥重要作用,与许多疾病密切相关[2]。根据白细胞细胞质的颗粒信息和形状信息将白细胞分为5类:中性粒细胞、嗜酸性粒细胞、嗜碱性粒细胞、单核细胞和淋巴细胞[3]。不同种类的白细胞的总数和比例都对应着不同的疾病,同时也是临床医生诊断和治疗疾病的重要参考资料,因此需要对白细胞更加快速准确地进行分类。
传统的白细胞分类方法主要是血液学专家将血细胞染色并在光学显微镜下对它们进行识别计数[4]。这种方法工作量大,效率低,对专业人员素养要求高,而且分类和计数的结果易受人为因素的影响[2]。白细胞分类是一个热点问题,很多专家学者都进行了这方面的研究。随着计算机技术的发展,通过机器学习建立的分类模型被广泛用于白细胞分类[5]。目前主要包括K-近邻(KNN)算法[6]、支持向量机算法(SVM)[7]、随机森林算法[8]、贝叶斯算法[9]。
近年来随着计算机性能和人工智能的快速发展[10],深度学习技术得到了广泛的应用,并在图像处理方面取得了很大的进展[11,12]。深度神经网络在处理数百万图像数据方面显示出了良好的优势,并且可以更准确地检测,识别图像中的对象和区域[13]。很多专家学者利用神经网络对白细胞图像进行特征提取,然后进行识别分类,取得了不错的效果[14,15]。
通过目前的研究结果可以发现,无论是利用机器学习还是深度学习来对白细胞分类,其使用的样本量很少或者使用自己的数据库,而且利用机器学习建立的模型需要在分类之前对白细胞图像进行分割预处理,分割算法的质量直接影响提取特征的质量最终影响分类的效果。为了解决上述问题,本文提出了一种白细胞分类框架。图1显示了模型的流程图。通过迁移学习来提高模型的鲁棒性。改进的残差神经网络ResNet(residual neural network)架构可以提高训练的速度,加强模型的泛化能力。改进的损失函数采用硬样本挖掘战略,过滤容易分类的样本,缩小同类别样本的差距,增大不同类别的差异,从而提高分类器的分类效果。

图1 模型流程
1 理论和方法
本文提出了一种基于残差神经网络的白细胞分类架构。图2显示了模型的总体框架。该框架由两个主要部分组成:①采用基于ImageNet数据集的迁移学习,进行参数迁移。②ResNet网络通过对网络层和分类器损失函数的改进对图像提取特征并分类。

图2 模型的总体框架
1.1 迁移学习
迁移学习的总体思路是利用已有的知识系统,即模型从任务中学习到的许多有标记的训练数据可以用在另一个具有不同数据样本的新任务中。通过这种方式,可以降低用大量资源标记数据的成本,同时可以显著提高的学习性能[16]。目前的迁移学习方法主要有实例迁移、特征迁移、共享参数迁移和关系知识迁移[17]。本文采用参数迁移方法进行学习,发现了源域和目标域模型之间共享的参数或先验,从而大大提高了学习效率[18]。
我们使用除了完全连接层外的ResNet体系结构[19]对1000个类别的ImageNet数据集[20]的模型参数进行了预训练。这样,有效地调整了卷积层的预训练参数,以初始化网络参数。最后,利用新的全连通层对整个网络进行再培训。通过迁移学习,可以显著提高模型的精度,增强模型的鲁棒性。
1.2 ResNet
本文以34层的ResNet为主要学习网络。ResNet是由He等[21]提出的。这些网络克服了网络深度增加带来的退化问题。ResNet的主要思想是在网络中添加一个直接连接通道,允许保留一定比例的前一层网络输出。ResNet解决了传统的卷积网络或全连通网络在传输信息时会丢失信息的问题,同时也存在梯度消失或梯度爆炸的问题,导致深度较深的网络无法训练[22]。ResNet的思想与公路网非常相似,可以将原始输入的信息直接传递到后面的层。如图3所示,这一层神经网络可以学习之前的网络输出,而不需要学习整个输出。所以ResNet也被称为残差网络。

图3 残差块对比
为了减少转换网络的计算成本,增强模型的泛化能力,本文运用IBN(instance-batch-normalization)残差块设计了一个结构更为精简的图像分类网络。在原始的残差网络中每个卷积层后面都会添加一个BN(batch-normalization)层,BN层的使用可以加快网络的训练速度,同时提高网络的泛化能力
(1)
其中,x∈N×C×H×W为BN层输入,γ∈C和β∈C是从数据中学习到的映射参数,u(x)∈C和σ(x)∈C分别为输入的平均值和标准差
(2)
(3)
Huang等[23]通过实验验证,BN层模型可以增强语义特征之间的差异性,保留图片中的语义信息,IN层(instance-normalization)模型具有更快的训练速度。IN层的计算公式为
(4)
(5)
(6)
本文将相同通道数的IN层和BN层添加到残差块的低层网络中,将IN层添加到残差块的高层网络中。使用基于IBN残差块替换原网络的所有基于BN残差块,充分利用IN层和BN层的优点。
图3(a)为原始的基于BN层的残差块结构,图3(b)中的IBN层由32通道的IN层和32通道的BN层组成,由于IBN层的通道数等于原网络BN层的通道数,所以网络的计算量没有增加。在网络低层中添加部分BN层可以保留特征的语义信息,在网络高层添加IN层加速损失的收敛,能够提升网络图片分类的能力。
1.3 损失函数的改进

(7)
其中,fi∈d表示xi从DNN中学习得到的深度特征向量,Wj∈d表示第j类的权重向量,bj是对应的偏置项。所以从概率的角度,softmax损失给出了属于所有类别的每个样本的概率分布。虽然softmax损失可以有效地惩罚分类,但它忽略了类内和类间的关系。
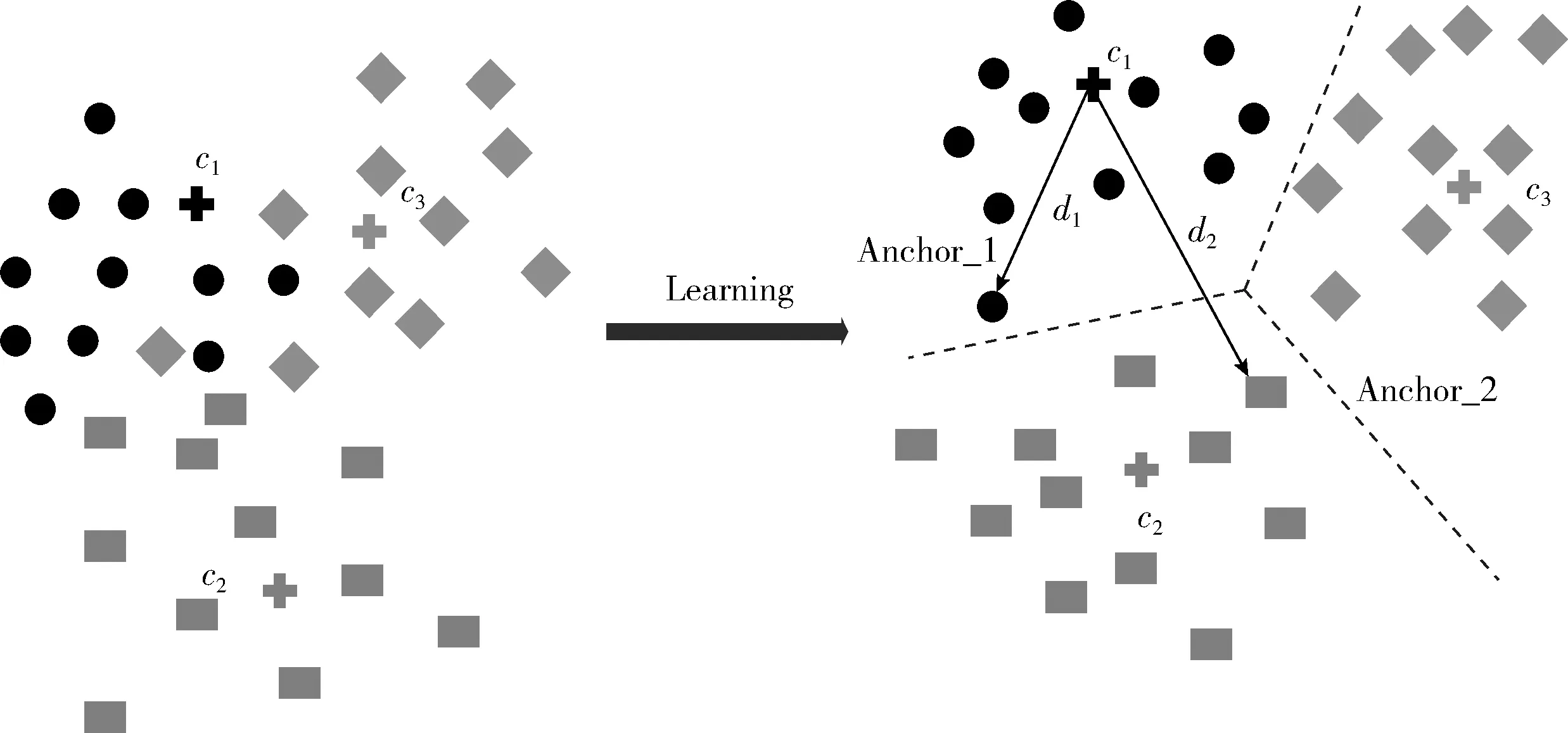
为了提高深度学习特征的判别能力,我们定义了一个新的损失函数来减小类内样本间的差异,扩大类间样本的差异。我们考虑一个新的三重态(fi,cyi,fj),其中fi为锚点,与其它类别的中心cyi相关联,fi表示来自其它类别的样本,m>0是边缘距离。我们期望在样本fi与其类中心cyi之间的距离应该小于fi与cyi之间的距离。它们的相对距离关系表示为
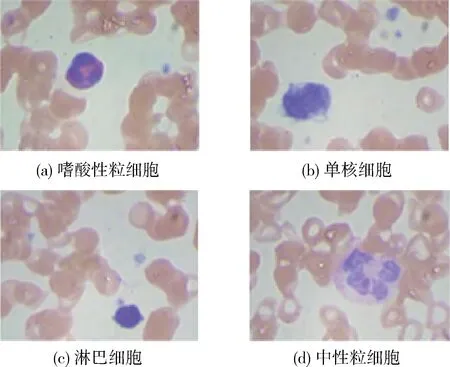
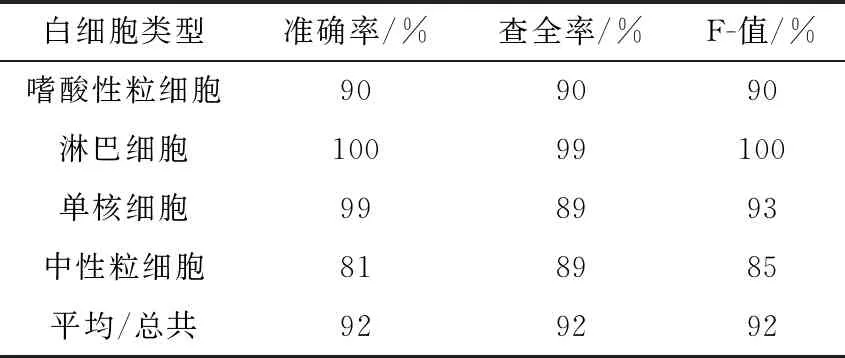
d(fi,cyi)+m (8) (9) 图4 提出的方法说明 (10) 其中,(·)+=max(·,0)表示为铰链函数。在DNN训练阶段,可以根据反向传播(BP)算法更新损失函数的梯度。当Lf≤0时,fi的梯度为0,否则fi的梯度计算方法如下所示 (11) 因此,softmax损失侧重于将样本映射到离散的标签上,而我们的修改的损失则是使用度量学习来约束所学习的特征,具有更明显的类内和类间区分能力。因此,可以将这两个损失组合在一起,以实现更有鉴别力和鲁棒性的嵌入。最终损失函数可表示为 L=Lsoftmax+λLf (12) 其中,λ是平衡这两部分的超参数。 我们选择了BCCD的公共数据库,其中包含12 500张血细胞增强图像(JPEG),细胞类型标签(CSV)包含9957个训练图像和2487张测试图像。我们将这些细胞图像分为4种类型:嗜酸性粒细胞、淋巴细胞、单核细胞和中性粒细胞。原始训练图像中有2497个嗜酸性粒细胞,2483个淋巴细胞,2478个单核细胞,2499个中性粒细胞。测试图像包括623个嗜酸性粒细胞、623个淋巴细胞、620个单核细胞和624个中性粒细胞。图5显示了不同类型的细胞图像。 图5 不同种类的细胞图像 为了提高模型的精度并减少过度拟合,我们使用矩阵变换来增加图像样本的数量。图6显示了矩阵变换后的细胞图像。其它数据增强方法,比如比例变换和颜色变换,不使用,因为细胞核的大小和强度是区分细胞的基本特征。虽然旋转细胞图像可能会略微降低图像的质量,但通过实验结果表明本文提出的框架模型对增强细胞图像是有意义的。 图6 矩阵变换后的细胞图像 为了训练分类模型,我们的模型的卷积层的权值由ImageNet数据集上预训练网络的传输参数初始化。并利用随机初始化器对模型中完全连通层的参数进行初始化。 在实验中,改进的ResNet对图像特征进行提取并分类,所有的训练数据归一化成224*224*3,然后分为小批次训练。最小批量大小设为128。采用随机梯度下降法作为模型训练的优化策略。初始学习率设置为0.001,动量设置为0.9。整个网络使用ReLU作为默认的激活函数。 本文的实验环境是python3.6。处理器为i7-6800k,内存为32 G,显卡为GTX1080Ti,操作系统为Linux。整个实验基于开源的深度学习框架pytorch 0.4。 为了验证我们的模型具有更好的分类效果,我们选取了一些现有的模型架构对我们的公共数据集进行了实验[19,21,26]。表1 给出了不同类型细胞的分类准确率、查全率和F-值,表2给出了不同模型的分类精度。结果表明,本文使用的分类模型比卷积神经网络(CNN)准确率高5%,比卷积神经网络-对抗神经网络(CNN-RNN)准确率高2%,总体达到了较好的分类效果,准确率达到92%。 表1 不同类型细胞的分类准确率、查全率和F-值 表2 不同模型的分类精度 本文提出了一种白细胞图像分类框架。首先利用矩阵变换扩大可靠的训练样本,然后通过改进的ResNet架构和改进的损失函数来对白细胞图像分类。其中在公共数据集上进行的大量实验结果表明,结合不同的神经网络模型,样本三倍中心损失是有效的。采用硬样本挖掘战略,过滤器易分类样本和增大类间差异,减小类内差异,能显著提高残差神经网络的图像鉴别分类能力。该模型解决了数据样本不足、数据不平衡等问题,可以充分利用图像特征进行分类,不需要图像分割。与现有的网络结构相比,该方法对WBCs图像分类的准确率最高。本文的模型也存在一定的缺陷,白细胞总共有5类,本文只对其中的4类进行了识别分类,因此还需要更加完善的数据库系统。在实际临床中,细胞往往可能不都是在成熟期,不同阶段的细胞状态,还需要做进一步的模型分析。在未来,我们希望继续完善现有的网络架构,进一步加强,并开发一个自动分类系统,不仅能识别白细胞,还能识别其它血细胞,从而更好地辅助医生诊断。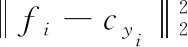
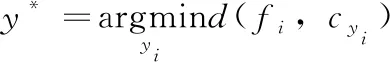
2 实验和结果分析
2.1 数据及其预处理
2.2 模型训练
2.3 实验环境
2.4 实验结果

3 结束语

