单细胞测序技术在微生物生态领域中的应用
王丹蕊 沈文丽 魏子艳 王尚 邓晔,,3
(1. 中国科学院生态环境研究中心,北京 100085;2. 山东大学海洋研究院,青岛 266237;3. 中国科学院大学资源与环境学院,北京 100049)
单细胞测序技术是指在单细胞水平上,通过全基因组或转录组扩增,对核酸分子进行高通量测序的技术。该技术能够揭示单个细胞的基因结构和基因表达水平,反映细胞间的异质性,剖析单个细胞对生态系统或有机体的贡献[1-2]。1990 年,Iscove等[3]首次提出对单细胞进行转录组分析的构想,并用 PCR技术实现了对 cDNA 分子的指数级扩增。1992年,Telenius等[4]开发出寡核苷酸引物PCR(Degenerate oligonucleotide primed PCR,DOPPCR)的方法,用简并寡核苷酸序列扩增基因组,为单细胞测序提供了思路。直到2001年,Dean等[5]首次使用随机六聚体引物和φ29 DNA聚合酶进行反应实现了DNA的滚环扩增,随后Raghunathan和Lasken等[6-7]于2005年发明了多重置换扩增技术(Multiple displacement amplification,MDA),实现了对单细胞全基因组的扩增与测序。但此时的单细胞扩增技术在覆盖度和扩增偏好性方面有明显的局限性,随后一些研究者致力于克服这些问题。例如,Stepanauskas等[8]于2017年 使 用φ29 DNA聚合酶的热稳定突变体提高了单细胞基因组测序的覆盖度;哈佛大学谢晓亮团队[9]于2012年发明了多次退火环状循环扩增技术(Multiple annealing and looping based amplification cycles,MALBAC),通 过拟线性的扩增过程降低了指数扩增的序列偏好性。发展至今,单细胞测序技术在神经生物学、微生物学、胚胎发育、器官发生和免疫学研究中取得了广泛应用,临床上也已用于辅助生殖和肿瘤的诊断与治疗[10]。Nature Methods于2011年将单细胞研究方法列入最值得关注的技术领域,又于2013 年将相关应用列为年度最重要的方法学进展;近日Nature再次将单细胞测序技术评为2020年度最受期待的技术之一。
相比扩增子测序和宏基因组测序,单细胞扩增和测序技术有其独特的不可替代的优点。扩增子测序是指对微生物的特定基因进行测序[11],传统针对16S rDNA、18S rDNA或ITS(内转录间隔区)基因进行的扩增子测序虽然可以满足检测微生物群落多样性的需求,但这种方法很难准确鉴定到属以下的分类等级,也无法深入探究物种的功能信息[12]。宏基因组测序又称环境基因组测序或群落基因组测序,直接对样本中所有微生物的全基因组进行测序,可以同时对物种和功能基因做出鉴定,也有助于发掘潜在代谢途径。然而该方法容易忽视某些稀有种[13],且测序结果的组装也始终是一大难题[14-15]。如果说宏基因组数据集是捕获整个群落信息的一张巨网[16],那么单细胞测序方法则是分离目标基因组的“手术刀”和深入探究目标群落的“放大镜”,能不断细化、深化我们对微生物群落的认识[17]。
本综述重点介绍了在微生物生态学领域使用过或具有应用前景的单细胞分离技术和单细胞基因组扩增技术,并详细论述了单细胞测序技术在获得单细胞基因组、剖析种群异质性、探究种间关系、联合系统发育和功能信息等方面的应用,最后在此基础上对其在微生物生态学领域的发展做出了一些展望。
1 单细胞分离与全基因组扩增
通常单细胞测序的完整流程包括样品保存与制备、单细胞分离、细胞裂解、全基因组扩增(WGA)与产物的系统发育筛选、文库制备与测序、测序结果的质量控制[18],其中单细胞分离与全基因组扩增是最为关键的步骤,诸多技术已被开发并应用于微生物的相关领域,而如何根据科学问题对分离和扩增的方法进行选择或改进对科学研究具有重大意义。
1.1 细胞分离技术
细胞分离是单细胞测序的第一步,其准确性将直接影响后续的测序和分析。提高通量、减少样品与试剂的消耗、提高细胞分离捕获的灵敏度和精确性一直是研究者的目标。常用的细胞分离技术包括有限稀释法(Limited dilution)、显微操作法(Micromanipulation)、激光捕获显微分离技 术(Laser capture microdissection)、拉 曼 镊 子(Raman tweezers)、涡旋与相分隔(Vortex and phaseseparation)、荧光激活细胞分选技术(Fluorescenceactivated cell sorting,FACS)和微流控技术(Microfluidics)[19-21](表1)。其中微流控技术因其较低的成本、较高的通量和理想的分离效果在近10年发展迅速,成为细胞分离技术的主流方向[22]。
1.2 单细胞基因扩增技术
单细胞分离之后需要通过单细胞基因扩增技术使单个细胞内极微量的DNA放大至纳克或微克水平,同时也将较长的DNA片段化,满足下一步测序的需要。目前常用的方法包括简并寡核苷酸引物PCR、多重置换扩增技术、多次退火环状循环扩增技术、Tn5转座酶技术[28],以及新兴的细胞内融合基因技术(Emulsion,paired isolation and concatenation PCR,epicPCR)[20]。
简并寡核苷酸引物PCR(图1-A)使用随机简并引物对全基因组进行扩增。由于随机引物退火温度的不均一性,不同序列扩增效率的差异被PCR反应成倍放大,产生大量扩增不足的区域,导致基因 组 覆 盖 率 较 低[4]。2017年,Blagodatskikh等[29]改进了该技术并命名为iDOP-PCR(Improved DOPPCR),新的方案使用具有热稳定性的聚合酶,并调整了引物设计,可显著提高文库质量。

表1 常见单细胞分离技术一览
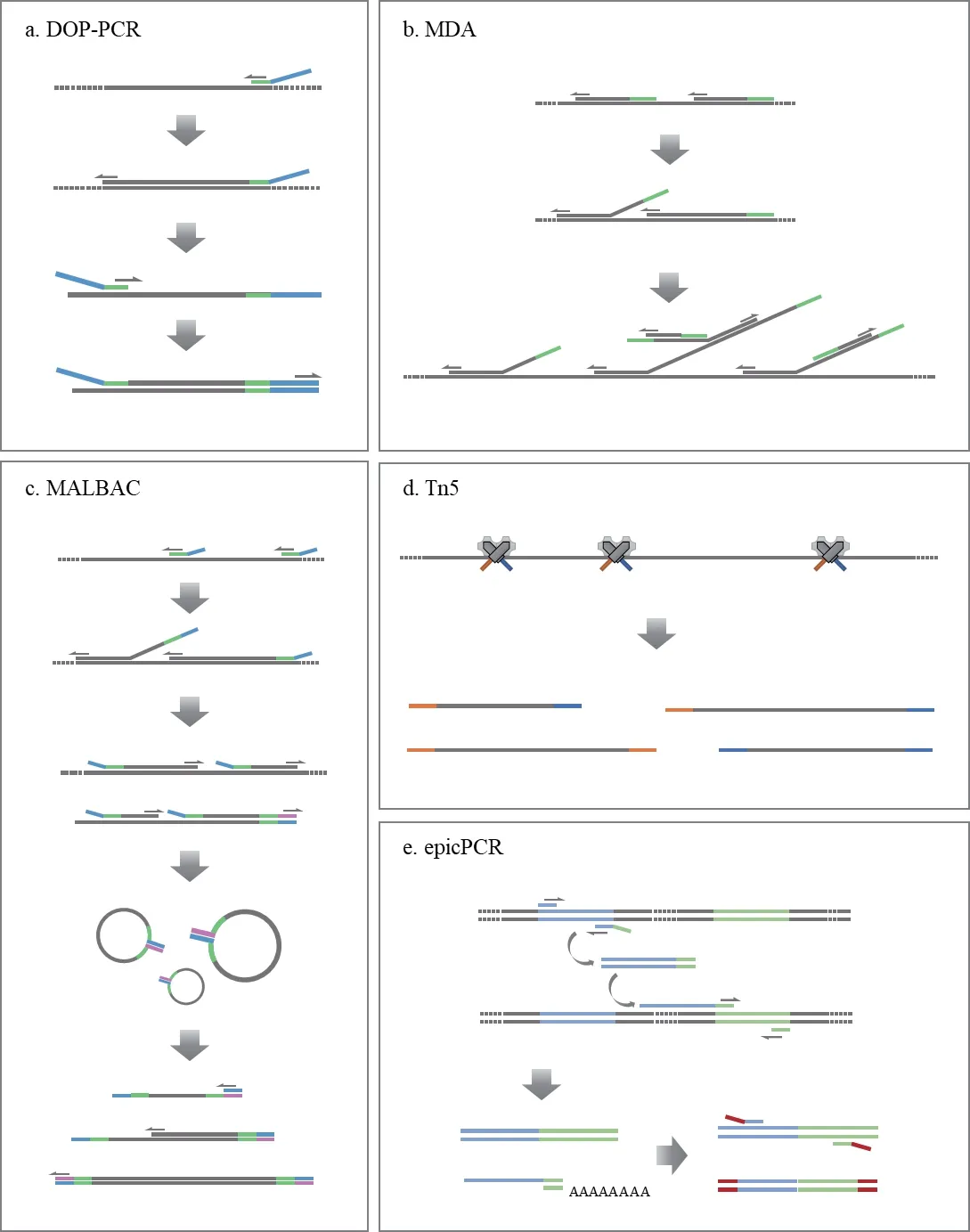
图1 常见单细胞基因组扩增技术原理示意图
多重置换扩增技术(图1-B)是目前环境微生物领域最为成熟也是应用最广的单细胞基因组扩增技术。由于φ29 DNA聚合酶具有3'-5'核酸外切酶活性和校正活性,因此与DOP-PCR相比,多重置换扩增技术(Multiple displacement amplification,MDA)具有更高的序列覆盖度和保真度[30],但也存在外源DNA污染、序列覆盖不均、嵌合体干扰、序列组装与分析困难等不足[31]。由于MDA的偏差在一定程度上是随机的,所以通常可以通过多个数据集的混合拼接来减小这种偏差同时提高组装的完整性[32]。研究表明,2-5个单细胞扩增基因组数据集混合组装得到的基因组完整性中位数>97%,高于单个单细胞组装完整性的中位数(30%-90%)[33]。随后Povilaitis等[34]基于多重置换扩增的原理改进得到全基因组扩增技术WGA-X,使用耐热的突变体φ29DNA聚合酶将延伸温度从30℃提高到45℃,大大提高了从CG含量高的单个环境细胞或病毒体回收基因组的能力。
多次退火环状循环扩增技术(Multiple annealing and looping-based amplification cycles,MALBAC)(图1-C)通过准线性预放大来减少与非线性放大相关的偏差。该技术利用特殊引物,使得扩增子的结尾互补而成环,从而很大程度上防止了DNA的指数性扩增[9]。MALBAC所使用的引物3'端是8个随机的核苷酸序列,5'端是27个固定的核苷酸序列,最大的特点在于它是准线性扩增而非指数扩增,因此拷贝数变异(Copy number variation,CNV)检测的准确性高且单核苷酸变异(Single nucleotide variants,SNV)检测的假阴性率低;而且,MALBAC的偏差具有可重复性,因此可进行降噪和归一化处理。然而,由于该技术使用的DNA聚合酶保真度低于φ29 DNA聚合酶,SNV检测的假阳性率高于MDA。目前MALBAC主要用于医疗诊断[35],在微生物单细胞的准确组装方面相比MDA优势不明显,在微生物生态学领域的应用前景不及MDA[36]。
Tn5转座酶(图1-D)最初用于二代测序的文库构建,将DNA片段化、末端修复、接头连接等简化为一步,大大简化建库步骤的同时为单细胞测序提供了有力工具。基于Tn5转座酶,谢晓亮团队[37]于2017年提出了改良的单细胞全基因组扩增方法(Linear amplification via transposon insertion,LIANTI),用Tn5转座子结合T7启动子形成的转座复合体随机插入单细胞基因组DNA,将基因组片段化并与T7启动子连接。随后T7启动子行使体外转录功能,用转录获得大量线性扩增的转录本,再经逆转录得到大量扩增产物,进行建库测序。该过程进行的是线性扩增,大大增强了扩增的稳定性,使LIANTI在遗传疾病的检测方面更加有效和精确。同年,Lan等[21]利用微流控技术将单细胞测序技术的通量提高到50 000个细胞/次,使得转座酶适用于环境基因组研究。
细胞内融合基因技术(图1-E)是单细胞分离技术、融合PCR、巢式PCR和测序技术的结合,广义而言也可归于单细胞测序的范畴。首先,通过稀释和涡旋将样品分散成单细胞体系;然后对待研究的系统发育基因和功能基因进行融合扩增;随后进行巢式PCR,建库并进行二代测序[20]。该技术的成本远低于普通单细胞全基因组测序,单次实验的通量可高达107个细胞,在解决物种与功能、种间关系等科学问题方面有良好的应用前景[38]。
2 单细胞测序技术在微生态研究中的应用
目前,关于微生物生态的研究大多是群体水平,比如基于宏基因组学的研究能够获得群体的遗传潜力,尽管信息量很大,但无法探究个体的遗传潜力,细胞间的关联和异质性等问题也没有得到很好的解决[39]。单细胞测序与单细胞组学等新技术、新方法的出现为探索复杂环境中的单细胞生态学提供了崭新的机会[40]。单细胞基因组学的组装过程不依赖宏基因组数据的组装和分箱算法,因此能够较为准确地获得单个细胞内质粒、病毒等遗传信息[41],深入发掘不可培养的未知微生物的功能[42];该方法在个体细胞尺度上对样本进行研究[43],从而为探究种群异质性提供了有效手段。此外,单细胞基因组学也常用于研究基因之间的相互作用,破译微生物间或微生物与宿主的交流行为[44],揭示各种生态系统中微生物群落的结构和功能等[45]。
2.1 获得单细胞基因组
地球有丰富的微生物资源。根据比例法则结合生物多样性的对数正态模型进行的预测估计,全球微生物物种可能高达1012种[46]。其中大多数微生物是不可培养或还未被充分认识的,所以由于其未知性也被称作生态系统的暗物质[47]。无需培养的微生物分析鉴定方法(如宏基因组测序),自2004年出现以来一直广受欢迎[48]。然而,环境中微生物通常由个别丰度高的物种和大量丰度低的物种组成[49],由于采样方法、测序深度等限制,大多数研究都是针对丰度较高的种群开展的。Schlos等[50]对近11.3万个细菌和古菌OTU进行普查后发现环境中存在大量的稀有物种,且基于传统方法得到的信息非常有限。单细胞测序技术为获取稀有物种的基因组、发现新的代谢通路提供了支持[32]。
单细胞测序技术对细菌和古菌的暗物质基因组百科(GEBA-MDM)项目有很大贡献,推动了微生物的功能预测和系统发育鉴定[51],极大拓展了系统发育树上的微生物多样性[52]。2017年,Yu 等[53]基于微流控技术开发出一种具有单细胞分辨率的微型宏基因组方法,并用它分析了黄石国家公园的两个温泉样本,从中获得了29个新的基因组草图。Sieracki等[54]用单细胞基因组学方法研究了一系列海洋样品中小型原生生物的多样性,揭示了生态系统中新的相互作用和代谢途径。Ahrendt等[55]使用单细胞基因组学方法对8种不可培养真菌的基因组进行了测序,结果表明,通过结合多个扩增基因组,可以获得90%以上的单细胞基因组。
2.2 剖析种群异质性
常用于物种鉴定的系统发育基因是高度保守的,仅可在属水平上作为物种鉴定的参考,不能反映生态系统中单细胞物种基因组的多样性[56];宏基因组测序中,应用最普遍的二代测序都基于短片段序列,通常会破坏种内变异,经拼接得到的也都是忽略个体差异的泛基因组[57];三代测序可读取较长片段,但目前准确性和通量都比较低[58-59]。相较之下,单细胞测序可以通过捕捉细胞间的基因组变异来剖析自然环境中的种群异质性。因此,它可以阐明微生物多样性和生态位划分,也能够用于基因水平转移的研究[32]。
Kashtan等[60]利用从1 000个原氯球菌细胞产生的大型单细胞扩增基因组文库来确定同一物种的不同生态型在整个季节变化中的基因组变异。测序结果显示,该种群由数百个具有不同“基因组骨架”(Genomic backbones)的亚种群组成,每个骨架包括一组不同的关键等位基因和一些特有的可变基因。Yoon等[61]对3个海洋原生生物(皮胆虫)进行单细胞鸟枪法测序,发现这些细胞代表了3种不同的微生态系,也为红藻亚界存在异养门提供了证据。Engel等[62]成功地应用单细胞基因组学评估了蜜蜂肠道微生物群中两个共生菌在物种水平上的异质性,揭示了菌株和生态位在代谢方面的特异性。2018年,Jochum等[63]对来自奥胡斯湾沉积物中的7个单细胞基因组进行了测序和分析,以了解它们在芳香化合物降解和能量代谢方面的潜力。研究证实了该种群具有代谢多样性,反映出微生物应对不同的能量条件和硫酸盐限制的生存策略。微生物所表现的这种种群异质性是一种适应性特征,可以提高微生物对多变或非均质环境条件的适应能力[64]。
2.3 探究种间关系
对微生物而言,每个细胞都是一个完整的遗传个体,包含了自身染色体、质粒、细胞器、病毒和其他内共生体的全部遗传信息。单细胞基因组学可以无差别地获得细胞内包含的所有遗传物质,从而在染色体DNA和其他遗传信息之间建立联系[65]。近五年的研究表明噬菌体与30多种细菌或古菌有关[66];还发现了一些能感染真核细胞的巨病毒[67]。2011年,Yoon等[61]在从海洋中分离出的单细胞中检测到了内生菌和病毒的遗传信息,揭示了单个细胞间的相互作用;于2014年,Roux等[68]结合单细胞测序和宏基因组测序取得了127个不可培养细菌的单细胞扩增基因组发现,SUP05病毒会使其宿主的能量代谢重构;2019年,Martinez-Hernandez等[69]发现远洋杆菌是海洋中数量极其丰富但未经培养的37-F6病毒的宿主;Labonte等[70]使用单细胞基因组学研究了扩散流、低温热液喷口中噬菌体与宿主的相互作用,结果表明,噬菌体感染后发生溶原化但不裂解宿主,与宿主共进化且增强了宿主对有毒金属和抗生素的抗性。
除此之外,单细胞测序也可用于探究细胞间的相互作用,发现微生物间的共生体,例如Nanoarchaeota和Ignicoccus的 共 生 关 系[71]、Actinomyces odontolyticus和Candidatus Saccharibacteria的寄生关系等[72]。低温透射电镜显示,酸性矿山废水中古菌之间常常存在物理性的胞间连接,例如细胞质桥、菌毛等[73]。如果胞间的相互作用足够强,不会在细胞分离过程中被破坏,单细胞测序技术则可将两个或多个细胞视作一个整体进行测序。Munson-McGee等[41]结合单细胞基因组测序和宏基因组测序发现,高温酸性温泉中专性共生纳米古菌与宿主的物理关联。2019年,Nakayama等[74]为了深入了解海洋蓝藻共生,对宿主远洋鞭毛藻进行单细胞测序并分析其中的蓝藻基因组。系统发育分析显示,样本中蓝藻属于新的分支,它与宿主鞭毛藻严格共存且经历了独立的进化,这种密切的共生关系导致它无法被传统宏基因组学检测到。因此,单细胞测序对发掘物种的多样性、生活方式、代谢途径和进化过程具有重大意义。
随着细胞内融合技术的发展成熟,2019年,Florenza等[38]建议使用epicPCR探究环境中真核生物和原核生物的相互作用关系,如固氮菌和浮游生物之间的互利共生[75],藻细胞周围的微型“生物圈”中有以碳水化合物为食的异养细菌[76],还有病原菌感染细胞并在宿主细胞中增殖[77]。该方法将单个真核细胞和伴生的原核细胞形成的单个细胞簇分散到液滴中,既实现了单个真核细胞的分隔,又保持了真核生物和相关原核生物之间紧密的联系。该方法也有一定局限性:只能揭示物理上紧密结合的相互作用,即在封装时紧密相连的相互作用,包括共生、捕食、寄生等,但无法区分相互作用关系发生在真核细胞的内部还是外部,也不能探究相互作用的分子机制。
2.4 联合系统发育和功能信息
2016年,Spencer等[20]首先开发了epicPCR技术,并将其用于硫酸盐还原细菌的研究,拓展了硫酸盐还原菌(Sulfate-reducing bacteria,SRB)的系统发育多样性。2019年,Qin等[78]用该技术对青藏高原盐湖沉积物中硫酸盐还原原核生物(Sulfatereducing prokaryotes,SRPs)的系统发育进行了鉴定,研究表明西藏盐湖中有多种新的特有的SRP。随后,研究者将epicPCR技术用于抗性基因及其宿主的研究。由于抗性基因相对丰度较低且易在宿主间转移,传统的研究方法存在很大局限性,epicPCR技术的应用大大提高了抗性基因风险评估的精确度水平[79]。
2017年,Lan等[21]首先提出了结合微流控技术、片段化和序列标签的单细胞基因组测序Sic-seq,并使用这种方法来分析了环境样本中微生物耐药基因、毒力因子和噬菌体序列的分布。2020年,Chijiiwa等[80]发现借助单细胞基因组测序技术可以在没有参考基因组的情况下在物种水平上识别代谢应答者,为区分鉴定微生物群落中具有特定功能的未培养微生物提供了方法。Doud等[81]通过识别和捕获基于其原位功能或特征的单个微生物,扩充了功能驱动的单细胞基因组学。该方法先用荧光标记微生物含有的纤维素颗粒,然后通过荧光激活细胞分选方法分离单细胞进行宏基因组鸟枪法测序,结合16S rRNA基因扩增子测序进行系统发育定位,并对纤维素酶进行基因鉴定、表达检测和活性测试。这种功能驱动的单细胞方法可以将微生物分类直接与原位功能联系起来,具有广泛的应用前景和重大的实际意义。
3 总结与展望
2018年12月,英国皇家学会举办了一场以“单细胞生态学”为主题的跨学科会议,使用物理和分子领域相关的最新方法,在单细胞尺度研究生物现象,揭示同一物种的个体(或个体群)与其他个体、环境,以及不同种个体的相互作用[82]。操纵细胞的物理学家、研究微生物群落性质的微生物学家和开发新的单细胞方法的基因组学家齐聚一堂,产生了诸多新的见解与灵感。现在正是单细胞测序技术的飞速发展期,它的产生与完善推动了多个学科的进展,在各领域的广泛应用又促进了技术的成熟。目前已有一些较成熟的单细胞测序平台和商品化试剂盒上市,如10×genomic公司的10×Chromium Single Cell Gene Expression Solution 和Chromium Single Cell DNA Reagent Kits,BD公司的BD RhapsodyTMSingle-Cell Analysis System,Wafergen公司的ICELL8 Single-Cell System,Bio-Rad公 司 的SureCell ATACSeq Library Preparation Kit和与Illumina合作开发的llumina®Bio-Rad®Single-Cell Sequencing Solution等。但由于单细胞测序的成本依然较高而能够同时检测的细胞数量也较为有限,其主要的应用热点依然以医学领域为主,环境微生物和微生物生态的应用才刚刚起步。而根据微生物生态研究的特点,领域内也有专家对单细胞测序的仪器使用方法进行了一系列优化,如结合单细胞测序技术将复杂群落分割成多个微型亚群再进行宏基因组测序的微型宏基因组(Mini-metagenome)技术[83],该技术降低了样品复杂性,同时具有传统宏基因组测序所不具备的单细胞分辨率,与单细胞技术相比又提高了测序通量,解决了嵌合体问题,非常适用于环境样本。单细胞测序技术有望成为微生物生态研究强大有力的主流技术力量,对于深入研究不可培养的微生物、加深对微生物生命之树的探索具有重大意义。

