基于高通量测序的技术检测梨树病毒
杨洁萍,周 丽,马 丽,全绍文,覃 阳,牛建新
(石河子大学农学院园艺系/特色果蔬栽培生理与种质资源利用兵团重点实验室,新疆石河子 832003)
0 引 言
【研究意义】果树为多年生植物,以无性繁殖为主。病毒多为系统侵染,被侵染后树体周身带病毒且终生受害,轻者树体生长不良,果实产量下降,品质变劣;重者树体衰退枯死或造成果实畸形,失去商品价值[1]。建立高灵敏度、快速、高通量的病毒检测技术,对应用于植物病毒病的早期预警预防有重要意义。【前人研究进展】自发现第一种植物病毒病以来,植物病毒的检测技术也在不断发展,常规的检测手段包括生物学鉴定法、电子显微镜观察法、血清学检测法和分子生物学检测法等[2-3]。分子生物学检测方法已广泛的应用于梨、苹果等果树病毒的检测鉴定[4-7],大多数果树病毒和病原体的特异性和敏感性检测主要基于分子技术。近年来,推出和发展了第二代测序技术(Next-generation sequencing,NGS)[8-9],因其快速和灵敏的特点已应用于发现和鉴定已知和未知病毒,已在葡萄[10-12]、苹果、梨[13]和樱桃[14]等果树上应用,发现了多种感染果树的新病毒[15,16]。库尔勒香梨(Pyrusbrestschneideri)是新疆的名优特产,其独特的风味品质。但库尔勒香梨树体普遍携带病毒,已明确鉴定的梨病毒有5种[17],即苹果茎沟病毒(Apple stem groove virus,ASGV)、梨环纹花叶病毒(Pear ring pattern mosaic virus,PRPMV)即苹果褪绿叶斑病毒(Apple chlorotic leaf spot virus, ACLSV)、梨脉黄病毒(Pear vein yellow virus, PVYV)即苹果茎痘病毒(Apple stem pitting virus, ASPV)、梨石痘病毒(Pear stony pit virus, PSPV)和榅桲矮化病毒(Quince stunt virus, QSV)。卢永灿[18]建立了检测3种梨病毒ASGV、ASPV和ACLSV的实时荧光定量RT-PCR技术及ASPV和ACLSV的环介导等温扩增技术;牛建新等[19-20]利用原位PCR检测了库尔勒香梨的ACLSV、建立了ASPV、ACLSV和ASGV的多重RT-PCR检测技术;黄妍妍等[21]利用TC-RT-PCR、IC-RT-PCR、RT-PCR技术检测了ASGV,并比较3种检测技术的灵敏性;刘娜等[22]克隆了ASPV分离株KL1、KL9的全基因组;孙晓霞等[23-24]利用高通量测序技术获得了库尔勒香梨的ASGV的全基因组和啤酒花矮化类病毒的序列。【本研究切入点】库尔勒香梨树体普遍带有病毒,导致香梨产量降低、风味品质变劣,给香梨生产带来巨大隐患。已有利用生物学鉴定法、血清学检测法、分子生物学检测法等多种方法检测梨病毒的研究,但没有利用高通量测序技术来检测库尔勒香梨病毒相关研究,并验证其可靠性。传统检测方法耗时长、需要对病原物的生物学特征、血清学特征、理化特征、基因组结构特征有预先的了解[25],而高通量测序技术检测可以不依赖对已知病原物特征的了解而对病毒进行鉴定。研究基于高通量测序的技术检测梨树病毒。【拟解决的关键问题】以库尔勒香梨花朵为试材,通过高通量测序技术进行转录组测序分析,筛选与植物病毒相关的基因序列,采用RT-PCR技术对候选病毒有关的基因序列进行验证,研究检测梨病毒种类的新方法。
1 材料与方法
1.1 材 料
2014年、2017年、2018年4月中旬,在库尔勒沙依东园艺场香梨园随机选取若干库尔勒香梨树采集花朵,包于锡箔纸中立即投入液氮速冻保存,带回实验室存放于-80℃,用于转录组测序。2018年10月,在库尔勒沙依东园艺场香梨园随机选取若干库尔勒香梨树,采集枝条,用湿布包裹带回实验室存放于4℃,用于后续RT-PCR验证。
1.2 方 法
1.2.1 转录组测序及序列
将3年采集的库尔勒香梨花朵分别于当年送至诺禾致源公司,委托其利用Illumina HisSeqTM2500测序平台进行转录组测序。将获得的原始序列过滤剔除低质量数据得到干净序列(clean data)后,采用Trinity[26]对clean reads进行拼接。Trinity 拼接得到的转录本序列,采用Corset[27]利用比对上转录本的reads数和表达模式对转录本进行层次聚类,以Corset层次聚类后得到最长Cluster序列进行基因功能注释,筛选出注释为植物病毒的长片段序列作为候选病毒进行后续分析。
2014年、2017年、2018年库尔勒香梨花朵转录组测序分别记为T1、T2、T3。
1.2.2 总RNA提取
采用EASYspin Plant microRNA Kit (Aidlab)试剂盒,提取库尔勒香梨枝条的韧皮部总RNA,按照说明书操作。检验总RNA的质量用1.0%琼脂糖凝胶电泳和核酸蛋白浓度测定仪(Nanodrop 2000)测定。
1.2.3 RT-PCR 验证
采用PrimeScriptTMRT reagent Kit with gDNA Eraser(TaKaRa)试剂盒进行反转录反应,合成的cDNA立即放于-20℃冰箱保存。
利用Primer 5.0软件设计引物,引物序列见表1。PCR 引物委托生工生物工程(上海)股份有限公司合成,PCR 反应体系为20 μL,其中 cDNA 2 μL,2XTaqPCR Master Mix 10 μL,正反引物各1 μL,ddH2O 7 μL。 PCR扩增程序为 :94℃预变性90 s;94℃变性 30 s,X℃退火30 s(退火温度因引物而异),72℃延伸1 min,35个循环;72℃最后延伸5 min;4℃保存。PCR 产物取5 μL用 1%琼脂糖凝胶电泳检测。每对引物重复试验3次,确定结果的准确性。表1

表1 病毒 RT-PCR 检测引物Table 1 Primer of RT-PCR for virus detection
2 结果与分析
2.1 转录组测序
研究表明,T1转录组测序共获得107 202 492个原始序列,经过滤共得到103 466 288个clean reads,GC含量平均为47.04%,Q20和Q30 分别平均为 97.64%、93.18%。此次样品测序数据可作为后续转录本拼接的参考序列。对所获得的读序进行拼接得到了100 690个转录本,平均长度为 1 081 bp,对转录本序列进一步对序列进行层次聚类后,共获得48 894个基因序列,平均长度为 816 bp。表2,表3
T2转录组测序共获得190 704 600个原始序列,经过滤共得到186 196 858个clean reads,GC含量平均为46.58%,Q20和Q30分别平均为96.70%、91.87%。可以看出,此次样品测序数据可作为后续转录本拼接的参考序列。对所获得的读序进行拼接得到了154 020个转录本,平均长度为798 bp,对转录本序列进一步对序列进行层次聚类后,共获得96 662个基因序列,平均长度为 1 103 bp。表4,表5
T3转录组测序共获得316 287 950个原始序列,经过滤共得到308 085 228个clean reads,GC含量平均为47.11%,Q20和Q30 分别平均为 97.79%、93.67%。此次样品测序数据质量比较好,可为后续转录本拼接作为参考序列。对所获得的读序进行拼接得到了180 542个转录本,平均长度为1 200 bp,对转录本序列进一步对序列进行层次聚类后,共获得158 918个基因序列,平均长度为 1 326 bp。表6,表7

表2 T1拼接长度分布(bp)Table 2 T1 assembly length distribution statistics

表3 T1拼接长度频数分布情况Table 3 T1 assembly length frequency distribution statistics

表4 T2拼接长度分布(bp)Table 4 T2 assembly length distribution statistics

表5 T2拼接长度频数分布情况Table 5 T2 assembly length frequency distribution statistics

表6 T3拼接长度分布(bp)Table 6 T3 assembly length distribution statistics

表7 T3拼接长度频数分布情况Table 7 T3 assembly length frequency distribution statistics
2.2 病毒相关序列注释
运用BLAST程序将获得的转录本分别与NCBI的Nr 数据库和 Swiss Prot 数据库(http://www.ebi.ac.uk/uniprot/)进行比对(E-value<10-5),挑选与植物病毒有同源关系的序列进行分析。
2.2.1 T1转录组测序病毒相关序列注释
对T1测序结果筛选出的66条注释为植物病毒的基因序列进行分析,发现2种已报道的侵染梨的病毒,分别为ASPV分离物PA66 (txid:651356)、ASGV 分离物P-209(txid:36402)和ASGV 韩国分离物。表5
总共58条基因序列注释为ASPV。有1条基因序列注释为ASPV全基因组,序列长度为8 745 bp的comp54214_c0,相似性为81.4%;有41条基因序列注释为ASPV分离物PA66的RNA复制相关蛋白,序列平均长度为1 091 bp,比对到RNA依赖的RNA聚合酶基因(RdRp)上相似性最高的序列为comp52223_c0,相似性为99.5%,长度为3 301 bp ;有7条基因序列注释为ASPV分离物PA66的运动蛋白和基因沉默蛋白TGBp1,序列平均长度为845 bp,最大长度为1 671 bp的comp53041_c0,相似性为98.7%,比对到运动蛋白和基因沉默蛋白TGBp1上相似性也是最高;有2条基因序列注释为ASPV分离物PA66的运动蛋白TGBp2,序列长度为232 bp的comp41062_c0和223 bp的comp29651_c0,相似性分别为100%和95.9%;有7条基因序列注释为ASPV分离物PA66的衣壳蛋白,序列平均长度为835 bp,最大长度为 1 490 bp的comp52217_c0,相似性为96.6%,比对到衣壳蛋白基因上相似性也是最高。
总共8条基因序列注释为ASGV。有3条基因序列注释为ASGV分离物P-209的基因组多蛋白,序列长度为3 817 bp的comp53314_c2、691 bp的comp352883_c0和236 bp的comp6702_c0,相似性分别为90.5%、93.5%和94.7%;有2条基因序列注释为ASGV分离物P-209的推定的运动蛋白,序列长度为394 bp的comp6929_c0和202 bp的comp6929_c1,相似性都为100%;有3条基因序列注释为ASGV韩国分离物的基因组多蛋白,序列长度为280 bp的comp645322_c0、302 bp的comp272030_c0和6 460 bp的comp53 156_c0,相似性分别为94.6%、98.0%和100%。表8
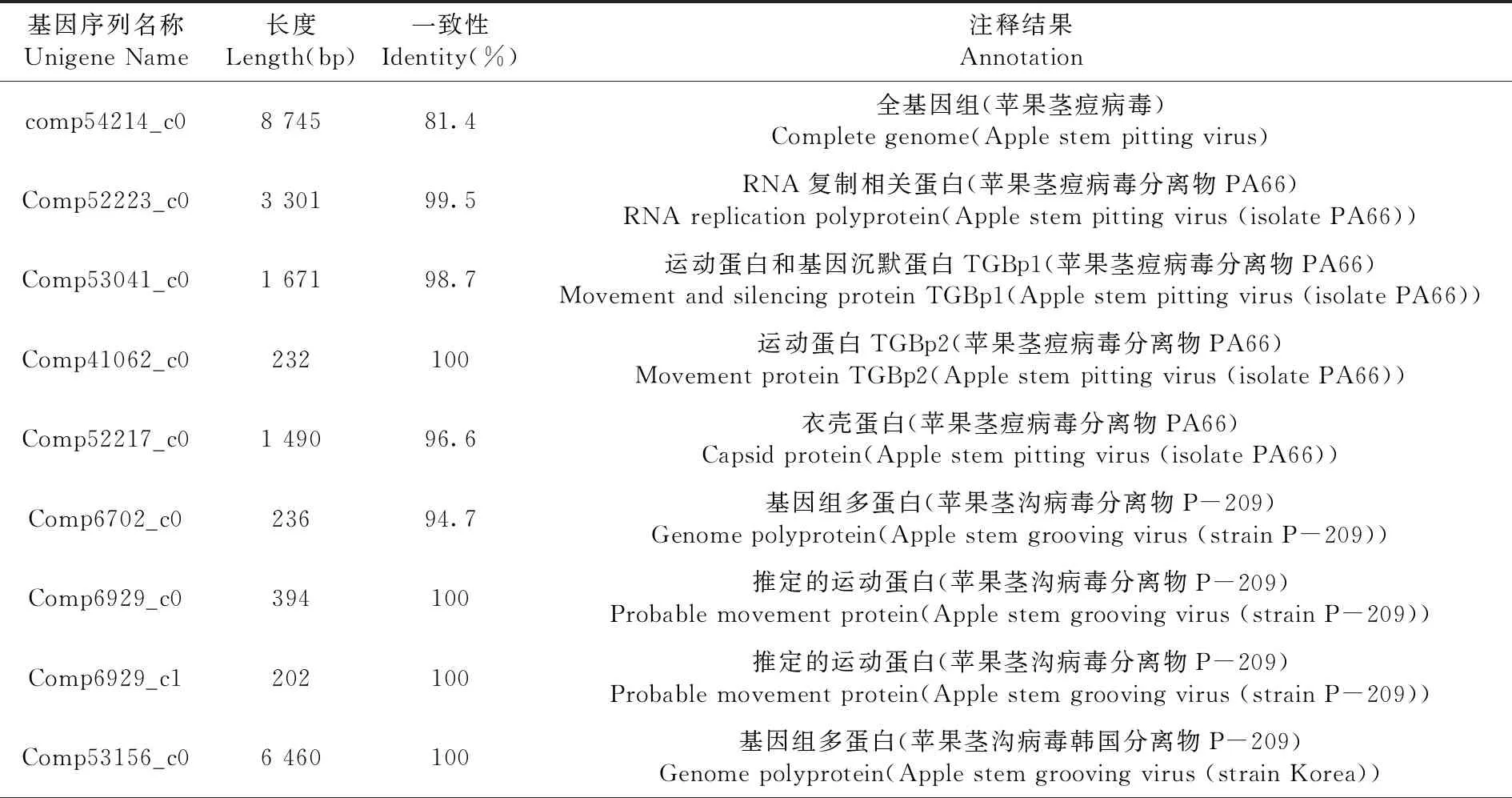
表8 T1候选病毒相关基因序列的注释Table 8 Summary of annotation results of candidate virus-related gene sequences in T1
2.2.2 T2转录组测序病毒相关序列注释
对T2测序结果筛选出的202条注释为病毒的基因序列进行分析,发现3种已报道的侵染梨的病毒病毒,分别为ASPV分离物PA66、ASGV 分离物P-209和ASGV 韩国分离物、ACSLV(txid:12175)和一种未报道侵染梨的芸薹黄化病毒(Brassica yellows virus,BrYV)。
总共185条基因序列注释为ASPV。有2条基因序列注释为ASPV全基因组,序列长度为9 294 bp(Cluster-2 341.456 70)和6 255 bp(Cluster-2 341.433 00),相似性分别为81.7%和97.8%;有127条基因序列注释为ASPV分离物PA66的RNA复制相关蛋白,序列平均长度为688 bp,比对到RNA依赖的RNA聚合酶基因上相似性最高的序列为Cluster-2 341.433 00,相似性为89.5%,长度为3 178 bp ;有19条基因序列注释为ASPV分离物PA66的运动蛋白和基因沉默蛋白TGBp1,序列平均长度为696 bp,最大长度为2 197 bp的Cluster-2 341.494 09,相似性为98.7%,比对到运动蛋白和基因沉默蛋白TGBp1上相似性也是最高;有13条基因序列注释为ASPV分离物PA66的运动蛋白TGBp2,序列平均长度为352 bp,最大长度为621 bp的Cluster-2 341.477 17,相似性分别为98.3%,比对到运动蛋白TGBp2上相似性也是最高;有24条基因序列注释为ASPV分离物PA66的衣壳蛋白,序列平均长度为578 bp,最大长度为1 473 bp的Cluster-2 341.472 07,相似性为97.8%,比对到衣壳蛋白基因上相似性也是最高。
总共12条基因序列注释为ASGV。有2条基因序列注释为ASGV全基因组上,序列长度为6 519 bp的Cluster-2 341.647 97和5 007 bp的Cluster-2 341.432 63,相似性分别为86.8%和80.4%;有5条基因序列注释为ASGV分离物P-209的基因组多蛋白,序列平均长度为360 bp,序列最大长度为443 bp的Cluster-2 341.331 23,相似性分别为95.2%,比对到241 kDa的多聚蛋白基因上相似性也是最高;有1条基因序列注释为ASGV分离物P-209的推定的运动蛋白,序列长度为528 bp的Cluster-2 341.394 14,相似性为100%;有4条基因序列注释为ASGV韩国分离物的基因组多蛋白,序列平均长度为354 bp,最大长度为435 bp的Cluster-2 341.392 52,似性为80.4%,比对到241 kDa的多聚蛋白基因上相似性最高的序列为Cluster-2 341.331 22,相似性为96.1%,序列长度为388 bp。
总共3条基因序列注释为ACLSV。有1条基因序列注释为ACLSV的外壳蛋白,序列长度为930 bp的Cluster-7 440.0,相似性为97.9%;有1条基因序列注释为ACLSV的RNA多聚酶,序列长度为392 bp的Cluster-12 813.0,相似性为100%;有1条基因序列注释为ACLSV的推定的运动蛋白,序列长度为1 080 bp的Cluster-2 341.679 88,相似性为98.2%。
总共2条基因序列注释为芸薹黄化病毒。有1条基因序列注释为芸薹黄化病毒的CP通读蛋白,序列长度为2 576 bp的Cluster-2 341.308 60,相似性为99.7%,E值为0;有1条基因序列注释为芸薹黄化病毒的p1蛋白,序列长度为1 298 bp的Cluster-4 524.0,相似性为98.7%,E值为0。表9
2.2.3 T3转录组测序病毒相关序列注释
对T3测序结果筛选出的921条注释为病毒的基因序列进行分析,发现2种已报道的侵染梨的病毒,分别为ASPV分离物PA66、ASGV 分离物P-209和ASGV 韩国分离物。
总共有775条基因序列注释为ASPV。有556条基因序列注释为ASPV分离物PA66的RNA复制相关蛋白,序列平均长度为 480 bp,最大长度为3 490 bp的Cluster-9 706.344 28,相似性为93.8%,比对到RNA依赖的RNA聚合酶基因上相似性最高的序列为Cluster-9 706.376 65,相似性为95.5%,长度为2 009 bp ; 有116条基因序列注释为ASPV分离物PA66的衣壳蛋白,序列平均长度为475 bp,最大长度为 2 051 bp的Cluster-9 706.763 78,相似性为97.3%,比对到衣壳蛋白基因上相似性也是最高; 有66条基因序列注释为ASPV分离物PA66的运动蛋白和基因沉默蛋白TGBp1,序列平均长度为460 bp,最大长度为 1 418 bp的Cluster-9 706.541 41,相似性为97.3%,比对到运动蛋白和基因沉默蛋白TGBp1上相似性最高的序列为Cluster-9 706.766 44,相似性为99.5%,长度为1 111 bp;有22条基因序列注释为ASPV分离物PA66的运动蛋白TGBp2,序列平均长度为557 bp,最大长度为 869 bp的Cluster-970 6.763 77,相似性分别为94.2%,比对到运动蛋白TGBp2上相似性最高的序列为Cluster-9 706.763 1,相似性为98.3%,长度为611 bp;有15条基因序列注释为ASPV分离物PA66的运动蛋白TGBp3,序列平均长度为407 bp,最大长度为 646 bp的Cluster-9 706.632 28和Cluster-9 706.632 29,相似性分别为98.6%、97.1%,比对到运动蛋白TGBp3上相似性最高的序列为Cluster-9 706.763 2,相似性为100%,长度为253 bp。

表9 T2候选病毒相关基因序列的注释结Table 9 Summary of annotation results of candidate virus-related gene sequences in T2
总共有146条基因序列注释为ASGV。有118条基因序列注释为ASGV分离物P-209的基因组多蛋白,序列平均长度为995 bp,最大长度为 5 419 bp的Cluster-9 706.428 9,相似性分别为82.3%,比对到241 kDa的多聚蛋白基因上相似性最高的序列为Cluster-9 706.129 673,相似性为92.8%,长度为3 331 bp;有11条基因序列注释为ASGV分离物P-209的推定的运动蛋白,序列平均长度为837 bp,最大长度为 1 130 bp的Cluster-9 706.129 564,相似性分别为96.5%,比对到36 kDa蛋白基因上相似性最高的序列为Cluster-9 706.127 953,相似性为96.6%,长度为1 088 bp;有17条基因序列注释为ASGV韩国分离物的基因组多蛋白,序列平均长度为403 bp,最大长度为727 bp的Cluster-9 706.128 358,相似性分别为98.5%,同时也是比对到241 kDa的多聚蛋白基因上相似性最高的序列。表10
2.3 RT-PCR验证
针对注释获得的ASPV、ASGV、ACLSV 和BrYV 4 种病毒序列,设计特异引物进行 RT-PCR扩增验证。用所提取的RNA进行反转录的cDNA为模板,经过PCR扩增,ASPV、ASGV 2 种病毒序列均能扩增出预期大小的目的条带。ACLSV和BrYV病毒基因序列均未能扩增出目标条带。图1

表10 T3候选病毒相关基因序列的注释结果汇总Table 10 Summary of annotation results of candidate virus-related gene sequences in T3

注:M:Maker Ⅰ, 1-3:苹果茎沟病毒的扩增条带; 4-6:苹果茎痘病毒的扩增条带
3 讨 论
ASPV、ASGV和ACLSV可危害多种果树,在大多数梨树中呈潜伏侵染和混合侵染,多数品种上不表现明显症状,早期多采用指示植物进行鉴定。随着果树病毒分离提纯技术的提高,制备出部分病毒特异抗血清,采用酶联免疫吸附法(ELISA)对梨病毒进行快速检测[28]。近年来,分子生物学检测技术的快速发展,使果树病毒检测效率高、操作简便、特异性强。与传统的血清学、分子生物学和生物学病毒检测方法相比,高通量测序技术在果树病毒诊断和特定病毒检测方面具有许多优势。一是不需要对被感染植物样本的病毒病原体有预先的知识经验,例如用PCR检测法要知道目标病毒种类的基因序列,以便设计用于其分子检测的引物,或者用ELISA检测法要制备用于血清学检测的特异性抗体。除了生物学检测法,经典的病毒检测方法具有高度特异性,无法识别可能与植物病害病因有关的未知病毒。高通量测序技术的应用减少了识别病毒病原所需的大量时间,该方法可识别植物的病毒基因组,还可能发现新的病毒病原体的存在。
试验利用高通量测序技术转录组分析库尔勒香梨中病毒的种类,通过分析三组转录组测序数据,获得了ASPV、ASGV、ACLSV和BrYV 4 种病毒核酸序列,并且获得了ASPV、ASGV的全基因组。用RT-PCR 技术对转录组测序数据进行验证时发现,只检测出了ASPV、ASGV这2种病毒。对每种病毒的基因序列数量进行统计,能在一定程度上反映每种病毒的相对含量,可以看出ASPV、ASGV这2种病毒在库尔勒香梨植株内含量比较高,并且在三组测序数据中都含有这2种病毒,也能反映出库尔勒香梨被ASPV、ASGV侵染比较普遍。ACLSV和BrYV根据设计的特异性引物都没有扩增出目标条带。被注释为ACLSV的3条基因序列比对到已知的ACLSV的不同蛋白基因上相似性都非常高,RT-PCR技术没有检测到可能是因为其含量太低,或者用于RT-PCR验证的材料中不带此病毒。BrYV属于马铃薯卷叶病毒属黄症病毒科单链RNA病毒,含有6个ORFs,试验获得的1条BrYV基因序列与已知的BrYV的CP通读蛋白基因相似性高达99.7%,通读蛋白与介体传毒和病毒韧皮部局限特性相关[29];另一条注释为BrYV基因序列与已知的BrYV的p1蛋白相似性也很高,此蛋白与病毒复制相关[29]。库尔勒香梨中是否携带BrYV尚有待进一步研究。
高通量测序技术可以鉴定混合样品病毒变异的基因型以及同一植株体内病毒的不同分离物的快速检测方法,试验检测出了ASGV分离物P-209和ASGV韩国分离物,利用该技术将促进不同病毒或病毒基因型之间相互作用的研究,揭示病毒发病和发展机制。高通量测序技术应用于病毒检测重要部分之一是测序数据的生物信息学分析,这一步骤在很大程度上依赖于与已知病毒的同源性识别。因此,公共数据库中公布的越来越多的病毒基因组序列是高通量测序技术用于病毒检测成功诊断的一个关键因素。NCBI(http://www.ncbi.nlm.nih.gov/genome)现已公布超过3 500种病毒(和类病毒)基因组参考序列,综合植物病原学基因组资源数据库有623种植物病毒基因组[30],提高了高通量测序数据与病毒序列的识别,促进植物病毒研究的发展进程。
4 结 论
利用库尔勒香梨花朵三组的转录组测序数据,分别对筛选出注释为来源于植物病毒的66条、202条、921条基因序列进行分析。三组的转录组测序数据均检测注释到了苹果茎痘病毒、苹果茎沟病毒的大量病毒序列,注释到了3条苹果褪绿叶斑病毒的部分序列和2条芸薹黄化病毒的序列,并且与已知病毒序列相似性非常高。针对注释获得的ASPV、ASGV、ACLSV 和BrYV 4 种病毒序列,利用RT-PCR检测随机采集的库尔勒香梨枝条样品,均扩增出苹果茎痘病毒和苹果茎沟病毒的特异条带,没有扩增出苹果褪绿叶斑病毒和芸薹黄化病毒的特异条带。

