槟榔不同发育时期果实转录组特征分析
押辉远 陈叶 张岩松 许启泰


摘 要:檳榔(Areca catechuL.)果实是四大南药之一。槟榔果的研究主要集中在生理生化、生防菌、有效成分及药理、加工和利用等方面,对槟榔果的发育及其次生物质形成的分子机制尚不清楚。本研究对不同发育时期的槟榔果皮和果核进行转录组测序,鉴定槟榔果不同发育时期的关键基因,以探讨果实发育相关基因的表达特征及次生物质形成有关的基因调控。结果显示,槟榔果皮中检测到4491个差异基因,其中617个差异基因共参与了111条KEGG代谢通路,生物过程代谢类有82个通路,共257个差异基因被注释,参与次生代谢途径共有5个,共27个差异基因。槟榔果核中检测到5443个差异基因,其中898个差异基因共参与了118条通路,466个差异基因被注释在生物代谢类通路上,共涉及89条通路,参与次生代谢相关的基因有53个,参与次生代谢途径共7条。进一步分析表明:随着果实的发育,果皮中80%次级代谢通路差异相关基因呈下调表达趋势;而果核中71.4%次级代谢通路差异相关基因呈上调表达趋势。本研究结果在转录组水平揭示了槟榔果发育的生物学过程,发现了不同时期槟榔果皮和果核中次级代谢相关调控基因表达的变化规律,也为槟榔的遗传育种研究奠定了基础。
关键词:槟榔;果实发育时期;转录组中图分类号:S31 文献标识码:A
Analysis of Transcriptome Characteristics of ArecaatDifferent Developmental Stages
YA Huiyuan1, CHEN Ye1, ZHANG Yansong1, XU Qitai1,2
1. School of Food and Medicine, Luoyang Normal University, Luoyang, Henan 471934, China; 2. Hainan Green Areca Science & Technology Development Co., Ltd., Dingan, Hainan 571200, China
Abstract: Areca(Areca catechu L.) is one of the four primary medicinal plants in south China. In the study, the high-throughput sequencing technology was used to sequence the transcriptome of the peel and kernel in different periods to find differentially expressed genes in different developmental stages. Among the peels, 4491 differential genes were divided, of which 617 differential genes were involved in 111 KEGG metabolic pathways,257 differential genes with 82 pathways in the biological process metabolism class, and a total of 27 genes with 5 genes involved in the secondary metabolic pathway. There were 5443 differential genes in the betel nut kernel, according to the KEGG pathway annotation results, 898 differential genes were involved in 118 pathways, 466 differential genes were annotated on biological metabolic pathways for 89 pathways, 53 genes involved in secondary metabolism with 7 secondary metabolic pathways involved. Further analysis showed that with the development of fruit, 80% of the secondary metabolic pathways in the pericarp showed a down-regulated expression of the genes, while 71.4% of the secondary metabolic pathways in the kernel showed up-regulated expression. The results of the study preliminarily revealed the overall characteristics of the transcriptome of different tissues and different developmental stages of areca nut, and found that the expression of secondary metabolism-related regulatory genes in betel nut and pit were observed in different stages, which was the medicinal development and secondary of betel nut.
Keywords: Areca catechuL.; fruit development period; transcriptome
DOI: 10.3969/j.issn.1000-2561.2020.07.001
槟榔(Areca catechuL.)是棕榈科槟榔属常绿乔木,广泛分布于南亚和东南亚等国家[1]。在我国,槟榔主要种植于云南、海南和台湾等地,种植面积居世界第五,产量居世界第三,是我国热带、亚热带地区仅次于橡胶的第二大产业[2-4]。槟榔果营养价值丰富,是一种天然的药食两用食品,被称为四大南药之首[5]。
目前已从槟榔中分离鉴定出生物碱、黄酮类、单宁、三萜类、类固醇、脂肪酸等多种化学成分。生物碱具有显著的生物活性,有驱虫、消积、利气利水、利湿除疸等功效[6]。植物生物碱分为萜类吲哚生物碱、哌啶生物碱、苄基异喹啉生物碱、茛菪碱和烟碱、嘌呤生物碱等。已有研究表明,槟榔碱是槟榔中的主要生物碱,属于哌啶类生物碱,其含量为0.3%~0.7%[7-8]。目前,国内外对槟榔生物碱的研究主要集中在槟榔碱及其衍生物的分离鉴定与药理、生理作用、免疫等方面[5]。现代医学研究表明,槟榔碱在促进胃肠道运动、抗肿瘤、镇痛、消炎、抗氧化、保护心血管系统等方面均有作用[1]。
迄今为止,槟榔碱及其多种衍生物已被分离和鉴定[9]。槟榔的生物碱含量与其果实成熟度相关[10],成熟果胚乳中的槟榔碱含量高于青果胚乳,而成熟果果皮的含量低于青果果皮[11]。不同发育时期槟榔花序的槟榔碱含量也存在显著差异,其含量随着槟榔花的成熟而不断降低[12]。然而,不同的槟榔品种由于成分也有较大差异,所以其药用成分含量差异也较大,这种差异性极大地影响了槟榔用于中药的药效稳定性[13-15]。关于槟榔生物碱等次级代谢产物生物合成的遗传控制和分子调控至今还不清楚,这种状况极大地阻碍了槟榔新品种的选育和药用成分的有效利用。
转录组测序能够从整体水平研究基因表达量以及基因结构,揭示特定生物学过程中的分子机理,目前已广泛应用于基础研究、临床诊断、药物研发和分子育种等领域。槟榔果是药食两用的组织部位,研究槟榔果果实发育的遗传基础、果实内次生物质合成的分子调控,将十分有助于槟榔分子育种的快速发展。本研究对海南槟榔的槟榔果皮和槟榔果核进行转录组测序,分析筛选不同时期果皮和果核的差异基因,探究槟榔果生长发育过程中生物碱等次级代谢产物的分子调控机理。
1 材料与方法
1.1材料
本研究于海南省定安县采集坐果30 d和坐果180 d的槟榔(图1)作为实验材料用于转录组测序,每组样品设置3个实验重复(表1)。
1.2方法
1.2.1 槟榔转录组RNA提取及转录组测序 材料总RNA提取方法参照天根(TIANGEN)公司的TRNzol Universal总RNA提取试剂使用说明书,分别提取槟榔果的果皮和果核的总RNA。用安捷伦2100系统检测总RNA完整性,Qubit RNA测定试剂盒纯度质量,将样品送至百迈客生物公司在Illumina Hiseq平台进行转录组的测序。
1.2.2 槟榔转录组数据的功能注释和代谢通路分析 使用BLAST[16]軟件将Unigene序列与NR[17]、Swiss-Prot[18]、Gene Ontology(GO)[19]、Clusters of Orthologous Groups(COG)[20]、euKaryotic Orthologous Groups(KOG)[21]、eggNOG 4.5[22]、Kyoto Encyclopedia of Genes and Genomes(KEGG)[23]数据库比对,使用KOBAS 2.0软件[24]得到Unigene在KEGG中的KEGG Orthology结果,预测Unigene的氨基酸序列后,使用HMMER[25]软件与Protein family(Pfam)[26]数据库比对,获得Unigene的注释信息。
1.2.3 槟榔转录组表达差异分析 将皮尔逊相关系数r(pearson correlation coefficient)作为样品间相关性的评估指标[27]。r2越接近1,说明2个样品的相关性越强。采用DESeq[28]进行样品组间的差异表达分析,获得2个条件之间的差异表达基因集;在差异表达分析过程中采用了公认有效的Benjamini-Hochberg方法对原有假设检验得到的显著性P值(P-value)进行校正,并最终采用校正后的P值,即False Discovery Rate(FDR)作为差异表达基因筛选的关键指标,以降低对大量基因的表达值进行独立的统计假设检验带来的假阳性。在筛选过程中,将FDR小于0.01且差异倍数Fold Change(FC)大于或等于2作为筛选标准。其中,FC表示两样品(组)间表达量的比值。
1.2.4 差异基因富集分析与关键功能基因筛选 差异表达基因GO功能富集,差异表达基因COG分类,差异表达基因KEGG注释,差异表达基因KEGG通路富集分析,差异表达基因蛋白互作网络。
2 结果与分析
2.1转录组测序数据统计
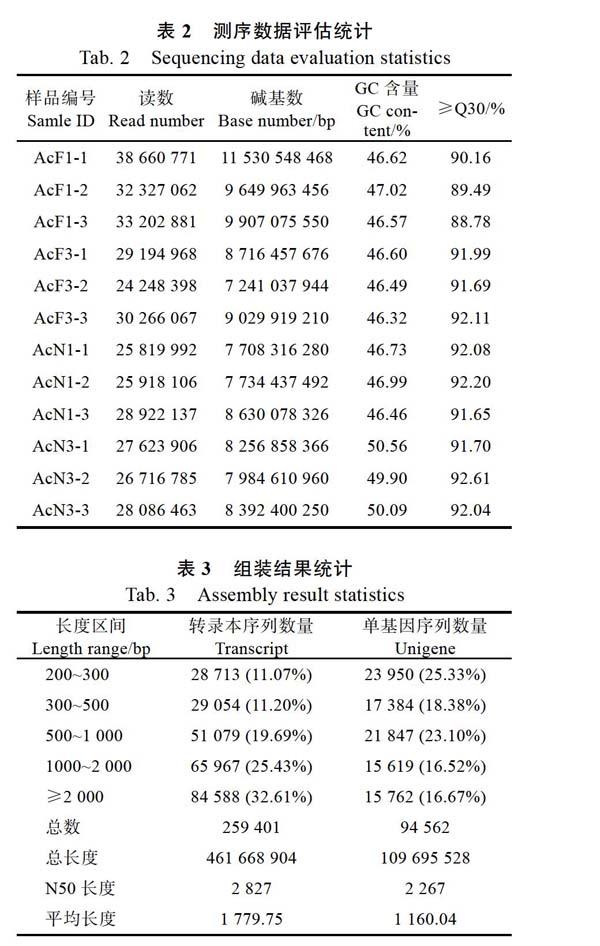
选择坐果30 d和180 d的槟榔果果皮和果核各3次重复,共12个样品,获得104.78 Gb Clean Data,各样品Clean Data均达到8.73 Gb,Q30碱基百分比在88.78%及以上(表2)。
转录组数据集已保藏在NCBI SRA数据库中,编号:PRJNA590547。
组装后共获得259 401条转录本序列,平均长1779.75 bp,N50长2827 bp。获得94 562条转录本序列组装单基因序列,其中长度在1 kb以上的Unigene有31 381条,对转录本序列组装单基因序列进行功能注释,包括与NR、Swiss-Prot、KEGG、COG、KOG、GO和Pfam数据库的比对,共获得35 806条单基因序列的注释结果(表3)。
2.2单基因功能注释
通过BLAST软件将Unigene序列与NR、Swiss-Prot、GO、COG、KOG、eggNOG 4.5、KEGG数据库比对,共得到35 806个基因获得注释(表4)。有8664(24.20%)条基因在COG数据库中获得了注释,有17 155(47.91%)条基因在GO数据库中获得了注释,有10 510(29.35%)条基因在KEGG数据库中获得了注释,有17 917(50.04%)条基因在KOG数据库中获得了注释,有18 072(50.47%)条基因在Pfam数据库中获得了注释,有17 224(48.10%)条基因在Swiss-Prot数据库中获得了注释,有30 231(84.43%)条基因在eggNOG 4.5数据库中获得了注释,有34 334(95.89%)条基因在NR数据库中获得了注释。
2.3槟榔果皮不同发育时期差异表达基因筛选和富集分析
以AcF3为实验组,AcF1为对照组,共筛选到4491个差异基因,上调差异基因1991个,下调差异基因2500个。1563个差异基因在GO功能得到注释,共分成3大类,45个小类。生物学过程(biological process)主要涉及细胞过程、代谢过程、单一机体过程、应激反应和生物调节5种生物学过程;细胞组分(cellular components)主要涉及细胞核、细胞器和质膜3种细胞组分;分子功能(molecular function)主要涉及蛋白结合和催化活性2种分子功能(表5)。
一般认为,经过校正的P值(P-FDR)≤0.05 时,此GO功能存在显著富集情况。在槟榔果皮差异基因的GO注释结果显示,生物学过程中差异基因显著有单一生物体过程、生物调节、细胞成分的组织或者生物合成、信号、多生物过程、免疫生物进程和生物粘附。细胞组分中细胞器、细胞器部分、大分子复合物、细胞外区和细胞连接的差异基因有显著差异。分子功能的催化活性、结构分子活性和转运活性的差异基因数有显著差异。
将果皮差异基因与KEGG数据库进行比对,共有617个差异基因注释在111个通路上,包括138个上调表达基因,479个下调表达基因。KEGG生化代谢通路共分成5个生物过程,包括代谢(metabolism)、遗传信息过程(genetic informa tion processing)、细胞学过程(cellular processes),环境信息过程(environmental information processing)和有机体系统(organismal systems)。
代谢类通路共257个差异基因被注释,参与了82个通路,主要涉及氨基酸代谢、碳水化合物代谢、能量代谢、脂质代谢和其他次级代谢产物的生物合成等。其中,参与次生代谢相关的基因有27个,参与次生代谢途径共有5个,苯丙烷生物合成途径(phenylpropanoid biosynthesis),类黄酮生物合成途径(flavonoid biosynthesis),芪类、二芳基庚烷和姜醇生物合成途径(stilbenoid, diarylheptanoid and gingerol biosynthesis),异喹啉类生物碱生物合成途径(isoquinoline alkaloid biosynthesis),萜类、哌啶和吡啶生物碱合成途径(tropane, piperidine and pyridine alkaloid biosynthesis)。其中,上調差异表达只有苯丙烷生物合成途径,共3个基因包含的酶有4-香豆酸-CoA连接酶、肉桂酰辅酶A还原酶。下调差异表达基因分布在5个次生代谢途径中:苯丙烷生物合成途径,类黄酮生物合成途径,芪类、二芳基庚烷和姜醇生物合成途径,异喹啉类生物碱生物合成途径,萜类、哌啶和吡啶生物碱合成途径。下调基因共10个。调控酶:反式肉桂酸4-单加氧酶、咖啡酰-CoA O-甲基转移酶、双功能天冬氨酸氨基转移酶和谷氨酸/天冬氨酸-预苯酸氨基转移酶、伯胺氧化酶;协同调控苯丙烷生物合成途径和萜类、哌啶和吡啶生物碱合成途径共17个基因,包含β-葡萄糖苷酶、肉桂醇脱氢酶、过氧化物酶、托品酮还原酶I(表6)。
2.4槟榔果核不同发育时期差异表达基因筛选和富集分析
以AcN3为实验组,AcN1为对照组,共筛选到5443个差异基因,上调差异基因2958个,下调差异基因2485个。这些差异基因有2452个在GO数据库得到注释,共分成3大类,48个小类。生物学过程(biological process)主要涉及细胞过程、代谢过程、单一机体过程、应激反应和生物调节基因5种,其中,呈显著差异的有单一生物体过程、对刺激的响应、生物调节、复制、生物学阶段。细胞组分(cellular components)主要涉及细胞核、细胞器和质膜3种,细胞器和大分子复合物的差异基因数有显著差异。分子功能(molecular function)主要涉及蛋白结合和催化活性2种,有显著差异的包含催化活性和转运活性(表7)
将果核差异基因与KEGG数据库进行比对,共有898个差异基因注释在118个通路上,包括595个上调表达基因,303个下调表达基因,其中466个差异基因被注释在代谢类通路上,共涉及89条通路。
参与次生代谢相关的基因53个,参与次生代谢途径共7条,包括咖啡因生物合成途径(caffeine metabolism),黄酮和黄酮醇生物合成途径(flavone and flavonol biosynthesis),類黄酮生物合成途径,异喹啉类生物碱生物合成途径,苯丙类生物合成途径,芪类、二芳基庚烷和吡啶生物合成途径,萜类、哌啶和吡啶生物碱合成途径。其中,参与上调表达的基因41个,1个咖啡因生物合成途径基因,12个苯丙类生物合成途径基因,15个类黄酮生物合成途径基因,5个黄酮和黄酮醇生物合成途径基因,4个异喹啉类生物碱生物合成途径基因,3个芪类、二芳基庚烷和吡啶生物合成途径基因,3个萜类、哌啶和吡啶生物碱合成途径基因,上调表达涉及的酶有尿酸氧化酶、伯胺氧化酶、4-羟基苯丙酮酸双加氧酶、3-羟基丁酰基-CoA脱氢酶、苯丙氨酸氨裂解酶、反式肉桂酸4-单加氧酶、4-香豆酸-辅酶A连接酶、反式肉桂酸4-单加氧酶、反式肉桂酸4-单加氧酶、查尔酮合成酶、查尔酮异构酶类、黄酮3-单加氧酶、类黄酮3,5-羟化酶、白细胞花青素还原酶、花青素还原酶、类黄酮3,5-羟化酶、类黄酮3-单加氧酶、酪氨酸氨基转移酶、酪氨酸脱羧酶(表8)。
参与下调表达的基因2个,一个是苯丙类生物合成途径基因,另一个是萜类、哌啶和吡啶生物碱合成途径基因。下调表达涉及的酶为组氨醇-磷酸氨基转移酶。协同调控的基因共19个,涉及的酶有双功能天冬氨酸氨基转移酶和谷氨酸/天冬氨酸-预苯酸氨基转移酶、过氧化物酶、天冬氨酸氨基转移酶。
3讨论
槟榔果是槟榔的药用部位。本研究利用高通量测序技术对2个不同时期的槟榔果皮和果核的转录组进行测序,初步揭示了槟榔转录组的整体表达特征,获得了不同时期槟榔果皮和果核中次级代谢相关调控基因表达的变化规律。本研究结果为槟榔的遗传多样性、分子遗传育种、遗传进化等研究提供大量的参考信息。
目前,已有研究表明在槟榔果实或种子中分离和鉴定出59种化合物,涉及生物碱、黄酮类化合物、单宁、三萜类和类固醇、脂肪酸等化合物,并证实其生物碱为槟榔药理活性主要成分[29-32]。本研究通过转录组测序技术对其次级代谢物相关基因进行了检索分析,从果皮中检索到次级代谢相关差异基因共33个基因,主要涉及苯丙烷生物合成途径,类黄酮生物合成途径,芪类、二芳基庚烷和姜醇生物合成途径,异喹啉类生物碱生物合成途径和萜类、哌啶和吡啶生物碱合成途径。从果核中检索到次级代谢相关差异基因共78个基因,主要涉及咖啡因生物合成途径,苯丙烷生物合成途径,类黄酮生物合成途径,黄酮和黄酮醇生物合成途径,芪类、二芳基庚烷和姜醇生物合成途径,异喹啉类生物碱生物合成途径和萜类、哌啶和吡啶生物碱合成途径。在槟榔果皮和果核中分别找到了参与生物碱代谢相关的差异基因,果皮中参与萜类、哌啶和吡啶生物碱合成途径的基因有4个(2个基因协同表达,2个基因呈下调表达);果核中参与萜类、哌啶和吡啶生物碱合成途径的基因为5个(2个基因协同表达,2个基因呈上调表达,1个下调表达)。这些研究对槟榔生物碱的生成调控机理研究具有重要意义。
有关槟榔转录组测序分析的研究目前仅有一则相关报道。Manimekalai等[33]对槟榔叶片进行了转录组分析,在叶片中发现了7种高度表达的类黄酮和萜类化合物生物合成的酶。本研究在果核中仅检测到查尔酮异构酶、类黄酮3-单加氧酶、白细胞花青素还原酶随时间的变化呈上调趋势。此外,Manimekalai等[33]在叶片中检测到泛醌和其他萜类代谢途径、黄酮类代谢途径和苯丙氨酸途径的部分基因呈现高表达量,而本研究中果皮和果核的差异基因所涉及的酶类并没有找到相应的酶,这可能与所用研究材料不同相关。
Wang等[10]在1997年就曾报道过槟榔的生物碱含量与其果实成熟度相关,刘蕊[11]的研究结果显示,成熟果胚乳中的槟榔碱含量高于青果胚乳,而成熟果果皮的含量低于青果果皮,这与本研究中萜类、哌啶和吡啶生物碱合成途径差异基因表达的结果基本一致。根据表6和表8的统计结果显示:随着果实的发育,果皮中次级代谢调控相关的下调表达基因数明显多于上调表达的基因,80%次级代谢通路差异相关基因呈下调表达趋势;而果核中的次级代谢调控相关基因上调表达基因数多于下调表达的基因,71.4%次级代谢通路差异相关基因呈上调表达趋势。上述比对结果表明,实验从转录组水平很好地验证了前人的研究结果,为槟榔生物碱合成的分子调控机理的下一步研究提供了数据支持。
[20] Tatusov R L, Galperin M Y, Natale D A,et al. The COG database: a tool for genome scale analysis of protein functions and evolution[J]. Nucleic Acids Research, 2000, 28(1): 33-36.
[21] Koonin E V, Fedorova N D, Jackson J D,et al. A comprehensive evolutionary classification of proteins encoded in complete eukaryotic genomes[J]. Genome Biology, 2004, 5(2): R7.
[22] Jaime H C, Damian S, Kristoffer F,et al. eggNOG 4.5: a hierarchical orthology framework with improved functional annotations for eukaryotic, prokaryotic and viral sequen ces[J]. Nucleic Acids Research, 2016, 44(Database issue): D286-D293.
[23] Kanehisa M, Goto S, Kawashima S,et al. The KEGG resource for deciphering the genome[J]. Nucleic Acids Research, 2004, 32(Database issue): D277-D280.
[24] Xie C, Mao X, Huang J,et al. KOBAS 2.0: a web server for annotation and identification of enriched pathways and diseases[J]. Nucleic Acids Research, 2011, 39: W316- W322.
[25] Eddy S R. Profile hidden Markov models[J]. Bioinformatics, 1998, 14(9): 755-763.
[26] Finn R D, Bateman A, Clements J,et al. Pfam: the protein families database[J]. Nucleic Acids Research, 2014, 42(Database issue): D222-230.
[27] Anders S, Huber W. Differential expression analysis for sequence count data[J]. Genome Biology, 2010, 11(10): R106.
[28] Leng N, Dawson J A, Thomson J A,et al. EBSeq: An empirical bayes hierarchical model for inference in RNA-seq experiments[J]. Bioinformatics, 2013, 29(16): 2073.
[29] Holdsworth D K, Jones R A, Self R. Volatile alkaloids fromAreca catechu[J]. Phytochemistry, 1998, 48(3): 581-582.
[30] 張 兴, 梅文莉, 曾艳波, 等. 槟榔果实的酚类化学成分与抗菌活性的初步研究[J]. 热带亚热带植物学报, 2009, 17(1): 74-76.
[31] 杨文强, 王红程, 王文婧, 等. 槟榔化学成分研究[J]. 中药材, 2012, 35(3): 400-403.
[32] 何细新, 李亚军, 胡小鹏, 等. 槟榔壳中三萜及蒽醌类成分的分离与结构鉴定[J]. 中药新药与临床药理, 2010, 21(6): 634-636.
[33] Manimekalai R, Nair S, Naganeeswaran A,et al. Transcriptome sequencing and de novo assembly in arecanut,Areca catechuL. elucidates the secondary metabolite pathway genes[J]. Biotechnology Reports, 2018, 17: 63-69.

