四川鲟源海豚链球菌的毒力基因谱及分子分型
白明焕,耿 毅,赵若璇,郭向辉,黄小丽,陈德芳,欧阳萍,汪 蕉
(1 四川农业大学 动物医学院,四川 成都 611130;2 四川农业大学 动物科技学院,四川 成都 611130)
海豚链球菌Streptococcus iniae作为一种重要的鱼类病原菌,其感染常致高发病率和病死率。自1972年首次从亚马逊淡水海豚Inia geoffrensis中分离得到以来,S.iniae先后在美国[1]、日本[2]、以色列[3]等地被报道,对全球的温水鱼养殖造成了巨大的影响并持续至今[4-8]。各国相继开展对海豚链球菌的相关研究,目前海豚链球菌至少存在2种生物型,且各型菌株的致病性有所差异[9-10],同时海豚链球菌还有scpI、simA、cpsD、sagA、pdi、pgm和cfi等多种毒力因子[11-14],各毒力因子之间的相互作用也是导致其致病性差异的重要原因,因此开展对海豚链球菌毒力谱及分型的研究对海豚链球菌病的防控,尤其是疫苗的研制具有重要的意义。
四川作为一个鲟科Acipenseridae养殖大省,在鲟鱼养殖过程中海豚链球菌病的危害较为严重[15-16],尽管对部分菌株进行了毒力基因谱与分型的研究,但对于四川地区各流行菌株的基因型、主要流行基因型、毒力基因谱,及是否发生变异等都不清楚。本研究采用随机扩增多态性DNA(Random amplified polymorphic DNA,RAPD)、重复序列 PCR(Repetitive element polymerase chain reaction,REPPCR)和多重PCR对17株鲟源海豚链球菌进行毒力基因谱与分子分型研究,以探明四川鲟源海豚链球菌分子流行病学特点,为有针对地筛选四川鲟海豚链球菌病疫苗候选株及防控提供参考。
1 材料与方法
1.1 材料和试剂
BHI培养基购自北京欣经科生物技术有限公司;PCR master mix、DL2000 DNA marker、细菌基因组DNA提取试剂盒、PCR产物纯化试剂盒购自天根生化科技 (北京)有限公司;API STREP 20 鉴定系统购自法国生物梅里埃公司;海豚链球菌ATCC29178株,购自ATCC菌种中心。
1.2 病原菌的分离和纯化
2016—2018 年从四川雅安、彭州、邛崃、蒲江等地送检的发病鲟鱼,首先用生理盐水冲洗体表,并用含体积分数75%的乙醇溶液的棉球进行体表消毒,然后用无菌的剪刀打开腹腔,分别从肝、脾、肾接菌至BHI培养基,并放置于28 ℃恒温培养箱进行细菌培养18~24 h,挑取BHI平板上菌落形态、大小一致的可疑菌落划线接种于BHI平板,28 ℃恒温培养18~24 h,挑取单菌落进行后续鉴定。获得纯化后的菌株4 ℃保存备用。共分离得到17株海豚链球菌。
1.3 病原菌的形态观察及lctO基因的特异性鉴定
取对数生长期的新鲜菌液观察其菌体形态,并按照细菌基因组DNA提取试剂盒说明书提取DNA。根据Mata等[17]的方法,设计合成S.iniae lctO基因的一对特异性扩增引物(表1)对其进行检测。PCR反应条件参照文献[15]。取5 μL扩增产物,10 g/L的琼脂糖凝胶,在恒压125 V下电泳35 min,置于凝胶成像系统观察PCR扩增结果并拍照记录。PCR产物经DNA纯化试剂盒纯化后,送成都擎科梓熙生物测序,并将分离株lctO基因序列与GenBank中己知核酸序列进行BLAST比对以确定其相似性。将已鉴定纯化后的细菌接种于BHI肉汤,28 ℃ 条件下震荡培养 18~24 h,将部分菌液用甘油保存于−20 ℃,部分菌液用脱脂奶粉冻干保存至−80 ℃备用。

表1 链球菌相关基因的引物信息Table 1 Primer information of related gene ofStreptococcus
1.4 病原菌的毒力基因谱检测
参照熊向英等[11]的方法设计合成S.iniae cfi毒力基因的引物,其余6个毒力基因scpI、simA、cpsD、sagA、pdi和pgm的扩增引物则参照Baums等[13]和邓梦玲等[16]的方法设计合成(表1),对分离株这些毒力基因进行多重PCR扩增。PCR体系及循环条件参照邓梦玲等[16]的程序进行。PCR扩增产物经20 g/L的琼脂糖凝胶电泳检测,将纯化回收的PCR产物送成都擎科梓熙生物测序。将获得的分离菌毒力基因序列和GenBank中已知核酸序列进行BLAST比对。
1.5 病原菌的分型
对17株鲟源海豚链球菌以及海豚链球菌ATCC 29178进行以下分型:
精氨酸双水解酶(Arginine dihydrolase,ADH)分型参照 API STREP 20鉴定系统使用说明书进行。
RAPD分型根据Bachrach等[10]的方法,以5′-GATCAAGTCC-3′为引物,对海豚链球菌进行随机扩增,25 μL 反应体系中含 2 μL 模板 DNA,2 μL 引物,12.5 μL PCR Master Mix(2×),8.5 μL ddH2O。PCR 反应条件:95 ℃ 预变性 5 min;95 ℃ 变性 30 s,38 ℃ 退火 1 min,72 ℃ 延伸 1 min,30 个循环;72 ℃延伸 10 min。
基因组REP-PCR参照Malathum等[18]的方法,以 5′-ACGTGGTTTGAAGAGATTTTCG-3′为引物,PCR 反应条件:95 ℃ 预变性 7 min;90 ℃ 变性 30 s,40 ℃ 退火 1 min,65 ℃ 延伸 8 min,35 个循环;65 ℃延伸16 min。PCR扩增产物用15 g/L琼脂糖凝胶于90 V电压电泳20 min,溴化乙锭染色,凝胶成像系统观察、拍照。获得的图像运用Quantity One v.4.62软件处理,选择UPGAMA聚类方法和条带位置差异容许度(Position tolerance)方法,条带位置差异容许度选择0.85%,优化值选择0.5%。
2 结果与分析
2.1 病原菌的分离与鉴定
2016—2018 年从四川雅安、彭州、邛崃、蒲江等鲟鱼养殖地分离到17株海豚链球菌,菌株详细信息见表2。在BHI平板28 ℃条件下恒温培养细菌24 h后,形成边缘光滑的乳白色菌落,为革兰阳性链状球菌(图1),lctO基因特异性检测为阳性(图2),扩增产物测序比对结果显示分离菌与GenBank数据库中S.iniae的相似性最高,达99%以上。
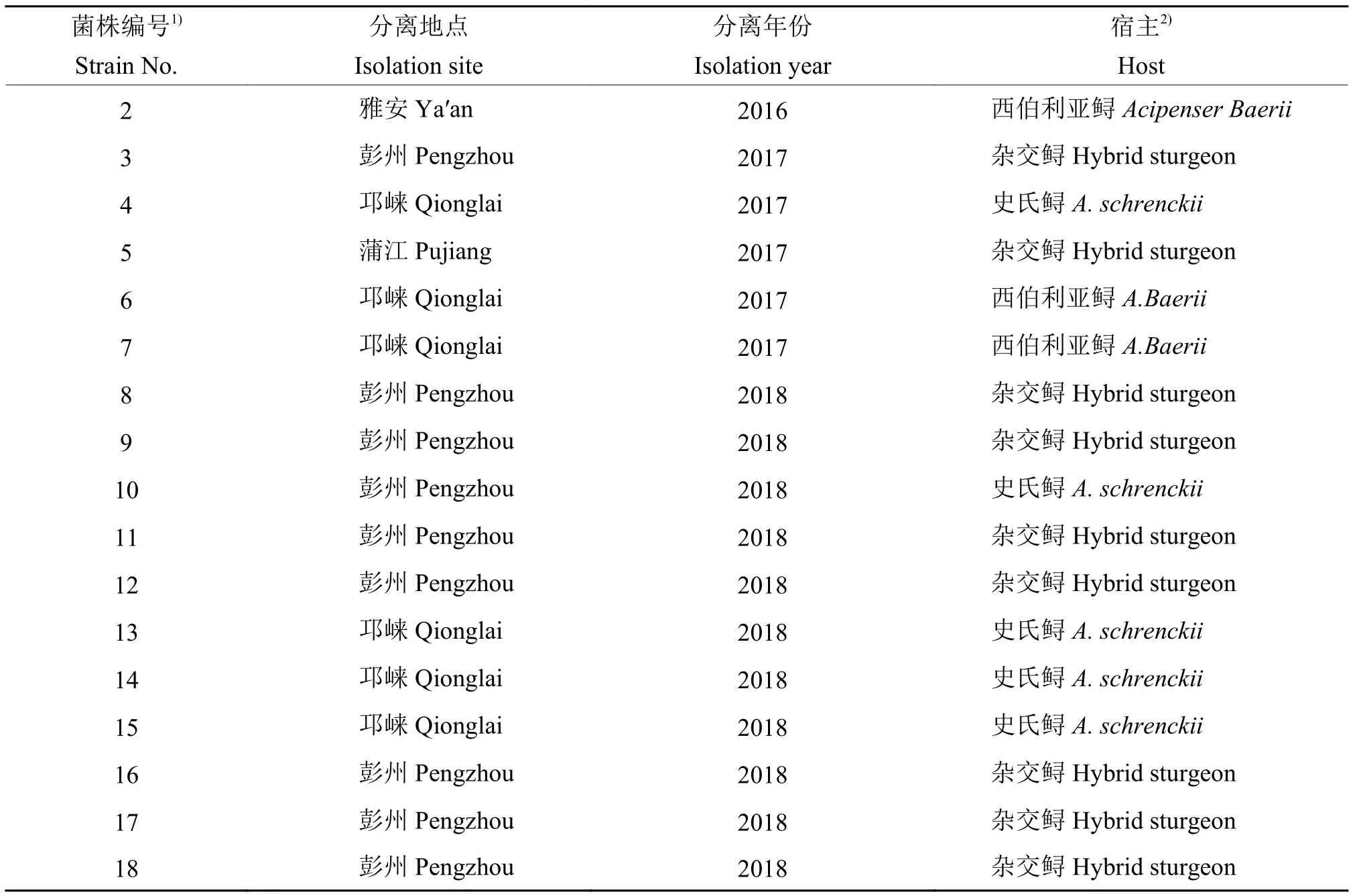
表2 四川患病鲟鱼中分离的海豚链球菌的信息Table 2 The information ofStreptococcus iniae isolated from diseased sturgeon in Sichuan
2.2 病原菌的分型
ADH 反应结果显示菌株 1、6、9、11、12、16、17和18为ADH阳性,其余菌株为ADH阴性,说明彭州地区的分离株大部分为ADH阳性,而雅安、蒲江以及大部分来自邛崃的分离株则为ADH阴性。
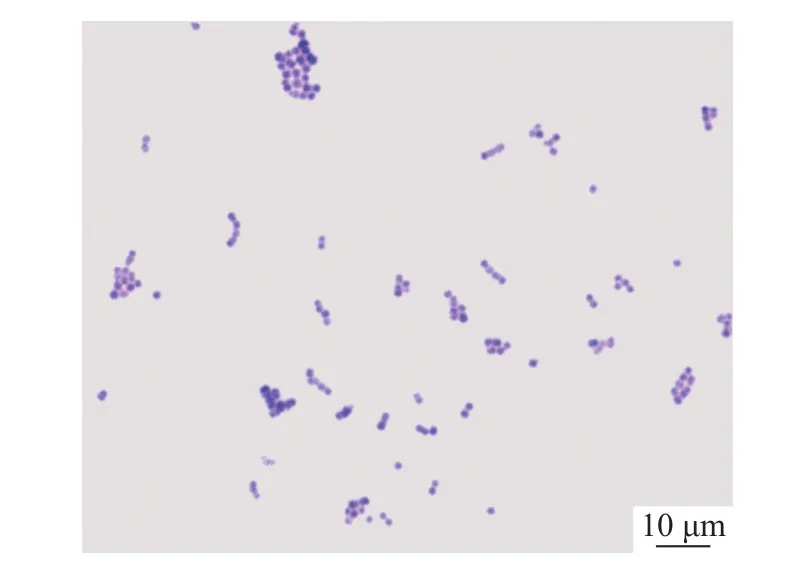
图1 海豚链球菌分离株革兰染色的显微形态Fig.1 Micromorphologies of isolated strains ofStreptococcus iniae in Gram staining
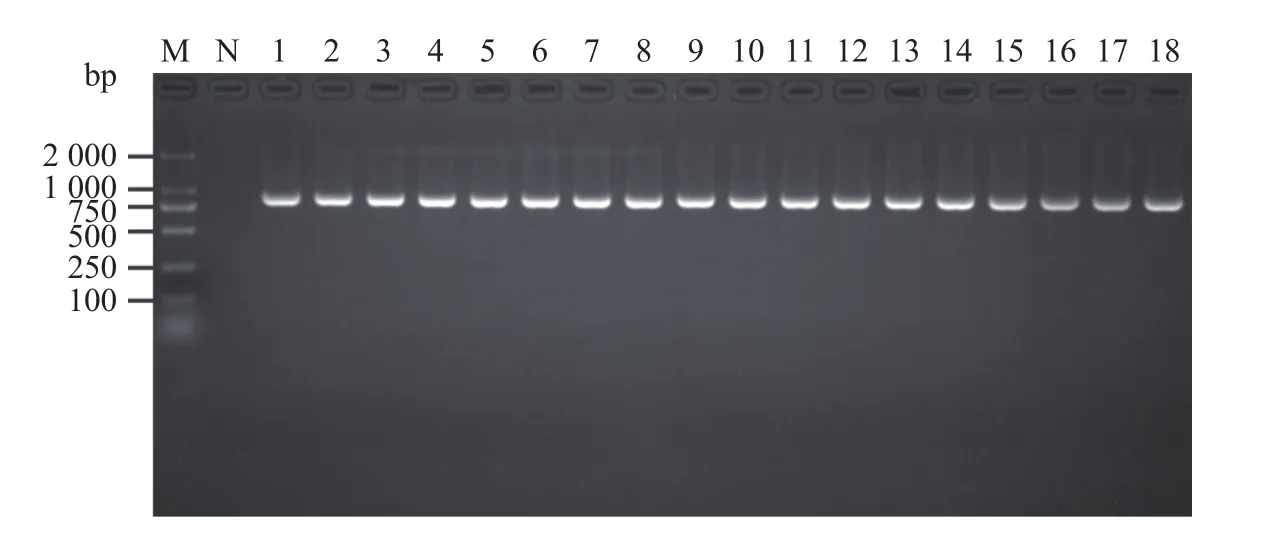
图2 海豚链球菌分离株lctO基因的PCR扩增产物电泳图Fig.2 Electrophoresis of PCR-amplifiedlctO genes from isolated strains ofStreptococcus iniae
通过对试验株进行RAPD分析显示出与ADH分型相似的结果,18株菌可分成2个基因型:Ⅰ型和Ⅱ型。其中雅安、蒲江、邛崃等地的分离株扩增出750 bp条带,属于Ⅰ型;而彭州地区的分离株未扩出750 bp条带,属于Ⅱ型(图3)。
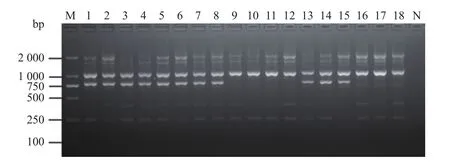
图3 海豚链球菌分离株的RAPD扩增图谱Fig.3 RAPD profiles of isolated strains ofStreptococcus iniae
通过对菌株进行REP-PCR(图4)聚类分析,18株菌共分为4个基因型:A、B、C、D型。菌株1、3、4、5、6、7、8、9、12、13、14、15、16、17 号为 D 型,2号菌株为B型,10、11号菌株为C型,18号菌株为A型,由此可见,D型为四川地区的优势流行型。
2.3 病原菌的毒力谱检测
对18株海豚链球菌进行毒力谱检测,结果(图5、图 6)显示pgm、scpI、pdi、cpsD、sagA和cfi基因符合预期片段,经测序鉴定为S.iniae。simA基因检测1号和2号菌株扩增出的条带符合预期结果,而3~18号菌株扩增出255 bp左右的条带,2号菌株和其他随机选取的3个菌株进行测序鉴定,4株菌扩增出的条带均为simA基因,相似性高达98%以上。将随机挑选菌株2、3、9和18的simA基因扩增产物纯化测序后上传NCBI数据库,并获得登录号MK959355、MK973066、MK973067 和 MK973068。
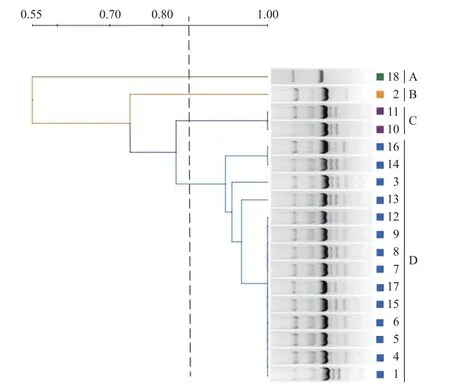
图4 基于REP-PCR对18株海豚链球菌进行聚类分析Fig.4 Clustering analysis of 18Streptococcus iniae strains based on REP-PCR
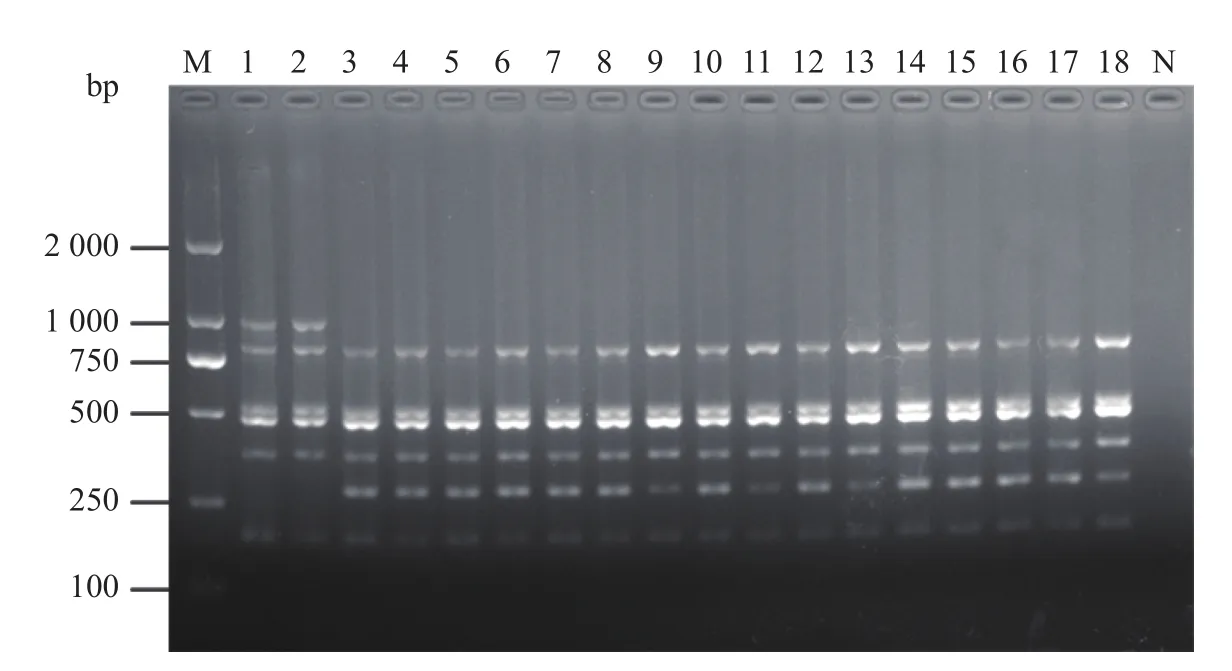
图5 海豚链球菌毒力基因的多重PCR检测Fig.5 Multi-PCR analysis of virulence genes fromStreptococcus iniae
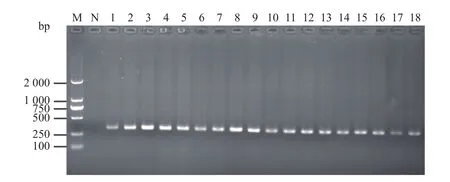
图6 海豚链球菌毒力基因cfi的PCR检测Fig.6 PCR analysis ofcfi virulence gene fromStreptococcusiniae
3 讨论与结论
S.iniae作为鱼类养殖中的主要致病菌[19],对世界鲟鱼养殖造成重大影响,目前有关S.iniae致病机制的研究主要包括动物感染模型的建立[20-21],感染后海豚链球菌在各组织的分布情况等[22-23]。现已证实S.iniae具有多种毒力因子[14]:主要通过调理作用使海豚链球菌免于巨噬细胞吞噬的荚膜和M样蛋白[14];参与细胞壁和荚膜的生物合成,并能抵抗正电性抗菌肽的磷酸葡萄糖苷酶(pgmA)[24];损伤宿主红细胞、粒细胞和单核细胞的细胞溶血素(sagA)[25];水解中性粒细胞化学诱导物补体因子C5a,从而损伤宿主具抗侵袭能力的C5a肽酶[26];增强细菌的抵抗力,并对宿主上皮细胞进行黏附和入侵的脱乙酰基酶[27];拦截抗体向补体呈递抗原的CAMP因子[28]等。这些毒力因子相互作用帮助S.iniae侵入宿主,逃避宿主的防御,引起宿主发病[29]。本研究对四川地区分离的17株鲟源海豚链球菌以及参考菌株进行7种主要毒力基因的检测,所有菌株均为阳性,其毒力谱均为pgm/scpI/simA/cpsD/saga/pdi/cfi,这表明了四川地区的鲟源海豚链球菌分离株均有较强的毒力。相关研究表明编码M样蛋白的simA基因是高度保守的毒力因子[30],其主要作用是帮助细菌黏附并入侵宿主各器官,同时还能抵抗宿主巨噬细胞的吞噬,并引起炎症反应。S.iniae感染往往造成鱼类宿主的急性死亡,而缺失simA基因的突变株抗吞噬能力明显降低[31]。本研究发现2016年后四川地区的分离株simA基因均发生了变异,经测序比对发现变异菌株的simA基因有部分序列的缺失,但simA基因变异发生的原因,以及变异对菌株毒力的影响还不清楚,这值得进一步的探究。
由于S.iniae各菌株分型复杂,又无法用Lancefield链球菌血清分类法进行分型[32],因此S.iniae究竟有多少种血清型尚不清楚。Barnes等[33]根据S.iniae对ADH的反应将以色列的分离株分成2种生物型,但随着研究的深入,该分型已无法满足需要。RAPD分析是建立在PCR基础上的一种可对整个未知序列的基因组进行多态性分析的分子技术,它不仅能反映生物个体间的遗传稳定性,也能敏感地反映出遗传差异。Bachrach等[10]通过研究发现生物Ⅰ型(ADH阳性)菌株在RAPD扩增中出现750 bp大小的条带,而生物Ⅱ型则不能扩增出,但我国多位学者先后对各地的S.iniae分离株进行分析的结果却不完全支持上述观点。黄婷等[34]和熊向英等[11]研究发现广西的分离株ADH反应为阴性,在RAPD扩增中却有750 bp大小的条带;周素明[35]的研究中分离株属于2个生物类群,但ADH反应均为阳性。本研究中部分菌株虽然为ADH阳性,但RAPD不能扩增出750 bp大小的条带,而有部分ADH阴性菌株却能扩增出750 bp的条带。为了进一步从分子水平上比较分离株,本研究采用REP-PCR进行分析,该方法是通过扩增细菌基因组中广泛分布的短重复序列,来揭示基因组间的差异。与RAPD相比,该方法可以更为准确地反映菌株间的差异。通过该方法将试验菌株明显分成了4个基因型。其中菌株9~12以及16~18在RAPD分型中为同一型,而REP-PCR分析则分成3个基因型。值得注意的是菌株9~12为不同时间同一地点的不同分离株,其遗传特征存在明显的差异,菌株16~18为同一地点相同时间的不同分离株,其遗传特征也存在多样性,这也进一步证实了四川地区存在多种基因型的菌株。同一环境中的多种血清型或基因型的菌株有利于菌株的基因重组或变异,而这种基因的重组或变异则可能增加病原菌突破宿主防御的能力和致病力,四川地区近年海豚链球菌对鲟鱼的危害越来越大是否与不同基因型的菌株共存导致基因突变有关,还需要探索。我们的研究发现四川海豚链球菌simA基因发生了变异,至于其他功能性基因有无变异,从而影响其致病力值得进一步研究。

