烟草TIR-NBS基因家族的生物信息学分析
董亚萍 杨仕梅 赵德刚 宋莉

摘 要: TIR-NBS基因是一类与植物抗病调节和生长发育密切相关的基因,其特征解析有助于对植物免疫和发育调节的认识。该研究通过生物信息学分析方法,从基因鉴定、染色体分布、基因结构、蛋白性质和结构、信号肽、亚细胞定位和顺式作用元件等方面,对烟草TIR-NBS基因家族进行了鉴定和分子特性分析。结果表明:烟草TIR-NBS基因家族含有30条成员,分布在21条染色体上,含有3~8个保守基序;编码蛋白均为不稳定蛋白,无信号肽,主要分布于细胞核、细胞质和叶绿体中,二级结构以α-螺旋和无规则卷曲为主要构成元件;进化时主要受纯化选择作用,与番茄的TIR-NBS基因亲缘关系最近;顺式作用元件分析表明其可能受光照、温度以及生长素、茉莉酸甲酯、脱落酸、水杨酸、赤霉素等激素调控。该研究结果为进一步探究TIR-NBS基因的生物学功能及提高植物产量和品质提供了依据。
关键词: 烟草, 抗病调节, 生长发育, 植物免疫, TIR-NBS基因家族, 生物信息学分析
中图分类号: Q943 文献标识码: A
文章编号: 1000-3142(2020)06-0891-10
开放科学(资源服务)标识码(OSID) :
Abstract: TIR-NBS gene is a class of gene closely related to plant disease resistance regulation and growth and development, and its characteristic analysis contributes to the understanding of plant immunity and developmental regulation. Through bioinformatics analysis method, the TIR-NBS gene family of tobacco was identified and molecularly analyzed from the aspects of gene identification, chromosome distribution, gene structure, protein properties and structure, signal peptide, subcellular localization and cis-acting elements. The results were as follows: The TIR-NBS gene family contained 30 members, distributed on 21 chromosomes, containing 3-8 conserved motifs; The encoded proteins were unstable proteins, no signal peptides, mainly distributed in the nucleus, cytoplasm and chloroplast; The secondary structure was characterized by α-helix and random coiling; It was mainly selected by purification in evolution, and had the closest relationship with TIR-NBS gene of tomato; Analysis of cis-acting elements showed that it may be regulated by light, temperature, and hormones such as auxin, methyl jasmonate, abscisic acid, salicylic acid, and gibberellin. The results provided a basis for further exploring the biological functions of the TIR-NBS gene and improving plant yield and quality.
Key words: Nicotiana attenuata(tobacco), disease resistance regulation, growth and development, plant immunity, TIR-NBS gene family, bioinformatics analysis
植物在長期遭受真菌、细菌、病毒等各种病原体侵袭和危害过程中,逐渐形成保护自身的抗病免疫机制,由R抗病基因(resistance gene)介导的免疫反应就是其中一条主要的途径 (房卫平等,2015;孙岩等,2017)。R基因由白细胞介素-1受体(toll and in-terleukin-1 receptor,TIR)结构域、功能结构域核苷酸结合位点(nucleotide bindingsite, NBS)、富含亮氨酸重复(leucine rich repeat, LRR)位点等结构组成。其中,TIR和NBS结构与抗病调节相关(Meyers et al., 2002),TIR-NBS类抗病基因作为一类重要的植物免疫基因,是R基因抗病免疫的重要基础(Staal et al., 2008;谢冬微等,2015) 。
TIR、NBS结构域是抗病基因免疫反应必需的。当拟南芥的抗病基因RPS4或RPP1A在仅含TIR和NB结构域的截短蛋白TIR+45或TIR+80片段时,即使没有病原菌效应蛋白的诱导也能引起细胞的坏死(Zhang et al., 2004;尹玲等,2018)。烟草抗病毒N蛋白的免疫作用与其TIR结构域进行信号转导有关(Dinesh-Kumar et al., 2000)。还有研究表明,TIR-NBS 类基因RLM3参与了拟南芥广谱抗坏死致病真菌的免疫反应过程(Staal et al., 2008)。尽管TIR-NBS类蛋白在植物抗病过程中扮演着非常重要的角色,但人们对该基因家族及其成员的了解仍极为有限。本研究通过生物信息学分析方法,对烟草TIR-NBS基因家族的分子特征和功能进行预测分析,为烟草TIR-NBS 类抗病基因的深入探究提供依据。
1 材料与方法
1.1 TIR-NBS基因家族鉴定
在Pfam中下载NBS(NB-ARC)(http://pfam.xfam.org/family/PF00931#tabview=tab6)和TIR(http://pfam.xfam.org/family/PF01582#tabview=tab6)隐马尔可夫模型。通过 hmmsearch 程序用下载的 NB-ARC.hmm和TIR.hmm文件对烟草全基因组蛋白数据库中的含TIR-NBS结构域的蛋白家族进行预测,选取的期望值E为1×10-20。
1.2 染色体定位分析
从Ensembl植物全基因组数据库(ftp://ftp.ensemblgenomes.org/pub/plants/release-42/fasta/nicotiana_attenuata)获得TIR-NBS基因在染色体上的位置信息,并用在线工具MG2C(http://mg2c.iask.in/mg2c_v2.0/)制作染色体定位图。
1.3 基因结构与motif分析
利用在线工具GSDS(http://gsds.cbi.pku.edu.cn/)对烟草TIR-NBS基因家族成员进行基因结构分析。通过在线工具meme(http://meme-suite.org/tools/meme)对烟草TIR-NBS蛋白motif进行预测分析。
1.4 蛋白结构与亚细胞定位分析
利用在线软件SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)分析TIR-NBS蛋白二级结构。利用在线工具SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)对烟草TIR-NBS基因进行信号肽分析。利用在线工具DeepLoc(http://www.cbs.dtu.dk/services/DeepLoc/)对烟草TIR-NBS基因进行亚细胞定位分析。
1.5 TIR-NBS蛋白的理化性质分析
利用ExPASy中的ProtParam (http://web.expasy. org/protparam/)软件对烟草TIR-NBS蛋白基本特性进行分析, 包括蛋白质大小、相对分子质量、理论等电点、不稳定系数、亲水性指数和脂溶性指数等指标。
1.6 进化选择压力分析
利用Blast建库比对和Calculator工具(Wang et al., 2010),对烟草TIR-NBS基因核苷酸的同义替换率(Ks)和非同义替换率(Ka)进行计算,获得基因Ka/Ks比率,进行选择压力分析。
1.7 系统发育分析
将检索得到的烟草(Nicotiana attenuata)、拟南芥(Arabidopsis thaliana)、番茄(Solanum lycopersicum)的TIR-NBS蛋白的氨基酸序列通过软件Clustal X进行TIR-NBS蛋白序列比对,然后利用MEGA-7.0 (Kumar et al., 2016)软件构建TIR-NBS基因家族邻接(NJ, neighbor-joining)系统发育树,Boostrap值设置为1 000,其他参数为系统默认值,最后使用FigTree(http://tree.bio.ed.ac.uk/software/figtree/)軟件展示系统发育树。
1.8 顺式作用元件分析
利用工具bedtools(Quinlan, 2014)获得TIR-NBS基因上游2 000 bp序列,利用在线网站PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对获得的序列进行预测,并对主要的顺式作用元件进行探讨。
2 结果与分析
2.1 TIR-NBS基因家族成员
根据Pfam数据库中TIR-NBS基因家族主要含有NB-ARC、TIR-NB-ARC-LRR、TIR-NB-ARC功能域的特征,通过构建隐马尔可夫模型,设置E-value为1×10-20,共鉴定到217条基因,去掉不含有TIR-NB-ARC结构的基因,最终获得30条烟草TIR-NBS基因。
2.2 TIR-NBS基因家族的染色体分布
对TIR-NBS基因染色体定位分析,图1结果显示,30条TIR-NBS基因不均匀地分布在21条染色体上。其中,有4条 TIR-NBS 基因在scaffold01866上,数量最多且以串联重复形式出现;其次是 1、10、11、scaffold00577、scaffold01435、scaffold02060号染色体分别有2条TIR-NBS 基因;其余染色体仅有1条TIR-NBS基因。
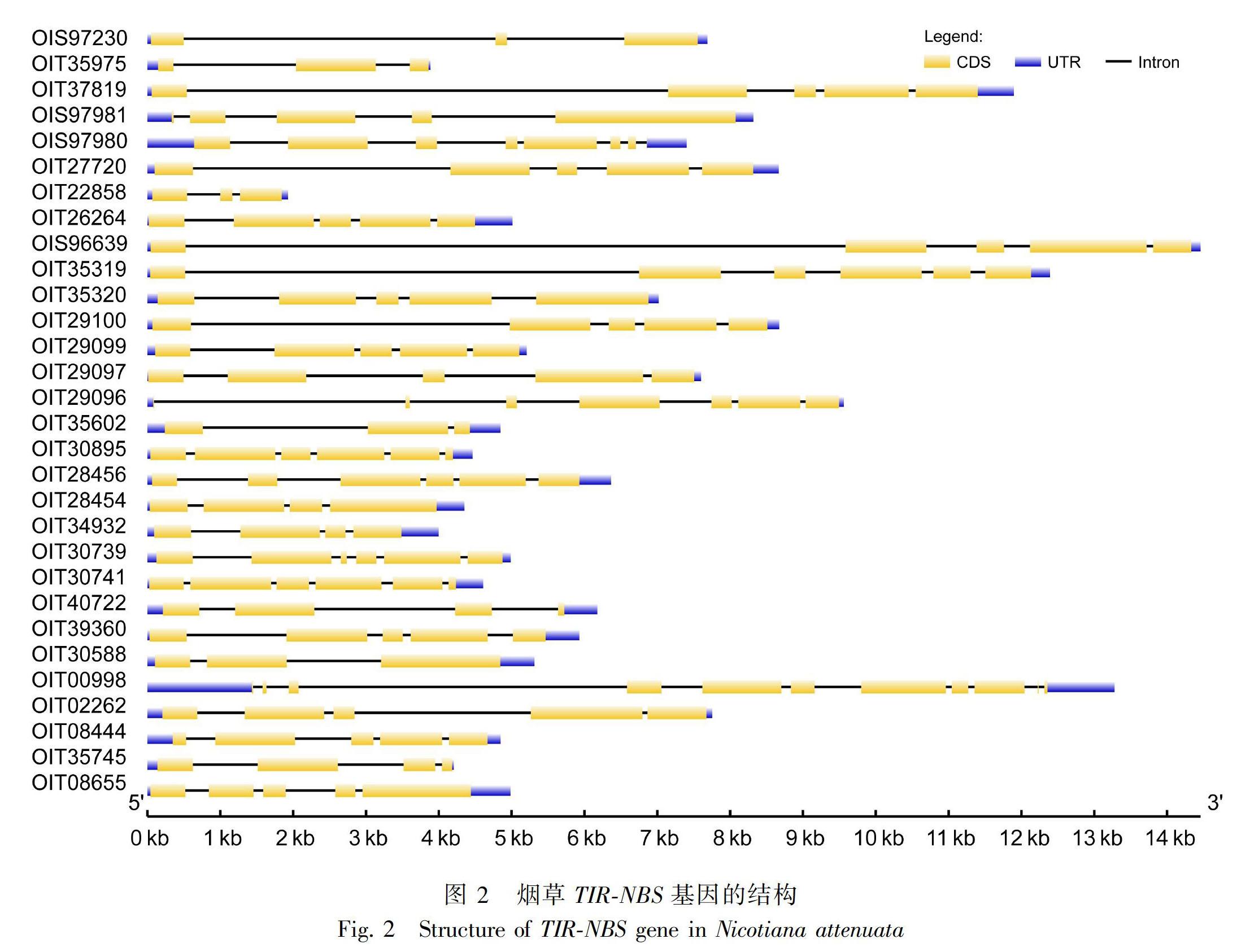
2.3 TIR-NBS基因的结构组成
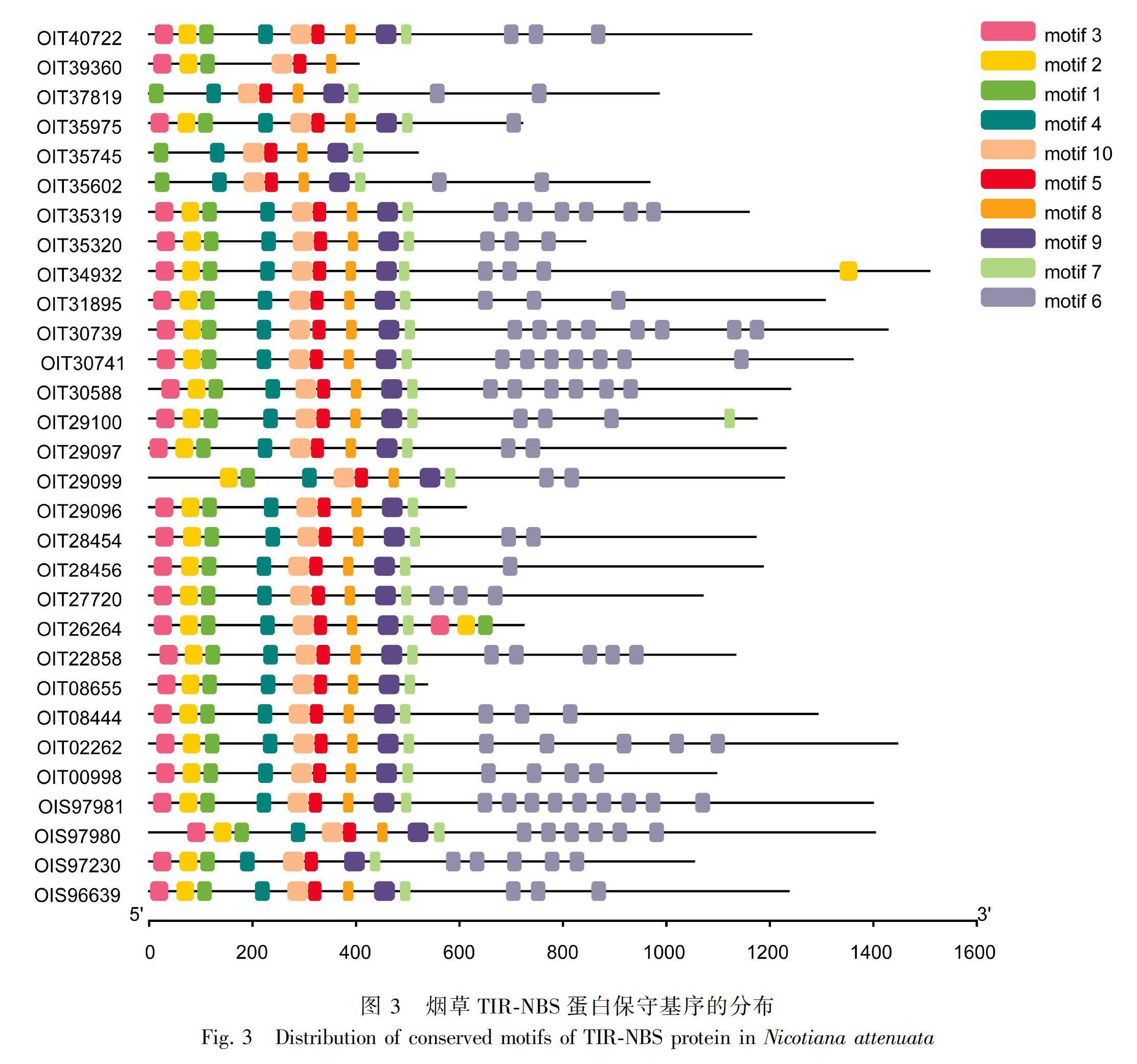
利用在线网站工具GSDS对30条烟草TIR-NBS 基因家族成员进行基因结构分析,图2结果显示,TIR-NBS基因家族外显子数量主要分布在3~8之间。利用meme对30条烟草TIR-NBS蛋白序列进行分析(图3),发现OIT39360所含基序最少,仅6个基序,其余所含基序都较多,最多为10个。说明该基因家族成员间在功能上可能存在差异。
2.4 TIR-NBS蛋白结构特点、细胞位置和信号肽
对30条烟草TIR-NBS蛋白的二级结构的预测结果表明,TIR-NBS蛋白由α-螺旋、延伸链、β-转角、无规则卷曲组成。其中,TIR-NBS蛋白的α-螺旋比例最高,其次是无规则卷曲。根据亚细胞定位分析,表1结果显示烟草TIR-NBS基因家族主要定位在细胞核、细胞质、叶绿体。信号肽分析结果表明,TIR-NBS基因家族成员均不含有信号肽。
2.5 TIR-NBS蛋白的理化性质
对烟草TIR-NBS家族蛋白的特征进行分析,表2结果表明烟草TIR-NBS蛋白长度范围在505~1 508 aa之间,相对分子质量大小范围为46 733.23~172 528.71 Da,理论等电点范围在5.78~9.40之间,编码氨基酸序列最长的是OIT34932,其相对分子质量也最大,为172 528.71 Da;OIT39360相对分子质量最小,为46 733.23 Da。分析表明,TIR-NBS蛋白为不稳定蛋白,其中OIT35745的不稳定系数最小,为33.98。TIR-NBS蛋白脂溶指数为90.09~104.23,总平均亲水性为-0.445~-0.115,均为亲水性蛋白。
2.6 TIR-NBS基因的进化选择
非同义替换(Ka)和同义替换(Ks)比值Ka/Ks可反映生物进化的过程中所受到的选择压力。对来源于TIR-NBS亚家族间亲缘关系最近的10条基因成员进行Ka/Ks分析,表3结果显示该基因对间的Ka/Ks比值均小于1,表明烟草TIR-NBS基因在进化时主要受纯化选择作用。紫色为拟南芥,青色为番茄,红色字体标记为烟草。
2.7 TIR-NBS基因家族的系统发育树
为了进一步了解烟草TIR-NBS基因家族进化关系,利用烟草(30条)、番茄(27条)、拟南芥(176条)共233条TIR-NBS蛋白序列构建NJ系统发育树(图4),由图可知,30条TIR-NBS基因家族成员与番茄的TIR-NBS基因家族亲缘关系相近。
2.8 TIR-NBS基因家族的顺式作用元件组成
通过对基因上游2 000 bp序列进行分析, 预测基因的顺式作用元件(图5)。TIR-NBS 基因家族除了存在大量的基本元件CAAT-box和TATA-box外,还存在参与光响应顺式作用调节元件ACE、AE-box、AT1-motif、Box 4、CAG-motif、GA-motif、GT1-motif、G-box,胚乳表达所需顺式作用调节元件GCN4-motif,参与种子特异性调节的顺式作用调节元件RY-element。此外,TIR-NBS基因家族中还含有与分生组织表达相关的顺式作用调节元件CAT-box、茉莉酸甲酯反应的顺式作用调节元件CGTCA-motif、水杨酸反应顺式作用元件TCA-ele-ment、低温响应顺式元件LTR、防御和应激反应顺式作用元件TC-rich repeats、昼夜节律控制顺式作用调节元件circadian、生长素反应顺式作用元件AuxRR-core、TGA-element以及赤霉素反应顺式作用元件TATC-box、P-box等。分析表明,TIR-NBS基因可能受光、 温度及生长素、 茉莉酸甲酯、脱落酸、水杨酸、赤霉素等激素调控,并可能通过这些顺式作用元件参与了烟草中的防御机制,在植物生长过程中起到防御和保护植物的作用。
3 讨论
TIR-NBS类抗病相关基因是植物抗病免疫系统中的一个重要组成部分。它主要的抗病调节结构域包括TIR和NBS两部分,其中植物抗病蛋白(R蛋白)中TIR结构域主要存在于拟南芥和其他双子叶植物中(Meyers et al., 2002),而在单子叶植物中,仅发现了非TIR序列(Tarr, 2009)。TIR结构域是大约有200个氨基酸的蛋白质结构域,包括三个高度保守的基序(Slack et al, 2000)。目前,已经在部分生物中鉴定了TIR-NBS结构域,并证明它与植物致病诱导蛋白和其他植物信号转导蛋白的相互作用(Nandety et al., 2013)。但到目前为止,在烟草基因组水平上,仅有少数TIR-NBS类基因家族被鉴定,对于大多数该类基因鲜有报道。
对烟草与拟南芥、番茄TIR-NBS类基因家族进化关系分析表明,烟草和番茄的TIR-NBS基因同源性较高,因此烟草和番茄的TIR-NBS基因家族在生物学功能、生长调控机制上可能存在相似性。当植物受到病原菌侵害时,水杨酸作为发生超敏反应及形成系统获得性抗性所需要的内源信号分子(陈冲等,2018),其通过诱导病原相关蛋白(pathogenesis-related protein, PR蛋白)等多种抗病蛋白产生SAR起作用(汪尚等,2016)。茉莉酸甲酯参与种子萌发、根生长、生育、果实成熟和衰老等多种发育过程的调节(丛汉卿,2015)。此外,茉莉酸甲酯还能激活植物防御机制,对昆虫引起的伤害、各种致病菌和环境胁迫(如干旱、低温和盐度)作出反应(Cheong & Choi, 2003)。通过对TIR-NBS基因顺式作用元件分析表明,TIR-NBS基因家族中含有防御和应激反应、低温响应、光响应、脱落酸反应、MeJA反应、水杨酸响应、赤霉素响应、分生组织表达等顺式作用元件,可知其不但与植物生长发育有关,还参与免疫应激反应调节,这与上述报道是相符的。
在烟草TIR-NBS类抗病基因家族中,被报道的基因数目很少,大多数基因的功能到目前为止还不清楚。本研究通过生物信息学方法概括了烟草基因组中的TIR-NBS类抗病基因的鉴定、基因结构及顺式作用分析等,对于发掘新的与植物抗病性相关的基因,加快烟草中抗病机制的研究具有十分重要的意义,也为烟草的抗病育种研究奠定了基础。
参考文献:
CHEN C, LIU S, WANG DD, et al., 2018. Identification of cucumber PCD induced by salicylic acid and expression analysis of related genes [J]. Acta Agric Boreal-Sin, 33(6): 56-63. [陳冲, 刘双, 王丹丹, 等, 2018. 水杨酸诱导黄瓜PCD的鉴定及相关基因的表达分析 [J]. 华北农学报, 33 (6): 56-63.]
CHEONG JJ, CHOI YD, 2003. Methyl jasmonate as a vital substance in plants [J]. Trends Genet, 19(7): 409-413.
CONG HQ, QI YY, ZHANG ZW, et al., 2015. Sequence ana-lysis of cassava EIN3 gene and its induced expression characteristics of ethylene and methyl jasmonate [J]. Mol Plant Breed, 13(12): 2705-2712. [丛汉卿, 齐尧尧, 张振文, 等, 2015. 木薯EIN3基因的序列分析及其乙烯和茉莉酸甲酯诱导表达特性 [J]. 分子植物育种, 13(12): 2705-2712.]
DINESH-KUMAR SP, THAM WH, BAKER BJ, 2000. Structure-function analysis of the tobacco mosaic virus resistance gene N [J]. Proc Natl Acad Sci USA, 97: 14789-14794.
FANG WP, XIE DY, LI ZF, et al., 2015. NBS-LRR-like disease-resistant protein-mediated molecular mechanisms of plant disease resistance [J]. Mol Plant Breed, 13(2): 469-474. [房卫平, 谢德意, 李志芳, 等, 2015. NBS-LRR类抗病蛋白介导的植物抗病应答分子机制 [J]. 分子植物育种, 13(2): 469-474.]
KUMAR S, STECHER G, TAMURA K, 2016. Mega7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets [J]. Mol Biol Evol, 33(7): 1870-1874.
MEYERS BC, MORGANTE M, MICHELMORE R, 2002. TIR-X and TIR-NBS proteins: Two new families related to disease resistance TIR-NBS-LRR proteins encoded in Arabidopsis and other plant genomes [J]. Plant J, 32(1): 77-92.
NANDETY RS, CAPLAN JL, CAVANAUGH K, et al., 2013. The role of TIR-NBS and TIR-X proteins in plant basal defense responses [J]. Plant Physiol, 162(3): 1459-1472.
QUINLAN AR, 2014. BEDTools: The Swiss-Army tool for genome feature analysis [J]. Curr Protoc Bioinform, 47(1): 11.12.11-11.12.34.
SLACK JL, SCHOOLEY K, BONNERT TP, et al., 2000. Identification of two major sites in the type I interleukin-1 receptor cytoplasmic region responsible for coupling to pro-inflammatory signaling pathways [J]. J Biol Chem, 275: 4670-4678.
STAAL J, KALIFF M, DEWAELE E, et al., 2008. RLM3, a TIR domain encoding gene involved in broad-range immunity of Arabidopsis to necrotrophic fungal pathogens [J]. Plant J, 55(2): 188-200.
SUN Y, LIU BL, YUAN LX, et al., 2017. Identification and analysis of NBS resistance gene family of flax carp [J]. Mol Breed, 15(4): 1417-1425. [孙岩, 刘宝玲, 苑丽霞, 等, 2017. 亚麻荠NBS类抗病基因家族的鉴定与分析 [J]. 分子植物育种, 15(4): 1417-1425.
TARR DEK, ALEXANDER HM, 2009. TIR-NBS-LRR genes are rare in monocots: Evidence from diverse monocot orders [J]. BMC Res Notes, 2(1): 197.
WANG DP, ZHANG YB, ZHANG Z, et al., 2010. Ka Ks_Calculator 2.0: A toolkit incorporating gamma-series methods and sliding window strategies [J]. Genom Proteom Bioinform, 8(1): 77-80.
WANG S, XU LQ, ZHANG YX, et al., 2016. Research progress of salicylic acid-mediated disease resistance in plants [J]. Acta Physiol Sin, 52(5): 581-590. [汪尚, 徐鹭芹, 张亚仙, 等, 2016. 水杨酸介导植物抗病的研究进展 [J]. 植物生理学报, 52(5): 581-590.
XIE DW, LU Y, ZHAO DB, et al., 2015. Genome-wide analysis of flax NBS disease resistance gene families [J].Plant Fiber Sci Chin, 37(3): 113-119. [谢冬微, 路颖, 赵德宝, 等, 2015. 亚麻NBS类抗病基因家族全基因组分析 [J]. 中国麻业科学, 37(3): 113-119.]
YIN L, FANG H, HUANG Y, et al., 2016. Research progress on various domains of plant TIR-NB-LRR type disease resistance genes [J]. Guihaia, 37(2): 186-190. [尹玲, 方辉, 黄羽, 等, 2016. 植物TIR-NB-LRR类型抗病基因各结构域的研究进展 [J]. 广西植物, 37(2):186-190.]
ZHANG Y, DOREY S, SWIDERSKI M, et al., 2004. Expression of RPS4 in tobacco induces an AvrRps4-independent HR that requires EDS1, SGT1 and HSP90 [J]. Plant J, 40(2): 213-224.
(責任编辑 周翠鸣)

