小麦抽穗和开花期相关QTL定位与分析
王克森 董爽爽 李法计 郭军 台述强 王利彬 程敦公 穆平 刘建军 李豪圣 赵振东 曹新有 张玉梅

摘要:抽穗期和开花期是衡量小麦发育快慢及稳定性的两个重要时期,发掘相关基因位点并应用于育种选择,可为小麦稳产性改良提供指导。本研究以人工合成六倍体小麦(Turtur)和Triticum spelta L.衍生系(Bubo)为亲本杂交构建的包含186个家系的重组自交系(recombinant inbred line,RIL)群体(F6)为材料,于2014—2017年连续种植于山东省农业科学院作物研究所济南试验基地,结合已构建的包含5 301个标记、长度为2 464 cM的高密度遗传连锁图谱对抽穗期和开花期QTL进行定位,结果表明,在1B(3)、2B、4B(2)、5A、5B和7B染色体上共检测到9个抽穗期相关QTL,可解释4.55%~13.40%的表型变异;在1D、2A、3D、4B、5A、6B和7B染色体上共定位到7个开花期相关QTL,可解释3.48%~16.93%的表型变异。其中,7B染色体上4409103~1233594标记区间内控制抽穗期的QTL连续两年被检测到;4B和7B染色体上控制开花期的QTL分别连续2年和3年被检测到。这将为下一步分子标记辅助育种和品种稳产性改良提供理论依据。
关键词:小麦;抽穗期;开花期;QTL
中图分类号:S512.103.2 文献标识号:A 文章编号:1001-4942(2020)01-0017-07
Abstract Heading time and flowering time are two important traits to measure the growth and stability of wheat. Discovering relevant gene loci and applying them to breeding can provide guidance for wheat yield improvement. In this study, a RIL (recombinant inbred line) population (F6) consisting of 186 families was constructed by artificially synthesizing hexaploid wheat (Turtur) and T. spelta L. derived line(Bubo) as parents. The materials were continuously planted in Jinan Experimental Base of the Crop Research Institute of Shandong Academy of Agricultural Sciences in 2014-2017. The QTL mapping was carried out at the heading and flowering stages in combination with the constructed high-density genetic linkage map containing 5 301 markers and a length of 2 464 cM. The results showed that 9 heading QTLs were mapped on the chromosomes 1B (3), 2B, 4B (2), 5A, 5B and 7B, which explained 4.55% ~ 13.40% of phenotypic variation, and 6 flowering QTLs were mapped on the chromosomes 1D, 2A, 3D, 4B, 5A, 6B and 7B, explaining 3.48% ~ 16.93% of phenotypic variation. Among them, the QTL of heading time in the 4409103~1233594 marker interval on chromosome 7B was detected for 2 consecutive years; the QTL of flowering time on chromosomes 4B and 7B were detected in 2 and 3 years respectively. It could provide theoretical references for molecular maker-assisted breeding and stable-yielding improvement.
Keywords Wheat; Heading time; Flowering time; QTL
小麥是世界上最重要的谷物资源之一,是我国第三大粮食作物,其产量对我国的粮食安全有着重要影响。抽穗期及开花期可反映小麦的生长发育速率及稳定性,并影响成熟期、产量、抗病性等。抽穗期及开花期标志着小麦由营养生长转向生殖生长,是决定小麦产量的关键时期[1]。
大量研究表明,小麦抽穗期及开花期是受多基因控制的数量性状,遗传基础复杂,易受环境影响。小麦抽穗期受春化(Vrn)、光周期(Ppd)和早熟性(Eps)基因的协同调控[2]。其中,春化和光周期基因都会与环境发生互作,而早熟性基因独立于春化和光周期基因之外,在满足春化和光周期反应的情况下,控制花原基细胞的起始及发育[3]。王羽等[4]通过多世代联合分析,认为小麦抽穗期符合一对主基因+多基因的遗传模型。近年来,关于分析定位小麦抽穗期QTL的研究已有较多报道。姚琴等[5]在两年两点环境下采用重组自交系群体在5DL和7B染色体上检测到3个抽穗期QTL,可以解释表型变异的8.30%~49.80%。Sourdille等[6]利用DH群体将抽穗期QTL定位在1A、2B、4D、4BS、5AL、5DL、7BS、2D、5A、4A、2A和2D染色体上。吴旭江等[7]利用重组自交系群体在3年9次试验中将抽穗期QTL定位在1A、4D、5B、6B和7A染色体上,可以解释表型变异的5.76%~15.26%。宋彦霞等[8]利用三个作图群体,在不同环境下检测到5个抽穗期QTL,分别位于3A、4D、5B、5D、6A染色体上,可以解释表型变异的3.97%~22.91%。开花期是小麦对水分较敏感的时期[9],直接影响小麦产量。闫雪等[10]采用DH群体,在干旱胁迫下检测到2个分别位于1B和1D染色体上的开花期QTL,分别解释表型变异的12.35%和10.79%;正常灌溉条件下检测到2个位于1D和5B染色体上的开花期QTL,分别解释表型变异的9.11%和9.65%。虽然已报道小麦抽穗期和开花期位点较多,但少有位点用于育种选择,且控制抽穗期和开花期的位点存在差异。
本研究选用人工合成六倍体小麦和T. spelta L.衍生系杂交构建的RIL群体为材料,对小麦抽穗期和开花期QTL进行定位,旨在发掘稳定遗传的抽穗期和开花期位点,并为分子标记辅助育种和品种稳产性改良提供理论依据。
1 材料与方法
1.1 试验材料
供试材料选用CIMMYT提供的以人工合成六倍体小麦(Turtur)和T.spelta L.衍生系(Bubo)为亲本(系谱见表1)构建的186个家系的RIL(F6)遗传群体。该群体由中国农业科学院作物研究所郝元峰博士提供。
1.2 试验方法
1.2.1 田间试验和性状调查 将RIL群体(F6)的186个家系及亲本于2014—2015、2015—2016和2016—2017年连续种植于山东省农业科学院作物研究所济南试验基地。试验采用单行区种植,行长1 m,行距25 cm,设3次重复,完全随机排列,两亲本作为对照。田间管理参照大田管理进行。
每行内半数穗子(不包括芒)由叶鞘中露出1/2时,即为进入抽穗期,以播种至抽穗的天数作为抽穗期;每行内半数穗子中上部小花的内外颖张开、花药散粉时,即进入开花期,以播种至开花的天数作为开花期。
1.2.2 统计分析 利用SPSS 17.0统计软件进行基本统计量分析,应用Microsoft Excel 2010绘制性状分布图。
1.2.3 DNA提取及分子标记检测 基因组DNA采用CTAB法[11]提取,通过琼脂糖凝胶电泳检测DNA质量,并用分光光度计检测OD260/280值,根据DNA浓度稀释保存。委托中玉金标记(北京)生物技术股份有限公司(http://sample.cgmb.com.cn)进行SNP标记分型,对质检后样品进行一系列的处理,包括线性扩增、片段化和沉淀、重悬、变性,然后利用GeneTitan MC Instrument进行杂交、洗染、扫描,产生的数据通过Axiom Analysis Suit进行分析。DarT标记参考Triticarte Pty. Ltd.整合在多个遗传群体上的信息(http://www.triticarte.com.au)。
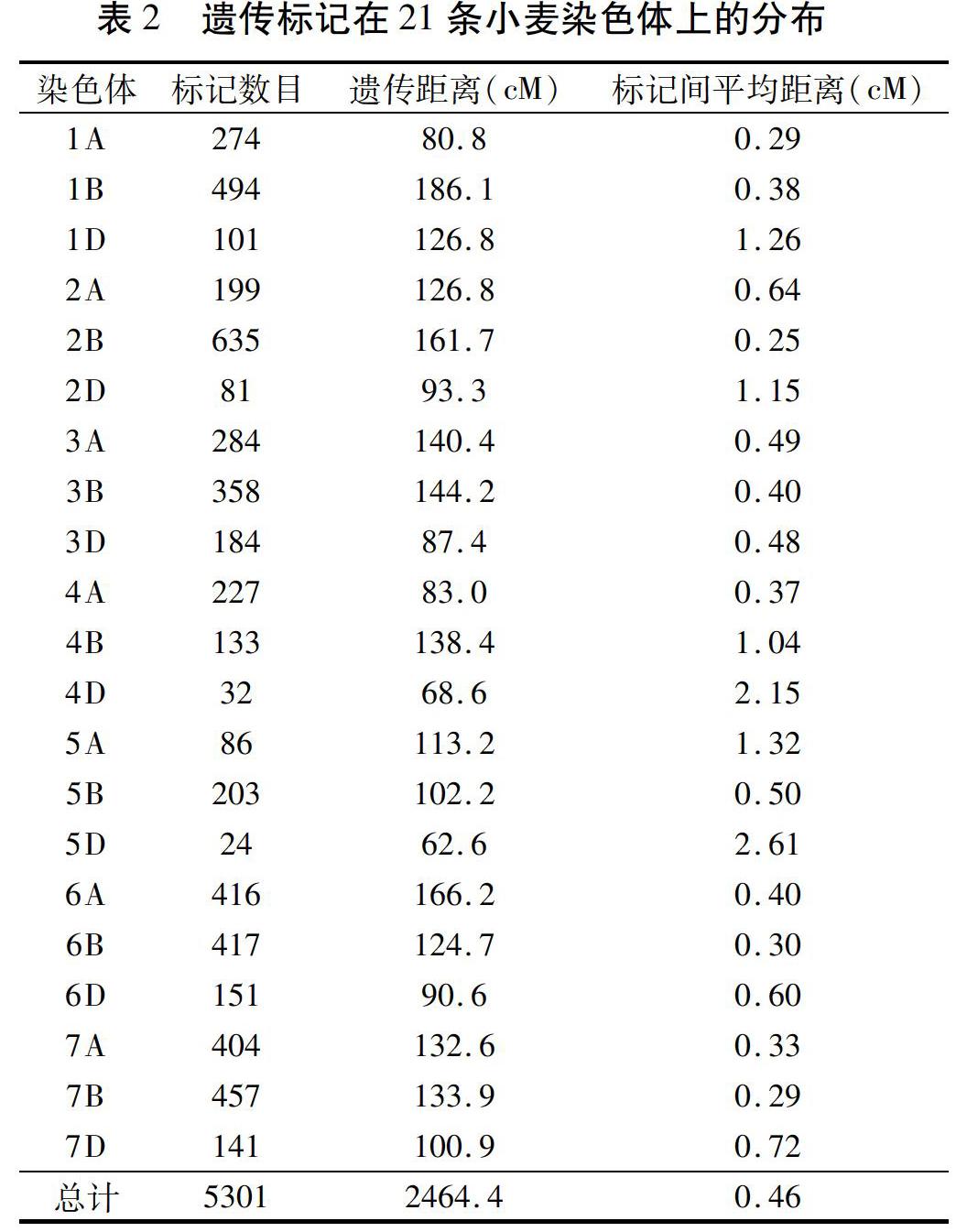
1.2.4 QTL定位 已构建了包含5 301个标记(4 120个DarT标记、621个SNP标记和560个传统的DarT标记)、总长度2 464 cM的遗传连锁图谱,标记间平均距离为0.46 cM[12](表2)。
根据构建的连锁图谱利用Windows QTL Cartograph 2.5软件采用复合区间作图法检测QTL,扫描步长设置为1.0 cM,对群体及亲本进行全基因组扫描,分析结果定位QTL的效应以及在染色体上的位置。根据Churchill等[13]的方法,在P<0.05水平下进行1 000次的随机性测验。当阈值(LOD)大于2.5时,认为存在一个QTL,并计算每个QTL的贡献率和加性效应值。QTL的命名采用McIntosh等[14]的方法命名:Q+性状名称缩写+染色体编号+QTL个数(如有多个QTL)。
2 结果与分析
2.1 RIL群体及其亲本抽穗期与开花期性状的表型分析
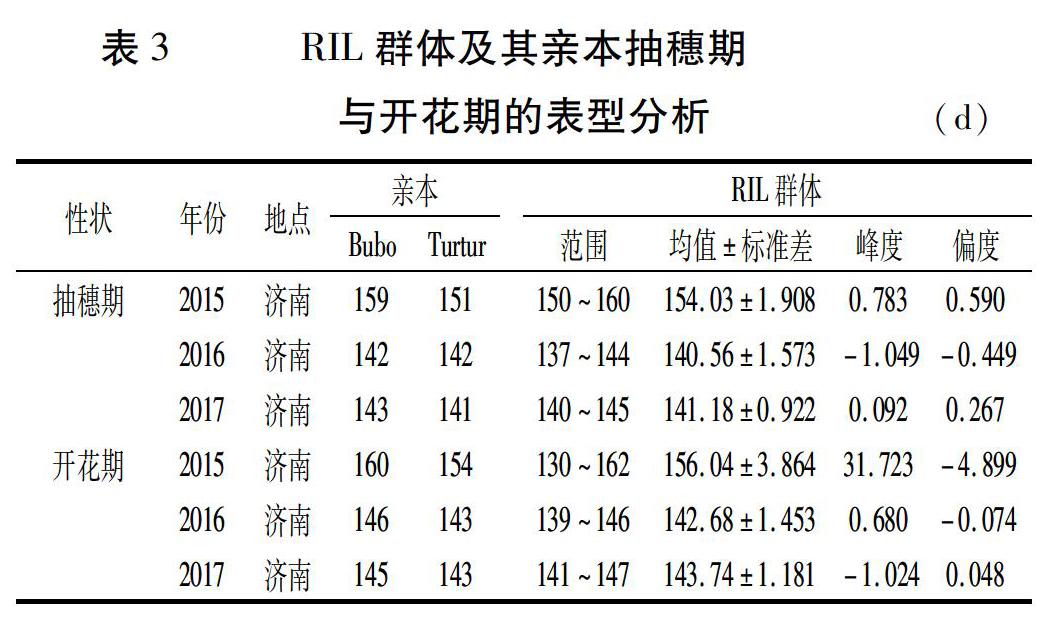
田间调查RIL群体及其亲本抽穗期与开花期,利用SPSS 17.0统计软件进行基本统计量分析,结果(表3)表明,亲本Turtur比Bubo早抽穗2~8 d,但2016年两亲本在济南的抽穗期相同,可能与当年小麦生长发育后期遭遇高温天气有关。亲本Turtur比亲本Bubo早开花2~6 d。RIL群体抽穗期和开花期性状具有明显的超亲分离现象,说明两亲本含有对表型变异起作用的基因。图1表明,抽穗期性状与开花期性状呈连续变化,且符合正态分布,具有数量性状遗传特点,因此,可以进行QTL定位分析。
2.2 QTL定位分析
2.2.1 抽穗期QTL定位分析 利用Windows QTL Cartographer 2.5軟件的复合区间作图法进行QTL检测,在LOD>2.5水平下,共定位到10个抽穗期QTL,分布于小麦的1B、2B、4B、5A、5B和7B染色体上(表4和图2)。
1B染色体上检测到2个效应值较大的QTL,一个位于1208560~3938149标记区间内,LOD值为6.14,加性效应为-0.68,可解释11.08%的表型变异,暂命名为qDTH-1B-1;另一个位于3021374~1136122标记区间内,LOD值为5.63,加性效应为0.63,可解释10.02%的表型变异,暂命名为qDTH-1B-2。4B染色体上检测到1个效应值较大的QTL,位于1126368F0-33CT~2303406标记区间内,LOD值为5.46,加性效应为-0.53,可解释10.36%的表型变异,暂命名为qDTH-4B-1。5A染色体上检测到1个效应值较大的QTL,位于2280217~2259418标记区间内,LOD值为5.20,加性效应为0.28,可解释8.28%的表型变异,暂命名为qDTH-5A。
另外,连续2年在7B染色体4409103~1233594标记区间内检测到抽穗期相关QTL,平均解释了9.56%的表型变异,暂命名为qDTH-7B。
2.2.2 开花期QTL定位分析 利用Windows QTL Cartographer 2.5软件的复合区间作图法进行QTL检测,在LOD>2.5水平下,共定位到10个开花期相关QTL,分布于小麦的1D、2A、3D、4B、5A、6B和7B染色体上(表5和图2)。
6B染色体上检测到1个效应值较大的QTL,位于1115431~2321040标记区间内,LOD值为5.15,加性效应为-1.26,可解释10.51%的表型变异,暂命名为qDTF-6B。连续2年在4B染色体3064397~1106144区间检测到开花期QTL,平均解释了4.65%的表型变异,暂命名为qDTF-4B。另外,连续3年在7B染色体wPt-7318~1278402标记区间内检测到开花期QTL,平均解释了9.93%的表型变异,暂命名为qDTF-7B。
3 讨论
小麦的抽穗期和开花期作为数量遗传性状,遗传基础复杂,表型易受环境影响。前人有关抽穗期和开花期的QTL分析,也发现同一作图群体在不同年份很难得到完全相同的QTL[15]。本研究结合SNP标记与DarT标记对人工合成六倍体小麦(Turtur)和T. spelta L.衍生系(Bubo)构建的RIL遗传群体进行全基因组扫描,构建了总长为2 464 cM的遗传连锁图谱,共包括5 301个标记,采用复合区间作图法对小麦抽穗期和开花期的QTL进行初步定位。利用3年表型数据在1B、1D、2A、2B、3D、4B、5A、5B、6B和7B染色体上共检测到20个与抽穗期和开花期性状相关的QTL位点,且多个位点与遗传标记近乎共分离。
对抽穗期性状定位发现,位于1B染色体1208560(45.8 cM)~3938149(46.0 cM)区间内的qDTH-1B-1(45.81 cM)与1208560标记遗传距离为0.01 cM,近乎共分离,可解释11.08%的表型变异;3021374(149.6 cM)~1136122(149.7 cM)区间内的qDTH-1B-2(149.61 cM)与3021374标记遗传距离为0.01 cM,近乎共分离,可解释10.02%的表型变异。另外,qDTH-7B(50.51 cM)连续两年在7B染色体4409103(50.5 cM)~1233594(51.0 cM)区间内被检测到,与4409103标记遗传距离为0.01 cM,近乎共分离,平均解释了9.56%的表型变异。Zhang等[16]在不同环境中对光温敏不育系BS366的抽穗期进行研究,检测到8个QTL,其中1B染色体30.54 cM上存在一个抽穗期QTL(2年被检测到),可解释3.06%和2.42%的表型变异;4B染色体23.76 cM上存在一个抽穗期QTL,可解释2.43%的表型变异;7B染色体2.01 cM和4.01 cM位置上检测到2个抽穗期QTL,分别解释了2.41%和2.54%的表型变异。Huang等[17]利用German elite×synthetic BC群体检测到13个抽穗期相关QTL,分别位于2A、2D、3A、3B、4A、5A、5B、6A、7A、7B和7D染色体上。师翠兰[18]采用山农01-35×藁城9411构建的RIL群体,检测到12个抽穗期QTL。其中QHt1B.1-87两年被检测到,分别解释了17.08%和30.32%的表型变异;QHt1B.2-44两年被检测到,分别解释了9.54%和9.43%的表型变异,与本研究中qDTH-1B-1(45.81 cM)位置接近。茹京娜等[19]利用DH群体,检测到6个位于1B、1D、4D、6B、7B和7D染色体上的抽穗期QTL,解释了表型变异的1.82%~6.72%。
研究发现,小麦抽穗期QTL多分布在1B、4B和7B等B組染色体上[5],本试验在1B、2B、4B、5B和7B染色体上检测到抽穗期相关QTL,说明B组染色体上可能富含控制抽穗期的QTL,对抽穗期性状的调控有较大的影响。本次试验中定位的qDTH-1B-1、qDTH-1B-2和qDTH-7B均与可用遗传标记近乎共分离,有望应用于分子辅助选择育种。
对开花期性状定位发现,7B染色体wPt-7318(51.6 cM)~1278402(51.8 cM)区间内的qDTF-7B(51.61 cM)连续3年被检测到,与wPt-7318标记的遗传距离为0.01 cM,近乎共分离,可解释6.12%~16.93%的表型变异。Bomer等[20]利用ITM Ⅰ中的一个包含有140个家系的RIL群体在2DS(54.2~73.9 cM)、5DS(126.0~141.7 cM)和3AL(258.0~284.0 cM)上检测到开花期主效QTL,在2BS上检测到1个开花期的微效QTL。杨睿等[3]利用波兰小麦和普通小麦品系中13个杂交后代的F8重组自交系检测到6个位于2B、4B、5A、5B、6A、7A染色体上的开花期QTL,解释了表型变异的9.64%~20.27%,并发现4B染色体与小麦成熟期密切相关。同时,在小麦染色体7BS上还存在控制小麦开花的基因TaFT,定位在染色体短臂GWM569~ABC158区间内,与标记ABC158的遗传距离为1 cM[21]。本试验在7B染色体不同区间内定位的qDTF-7B(51.61 cM)位于TaFT基因附近,可连续3年被检测到,我们通过分析发现1278402标记与TaFT基因遗传距离为1 Mb,极有可能是同一个基因,有待于进一步进行验证,表明7B染色体与小麦生育期性状的相关性,并可能还存在控制生育期的微效基因,今后工作中应重视7B染色体对小麦生育期的调控作用。
4 结论
本研究利用构建的包含5 301个标记、长度为2 464 cM的高密度遗传连锁图谱对小麦抽穗期和开花期进行QTL定位。多个位点与遗传标记近乎共分离,其中,抽穗期共检测到9个QTL位点,分布于1B、2B、4B、5A、5B和7B染色体上;开花期共检测到7个QTL位点,分布于1D、2A、3D、4B、5A、6B和7B染色体上。7B染色体上同时检测到抽穗期与开花期主效QTL,且qDTF-7B 与小麦开花基因TaFT位置接近,疑为同一基因。本研究结果可为小麦抽穗期和开花期QTL精细定位以及相关育种标记选择提供参考。
参 考 文 献:
[1] 王建革, 孙宝启, 黄友志.小麦抽穗期的遗传控制[J].遗传,2002, 24(2):86-89.
[2] Hoogendoorn J. A reciprocal F1 monosomic analysis of the genetic control of time of ear emergence, number of leaves and number of spikelets in wheat (Triticum aestivum L.)[J]. Euphytica,1985,34(2):545-558.
[3] 杨睿, 杨兴圣, 刘联正, 等.小麦熟期相关性状的QTL定位分析[J].麦类作物学报,2012,32(3): 398-403.
[4] 王羽, 樊庆琦, 张利, 等.小麦K35早熟特性的遗传分析[J].麦类作物学报, 2007, 27(6):957-960.
[5] 姚琴, 周荣华, 潘昱名, 等.小麦品种偃展1号与品系早穗30重组自交系群体遗传连锁图谱构建及重要农艺性状的QTL分析[J].中国农业科学, 2010, 43(20):4130-4139.
[6] Sourdille P, Snape J W, Cadalen T, et al. Detection of QTLs for heading time and photoperiod response in wheat using a doubled-haploid population[J].Genome, 2000, 43(3):487-494.
[7] 吴旭江, 臧淑江, 程凯, 等. 小麦重组自交系群体抽穗期QTL分析[J]. 江苏农业学报, 2015, 31(6):14-20.
[8] 宋彦霞, 景蕊莲, 霍纳新, 等.普通小麦(T. aestivum L.)不同作图群体抽穗期QTL分析[J].中国农业科学, 2006, 39 (11): 2186-2193.
[9] 谷俊涛, 刘桂茹, 屈平, 等.不同抗旱类型小麦品种开花期光合特性的研究[J].河北农业科学, 2001, 5(1):9-15.
[10] 闫雪, 史雨刚, 李晓宇, 等.小麦开花期QTL分析[J].山西农业科学, 2015, 43(8):919-921.
[11] Saghai-Maroof M A,Soliman K M,Jorgensen R A,et al.Ribosomal DNA spacer-length polymorphisms in barley:Mendelian inheritance, chromosomal location, and population dynamics[J]. Proceedings of the National Academy of Sciences of the United States of America, 1984, 81(24):8014-8018.
[12] Velu G, Tutus Y, Gomez-Becerra H F, et al. QTL mapping for grain zinc and iron concentrations and zinc efficiency in a tetraploid and hexaploid wheat mapping populations[J]. Plant and Soil, 2017, 411(1/2):81-99.
[13] Churchill G A, Doerge R W. Empirical threshold values for quantitative trait mapping[J]. Genetics,1994,138(3):963-971.
[14] McIntosh R A. Catalogue of gene symbols for wheat[J]. Cereal Research Communications, 1975, 3(1):69-71.
[15] 宋彥霞. 小麦抽穗期及其它农艺性状的QTL分析[D]. 雅安:四川农业大学,2005.
[16] Zhang L P, Xu X Q, Zhao C P,et al. QTL mapping of heading stage for photoperiod-thermo sensitive male sterile line BS366 in wheat [J]. Agricultural Science & Technology,2010,11(6):45-48.
[17] Huang X Q , Kempf H , Ganal M W , et al. Advanced backcross QTL analysis in progenies derived from a cross between a German elite winter wheat variety and a synthetic wheat (Triticum aestivum L.)[J]. Theoretical and Applied Genetics, 2004, 109(5):933-943.
[18] 师翠兰. 小麦“山农01-35×藁城9411”RIL群体遗传图谱构建及主要产量和品质性状QTL分析[D]. 泰安:山东农业大学, 2012.
[19] 茹京娜, 于洋, 董凡凡, 等.小麦抽穗期QTL及其与环境的互作[J].麦类作物学报, 2016, 34(9):1185-1190.
[20] Bomer A, Schumann E, Furste A, et al. Mapping of quantitative trait locus determining agronomic important characters in hexaploidy wheat (Triticum aesticum L.) [J].Theoretical and Applied Genetics,2002,105(6/7):921-936.
[21] Yan L, Fu D , Li C , et al. The wheat and barley vernalization gene VRN3 is an orthologue of FT[J]. Proceedings of the National Academy of Sciences, 2006, 103(51):19582-19586.

