小麦耐盐相关基因TaRSTR的克隆与功能分析
王玉 连娟 王聪 赵利利 赵宝存
摘要:高盐是限制植物生长和发育的重要非生物胁迫因子。以耐盐小麦RH8706-49根部基因表达谱芯片结果为基础,利用电子克隆和RT-PCR方法克隆了一个盐胁迫时上调表达的基因,命名为TaRSTR(登录号:EU263918)。荧光定量PCR分析证实该基因受盐胁迫诱导表达,亚细胞定位结果显示TaRSTR蛋白定位在细胞核里。TaRSTR过表达提高了转基因拟南芥在盐胁迫下的种子萌发率及成年植株的耐受性。TaRSTR基因過表达可显著提高已知耐盐相关基因AtFRY1、AtP5CS1和AtRD29B的表达量,推测TaRSTR基因通过这些标记基因提高了转基因拟南芥的耐盐性。上述结果表明TaRSTR基因过表达能提高植株的耐盐性,是植物耐盐的正调节子。
关键词:小麦;TaRSTR基因;耐盐;实时定量PCR
中图分类号:S512.103.4:Q781 文献标识号:A 文章编号:1001-4942(2020)01-0010-07
Abstract High salinity is a very important abiotic stress factor that inhibits the growth and development of plant. The gene expression profile of salt-tolerant wheat mutant RH8706-49 under salt stress was investigated by microarray chip, and according to the results, a gene induced by salt stress was cloned and named TaRSTR (accession number: EU263918). The real-time PCR analysis results confirmed that TaRSTR was indeed induced by salt stress. TaRSTR was located in nuclear. TaRSTR overexpression improved the seed germination rate and the salt tolerance of adult transgenic Arabidopsis plants under salt stress. TaRSTR overexpression increased the expression levels of abiotic stress related marker genes, such as AtFRY1, AtP5CS1 and AtRD29B. It was predicted that TaRSTR improved the salt tolerance of transgenic Arabidopsis thaliana via these marker genes. In summary, TaRSTR gene was invovled in salt-stress response and could positively regulate the salt tolerance in plant.
Keywords Triticum asetivum L.; TaRSTR gene; Salt tolerance; Quantitative real-time PCR
高盐是严重影响农作物产量的非生物胁迫因子之一。在漫长的进化过程中,植物进化出了对盐的耐受机制。植物耐盐是一个涉及多基因的遗传性状[1,2]。植物通过改变耐盐相关基因的表达来调整细胞的生理活动过程,减轻盐胁迫造成的伤害或提高植物对高盐的耐受力[3.4]。在众多的耐盐相关基因中,RING/U-box家族基因在植物耐盐方面起到重要作用。RING是Really Interesting New Gene的缩写,RING结构域(RING finger domain,RFD)是一种特殊类型的锌指结构,具有40至60个残基,可结合两个锌原子。含RFD的蛋白不都具有规则的锌指特征,其中,U-box就是RING结构域的一种修饰形式,是由三个β-折叠和一个α-螺旋组成的RING结构。RING/U-box家族蛋白种类繁多、功能多样,在植物中分布最为广泛[5]。RING/U-box蛋白参与蛋白质之间的相互作用及泛素化[6,7],并在各种细胞生命活动过程中发挥关键作用,包括植物对生物和非生物胁迫的耐受性[8,9] 。
对拟南芥中RING/U-box蛋白的研究显示该家族在多种非生物胁迫过程中起作用,包括干旱、高盐、冷和热等[10,11]。油菜的RING/U-box蛋白也参与了冷、盐和脱氢胁迫过程[8]。最近的研究表明,RING/U-box蛋白在植物应对盐胁迫的响应中起着重要作用。番茄中的一个RING/U-box蛋白编码基因SpRing是胁迫诱导型基因,参与了野生番茄(Solanum pimpinellifolium)的盐胁迫信号传导。SpRing在拟南芥中过表达增强了种子在发芽期间对盐的耐受性,同时使某些关键胁迫相关基因的表达水平发生了变化[12]。水稻OsSIRP2基因编码RING/U-box蛋白,其受盐诱导表达,OsSIRP2过表达提高了水稻对盐和渗透胁迫的耐受性,该蛋白介导植物在非生物胁迫和信号转导过程中的反应[13]。水稻RING/U-box蛋白编码基因OsRMT1在拟南芥中过表达提高了转基因拟南芥对盐胁迫的耐受性,OsRMT1的降解受盐胁迫抑制,推测OsRMT1参与了水稻的盐耐受机制[14]。拟南芥中一个受ABA和其他胁迫处理诱导的AtAIRP4基因编码RING/U-box蛋白。与野生型和atairp4突变株系相比,AtAIRP4过表达植物在种子萌发期间对盐和渗透胁迫更加敏感,表明AtAIRP4是耐盐的负调控因子[15]。
小麦(Triticum aestivum)作为一种重要的农作物被广泛种植,高盐环境是其生长、发育、繁殖的一个重要限制因素,会造成小麦产量和品质降低。因此,克隆小麦耐盐相关基因,探索小麦在盐胁迫下的应激机制以及抗逆性适应机理,对提高小麦耐盐性有极其重要的意义。本研究根据小麦耐盐突变体RH8706-49进行NaCl胁迫处理后根部基因的表达谱芯片分析结果,得到一个盐胁迫初期表达上调的EST探针,根据EST序列克隆了该基因,并对其表达模式、亚细胞定位和耐盐功能进行初步分析,为该基因的功能研究奠定理论基础。
1 材料与方法
1.1 试验材料及植物培养方法
小麦耐盐突变体RH8706-49(本研究室保存),耐盐指数≥1.3[16]。将小麦种子在水中浸泡过夜,萌发后25℃光照(16 h光/8 h暗)培养,长至二叶一心时用175 mmol/L NaCl处理,于处理0、1、6、24、72 h时分别取叶片和根为材料,用于基因表达模式分析。野生型拟南芥(Arabidopsis thaliana)Col-0种子(本研究室保存)用75%乙醇和2.5%次氯酸钠表面消毒,铺于MS培养基上,4℃春化3~4 d后于22℃培养箱中培养(16 h光/8 h暗),9 d后移到蛭石营养土中,于22℃温室光照(16 h光/8 h暗)培养。
大肠杆菌(Escherichia coli)菌株DH5α、根癌农杆菌(Agrobacterium tumefaciens)菌株GV3101、真核表达载体pRTL2-AN-mGFP和pCAMBIA1300-35S均为本实验室保存。
1.2 TaRSTR基因克隆及生物信息学分析
根据小麦RH8706-49盐胁迫下根部基因的表达谱芯片结果,我们发现探针(Ta.3452.3.S1_a_at)在盐胁迫早期上调表达,上调至最高值之后下调[17]。以该探针的EST序列作为起始序列,利用电子拼接获得全长cDNA的逻辑序列,以胁迫后RH8706-49的根部cDNA为模板进行RT-PCR扩增,对扩增片段进行克隆测序。所用引物序列见表1。对克隆到的基因进行保守结构域分析(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi),预测该基因的生物学功能。
1.3 RNA的提取和实时定量PCR
利用Trizol (天根生化科技有限公司,北京)试剂分别提取不同植物材料的总RNA,参照TaKaRa公司的Prime ScriptTMⅡ Reverse Transcriptase试剂盒进行反转录,得到cDNA后进行实时定量PCR。实时定量PCR在Rotor-Gene 3000定量PCR仪(Gene Company Limited, USA)上进行,分析软件为RG3000 6.0版软件(Gene Company Limited, USA)。小麦β-actin基因(登录号:AB181991)为内参,分析方法为ΔΔCt比较定量法,每一样品3次生物学重复。定量引物序列见表1。
1.4 亚细胞定位载体的构建及转化拟南芥
参照Yoo等[18]的方法将TaRSTR 的cDNA序列连接到载体pRTL2-AN-mGFP中,得到TaRSTR∷GFP融合表达载体,通过PEG介导转化拟南芥叶肉细胞原生质体,对转化细胞进行激光共聚焦显微镜观察,根据荧光的位置确定蛋白质的亚细胞定位。空载体pRTL2-AN-mGFP转化的细胞作为对照。构建载体的引物序列见表1。
1.5 过表达载体的构建及转化拟南芥
将TaRSTR cDNA序列插入双元表达载体pCAMBIA1300-35S中,获得过表达重组载体。采用浸花法[19]将该重组载体转化拟南芥Col-0。构建载体的引物序列见表1。获得转基因植株后,分别进行DNA水平(基因组DNA的PCR)和RNA水平(半定量RT-PCR)鉴定。确定转基因是否成功。半定量RT-PCR以Atactin1(登录号:AT2G37620)为内参。引物序列见表1。
1.6 转基因拟南芥耐盐性分析
为了鉴定TaRSTR与耐盐的关系,我们对不同发育时期的转基因株系进行耐盐性鉴定。首先,将转基因拟南芥株系的种子消毒后分别接种于MS0和含有120 mmol/L NaCl的MS培养基上,7 d后统计萌发率。其次,将正常培养5周的植株用175 mmol/L NaCl溶液浇灌,每4 d浇灌一次,处理8 d后观察表型。每个处理均做3次生物学重复。
1.7 过表达TaRSTR基因对其他耐盐基因表达的影响
通过荧光定量PCR分析拟南芥中耐盐相关基因AtRD29B、AtP5CS1、AtFRY1和AtSOS3在TaRSTR过表达转基因植株和对照Col-0植株中的表达量,分析TaRSTR基因可能参与的盐胁迫应答信号途径,以Atactin2(登录号:AT3G18780)为内参。定量PCR应用的基因引物序列见表1。
2 结果与分析
2.1 TaRSTR基因的克隆及保守结构域分析
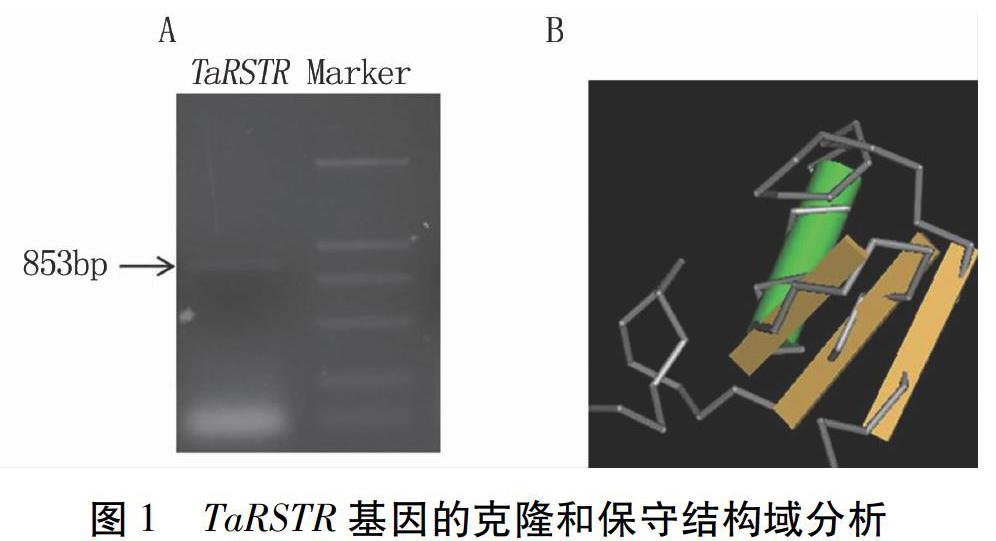
利用RT-PCR技术,我们扩增到一个853 bp的cDNA片段(图1A),对该序列进行克隆和测序,结果表明该序列包括756 bp的开放阅读框,编码包含251个氨基酸的蛋白质,该蛋白中存在一个RING/U-box结构域 (图1B)。该基因命名为TaRSTR (RING salt-tolerant relative gene),序列已提交到GenBank,登录号为EU263918。
2.2 TaRSTR基因在盐胁迫下的表达量分析
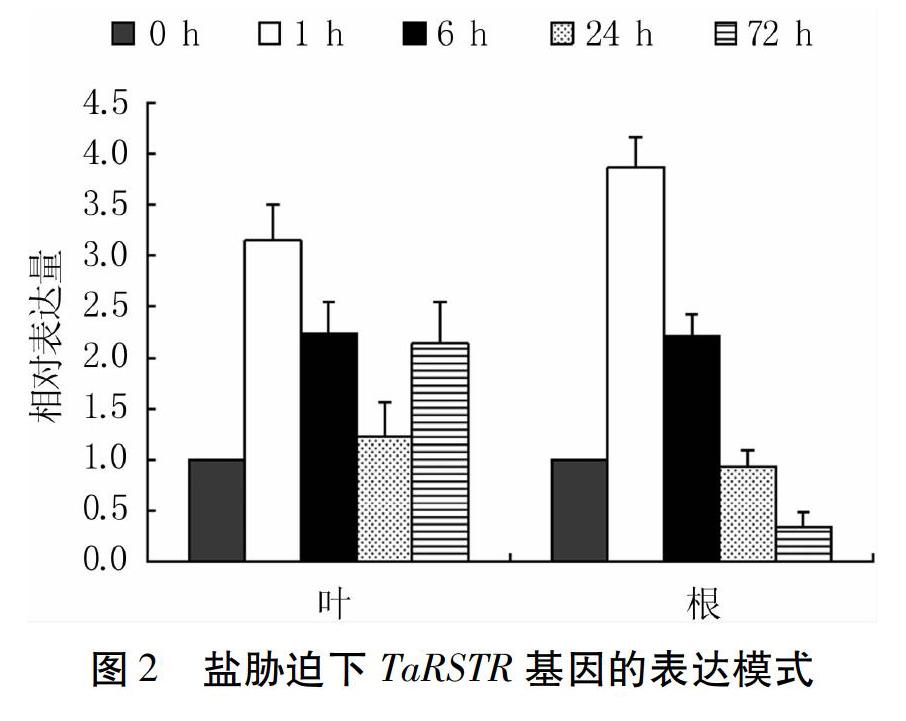
为了检测TaRSTR基因在盐胁迫下的表达量,以二叶一心期的RH8706-49小麦幼苗为材料,在175 mmol/L NaCl胁迫0、1、6、24、72 h時分别取叶片和根,进行实时定量PCR,分析TaRSTR基因在胁迫不同时间点的叶片和根中的表达量。结果表明,无论是在根中还是在叶中,TaRSTR基因在盐处理1 h时表达量最高;之后随处理时间延长,在叶中先下降后升高,胁迫72 h时TaRSTR基因的表达量是未胁迫(0 h)时的2倍;而在根中自1 h时达到最高值后,TaRSTR基因的表达量持续下调(图2),表明TaRSTR基因在盐胁迫初期即被诱导表达,但是在叶中和根中的表达模式不一样。
2.3 TaRSTR的亞细胞定位
以GFP空载体为对照,将TaRSTR∷GFP融合表达载体转化拟南芥叶肉细胞原生质体,用激光共聚焦显微镜观察TaRSTR蛋白的亚细胞定位。结果表明,对照GFP的荧光分布在细胞核、细胞质和细胞膜上(图3A),而TaRSTR∷GFP融合蛋白的荧光只分布在细胞核里(图3B),表明TaRSTR定位在细胞核内。
2.4 TaRSTR过表达提高转基因拟南芥的耐盐性
对获得的3个转基因纯合体株系(OE-1,OE-2,OE-3)进行基因组水平和mRNA水平的检测,结果(图4)发现,PCR和RT-PCR扩增结果中,对照Col-0中都没有目的条带出现,而3个转基因纯合体株系中都有TaRSTR基因的扩增条带,表明TaRSTR基因已经整合进拟南芥基因组(图4D),并已经表达(图4E)。
以Col-0为对照,对TaRSTR过表达转基因株系在NaCl处理下的萌发率进行检测。正常培养条件下,3个转基因株系与对照的种子萌发率没有明显差异(图4A、C),而120 mmol/L NaCl胁迫下,转基因株系的萌发率均显著高于对照Col-0(图4B、C)。
以Col-0为对照,对正常培养35 d的野生型和TaRSTR过表达转基因株系进行175 mmol/L NaCl浇灌处理,处理8 d后观察表型,结果发现盐处理前,对照植株和转基因植株的长势基本一致(图5A),而盐处理后对照Col-0的株高较矮、叶片枯萎,转基因植株的生长状态相对良好,植株更挺拔,果荚更多、更饱满,结实率更高(图5B)。上述结果表明,TaRSTR过表达能够提高转基因拟南芥在不同发育阶段的耐盐性。
2.5 TaRSTR过表达对拟南芥中耐逆相关标记基因表达量的影响
为了从分子水平探索TaRSTR的耐盐机理,对拟南芥中已报道的与植物非生物胁迫耐受性密切相关的4个基因(AtRD29B、AtP5CS1、AtFRY1和AtSOS3)进行表达量分析。选取耐盐性鉴定结果中耐盐性较强的过表达株系OE-1(图5B)为材料,以Col-0为对照,采用实时定量PCR鉴定不同标记基因的表达量,结果表明,在TaRSTR基因过表达的情况下,AtFRY1、AtRD29B、AtP5CS1基因的表达量均显著上调,而AtSOS3的表达量没有明显变化(图6)。可见,TaRSTR过表达上调了部分耐逆相关标记基因的表达。
3 讨论与结论
本研究克隆的小麦基因TaRSTR编码一个RING/U-box家族蛋白,该家族的蛋白参与泛素化过程,介导很多生物活动过程,包括对高盐等非生物胁迫的适应[20]。qRT-PCR试验结果表明,TaRSTR基因在175 mmol/L NaCl处理1 h时表达量即上调至最高,说明TaRSTR是一个早期响应盐胁迫的基因。分子特征分析表明该蛋白定位于细胞核内。已有报道表明,RING/U-box家族蛋白参与多种非生物胁迫的适应过程[15]。在植物对盐胁迫的响应过程中几种RING/U-box蛋白起着重要作用。例如拟南芥的AtAIRP4[15]、番茄的RING/U-box蛋白SpRing[12]、水稻的OsSIRP2[13]和OsRMT1[14]蛋白,均参与了植物的耐盐过程。本研究中,TaRSTR过表达提高了拟南芥在萌发期和生殖生长时期的耐盐性,表明TaRSTR是高盐胁迫耐受的正调节子。
MAPK和SOS通路是高盐、干旱和低温胁迫下激活的重要信号转导网络[21]。我们对TaRSTR过表达转基因植物的这两个重要耐逆胁迫信号通路中的标记基因AtFRY1、AtRD29B、AtP5CS1(MAPK通路)和AtSOS3(SOS通路)的表达情况进行分析,结果表明,TaRSTR过表达条件下,AtFRY1S、AtRD29B、AtP5CS1基因的表达量均显著上调,而AtSOS3的表达量没有明显变化。其中AtFRY1编码一种多磷酸肌醇-1-磷酸酶, 该酶可以提高三磷酸肌醇(inositol triphosphate,IP3)的浓度,提高信号传导效率[22]。AtRD29B通过应对渗透胁迫参与了盐胁迫和旱胁迫,该基因表达上调提高了细胞对渗透胁迫的耐受[23, 24]。高等植物中,AtP5CS1是脯氨酸合成的限速酶[25],增强该酶的表达能够提高脯氨酸的含量[26]。AtP5CS1基因的过表达可以增强转基因烟草[27]和水稻[28]对非生物胁迫的耐受性。本研究结果表明,在TaRSTR过表达的条件下,上述标记基因的表达量均有不同程度的上调,表明TaRSTR可以通过提高耐盐标记基因的表达来提高植物的耐盐性,这与我们的表型鉴定结果是一致的。综上所述,TaRSTR基因是一个受高盐胁迫诱导表达的基因,TaRSTR定位在细胞核里,通过影响MAPK耐盐通路的基因表达提高植物的耐盐性。
参 考 文 献:
[1] Munns R,Tester M.Mechanisms of salinity tolerance[J].Ann.Rev. Plant Biol., 2008, 59: 651-681.
[2] Sanchez D H, Lippold F, Redestig H, et al. Integrative functional genomics of salt acclimatization in the model legume Lotus japonicus[J]. Plant J., 2008, 53: 973-987.
[3] Zhu J K. Salt and drought stress signal transduction in plants[J]. Annu. Rev. Plant Biol., 2002, 53: 247-273.
[4] Koiwa H, Bressan R A, Hasegawa P M. Identification of plant stress-responsive determinants in Arabidopsis by large-scale forward genetic screens[J]. Exp. Bot., 2006, 57: 1119-1128.
[5] Marchler-Bauer A, Bo Y, Han L, et al. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures [J]. Nucleic Acids Res.,2007, 45(D1): D200-D203.
[6] Stone S L. Functional analysis of the RING-type ubiquitin ligase family of Arabidopsis[J]. Plant Physiol., 2005, 137: 13-30.
[7] Lim S D, Yim W C, Moon J C, et al. A gene family encoding RING finger proteins in rice: their expansion, expression diversity, and co-expressed genes[J]. Plant Mol. Biol., 2010, 72: 369-380.
[8] Jung Y J, Lee I H, Nou I S, et al. BrRZFP1 a Brassica rapa C3HC4-type RING zinc finger protein involved in cold, salt and dehydration stress[J]. Plant Biol., 2013, 15(2): 274-283.
[9] Tak H, Mhatre M. Molecular characterization of VvSDIR1 from Vitis vinifera and its functional analysis by heterologous expression in Nicotiana tabacum[J]. Protoplasma, 2013, 250(2): 565-576.
[10] Islam M M, Tani C, Watanabe-Sugimoto M, et al. Myrosinases, TGG1 and TGG2, redundantly function in ABA and MeJA signaling in Arabidopsis guard cells[J]. Plant Cell Physiol., 2009, 50(6):1171-1175.
[11] Yang Y, Ma C, Xu Y, et al. A zinc finger protein regulates flowering time and abiotic stress tolerance in chrysanthemum by modulating gibberellin biosynthesis[J]. Plant Cell, 2014, 26: 2038-2054.
[12] Qi S, Lin Q, Zhu H, et al. The RING finger E3 ligase SpRing is a positive regulator of salt stress signaling in salt-tolerant wild tomato species[J]. Plant Cell Physiol., 2016, 57(3): 528-539.
[13] Chapagain S, Park Y C, Kim J H, et al. Oryza sativa salt-induced RING E3 ligase 2 (OsSIRP2) acts as a positive regulator of transketolase in plant response to salinity and osmotic stress[J]. Planta, 2018, 247(4): 925-939.
[14] Lim S D, Jung C G, Park Y C, et al. Molecular dissection of a rice microtubule-associated RING finger protein and its potential role in salt tolerance in Arabidopsis[J]. Plant Mol., Biol., 2015, 89(4/5):365-384.
[15] Yang L, Liu Q, Liu Z, et al. Arabidopsis C3HC4-RING finger E3 ubiquitin ligase AtAIRP4 positively regulates stress-responsive abscisic acid signaling[J]. Integr. Plant Biol., 2016, 58(1): 67-80.
[16] 葛榮朝, 赵宝存, 陈桂平, 等.小麦耐盐相关基因TaSTK的克隆 [J]. 作物学报,2007,33(5):857-860.
[17] 赵宝存, 赵芊, 葛荣朝, 等.利用基因芯片研究小麦耐盐突变体盐胁迫条件下基因的表达图谱 [J]. 中国农业科学,2007,40(10):2355-2360.
[18] Yoo S D, Cho Y H, Sheen J. Arabidopsis mesophyll protoplasts: a versatile cell system for transient gene expression analysis [J]. Nat. Protoc., 2007, 2(7): 1565-1572.
[19] Bent A. Arabidopsis thaliana floral dip transformation method [J]. Methods Mol. Biol., 2006, 343:87-103.
[20] Ko J H,Yang S H,Han K H.Upregulation of Arabidopsis RING-H2 gene,XERICO,confers drought tolerance through increased abscisic acid biosynthesis[J]. Plant J., 2006, 47(3): 343-355.
[21] Li J M, Nam K H. Regulation of brassinosteroid signaling by a GSK3/SHAGGY-like kinase[J]. Science, 2002, 295:1299-1301.
[22] Chinnusamy V, Zhu J. Salt stress signaling and mechanisms of plant salt tolerance[J]. Genet. Eng., 2006, 27: 141-177.
[23] Msanne J, Lin J, Stone J M, et al. Characterization of abiotic stress-responsive Arabidopsis thaliana RD29A and RD29B genes and evaluation of transgenes [J]. Planta, 2011, 234: 97-107.
[24] Tezuka K, Taji T, Hayashi T, et al. A novel abi5 allele reveals the importance of the conserved Ala in the C3 domain for regulation of downstream genes and salt tolerance during germination in Arabidopsis [J]. Plant Signal Behav., 2013, 8: e23455.
[25] Hu C A, Delauney A J, Verma D P S. A bifunctional enzyme (Δ1-pyrroline-5- carboxylate synthetase) catalyzes the first two steps in proline biosynthesis in plants[J]. Proc. Natl. Acad. Sci. USA., 1992, 89: 9354-9358.
[26] Armengaud J, Urbonavicius J, Fernandez B, et al. N2-methylation of guanosine at position 10 in tRNA is catalyzed by a THUMP domain-containing, S-adenosylmethionine-dependent methyltransferase, conserved in Archaea and Eukaryota[J]. Biol. Chem., 2004, 279: 37142-37152.
[27] Kishor P, Hong Z, Miao G H, et al. Overexpression of Δ1-pyrroline-5-carboxylate synthetase increases proline production and confers osmotolerance in transgenic plants[J]. Plant Physiol., 1995, 108: 1387-1394.
[28] Zhu B C, Su J, Chang M C, et al. Overexpression of a Δ1-pyrroline-5-carboxylate synthetase gene and analysis of tolerance to water- and salt-stress in transgenic rice[J]. Plant Sci., 1998, 139:41-48.

