云南变绿红菇的遗传多样性与群体遗传分化研究
余金凤 陈正启 周 汐 吴素蕊 郭 相
云南变绿红菇的遗传多样性与群体遗传分化研究
余金凤 陈正启 周 汐 吴素蕊 郭 相*
(中华全国供销合作总社昆明食用菌研究所,云南 昆明 650221)
以昆明、曲靖、楚雄、玉溪、昭通、临沧、大理等7个变绿红菇地理居群的30个子实体样本为试验材料,基于ITS序列和LSU序列,对其分子变异、遗传多样性、群体遗传分化进行分析。结果:30个样品中,基于ITS序列检测到18个单倍型,基于LSU序列检测到10个单倍型;基于ITS序列和LSU序列分别构建的单倍型系统发育树及30个样品的系统发育树显示,各地理居群的遗传距离与地理距离之间没有形成对应关系,二者无明显相关性。基于两个序列的分析结果均显示,各地理居群内单倍型多样性较丰富,而核苷酸多样性低。表明变绿红菇在不同的地理居群中的适应能力强,遗传多样性较低,绝大部分遗传分化来自于个体间的差异。
变绿红菇;ITS;LSU;遗传多样性;群体遗传分化
变绿红菇()又名青头菌、绿菇,隶属担子菌亚门、层菌纲、红菇目、红菇科、红菇属,是云南常见野生菌之一,在全省各地州均有分布[1-5]。其富含蛋白质、氨基酸、多糖等营养成分,味道鲜美,深受消费者喜爱[3, 6-9]。近年来对变绿红菇的研究多集中于化学成分、抗肿瘤抗氧化活性分析,菌种分离及分子鉴定等,对其遗传多样性和遗传分化方面的研究较少。
遗传多样性通常指生物携带的遗传信息总和。一个物种的多样性是长期进化的结果,物种遗传多样性越高,遗传变异越丰富,其对环境的适应力越强;反之,对环境的适应力越弱。精确评估物种的遗传多样性是物种保护的基础,是确定优先保护种、筛选优良种质的关键[10-13]。
本研究利用核糖体DNA 中的非编码转录间隔区ITS(Internal Transcribed Spacer)序列[14-17]及28s核糖体大亚基LSU序列[18-21]作为DNA分子标记,分析云南省7个地区变绿红菇的遗传多样性及群体遗传分化,探讨地理分布对变绿红菇遗传特性的影响,为云南省变绿红菇野生资源的保育和开发利用提供参考。
1 材料与方法
1.1 材料
(1)供试菇体。30个变绿红菇样本分别采自云南昆明、曲靖、楚雄、玉溪、昭通、临沧、大理(表1)。新鲜子实体采集后去除泥沙杂物,装入干净的自封袋中,并加入变色硅胶干燥。

表1 变绿红菇样本信息
(2)试剂。DNA PCR扩增所有试剂均购自TaKaRa 生物有限公司,分析用的引物由生工生物工程(上海)股份有限公司合成。
(3)主要仪器设备。全自动基因扩增仪、高速冷冻离心机、Bio-Rad凝胶成像系统、电泳仪和核酸蛋白测定仪。
1.2 试验方法
(1)样品预处理。将干燥后的变绿红菇子实体表面杂物去除,取出部分子实体封装于1.5 mL离心管中存储备用。
(2)基因组DNA的提取。以干燥子实体为材料,采用基因组DNA快速提取试剂盒提取总基因组DNA。
(3)保守序列的扩增及检测。变绿红菇ITS、LSU 基因序列的PCR 扩增采用的特异性引物序列见表2。

表2 用于PCR扩增的引物
PCR反应采用50 µL反应体系:TaKaRa Taq 0.5 µL,dNTP Mixture 4 µL,10×PCR Buffer 5 µL,10 µmol/L的上下游引物各1 µL,DNA模板1 µL,ddH2O 37.5 µL。设置以无菌水代替模板的阴性对照。
PCR反应扩增程序:95 ℃预变性5 min;94 ℃变性30 s,55 ℃退火1 min,72 ℃延伸1 min,30次循环;再72 ℃延伸7 min;4 ℃保存。PCR结束后取5 μL 扩增产物在1%琼脂糖凝胶上进行电泳,观察Bio-Rad凝胶成像系统结果。
(4)ITS和LSU片段的回收。检测到PCR产物后,将剩下的产物用1.5%的琼脂糖凝胶电泳,从琼脂糖凝胶中切割下含有目的片段的凝胶,使用TIANGEN胶回收试剂盒进行DNA的回收纯化。
(5)筛选阳性克隆。①将含有目的片段的胶回收产物与质粒载体pMD19-T参照使用说明进行连接,连接体系为:胶回收产物(根据浓度而定)4.0 μL,pMD19-T 1.0 μL,2×Ligation I 5.0 μL,共10 μL,16 ℃连接30 min。②重组质粒的转化:将连接产物转化至感受态细胞DH5α中,涂布于含Amp的LB固体培养基上,37 ℃培养过夜。③克隆子的初步筛选:挑取单菌落于含Amp的液体LB培养基中,37 ℃摇床过夜培养。④PCR验证:取培养的菌液为模板,进行PCR扩增验证,扩增体系与程序设置同1.2(3)所述。
(6)测序。将PCR验证为阳性克隆的菌液送昆明硕擎生物科技有限公司测序。
1.3 序列处理
将已测得的目的片段序列和从GenBank中通过BLAST检索获得的参考序列进行多重对位排列,并手动去除排列结果两端的非对位排列区。
1.4 序列变异情况
用MEGA(Version5.1)软件计算变异位点、简约信息位点、单一变异位点、转换/颠换位点数比。
1.5 遗传多样性及遗传变异分析
用Arlequin3.5软件统计各地理居群样品的单倍型多样性、核苷酸多样性、核苷酸平均差异数、计算不同地理居群间的群体遗传差异指数,分析各地理居群的遗传变异情况。DnaSP5软件计算群体的核苷酸平均差异数及核苷酸歧义度,评估各地理居群的遗传多样性。
2 结果与分析
2.1 ITS序列及LSU序列的变异情况
本研究获得变绿红菇样本的ITS序列和LSU序列各30个,经处理去掉两端侧翼序列后ITS序列长度为697 bp,多态位点24个,其中简约信息位点5个,单一变异位点19个。4种碱基组成分别为:A=23.59%,T=27.71%,G=25.38%,C=23.33%,A+T含量(0.513)略高于G+C(0.487),转换/颠换R值为3.01。LSU序列长度为761 bp,多态位点10个,其中简约信息位点4个,单一变异位点6个。4种碱基组成分别为:A=22.65%,T=24.11%,G=31.17%,C=22.06%,A+T含量(0.468)低于G+C(0.532),转换/颠换R值为2。两个片段的测序统计结果均说明绝大部分核苷酸的替换方式为转换,都验证了亲缘关系近的分类阶元之间的替换主要以转换为主[22]。
2.2 基于ITS序列的群体遗传多样性及遗传变异分析
根据ITS分子标记数据,对变绿红菇不同地理居群的遗传多样性的分析结果(表3):7个地理居群的30个变绿红菇个体样品,共检测到单倍型18个。样品共享单倍型情况如表4所示。在物种水平上单倍型多样性(Hd)为0.949±0.021,核苷酸多样性(Pi)为0.003 69±0.000 045。可见单倍型多样性高而核苷酸多样性低,表明变绿红菇在不同的环境下有相应的适应对策,生存能力较强。
用NJ法构建单倍型系统发育树(图1)。联系图1和表4结果可以看出,各单倍型并未按地理分布在系统发育树上聚集到一起,支持率较低。说明单倍型之间的差异较小,不能形成可靠分支。且各地理居群是由多个单倍型组成,分属于不同地理居群的个体可共享一个单倍型。说明各地理居群间存在基因交流,遗传分化较小。说明不同地区的样品之间差异较小,相似度较高。
对不同地理居群间的Fst值统计结果(表5)显示,Fst值的变化范围为0.023 06~0.186 05。其中,昆明和大理群体分化较小,Fst值为0.023 06;大理和临沧群体分化较大,Fst值为0.186 05。此外,昭通与其他6个地理居群间的Fst值均≤0,说明其与各群体间基因交流频繁,无分化;楚雄除与大理群体之间存在很小程度的分化外,与其他各地理居群间均无分化;玉溪与曲靖群体间也无分化。由表6可知,不同地理居群间核苷酸差异数Kxy变化范围为1.781 82~4.250 00,核苷酸歧义度Dxy范围为0.002 58~0.006 16,大理和曲靖与其他群体差异较大。说明这两个群体相对于其他群体变异较大,遗传距离较远。
2.3 基于LSU序列的群体遗传多样性及遗传变异分析
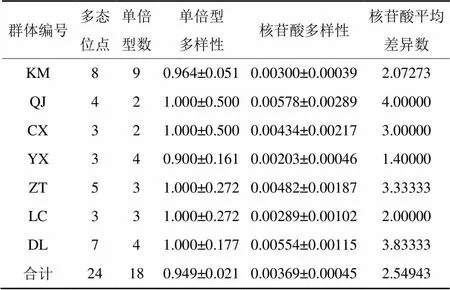
表3 基于ITS序列变绿红菇不同地理居群的遗传多样性
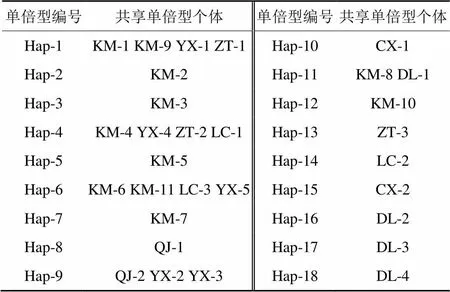
表4 基于ITS序列的变绿红菇单倍型及共享单倍型样本信息
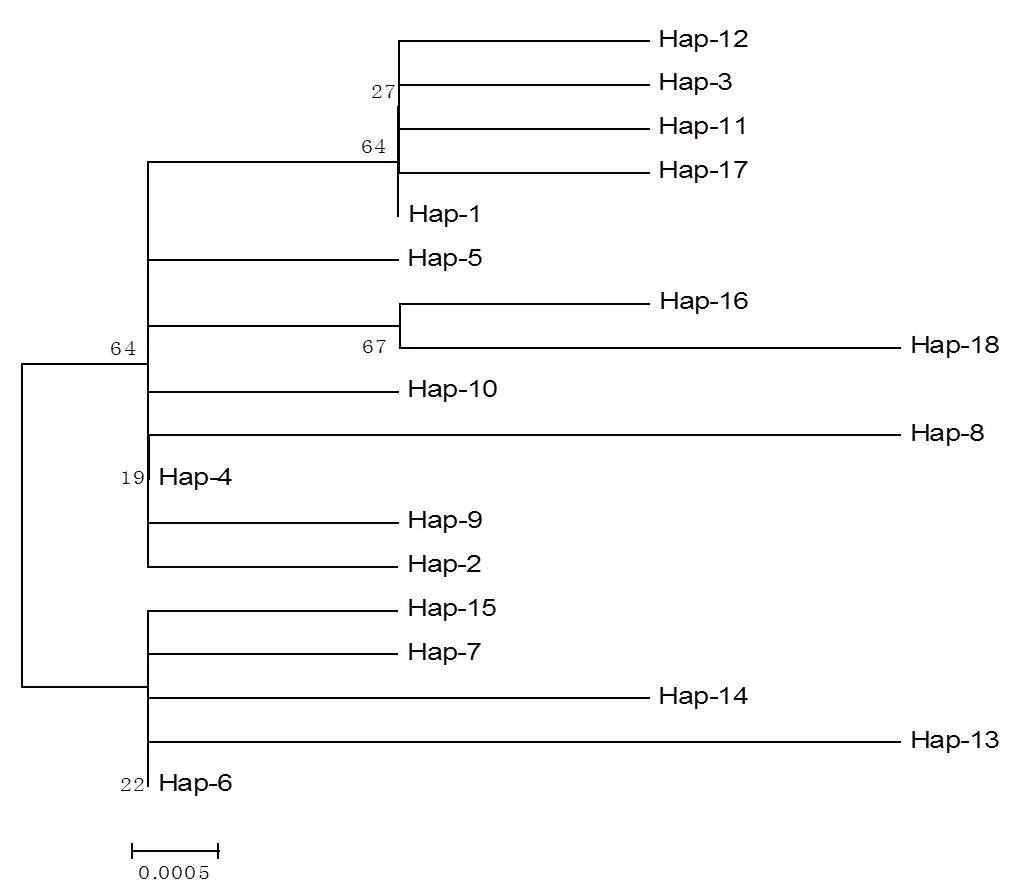
图1 基于ITS序列构建的变绿红菇单倍型系统发育树
依据LSU分子数据,对变绿红菇不同地理居群的遗传多样性的分析结果(表7):7个地理居群的30个变绿红菇个体样品中,共检测到10个单倍型。样品共享单倍型情况如表8所示,在物种水平上单倍型多样性(Hd)为0.710±0.087,核苷酸多样性(Pi)为0.001 58±0.000 37。
用NJ法构建单倍型系统发育树(图2)。联系树形和表8的统计结果可知,各单倍型并未按地理分布在系统发育树上聚集到一起,支持率较低。说明单倍型之间的差异较小,不能形成可靠分支。各地理居群是由多个单倍型组成,而分属于不同地理居群的个体可共享一个单倍型。说明各地理居群间存在基因交流,遗传分化较小。说明不同地区的样品之间差异较小,相似度较高。该结果与ITS序列的分析结果吻合。
不同地理居群间的Fst值范围为0.016 22~0.055 56(表9);大理和昭通、曲靖和大理群体分化较大,Fst值为0.055 56。楚雄除与大理群体之间存在很小程度的分化外,与其他各地理居群间均交流频繁,无分化;曲靖与昭通、临沧两个群体间的Fst值均为0,说明其间基因交流频繁,无分化;临沧与曲靖、昭通两群体间,大理与玉溪、临沧两群体间也无分化。不同地理居群间核苷酸差异数Kxy变化范围为0~2.300 00,核苷酸歧义度Dxy范围为0~0.003 04(表10),大理和玉溪与其他群体差异较大,说明这两个群体相对于其他群体变异较大,遗传距离较远。

表5 基于ITS序列的不同地理居群间的Fst值
注:Fst<0.05,表示群体间几乎没有遗传分化; 0.05≤Fst<0.15,表示分化程度较低;0.15≤Fst<0.25,为中度分化;Fst≥0.25,表明分化程度较高[23-26],表9同。
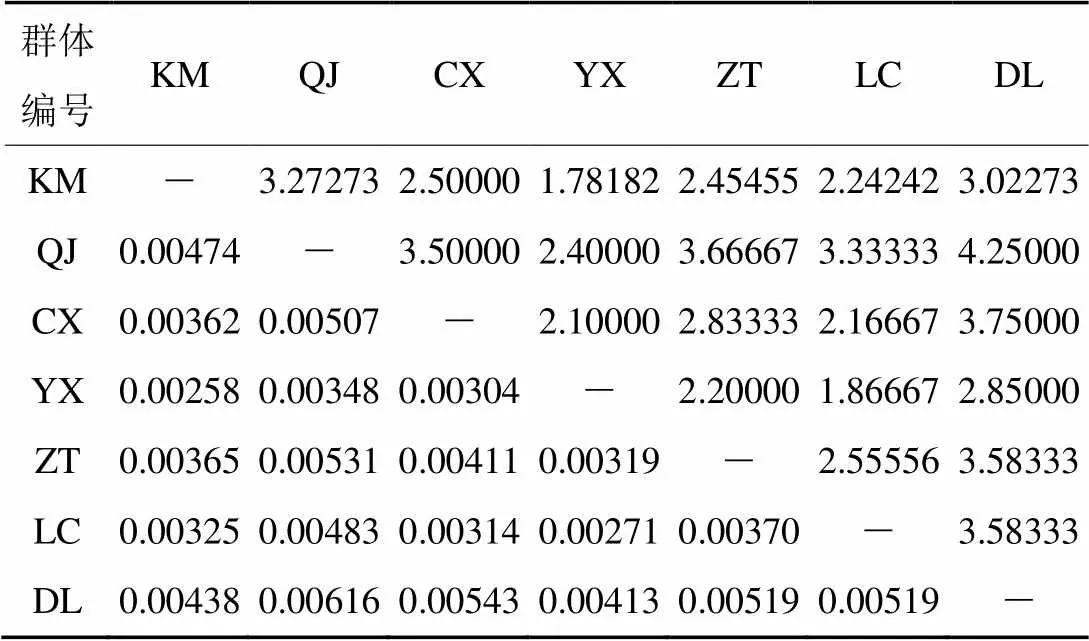
表6 基于ITS序列的不同地理居群间核苷酸平均差异数(Kxy,上三角)和核苷酸歧义度(Dxy,下三角)

表7 基于LSU序列的变绿红菇不同地理居群的遗传多样性指数
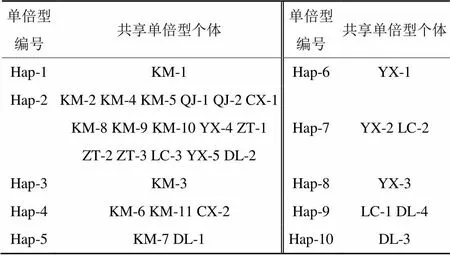
表8 基于LSU序列的变绿红菇单倍型及共享单倍型样本信息

图2 基于LSU序列构建的变绿红菇单倍型系统发育树

表9 基于LSU序列的不同地理居群间的Fst值

表10 基于LSU序列不同地理居群间的核苷酸平均差异数(Kxy,上三角)和核苷酸歧义度(Dxy,下三角)
3 讨 论
本研究基于云南7个地区野生变绿红菇的ITS序列及LSU序列信息,研究不同地理居群变绿红菇的遗传变异,统计其单倍型数并构建单倍型系统发育树,解析了云南部分地区变绿红菇群体的单倍型多样性及遗传多样性。
基于ITS序列的研究结果表明,单倍型Hap-1、Hap-4、Hap-6出现的频率较高(13.3%),而单倍型Hap-2、Hap-3、Hap-5、Hap-7、Hap-8、Hap-10、Hap-12~18均为独享单倍型,说明变绿红菇单倍型多样性较高。各地理居群的遗传多样性指数变化不大。曲靖群体单倍型多样性和核苷酸多样性明显高于其他群体,群体遗传多样性较高;玉溪群体的单倍型多样性和核苷酸多样性则明显低于其他群体,遗传多样性低。从单倍型系统发育树看,各进化分支之间的分歧度很低,没有形成明显的聚类分支,单倍型分布与地理居群分布之间没有明显的对应结构关系。同一物种不同群体间的遗传分化程度可以用遗传分化指数Fst来判别,以衡量群体间的遗传距离。本研究遗传分化结果显示,各群体间存在着较低程度的遗传分化[27]。
基于LSU序列的研究结果,30个样品中检测到10个单倍型,低于ITS序列检测结果的18个,这可能是由于LSU序列更加保守,产生突变和新单倍型的概率更低。10个单倍型Hap-2出现频率最高(53.3%),其可能是变绿红菇群体的祖先单倍型,是在种群中稳定存在、适应环境能力强的优势单倍型。从遗传多样性指数可知,曲靖和昭通的单倍型多样性为0,可能与用于实验的样本数太少有关。但与ITS序列的分析结果相比,其总体上的遗传多样性是低的。遗传分化指数结果与ITS序列的分析结果一致,同样表明群体间遗传分化较低。
综合两个序列片段的数据分析结果,云南地区变绿红菇群体整体上表现出较低的遗传多样性。各地理居群单倍型多样性较高,核苷酸多样性较低,说明变绿红菇在不同地理居群中的适应能力强,但遗传分化程度低,绝大多数分化来自于个体间的差异,可能原因是变绿红菇传播历史较短,没有积累过多的遗传变异。
变绿红菇作为云南省普遍食用的野生菌,具有显著的经济价值,深入了解变绿红菇的遗传多样性,了解其遗传分化现状,对于后期有针对性地开展资源保护培育及综合开发利用具有重要意义。本研究使用ITS标记和LSU标记评估云南变绿红菇的遗传多样性和遗传分化情况,获得了多态位点和关键的遗传学数据信息。在未来的研究中,需不断增加不同地理居群样本数量,使样品数能完全囊括云南各地区,再配合多种类型的分子标记,精确评估物种遗传多样性,为后续研发提供详实的数据支撑。
[1] HAWKSWORT D L, KIRK P M, SUTTON B C, et al. Ainsworth & Bisby's dictionary of the fungi[J]. Revista do Instituto de Medicina Tropical de São Paulo, 1996, 38(4): 272.
[2] 卯晓岚. 中国大型真菌[M].郑州: 河南科学技术出版社出版, 2000.
[3] 段玉云, 周家齐, 曾黎琼, 等. 野生食用菌的化学成分分析[J]. 食用菌, 1999, 21(5): 3-5.
[4] 吴兴亮. 中国贵州大型真菌资源及其利用[J]. 贵州科学, 2000(1): 71-76.
[5] 黄萍, 沈孝善. 绿菇子实体菌株的原生质体分离及再生菌株的获得[J]. 西南农业学报, 2001(1): 91-95.
[6] 徐丹先, 林佶, 段志敏, 等. 云南野生奶浆菌的化学成分分析[J]. 食用菌, 2012(4): 60-61.
[7] 王文辉, 彭永芳. 云南九种野生食用菌氨基酸的快速测定[D]. 中国食用菌, 1999(2): 29-30.
[8] 张相日, 李金灿, 陆辉, 等. 云南野生食用菌多糖含量测定及培养特性研究[J]. 广州大学学报 (自然科学版), 2005(2): 29-32.
[9] 赵玉堂,周玲仙. 云南食物成分表[M]. 昆明: 云南科技出版社, 2003.
[10] 蒋志刚, 马克平. 保护生物学的现状, 挑战和对策[J]. 生物多样性, 2009, 17(2): 107-116.
[11] 邱芳, 伏健民. 遗传多样性的分子检测[J]. 生物多样性, 1998, 6(2): 143-150.
[12] 马克平. 试论生物多样性的概念[J]. 生物多样性, 1993, 1(1): 20-22.
[13] 庞广昌, 姜冬梅. 群体遗传多样性和数据分析[J]. 林业科学, 1995(6): 543-550.
[14] 胡伟, 张跃新, 邓勋, 等. 基于 rDNA ITS序列和RAPD分子标记的桑黄菌遗传多样性分析[J]. 安徽农业科学, 2013, 41(29): 11597-11600.
[15] 陈剑山, 郑服丛. ITS序列分析在真菌分类鉴定中的应用[D]. 安徽农业科学, 2007, 35(13): 3785-3786, 3792.
[16] 燕勇, 李卫平, 高雯洁, 等. rDNA-ITS序列分析在真菌鉴定中的应用[J]. 中国卫生检验杂志, 2008, 18(10): 1958-1961.
[17] 白树猛, 田黎. ITS序列分析在真菌分类鉴定和分子检测中的应用[J]. 畜牧与饲料科学, 2009, 30(1): 52-53.
[18] SONNENBERG R, NOLTE A W, TAUTZ D. An evaluation of LSU rDNA D1-D2 sequences for their use in species identification[J]. Frontiers in zoology, 2007, 4(1): 6.
[19] 邱君志, 黄志鹏, 潘洁茹, 等. 虫生真菌座壳孢的RAPD与LSU rDNA序列分析[J]. 农业生物技术学报, 2004, 12(5): 578-582.
[20] 李雪玲. 贝盖侧耳的系统发育地位——基于nrDNA—LSU和ITS序列分析的研究[J]. 北京林业大学学报, 2005(3): 67-71.
[21] 谭著明. 红汁乳菇生物学特性与半人工栽培技术研究[D]. 长沙: 湖南农业大学, 2005.
[22] SIMON C, FRATI F, BECKENBACH A, et al. Evolution, weighting, and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers[J]. Annals of the entomological Society of America, 1994, 87(6): 651-701.
[23] WRIR B S, COCKERHAM C C. Estimating F‐statistics for the analysis of population structure[J]. evolution, 1984, 38(6): 1358-1370.
[24] GOUDET J. FSTAT (version 1.2): a computer program to calculate F-statistics[J]. Journal of heredity, 1995, 86(6): 485-486.
[25] ROUSSET F. Genetic differentiation and estimation of gene flow from F-statistics under isolation by distance[J]. Genetics, 1997, 145(4): 1219-1228.
[26] NEI M. F‐statistics and analysis of gene diversity in subdivided populations[J]. Annals of human genetics, 1977, 41(2): 225-233.
[27] WEIR B S, HILL W G. Estimating F-statistics[J]. Annual review of genetics, 2002, 36(1): 721-750.
Genetic variation and population genetic differentiation ofin Yunnan
Yu Jinfeng Chen Zhengqi Zhou Xi Wu Surui Guo Xiang*
(Kunming Edible Fungi Institute, All China Federation of Supply and Marketing Cooperatives, Kunming,Yunnan 650221, China)
In order to find out the genetic diversity and differentiation ofpopulation distributed in different areas of Yunnan.The fruiting bodies offrom 7 geographical populations, namely Kunming (KM), Qujing (QJ), Chuxiong (CX), Yuxi (YX), Zhaotong (ZT), Lincang (LC) And Dali (DL), were used as test materials. Molecular variation, genetic diversity and population genetic differentiation of 30 samples were analyzed based on ITS and LSU sequences. The results showed that 18 haplotypes were detected based on ITS sequence and 10 haplotypes were detected based on LSU sequence. The haplotype phylogenetic tree constructed based on ITS sequence and LSU sequence showed that there was no corresponding relationship between genetic distance and geographical distance in each geographical population, and there was no significant correlation between them. The results of the two sequences analysis showed that the haplotype diversity was rich in each geographic population, while the nucleotide diversity was low, indicating that the genetic diversity ofwas low; Fst values among different geographical populations were basically less than 0.15, indicating that the degree of genetic differentiation was low, and most of the differentiation came from differences among individuals.
; ITS; LSU; genetic variation; population genetic differentiation
S646
A
2095-0934(2020)03-178-07
云南省技术创新人才项目(2017HB094)
余金凤(1988—),女,硕士,助理研究员,主要从事食用菌分子生物学研究。E-mail:584905471@qq.com。
郭相(1980—),男,硕士,副研究员,主要从事珍稀野生食用菌持续利用技术研究。E-mail:guoxkm@yeah.net。

