利用发根农杆菌体系检测西瓜CRISPR/Cas9系统的靶位点
张月乔 葛洁 田树娟 袁黎

摘 要: 西瓜遗传转化技术周期长,过程复杂,CRISPR/Cas9基因编辑系统又存在着一定的脱靶效应,为了保障所选择的靶位点的可行性,本研究选择西瓜ClSPO11-1基因(ID:Cla003301)为靶标,利用CRISPR/Cas9基因编辑系统构建双靶点的基因敲除载体,利用发根农杆菌Ar. Qual菌株侵染西瓜子叶外植体,通过简单的组培过程,使西瓜外植体长出不定根,在不定根中检测到了在靶位点1处发生了不同程度的碱基缺失,经过与野生型氨基酸序列比对分析后发现均造成了翻译提前终止和氨基酸的突变,成功实现了对西瓜不定根的基因编辑,该方法周期短,操作简便,实现了快速鉴定在西瓜中选择的靶位点的靶向效率,为研究西瓜基因功能和遗传改良提供了保障。
关键词: 西瓜; CRISPR/Cas9系统; 发根农杆菌; 基因敲除
中图分类号:S651 文献标志码:A 文章编号:1673-2871(2020)04-007-05
Abstract: Watermelon genetic transformation technology is time consuming and process complicated. The CRISPR/Cas9 gene editing system has a certain off-target effect. In order to ensure the feasibility of the selected target site, the watermelon ClSPO11-1 gene (ID:Cla003301) was selected in this study as target, using the CRISPR/Cas9 gene editing system to construct a double-target gene knockout vector, using Agrobacterium rhizogenes Ar. Qual strain to infect watermelon cotyledon explants, and to make watermelon explants growing through a simple tissue culture adventitious roots. In the adventitious roots, different degrees of base deletions at target site 1 were detected. After comparison and analysis with amino acid sequences of wild-type , both premature translation termination and amino acid mutations was found. This method has a short cycle and simple operation. It achieves the rapid target identification efficiency of selected target sites in watermelon, and provides a guarantee for the study of watermelon gene function and genetic improvement.
Key words: Watermelon; CRISPR/Cas9 system; Agrobacterium rhizogenes; Gene knockout
西瓜(Citrullus lanatus)属于葫芦科西瓜属,是原产于非洲的一种重要经济作物,因其具有清热解暑,除烦止渴,通便利尿和降血压等多种功效,深受人们喜爱,并广泛栽培于世界各地。目前,我国西瓜的栽培面积、产量及人均消费量均居世界首位[1],北起黑龙江,南到海南岛,西从西北边疆,东到东南沿海,均有栽培。随着西瓜全基因组测序的完成,西瓜的遗传研究已经步入了后基因组的时代[2],对于西瓜重要的一些农艺性状相关的基因虽有预测,但对于其功能的验证仍然存在着一定的困难。
基因编辑技术作为合成生物学的核心技术,是近几年发展起来的对基因组进行定向精确修饰的技术,在特定的基因位点产生DNA双链断裂,借助编辑受体DNA自身的修复系统可以对基因组中的靶位点进行敲除、插入、碱基替换、点突变等定点人工修飾。由于构建载体的过程简单以及编辑效率高等优点,CRISPR/Cas系统已然成为当前应用的主流基因编辑系统,其中应用最为广泛的是CRISPR/Cas9系统。CRISPR/Cas9基因编辑系统不仅可以在人类和小鼠的细胞内进行基因编辑,也可以在拟南芥、水稻、烟草、番茄、玉米、西瓜、葡萄等模式植物和粮食作物以及园艺植物中实现定点基因编辑,但是要获得稳定的突变体植株仍还需要大量工作[3-5]。然而该系统在使用过程中由于靶位点在全基因组中可能存在了高度相似的序列从而引起Cas9在其他位置出现切割,导致了非预期的突变,即产生了所谓的脱靶效应[6-7]。
转基因技术是当今生物技术研究的热点,是研究基因功能及作物改良的重要技术。目前应用较广泛的转基因方法主要有以下几种类型,农杆菌介导法、基因枪法、聚乙二醇(PEG)法、电激法、花粉管通道法等,在西瓜中主要运用的是农杆菌介导的遗传转化体系。农杆菌是一种普遍存在于土壤中的革兰氏阴性细菌,发根农杆菌(Agrobacterium rhizogenes)它能在自然条件下趋化性地感染大多数双子叶植物的受伤部位,并诱导产生发状根[8]。其原理是发根农杆菌细胞中含有Ri质粒[9],其上具有一段T-DNA,农杆菌通过侵染植物伤口后进入细胞,可以将T-DNA插入到植物的基因组中[10]。已有研究表明利用发根农杆菌可以在多种植物中成功诱导出不定根,例如茄科、十字花科、豆科以及葡萄等植物[11-12]。
由于西瓜遗传转化周期长,遗传转化的过程复杂以及转化效率低,CRISPR/Cas系统自身存在脱靶效应这一现象,因此评估所选择的靶位点的可行性对西瓜实现成功的基因组编辑是至关重要的[13]。笔者通过建立一种新的发根农杆菌介导的西瓜子叶外植体产生不定根方法可以快速诱导西瓜产生不定根,为实现基因组编辑、西瓜的基因功能验证奠定了基础。
1 材料和方法
1.1 植物材料及载体菌株
野生种质‘YL,生长于北纬36°~39°,东经107°~111°,抗旱性强并且具备一定的抗枯萎病能力。由于‘YL可以实现高效且稳定的再生体系,因此本试验的供试材料为野生西瓜种质‘YL,试验材料于2019年3—6月在西北农林科技大学园艺学院杨凌西甜瓜试验示范基地的塑料大棚内种植,收种,该试验基地位于关中平原,属暖温带半湿润气候带。CRISPR/Cas9基因编辑载体PBSE402,pCBC-DT1T2载体由西北农林科技大学西甜瓜课题组提供;大肠杆菌(E. coli)DH5ɑ菌株和发根农杆菌(Agrobacterium rhizogenes)菌株Ar. Qual购于上海唯地生物技术有限公司。试验于2019年8—12月在西北农林科技大学西甜瓜课题组进行。
1.2 靶位点的选择
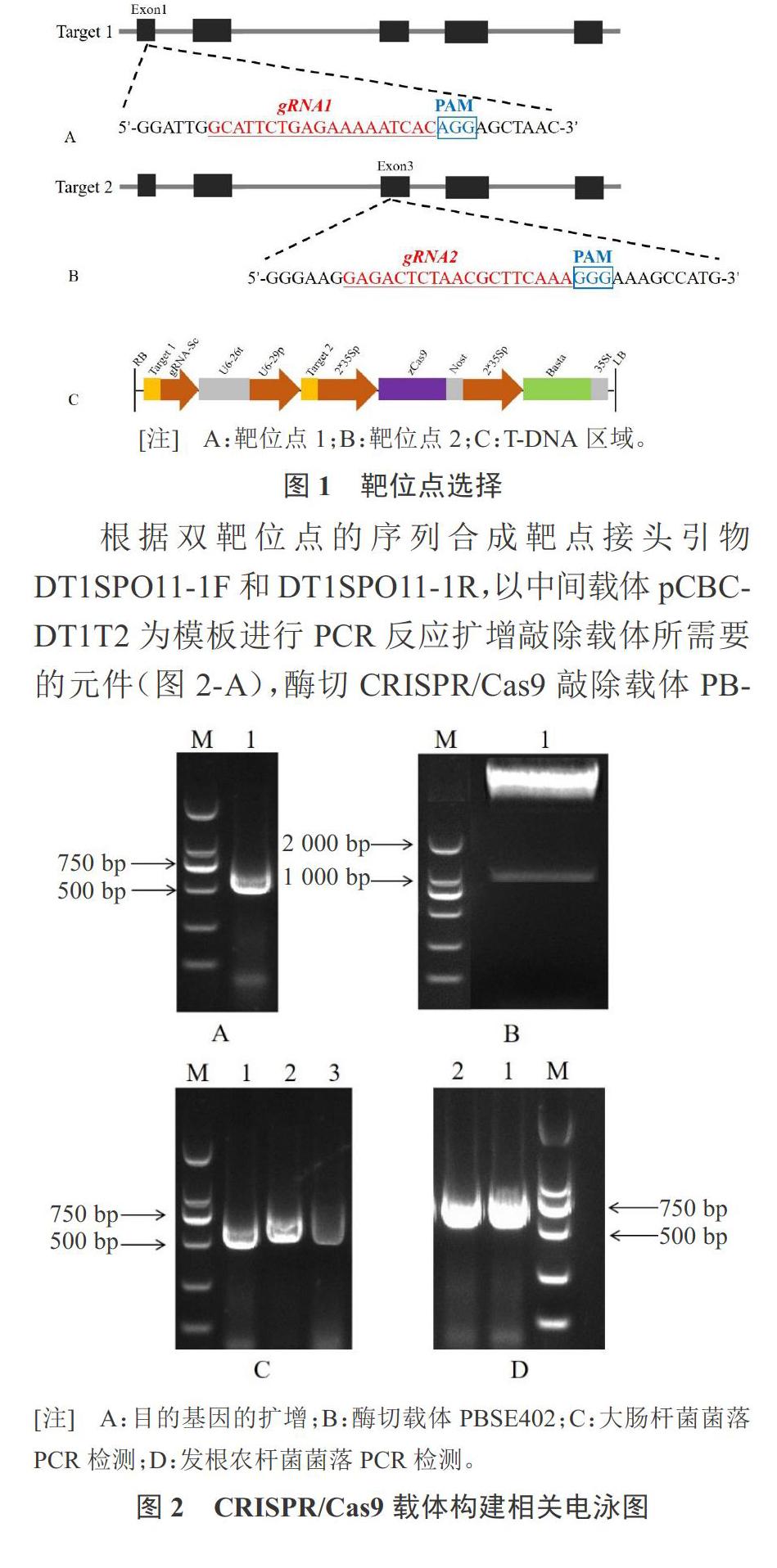
本试验所选取的敲除基因为西瓜减数分裂关键基因ClSPO11-1(Gene ID:Cla003301),使用在线靶位点设计软件(http://crispr.hzau.edu.cn/CRISPR2/)CRISPR-P2.0设计靶位点[14-16]。根据CRISPR/Cas9系统去识别目标序列中含有PAM序列的特点,通常选择的靶位点的GC含量不应低于40%,并且需要对靶位点进行特异性分析,避免实验中造成脱靶现象。为了提高编辑效率,选择2个靶位点进行编辑。
1.3 CRISPR/Cas9敲除载体的构建
(1)使用限制性内切酶BsaI酶切PBSE402载体。反应体系为:PBSE402质粒30 ?L,10×CutSmart Buffer 5 ?L,BsaI-HF 1.5 ?L,ddH2O 13.5 ?L,37 ℃下酶切2 h。使用天根DNA纯化回收试剂盒回收线性化载体。
(2)根据已选择的靶位点序列合成接头引物,上游引物SPO11-1F:TCGAAGTAGTGATTGGCAT TCTGAGAAAAATCACGTTTTAGAGCTAGAAAT AGC和下游引物SPO11-1R:TTCTAGCTCTAAAA CTTTGAAGCGTTAGAGTCTCCAATCTCTTAGTCGACTCTAC,將引物稀释到10 ?mol·L-1,以稀释100倍的中间载体pCBC-DT1T2为模板,使用KOD-Plus-Neo高保真酶进行PCR扩增,反应体系:10× PCR Buffer for KOD-Plus-Neo 5 ?L,2 mmol·L-1 dNTPs 5 ?L,25 mmol·L-1 MgSO4 3 ?L,DT1SPO11-1F 1.5 ?L,DT1SPO11-1R 1.5 ?L,pCBC-DT1T2 1 ?L,KOD-Plus-Neo 1 ?L,ddH2O 32 ?L。反应程序为:94 ℃预变性2 min;98 ℃变性10 s,60 ℃退火30 s,68 ℃延伸1 min,35个循环。将PCR产物经电泳检测后,切取胶块进行胶回收。
(3)通过TAKARA公司的In-Fusion无缝连接酶将酶切的PBSE402载体以及PCR回收产物进行连接。
(4)将连接产物导入大肠杆菌感受态DH5ɑ中,涂于含有50 mg·L-1卡那霉素的LB固体平板上,37 ℃过夜培养。挑取单菌落用特异性引物U6-26-IDF:TGTCCCAGGATTAGAATGATTAGGC和U6-29-IDR:AGCCCTCTTCTTTCGATCCATCA AC进行PCR检测,送公司进行测序验证。
(5)将测序正确的重组质粒导入发根农杆菌感受态Ar.Qual,每100 ?L感受态加入已验证正确的重组质粒1 μg,用手拨打管底混匀,依次于冰上静置5 min、液氮激冻5 min,37 ℃水浴5 min,冰浴5 min;再加入700 ?L无抗生素的TY液体培养基,28 ℃,200 r·min-1振荡培养2~3 h。取200 ?L Ar.Qual涂布于含Cmr抗性的TY平板上,倒置放于28 ℃恒温培养箱2~3 d。挑取单菌落进行PCR鉴定阳性克隆。
1.4 发根农杆菌Ar.Qual介导的西瓜遗传转化
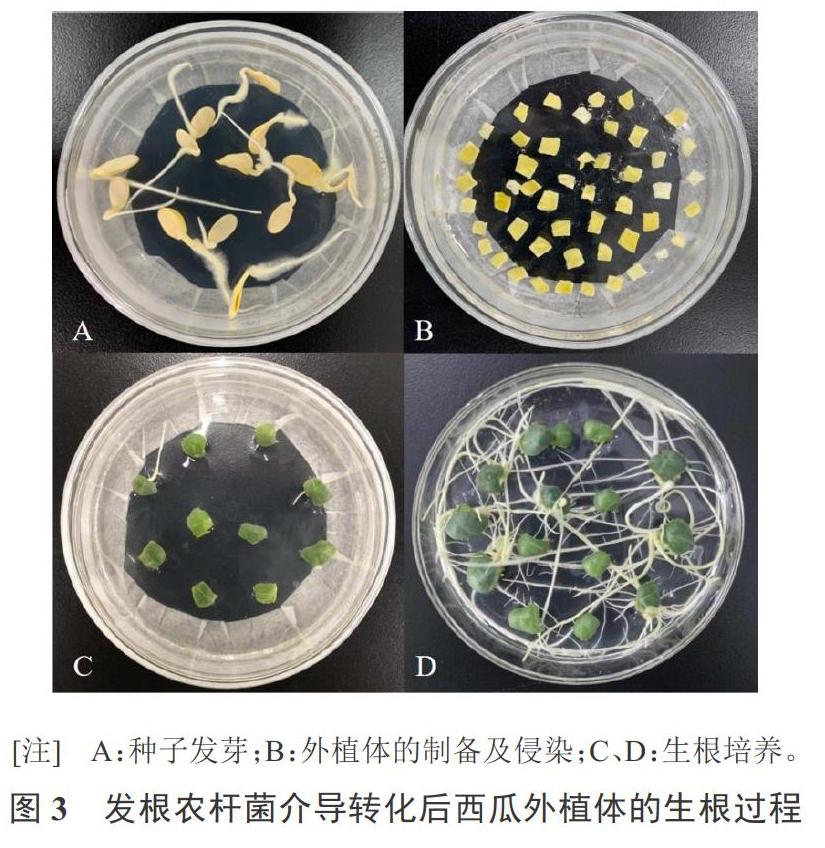
(1)取成熟的西瓜‘YL种子50~55 ℃蒸馏水浸泡种子约30 min,取出浸泡后的种子去除种壳。在超净工作台中将已剥取的种仁用75%的酒精清洗种仁约30 s,用无菌水清洗两遍之后再用3%次氯酸钠浸泡消毒15 min,再无菌水清洗5~7次之后晾干播种于MS固体培养基中,置于25 ℃黑暗培养3 d。
(2)待种子发芽后将其取出,切去子叶的两端,将剩余子叶部分均分成8块便于侵染。
(3)将鉴定正确的发根农杆菌Ar.Qual单菌落挑至含Cmr抗生素34 mg·L-1的TY液体培养基中,待菌液浓度摇至OD600=0.8时,用MS液体重悬菌液使其最终的OD600值为0.2。将切好的子叶浸泡在菌液重悬液中15 min,取出晾干放在垫有滤纸的MS固体培养基中进行共培养,25 ℃黑暗培养2 d。
(4)2 d天后将共培养的子叶取出,用无菌水洗去子叶表面多余的农杆菌菌液,取出晾干放置于含有200 mg·L-1特美汀和1.4 mg·L-1 Basta的MS固体培养基上,25 ℃光周期16 h/8 h培养,约2周之后观察根的生长情况。
1.5 西瓜不定根基因编辑检测
使用CTAB法分别提取发根农杆菌转化外植体诱导产生的不定根DNA,用SPO-1-F/R和SPO- 2-F/R对2个靶位点附近的序列分别进行PCR扩增,将能够扩增出条带的目的片段进行胶回收送测序,检测分析靶位点是否出现编辑。
2 结果与分析
2.1 双靶位载体的构建
根据CRISPR-P2.0在线工具设计了双靶位点(图1),分别位于第1个和第3个外显子上,gRNA1的GC含量为36.8%,gRNA2的GC含量为42.1%。
根据双靶位点的序列合成靶点接头引物DT1SPO11-1F和DT1SPO11-1R,以中间载体pCBC- DT1T2为模板进行PCR反应扩增敲除载体所需要的元件(图2-A),酶切CRISPR/Cas9敲除载体PBSE402后的PCR检测结果(图2-B)。将克隆的中间产物利用无缝克隆酶连接到酶切后的敲除载体PBSE402上,转化大肠杆菌,使用载体特异性引物U6-26-IDF和U6-29-IDR进行菌落PCR验证阳性克隆,PCR检测条带大小为626 bp(图2-C),表明CRISPR/Cas9敲除载体构建成功。提取阳性克隆质粒送公司使用测序引物EDIT-3进行测序,将测序正确的质粒转入农杆菌感受态Ar.Qual中,挑取单克隆进行菌落PCR验证(图2-D),证明该质粒已成功转入农杆菌中。
2.2 发根农杆菌介导的西瓜转化技术检测靶位点的可行性
通过发根农杆菌介导的西瓜转化体系对CRISPR/Cas9表达载体靶位点的可行性进行验证,首先对切好的西瓜子叶外植体进行侵染,在25 ℃下共培养3 d,用无菌水进行脱菌后,放置于MS筛选培养基上进行培养(图3)。
提取长出的不定根系DNA用SPO-1-F/R和SPO-2-F/R对2个靶位点附近的序列分别进行PCR扩增之后琼脂糖凝胶电泳对其进行检测(图4-A),将能够扩增出条带的目的片段进行胶回收送测序。测序结果表明(图4-B),仅在靶位点1的位置出现了2种类型的缺失,分别是:1类型发生了10个碱基的缺失;2类型发生了2个碱基的缺失。但是在靶位点2的位置没有出现编辑的情况(图中未显示),证明所选择的靶位点1有效。结果表明该方法成功实现了在西瓜不定根中的基因编辑,发生了碱基的缺失。
在获得的2个发生编辑的不定根中,进一步使用DNAMAN软件对其氨基酸序列进行分析(图5)。1植株由于在靶位点1处发生了10 bp碱基的缺失导致出现了7个氨基酸的突变以及在第13位氨基酸处翻译提前终止;2植株由于在靶位点1处发生了2 bp碱基的缺失导致在第17位氨基酸处出现突变。结果表明ClSPO11-1编码的氨基酸出现了不同程度的变异,成功敲除了ClSPO11-1基因。
3 讨 论
随着越来越多的生物基因组测序的完成以及模式生物基因功能的挖掘,利用反向遗传学来研究基因功能至关重要。传统的化学和物理等方法来获得突变体植株存在随机性[17],需要大规模的突变体库以及大规模的筛选[18],因此耗时耗力。CRISPR/Cas系统的出现实现了在植物在特定的位置引入突变,为基因功能的研究和遗传改良的应用提供了新的思路。目前北京市农林科学院许勇团队已成功研究出西瓜的遗传转化技术,并且在西瓜中利用CRISPR/Cas9对番茄红素合成相关基因ClPDS进行编辑,与农杆菌转化法结合得到转基因植株,并在其突变体中观察到明显的白化病表型[19],结果表明,CRISPR/Cas9系统可以在西瓜中进行基因组的编辑。
由于西瓜的遗传转化体系过程复杂、周期长以及效率低等问题,本研究建立了一种新的发根农杆菌介导的西瓜子叶外植体产生不定根的方法,该方法的優点是试验周期短、操作相对简单,可以快速测定基因敲除靶位点选择的可行性,避免在试验过程中出现脱靶的情况。利用CRISPR/Cas9系统构建了双靶位载体敲除西瓜ClSPO11-1基因,通过组培的方法诱导西瓜子叶外植体产生了不定根,最终获得了2株在靶位点1处发生编辑的西瓜不定根突变体,表明了该方法检测靶位点是可行的。有研究表明在烟草和蒲公英等植物中,利用发根农杆菌诱导产生的不定根进行脱分化和再分化可以培育成完整的转基因植株[20-21],在南瓜中也通过不定根成功诱导产生了南瓜愈伤组织[22],为诱导西瓜不定根分化形成完整的转基因植株提供了新的依据。
参考文献
[1] 马跃.透过国际分析看中国西瓜甜瓜的现状与未来[J].中国瓜菜,2011,24(2):64-67.
[2] GUO S,ZHANG J,SUN H,et al.The draft genome of watermelon (Citrullus lanatus) and resequencing of 20 diverse accessions[J].Nature Genetics,2013,45(1):51-82.
[3] 瞿礼嘉,郭冬姝,张金喆,等.CRISPR/Cas系统在植物基因组编辑中的应用[J].生命科学,2015,27(1):64-70.
[4] WANG F J ,WANG C L,LIU P Q,et al.Enhanced rice blast resistance by CRISPR/Cas9-targeted mutagenesis of the ERF transcription factor gene OsERF922[J].PLOS ONE,2016,11(4):e0154027.
[5] FENG C,YUAN J,WANG R,et al.Efficient targeted genome modification in maize using CRISPR/Cas9 system[J].Journal of Genetics & Genomics,2016,43(1):37-43.
[6] TSAI S Q,ZHENG Z L,NGUYEN N T,et al.GUIDE-Seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases[J].Nature Biotechnology,2015,33(2):187-197.
[7] GANTZ V M,BIER E.The mutagenic chain reaction:A method for converting heterozygous to homozygous mutations[J].Science,2015,348(6233):442-444.
[8] SMITH E F,TOWNSEND C O.A plant-tumor of bacterial origin[J].Science,1907,25(643):671-673.
[9] VAN L N,ENGLER G,HOLSTERS M,et al.Large plasmid in Agrobacterium tumefaciens essential for crown gall-inducing ability[J].Nature,1974,252(5479):169-170.
[10] CHILTON M D,DRUMMOND M H,MERIO D J,et al.Stable incorporation of plasmid DNA into higher plant cells:the molecular basis of crown gall tumorigenesis[J].Cell,1977,11(2):263-271.
[11] 戴均貴,朱蔚华.发根培养技术在植物次生代谢物生产中的应用[J].植物生理学通讯,1999,35(1):69-76.
[12] BOSSELUT N,GHELDER C V,CLAVERIE M,et al.Agrobacterium rhizogenes-mediated transformation of Prunusas an alternative for gene functional analysis in hairy-roots and composite plants[J].Plant Cell Reports,2011,30(7):1313-1326.
[13] LIN C S,HSU C T,YANG L H,et al.Application of protoplast technology to CRISPR/Cas9 mutagenesis:from single cell mutation detection to mutant plant regeneration[J].Plant Biotechnology Journal,2018,16(7):1295-1310.
[14] LEI Y,LU L,LIU H Y,et al.CRISPR-P:A web tool for synthetic single-guide RNA design of CRISPR-System in plants[J].Molecular Plant,2014,7(9):1494-1496.
[15] YUDUAN D,HONG L,LINGLING C,et al.Recent advances in genome editing using CRISPR/Cas9[J].Frontiers in Plant Science,2016,7(703):1-12.
[16] LIU H,DING Y,ZHOU Y,et al.CRISPR-P 2.0:an improved CRISPR-Cas9 tool for genome editing in plants[J].Molecular Plant,2017,10(3):530-532.
[17] COLBERT T.High-throughput screening for induced point mutations[J].Plant Physiology,2001,126(2):480-484.
[18] TILL B J,REYNOLDS S H,GREENE E A,et al.Large-scale discovery of induced point mutations with high-throughput TILLING[J].Genome Research,2003,13(3):524-530.
[19] TIAN S,JIANG L,GAO Q,et al.Efficient CRISPR/Cas9-based gene knockout in watermelon[J].Plant Cell Reports,2017,36(3):399-406.
[20] ALTAMURA M M,ARCHILLETTI T,CAPONE I,et al.Histological analysis of the expression of Agrobacterium rhizogenes rolB-GUS gene fusions in transgenic tobacco[J].New Phytologist,1991,118(1):69-78.
[21] LEE M H,YOON E S,JEONG J H,et al.Agrobacterium rhizogenes-mediated transformation of Taraxacum platycarpum and changes of morphological characters[J].Plant Cell Reports,2004,22(11):822-827.
[22] BALEN B,LELJAK-LEVANIC D,MIHAIJEVI? S,et al.Formation of embryogenic callus in hairy roots of pumpkin (Cucurbita pepo L.)[J].In Vitro Cellular & Developmental Biology Plant,2004,40(2):182-187.

