基于线粒体DNA标记对3个华鳈群体的遗传结构分析
朱传坤 潘正军 常国亮 吴楠 王辉 丁怀宇 强晓刚 余祥胜
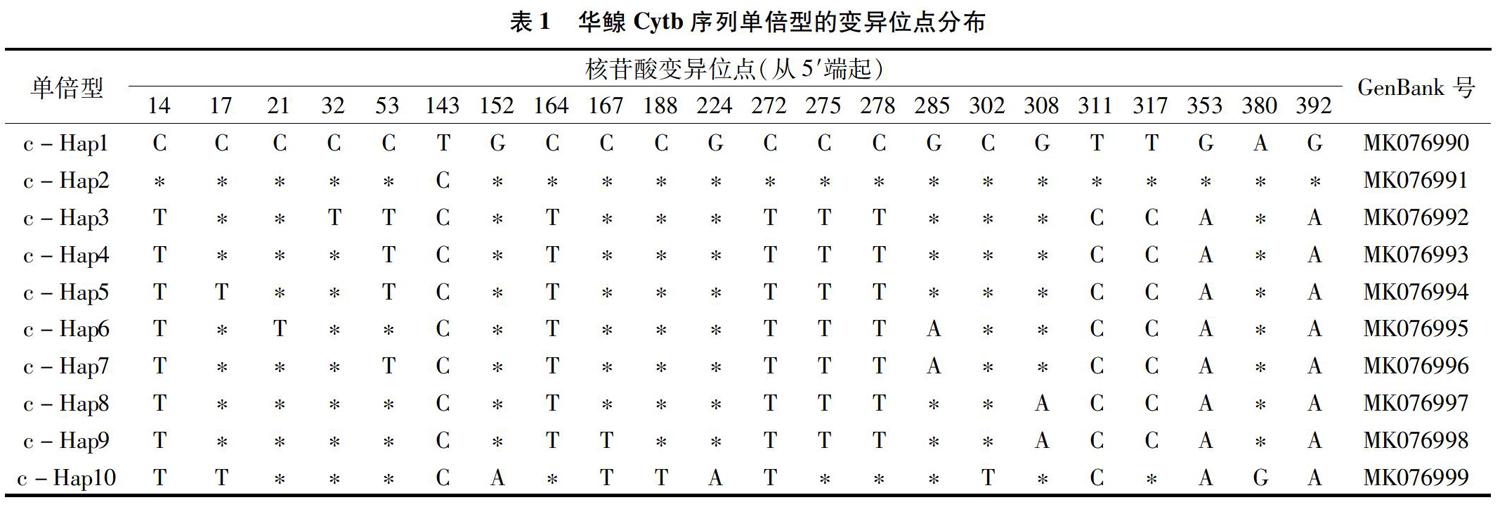


摘要: 华鳈(Sarcocheilichthys sinensis)是一种兼具食用和观赏价值的小型鲤科鱼类。采用Cytb和D-loop 2个线粒体DNA序列对洪泽湖、千岛湖和西湖3个群体的45尾华鳈样本进行了遗传变异与遗传结构分析。结果显示,在长度为404 bp的Cytb序列中,共检测出变异位点22个(5.4%),定义了10种单倍型;在998 bp的D-loop序列中,变异位点有40个(4.0%),构成了7种单倍型。2个标记在华鳈群体中的平均单倍型多样性、核苷酸多样性和平均核苷酸差异数分别为0.840±0.033、0.012±0.002、4.759±1.660(Cytb)和0.755±0.036、0.006±0.002、6.430±2.241(D-loop),表现了较高的遗传多态性。遗传多样性大小顺序为洪泽湖>西湖>千岛湖,3个群体间基因交流较少,表现出极大的遗传分化,此外,分子变异分析结果表明华鳈变异的主要来源为群体内变异。本研究结果将为了解我国东部地区华鳈的遗传多样性现状提供基础资料,同时为华鳈种质资源保护与合理利用提供参考。
关键词: 华鳈;线粒体DNA;Cytb;D-loop;遗传多样性;遗传分化
中图分类号: S917.4 文献标志码: A
文章编号:1002-1302(2020)05-0050-07
华鳈(Sarcocheilichthys sinensis Bleeker),俗称鳈、石鲫、花石鲫、山鲫鱼、山鲤子等,隶属于鲤科(Cyprindae)、[XCZ5.tif;%118%118]亚科(Gobioninae)、鳈属(Sarcocheilichthys)[1-2],广泛分布于我国平原地区大部分江河、湖泊及其附属水体中[2]。华鳈不仅可食部分含有率高,肌肉蛋白质含量高,且其风味氨基酸和不饱和脂肪酸含量丰富,必需氨基酸含量均衡,是一种高蛋白、低脂肪、低能量、味道鲜美的优质食用鱼类[3]。此外,因其色泽鲜亮、体型小、易饲养等优点,也具有较高的观赏价值,深受观赏鱼爱好者喜爱[4-5]。
一直以来,华鳈被视为野杂鱼类,并未引起重视,然而随着大型经济鱼类资源的日渐匮乏,包括华鳈在内的小型鱼类已成为人们的主要捕捞对象[6]。近年来,由于捕捞强度增加,水体污染,以及人类活动造成的栖息地破坏,华鳈野生资源量锐减[7-8]。目前,人们逐渐认识到了这种小型鱼类的经济价值,并开展了一系列关于其人工驯化[8]、繁殖生物学[9]、人工繁殖[4,10]、生长[5]及胚胎发育[10]等研究工作。然而,关于华鳈遗传学及基因组学的研究非常有限[11-13],而针对其群体遗传结构分析的研究尚未见报道。因此,本研究拟利用Cytb和D-loop 2个线粒体DNA标记对我国东部地区3个华鳈野生群体的遗传结构进行分析,以了解该地区华鳈自然资源遗传多样性现状,并为今后华鳈种质资源保护及优良苗种人工繁育提供参考。
1 材料与方法
1.1 样本采集及DNA抽提
课题组于2015年4月至2017年8月在洪泽湖(江苏省淮安市淮阴区韩桥乡)、千岛湖(浙江省杭州市淳安县富溪乡)和西湖(浙江省杭州市西湖北里湖)3个采样点各采集华鳈样本15尾。剪取每尾个体的尾鳍组织后浸泡于无水乙醇中,并于4 ℃冰箱中保存备用。采用传统的苯酚-氯仿法对各样本基因组DNA进行抽提,DNA质量采用Nanodrop 2000微量紫外分光光度计进行检测。
1.2 线粒体DNA片段扩增与测序
本研究选取了线粒体细胞色素b基因(Cytb)和控制区(D-loop)的部分片段作为遗传标记进行分析。2片段的扩增引物均参考已发表的鲤科鱼类通用引物,其中Cytb引物序列为F:5′-[JP9]GACTTGAAAAACCACCGTTG-3′,R:5′-[JP9]CCTCAGAAGGATATTTGTCCTC-3′[14];D-loop引物序列为F:5′-[JP9]CATCTTAGCATCTTCAGTG-3′,R:5′-[JP9]TCACCCCTGGCTCCCAAAGC-3′[15]。
PCR扩增体系总体积为50 μL,其中包含10×reaction buffer 5 μL,Taq DNA聚合酶4 U(大连宝生物),dNTP(2.5 mmol/L)1.6 μL,上下游引物混合液(2.5 μmol/L)1.6 μL,DNA模板80~200 ng,灭菌超纯水36.5 μL。PCR反应在BioRad公司的T-100型PCR仪中进行,PCR反应程序为:94 ℃预变性5 min;94 ℃变性45 s,52 ℃退火45 s,72 ℃延伸1 min,运行37个循环;72 ℃终延伸20 min。PCR产物通过1.5%琼脂糖凝胶电泳后,利用回收试剂盒Gel Extraction Kit(北京康為世纪)对目的片段进行回收和纯化,纯化后的产物送往武汉艾康健生物科技有限公司,利用ABI3730测序平台进行测序。
1.3 数据分析
测序所得序列利用Finch TV 1.5软件进行查看及人工矫正,之后将序列保存为fasta格式,并利用Clustal X 1.83软件[16]进行比对分析。比对分析完成后将比对结果中5′端和3′端参差不齐的序列删除,使各序列具有相同的长度。后利用DnaSP 4.50.3软件[17]分析Cytb和D-loop序列中的多态性核苷酸位点、平均核苷酸差异数(K)、核苷酸多态性参数(π)、单倍型个数、单倍型多态性(Hd)及平均基因流(Nm),此外,利用软件中的Tajimas D和Fus Fs检测法进行中性检测。
在Mega 4.0软件[18]中,利用Kimura 2-parameter遗传距离构建单倍型间的Neighbor-Joining (N-J)进化树,bootstrap值设置为1 000,并利用江西鳈(Sarcocheilichthys kiangsiensis)中相应同源序列作为外类群,GenBank号分别为AY952984.1(Cytb)和KY779851.1(D-loop)。为分析群体间的遗传差异和遗传距离,利用Arlequin 3.11软件[19]进行分子变异分析(AMOVA)和固定系数(Fst)计算。此外,利用IBDWS 3.23软件(http://ibdws.sdsu.edu)中的Mantel检测法对各群体遗传距离和地理距离间的关联性进行分析,参数设置均取默认值。
3 讨论
3.1 华鳈Cytb和D-loop碱基组成与变异程度
在鱼类群体遗传研究中,线粒体DNA因具有分子量小、进化速度快、母系遗传和复制相对独立等优点[20],在多种鱼类群体遗传结构和遗传多样性分析中得到广泛应用。在线粒体基因组中,不同区域之间的进化速率存在较大差异,一般编码蛋白质的基因序列进化较慢,而不编码蛋白的D-loop区域,由于所受选择压力较小,一般比其他线粒体基因的进化速率快2.8~5.0倍[21],是线粒体基因组中进化最快的区域[22],因此,D-loop包含了更为丰富的遗传多样性信息,在水产动物遗传分析中普遍应用。线粒体DNA上结构和功能解析最为透彻的蛋白质编码基因中的Cytb基因进化速度适中,在水产动物种群水平差异检测及种间和种内遗传分化研究中也被广泛应用。在水产动物种群遗传分析研究中,为确保遗传数据的可靠性,研究人员一般会选择2个以上的线粒体DNA片段作为遗传标记,其中利用进化速度快的D-loop序列和进化速度适中的Cytb序列作为标记的研究较多[23-25]。鉴于此,本研究选取D-loop和Cytb序列作为分析华鳈群体遗传结构的标记,以便更加全面地反映华鳈群体遗传结构现状。
在动物线粒体基因组中,A、T、G、C 4种碱基的分布具有明显的偏倚性,表现为A、T 2种碱基含量明显高于G、C含量[26]。本研究获得的Cytb和D-loop序列中,A+T含量均高于G+C含量,这与多种已报道鱼类中的比例[21,23-24,27]类似。插入、缺失位点在华鳈Cytb基因序列中未检测出,而在D-loop序列中共检测出4个,这也反映出2个序列在进化过程中承受了不同选择压力,Cytb基因由于编码蛋白质,插入、缺失突变极易造成移码突变而造成蛋白合成障碍,因此,发生这类突变的个体往往被淘汰[26];而D-loop序列由于不编码蛋白,承受的选择压力相对较小,因此,插入、缺失突变在自然选择过程相对较容易保留下来。正因如此,在已报道鱼类中,D-loop序列的多态位点比例一般均比Cytb序列的高[23-24],然而,本研究却得到与之相反的结果:D-loop序列的多态位点比例(4.0%)低于Cytb序列中的比例(5.4%),最终导致基于D-loop序列分析所得的单倍型数(7)、单倍型多样性(0.755±0.036)和核苷酸多样性(0.006±0.002)均小于基于Cytb序列所得数据(依次为10、0.840±0.033、0.012±0.002)。在另外一种鲤科鱼类拉氏[XCZ26.tif;%118%118](Phoxinus lagowskii)中,也得到了与本研究类似的结果[27],可见,虽然总体而言线粒体基因组中D-loop序列的变异程度高于蛋白编码基因序列,但也存在相反情况,而造成這种变化的原因有待进一步研究。
3.2 华鳈遗传多样性与群体遗传分化
遗传多样性与生物的生存能力、适应能力和进化潜力密切相关[28],物种的遗传多样性越高,其对环境变化的适应能力越强。而Hd和π是衡量物种或种群线粒体DNA遗传多样性程度的2个重要指标,并且有学者以Hd=0.5和π=0.005为界限,将鱼类种群分为分为4种类型:低Hd低π型(Hd<0.5,π<0.005)、高Hd低π型(Hd>0.5,π<0.005)、低Hd高π型(Hd<0.5,π>0.005)和高Hd高π型(Hd>0.5,π>0.005)[29]。本研究中,洪泽湖群体基于2个线粒体序列的分析结果均为高Hd高π型;西湖群体基于Cytb时为高Hd高π型,基于D-loop序列时为低Hd低π型;千岛湖群体基于Cytb时为高Hd低π型,基于D-loop序列时为低Hd低π型。以上分析结果显示,洪泽湖群体的遗传多样性最丰富,而千岛湖群体的遗传多样性最低,这从Cytb和D-loop序列在3个群体中的单倍型数目也可反映出(单倍型数目:洪泽湖>西湖>千岛湖)。虽然千岛湖和西湖华鳈群体的遗传多样性较低,但就本研究所取华鳈的总体样本而言,遗传多样性参数仍处于较高水平,这可能是由于不同湖泊的环境因子对华鳈种群的影响与选择压力不同所致。千岛湖和西湖2个群体基于D-loop序列的核苷酸多样性指数均处于低水平,说明栖息环境破坏、过度捕捞及人类活动等因素已影响了这2个湖泊华鳈种群规模恢复,急需加强种质资源保护工作。本研究结果与杨培民等在拉氏[XCZ26.tif;%118%118]中所得的研究结果[27]极为类似。
在群体遗传分化研究中,常用遗传分化指数Fst来衡量2个群体间的分化程度[30]。在表示群体间的遗传分化程度时,若Fst介于0~0.05,表示遗传分化极小;若Fst介于0.05~0.15,表示遗传分化中等;若Fst介于0.15~0.25,表示遗传分化较大;若Fst大于0.25,表示遗传分化极大[31]。本研究中,洪泽湖和千岛湖华鳈群体间、千岛湖和西湖间均表现为极大的遗传分化,而洪泽湖与西湖群体间利用Cytb序列的分析结果为中度分化,而基于D-loop序列时则为极大分化。此外,尽管千岛湖与西湖均位于浙江省,洪泽湖位于江苏省,但千岛湖与西湖华鳈群体间的遗传分化程度高于与洪泽湖群体间的分化程度,这可能是由于地形与地势等环境因素影响了千岛湖和西湖华鳈群体间的基因交流,从而表现出的地理距离与遗传距离间无显著关联关系。总体而言,华鳈3个群体间遗传分化程度较大,群体间基因交流较少,形成了各自的群体特征,其中较多的群体特异性单倍型就是一个直观的反映。尽管如此,A-MOVA分析结果显示,华鳈的主要变异来源仍然来自群体内部,这与先前报道的绝大多数鱼类的分析结果[23-25]相似。利用Cytb和D-loop序列进行中性检验的结果表明,华鳈未发生近期的群体扩张事件,其群体处于较稳定状态。
参考文献:
[1]张春霖. 中国系统鲤类志[M]. 北京:高等教育出版社,1959:73-74.
[2]伍献文. 中国鲤科鱼类志(下册)[M]. 上海:上海科学技术出版社,1982:470-471.
[3]李绍明,梁志强,伍远安,等. 华鳈肌肉营养成分分析及评价[J]. 安徽农业科学,2011,39(31):19233-19235.
[4]宣云峰,王荣泉,徐海华,等. 华鳈人工繁殖试验[J]. 水产养殖,2010,31(11):35-37.
[5]郭 健,邓其祥,许 明,等. 西河华鳈的年龄和生长研究[J]. 四川师范学院学报(自然科学版),1995,16(4):343-346.
[6]林明利,张堂林,叶少文,等. 洪泽湖鱼类资源现状、历史变动和渔业管理策略[J]. 水生生物学报,2013,37(6):1118-1127.
[7]施振宁. 华鳈的驯养与亲本培育试验[J]. 江西水产科技,2015(2):21-23.
[8]宣云峰,王荣泉,徐海华,等. 华鳈驯化养殖试验初探[J]. 科学养鱼,2010(12):32.
[9]宋天祥,马 骏. 华鳈的繁殖生物学[J]. 动物学研究,1994,15(增刊1):96-102,199.
[10]宋天祥,马 骏. 华鳈人工繁殖和早期发育的研究[J]. 湖泊科学,1996,8(3):260-267.
[11]Zhou Z,Zhang H,Wang G,et al. Complete mitochondrial genome of Chinese lake gudgeon Sarcocheilichthys sinensis (Cypriniformes:Cyprinidae)[J]. Mitochondrial DNA,2015,26(1):141-142.
[12]Li C,He L,Chen C,et al. Sequencing,description and phylogenetic analysis of the mitochondrial genome of Sarcocheilichthys sinensis sinensis (Cypriniformes:Cyprinidae)[J]. Mitochondrial DNA,2016,27(2):1056-1057.
[13]Yang S,Wei M,Wan Q,et al. The complete mitochondrial genome sequence of Sarcocheilichthys sinensis sinensis (Cypriniformes:Cyprinidae)[J]. Mitochondrial DNA,2014,25(4):286-287.
[14]Xiao W,Zhang Y,Liu H. Molecular systematics of Xenocyprinae (Teleostei:cyprinidae):taxonomy,biogeography,and coevolution of a special group restricted in East Asia[J]. Molecular Phylogenetics and Evolution,2001,18(2):163-173.
[15]Liu H Z,Tzeng C S,Teng H Y. Sequence variations in the mitochondrial DNA control region and their implications for the phylogeny of the Cypriniformes[J]. Canadian Journal of Zoology-Revue Canadienne de Zoologie,2002,80(3):569-581.
[16]Thompson J D,Gibson T J,Plewniak F,et al. The CLUSTAL_X Windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research,1997,25(24):4876-4882.
[17]Rozas J,Sánchez-Delbarrio J C,Messeguer X,et al. DnaSP,DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics,2003,19(18):2496-2497.
[18]Tamura K,Dudley J,Nei M,et al. MEGA4:molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution,2007,24(8):1596-1599.
[19]Schneider S,Roessli D,Excoffier L. Arlequin:a software for population genetics data analysis.V3.01.Genetics and Biometry Lab,Department of Anthropology[Z]. 2003.
[20]肖武漢,张亚平. 鱼类线粒体DNA的遗传与进化[J]. 水生生物学报,2000,24(4):384-391.
[21]袁 娟,张其中,李 飞,等. 铜鱼线粒体控制区的序列变异和遗传多样性[J]. 水生生物学报,2010,34(1):9-19.
[22]郭新红,刘少军,刘 巧. 鱼类线粒体DNA研究新进展[J]. 遗传学报,2004,31(9):983-1000.[HJ1.91mm]
[23]陈会娟,刘明典,汪登强,等. 长江中上游4个鲢群体遗传多样性分析[J]. 淡水渔业,2018,48(1):20-25,68.
[24]李大命,张彤晴,关浩勇,等. 基于线粒体Cytb基因和D-loop区序列的高邮湖湖鲚(Coilia nasus)遗传多样性分析[J]. 淡水渔业,2017,47(6):3-8.
[25]户 国,程 磊,马 波,等. 中国达氏鳇野生群体和两个养殖群体的线粒体遗传多样性分析[J]. 中国水产科学,2018,25(4):803-810.
[26]Brown W. Molecular evolutionary genetics[M]. New York:Plenum Press,1985:95-130.
[27]杨培民,胡宗云,金广海,等. 用線粒体D-loop和Cytb基因序列分析拉氏[XCZ26.tif;%88%88]3个群体的遗传结构和遗传分化[J]. 水产学杂志,2017,30(4):7-12,27.
[28]Féral J P. How useful are the genetic markers in attempts to understand and manage Marine biodiversity[J]. Journal of Experimental Marine Biology and Ecology,2002,268(2):121-145.
[29]Grant W S,Bowen B W. Shallow population histories in deep evolutionary lineages of Marine fishes:Insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity,1998,89(5):415-426.
[30]Kirk H,Freeland J R. Applications and implications of neutral versus non-neutral markers in molecular ecology[J]. International Journal of Molecular Sciences,2011,12(6):3966-3988.
[31]Balloux F,Lugon-Moulin N. The estimation of population differentiation with microsatellite markers[J]. Molecular Ecology,2002,11(2):155-165.
收 稿日期:2019-02-14
基金项目:江苏省高校自然科学基金面上项目(编号:15KJB240001);淮阴师范学院博士科研启动项目(编号:31ZCK00)。
作者简介:朱传坤(1984—),男,山东临沂人,博士,副教授,主要从事鱼类基因组学与分子标记辅助育种研究。E-mail:zhuchuankun@hytc.edu.cn。

