2019新型冠状病毒及其诊疗与防控:回顾与展望
叶子葳,金冬雁
·特邀综述·
2019新型冠状病毒及其诊疗与防控:回顾与展望
叶子葳,金冬雁
香港大学 李嘉诚医学院微生物学系 生物医学学院,中国香港
2019新型冠状病毒的暴发持续至今,导致了世界各地数以百万计的感染个例,更夺去了数十万人的生命。世界卫生组织在2020年2月将此病毒引起的疾病定名为2019冠状病毒病(Coronavirus disease 2019,COVID-19),而国际病毒分类委员会也将此病毒命名为SARS-CoV-2。COVID-19的典型临床症状类似感冒,少数病人可发展为重症甚至死亡。21世纪以来,人类冠状病毒有3次大暴发,分别是2003年暴发的严重急性呼吸综合征(SARS)、2012年暴发的中东呼吸综合征(MERS) 和本次的新型肺炎。自2003年以来,对SARS和MERS冠状病毒的研究从未间断,对其自然起源、致病机理、药物筛选及疫苗研发等已取得一定进展。鉴于SARS-CoV-2和SARS-CoV的基因组序列高度相似,以往对SARS-CoV的研究对深入探讨SARS-CoV-2生物学特性、诊断、治疗和防控有很强的借鉴性。文中通过回顾过往的研究进展,对比SARS-CoV和SARS-CoV-2的生物学特性,分析当前亟需的防控和诊疗措施,探讨疫苗研发所面对的一些难题,并展望疫情发展趋势及对本领域研究与开发的主要挑战,冀为我国和全世界有效控制COVID-19疫情提供参考。
2019冠状病毒病,严重急性呼吸综合征冠状病毒,2019新型冠状病毒,中东呼吸综合征冠状病毒,病 毒诊断,冠状病毒疫苗,抗病毒治疗
自2019年12月以来,突如其来的新型冠状病毒肺炎疫情席卷全球。此症主要通过人与人之间的飞沫及密切接触传播。其典型临床症状为发热、干咳、呼吸困难、头痛和肺炎。少数患者可发展为重症,因肺泡损伤而引起进行性呼吸衰竭,甚至死亡[1]。截止2020年3月24日,全国累计有超过8.2万个确诊病例,其中包括3 200多个死亡病例;而国外的确诊和死亡病例数则超过30万例和1.3万例。截止2020年4月21日,全球累积确诊和死亡超过244万例和17万例。2020年1月底,世界卫生组织(WHO)将新型冠状病毒的暴发定义为“国际突发公共卫生事件”,随后又在3月11日宣布新型冠状病毒疫情构成全球大流行。2020年2月WHO将新型冠状病毒引发的疾病正式命名为COVID-19,而国际病毒分类委员会则将原先的2019新型冠状病毒(2019-nCoV) 正式命名为SARS-CoV-2。面对猝不及防的疫情,我国学者迅速分离出该病毒并对其进行了测序,及时在国际上共享了新病毒序列并迅速开展相关的研究。除了隔离已感染的病人并抢救重症患者,有效遏制SARS-CoV-2的传播,研发安全有效 的抗病毒药物和疫苗将是下一步所面临的最大挑战。
冠状病毒最早发现于鸟类和哺乳动物[2],是一类有包膜的单股正链RNA病毒,可在人及各种动物中引起严重的呼吸系统、消化系统和神经系统疾病[3-5]。它可以分为α、β、γ和δ四个不同的属。在过去近20年中,世界上曾有3次较大规模的人类冠状病毒流行,包括2003年暴发的严重急性呼吸综合征(SARS)、2012年暴发的中东呼吸综合征(MERS) 和近期暴发的新型冠状病毒肺炎(COVID-19)。这3种病毒都属于β冠状病毒,且传染性和致病性较强。除此之外常见的人类冠状病毒还有229E、NL63、OC43和HKU1,能引起普通感冒,又称为社区获得性人类冠状病毒。229E和NL63均属α冠状病毒,而OC43和 HKU1属β冠状病毒[3-5]。2003年以来对于人类冠状病毒的研究有所加强,涉及其动物溯源、跨种传播、致病机理、与细胞相互作用、感染与免疫、抗病毒药物及疫苗研发等各个领域。由于SARS-CoV-2和SARS-CoV高度同源[6],而且SARS-CoV-2的某些特点与社区获得性人类冠状病毒也有相类似的一面,所以以往对各种人类冠状病毒的研究结果对SARS-CoV-2都有一定的参考价值。本文结合我们已知的对冠状病毒的认识,以SARS-CoV-2的全基因组序列为依据,比较及分析SARS-CoV-2的生物学特征,并就其防控、诊疗及疫苗研发的重点和难点提出意见和建议。
1 SARS-CoV-2的生物学特性
1.1 形态结构和序列特点
冠状病毒分类上属于巢病毒目、冠状病毒科(下分为冠状病毒亚科和环曲病毒亚科)。其基因组大小在26 000至32 000个碱基对之间,是基因组规模最大的一类RNA病毒。在电子显微镜下,我们可以看到冠状病毒呈圆球状,或者椭圆形,上面有规则排列的囊状胶原蛋白,看起来像是中世纪欧洲帝王的皇冠——“冠状病毒”因此得名[4]。冠状病毒粒子形状并不规则,直径约60–220 nm。在结构上,该病毒的囊膜是由双层脂质构成并穿插膜蛋白和纤突蛋白。在病毒的内部,分布着呈螺旋结构的病毒RNA,与衣壳蛋白共同组成核蛋白核心[4,7]。电镜下观察SARS-CoV-2颗粒一般呈球形,直径为60–140 nm。病毒颗粒有明显的纤突,导致病毒颗粒呈现日冕状。在人呼吸道上皮超薄切片中观察到胞外游离的病毒颗粒,以及胞质囊膜内充满病毒颗粒的包涵体。所观察到的形态与冠状病毒科完全相符[8]。
从结构上看,SARS-CoV、MERS-CoV和SARS-CoV-2这3种β冠状病毒,在它们多顺反子基因组的5′端编码不同的非结构蛋白(Non-structural proteins,NSP)[6],即ORF1a和ORF1b复制酶,可被加工为不同的非结构蛋白。紧接下来是多个结构蛋白(Structural proteins) 的编码区,依次是纤突蛋白(Spike,S)、包膜蛋白(Envelope,E)、膜蛋白(Membrane,M) 和核衣壳蛋白(Nucleocapsid,N),以及多个具有组特异性的辅助蛋白(Accessory proteins) (图1)。SARS-CoV-2的基因组是由29 891个碱基对组成,编码9 860个氨基酸。它和SARS-CoV的核苷酸序列一致性达82%[6]。SARS-CoV的NSP1、NSP3、ORF3b、ORF6、M和N蛋白对干扰素有拮抗作用[5],而它们在SARS-CoV-2中对应蛋白分别有84%、76%、32%、69%、91%和94%的氨基酸序列完全相同[6]。在SARS-CoV中被证明可诱导NLRP3炎症小体激活的E蛋白和ORF3a蛋白[9],在SARS-CoV-2的对应蛋白有高达95%和72%的氨基酸序列完全相同。SARS-CoV和SARS-CoV-2的差别主要表现在ORF3b、ORF8和S蛋白,特别是后两者[6]。这3个蛋白在SARS-CoV-2中的功能,已经成为后续研究的热点。它们在SARS-CoV-2和SARS-CoV之间的差异是否导致两种病毒的表型差异,需要通过各种实验进一步验证。

图1 SARS-CoV、MERS-CoV-2和SARS-CoV-2的基因组构成
SARS-CoV-2和SARS-CoV两种病毒在感染率及致病性方面有一定差异,SARS-CoV-2感染率较高而致病性较低,推测其抑制干扰素或激活NLRP3炎症小体导致病理性炎症反应的能力可能要比SARS-CoV稍弱。这一推断如果得到实验证实,可以继续通过比较SARS-CoV-2和SARS-CoV之间的几种干扰素拮抗蛋白及炎症小体激活蛋白的活性进一步阐明其分子基础。这几种蛋白有些很相似[6],如果其活性有差异可以迅速确定哪些氨基酸残基的不同对活性有关键影响。另有几个蛋白差异较大[6],通过比较其活性可能确定SARS-CoV-2是否通过改变其中的几种在不同组的病毒之间各不相同的辅助蛋白从而显著提高对人类的适应性。因此,比较SARS-CoV-2和SARS-CoV生物学特性的差异应是本领域中的一个研究重点和热点。
1.2 复制特性
除去最基本的遗传物质与蛋白质结构,冠状病毒外侧还有由脂质和糖蛋白组成的包膜。包膜的主要功能是维护病毒结构的完整性,并参与病毒入侵宿主细胞的过程。首先包膜上的S糖蛋白识别并结合位于宿主细胞表面的受体,包膜与宿主细胞的细胞膜融合,随后病毒衣壳与遗传物质进入宿主细胞内,完成病毒侵入细胞的过程[10]。SARS-CoV-2和SARS-CoV的基因组序列高度相似,其生命史和其他大多数RNA病毒也相似,总结起来就是附着、入侵、脱壳、合成、组装和释放的过程(图2)。冠状病毒表面的S蛋白可以被宿主细胞的受体蛋白识别,进而入侵释放出其遗传物质RNA,这些RNA直接被翻译成可被蛋白酶降解为多种NSP的多蛋白ORF1a及ORF1ab,并形成复制转录复合体(Replication-transcription complex,RTC),负责病毒蛋白的大量转录和翻译工作[11]。
很多研究证明冠状病毒的NSP对其复制有很重要的作用[12]。十六个非结构蛋白的已知功能总结详见表1。除了复制,病毒的包装和感染性依赖于它的4个结构蛋白。S蛋白,正如前面所讲的,在识别并结合宿主细胞表面受体及介导病毒包膜与细胞膜融合的过程中起到关键性作用[13];M和E蛋白则参与了病毒包膜的形成与出芽过程,M蛋白主要是负责包膜形成[14],而E蛋白更多地参与病毒蛋白的装配和释放[15]。对于N蛋白的多种不同功能,文献中曾有报道[12]。已知N蛋白可与病毒基因组RNA相互缠绕形成病毒核衣壳,在病毒RNA的合成过程中发挥重要作用。一方面,它可以通过和NSP3作用来帮助RTC更好地结合到病毒的RNA基因组进行复制;另一方面,它可协助将病毒核衣壳组装到病毒颗粒中[12]。此外,N蛋白还可以抑制干扰素以促进病毒的复制[16]。
理解冠状病毒的复制特性,可为研究和发现病毒特异性的抗病毒药物奠定重要基础。理论上,只要有效抑制SARS-CoV-2复制周期的任何一个关键步骤,都可收到抑制病毒复制的效果。病毒复制需要核苷酸,采用核苷和核苷酸类似物可以竞争性抑制很多步骤,它们也可以掺入正在合成的RNA链,阻断其继续延伸。病毒复制涉及很多酶包括蛋白酶及解旋酶,抑制其活性也可阻断病毒的生命周期,其中通过化学生物学方法筛选各种酶的抑制剂,是发现新的小分子抗病毒药物的常用方法[17-18]。了解病毒复制过程中的蛋白质-蛋白质相互作用,也可通过肽模拟技术,研究开发可抑制关键病毒蛋白与其他病毒或细胞蛋白相互作用的肽模拟分子(Peptide mimetics),从而有效抑制病毒复制[18]。以上3种策略,在设计和研发针对其他病毒特别是艾滋病毒的抗病毒药物方面已取得一定成果,其基本原理也适用于SARS-CoV-2的药物研发。
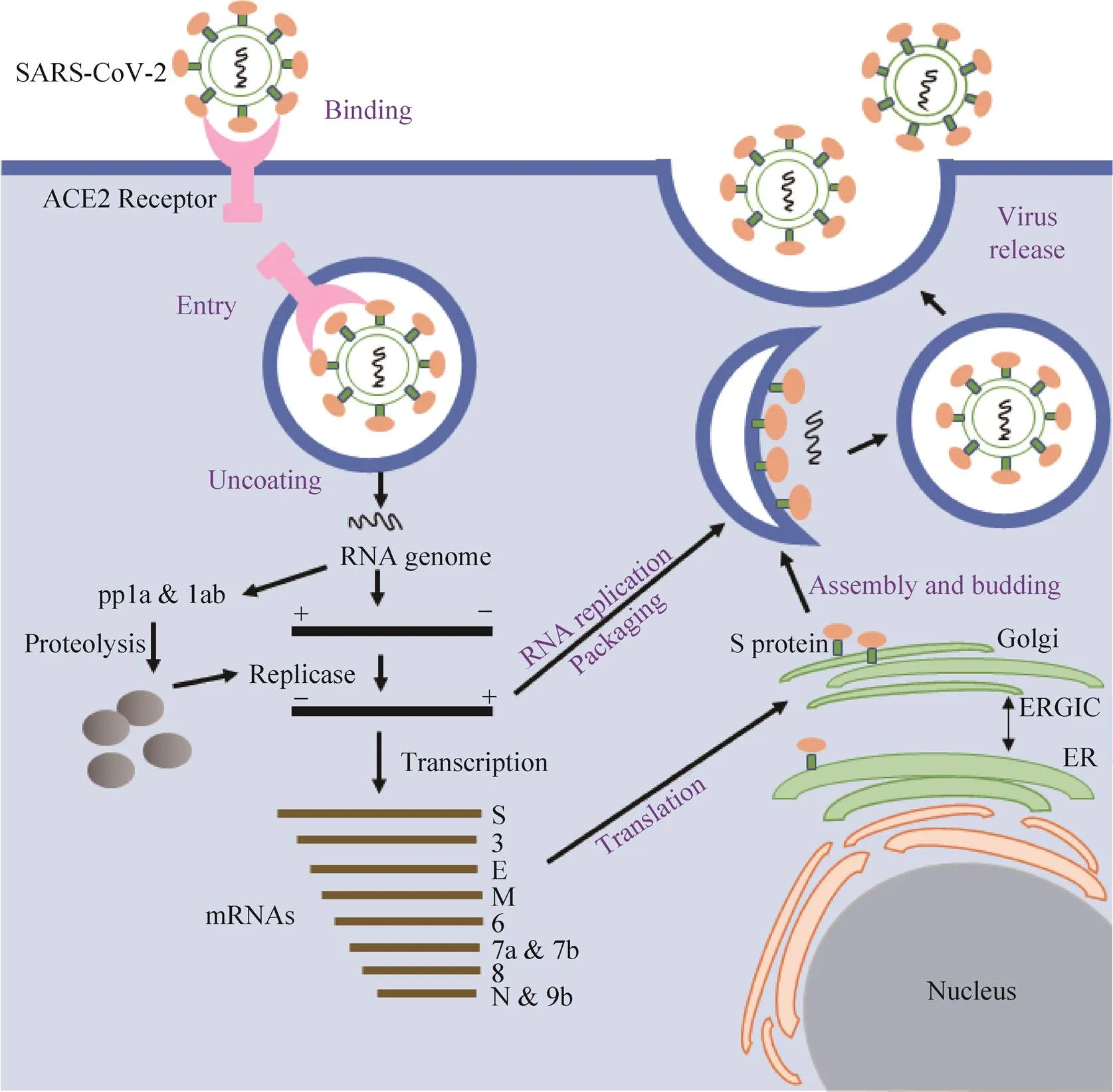
图2 SARS-CoV-2的生命史

表1 冠状病毒非结构蛋白的功能
1.3 细胞受体
SARS-CoV-2和SARS-CoV的S蛋白的氨基酸序列差异相对比较大,尤其是受体结合域只有73%相同[6]。但它们都可以与人类细胞受体血管紧张素转化酶2 (Angiotensin converting enzyme 2,ACE2)有效结合[19-20]。通过计算机模型对比SARS-CoV-2和SARS-CoV的S蛋白,发现其与ACE2相互作用的5个关键氨基酸残基中有4个都不同[20],但是SARS-CoV-2的S蛋白不仅仍可与人ACE2蛋白结合,而且根据冷冻电镜的研究结果其结合亲和力比SARS-CoV的S蛋白高10到20倍[21]。ACE2广泛地存在于黏膜细胞中,曾有研究对ACE2蛋白在人体器官组织中的表达水平进行检测,结果明确指出ACE2在肺部组织和小肠组织表达最丰富。此外ACE2在几乎所有器官的内皮细胞和平滑肌细胞上的表达也很丰富[22]。ACE2在消化系统的高表达量也解释了为何SARS-CoV-2除了呼吸道感染之外,消化道感染也比较常见,而腹泻也是SARS-CoV-2感染的主要临床表现之一,这种情况与SARS-CoV也十分相像。
S蛋白除与ACE2结合,也可与主要的中和抗体结合,因此S蛋白的主要中和表位也是疫苗研究的重要内容[23]。S蛋白的激活需要由宿主细胞蛋白酶切割为S1和S2[24],其中S1负责与ACE2的结合,而S2的一个重要功能就是介导病毒包膜与靶细胞质膜的融合[24-25]。这个过程与其他重要人类病毒表面糖蛋白所诱导的膜融合很相似,也是抗病毒药物研发的重要标靶[26]。负责切割的宿主细胞蛋白酶包括TMPRSS2,其抑制剂已经用 于临床,已被证明有抑制SARS-CoV-2复制的作用[24]。抑制艾滋病毒gp41蛋白与细胞质膜融合的肽模拟分子已经研制成功并应用于临床[27],其经验对于SARS-CoV-2也基本适用。同理,抑制艾滋病毒gp120蛋白与其细胞辅助受体CCR5结合的小分子抑制剂Selzentry也已批准用于临床,该化合物最初来源于以抑制两者结合为目标的高通量筛选,再经过多轮优化和试验而得到[28]。这些成功经验对SARS-CoV-2抗病毒药物的筛选具有一定的借鉴意义。
1.4 理化性质
SARS-CoV和MERS-CoV在干燥环境下可在体外存活48 h,在20 ℃和40%–50%的湿度下最多可存活5 d[29-30]。关于SARS-CoV-2的理化性质,目前还没有具体的报道,但是它极有可能与SARS-CoV和MERS-CoV相似。有报道称SARS-CoV-2对紫外线和56 ℃加热30 min敏感。乙醚、75%的乙醇、含氯消毒剂、过氧乙酸、氯仿和其他脂肪溶剂都能有效地灭活病毒。据此我们推测,温度和湿度对SARS-CoV-2的体外存活可能确有一定影响[31],应通过实验并结合流行病学资料作进一步的研究和分析。目前SARS-CoV-2有活跃传播的几个地区气温都较低、日照较充分而且相当干燥,包括意大利北部、韩国大邱、伊朗库姆和西班牙马德里,而气温较高但比较潮湿的新加坡则传播有限。其中是否有规律可循,值得采用科学方法分析判定,并据以推断是否疫情有可能在夏天来临后退却。
2 COVID-19的特点
2.1 临床表现
COVID-19的症状存在比较大的个体差异,其严重程度介乎无症状感染到危重不等。对疫情初期的47例患者进行的医学观察发现,SARS-CoV-2感染者以发热(98%)、乏力(44%)、干咳(76%)为主要表现。少数患者伴有咳痰(28%)、腹泻(3%)等症状[1,32]。重症病例多在一周后出现呼吸困难或者低血氧症,严重者会快速地发展为急性呼吸窘迫综合征(Acute respiratory distress syndrome,ARDS)、脓毒症休克、代谢性酸中毒和出凝血功能障碍[1]。值得注意的是重症、危重患者病程中可仅出现中低热,甚至没有明显发热(表2)。大部分患者仅表现为低热、轻微乏力等,无肺炎表现,多在一周后恢复[1]。此后还有一系列的研究说明了COVID-19的临床表现[33-35],其中一项关于143位COVID-19住院病人的研究表明病人中腹泻的比例高于前述研究,可达到14%[35]。对4万多确诊病例的流行病学分析表明,轻症及中症患者占80.9%,重症占13.8%而危重则有4.7%[36]。最近在欧洲部分患者中还发现丧失嗅觉和味觉的症状。COVID-19的病程多在两周之内,这一特点与SARS-CoV及其他人类冠状病毒基本相似。如果检测病毒核酸的灵敏度较高,从第一次检测阳性到第一次检测阴性的时间会较长,平均为20 d左右。根据临床症状,轻症及中症的COVID-19很难与流感甚至普通感冒区分。

表2 SARS-CoV-2病毒与SARS-CoV和MERS-CoV病毒的比较
临床上一般可根据体温升高、淋巴细胞减少、胸部计算机体层显像(CT) 发现肺部浸润或磨玻璃影等标准,将这些患者确定为病毒性肺炎。在重症COVID-19病人中发现,其血液中有很高浓度的白细胞介素2、7和10、IP10、MCP1和肿瘤坏死因子α等,显示患者体内正出现严重的细胞因子风暴。
造成COVID-19临床症状的根本原因是SARS-CoV-2感染,但人体固有免疫系统及抗病毒应答被激活后所引起的病理性炎症反应,则是肺及其他器官损伤的直接原因[33]。病毒及免疫是一对矛盾,正是它们之间复杂多变的相互作用造成COVID-19临床表现的多样化及严重程度差异。目前在临床上的一大挑战是如何尽早识别重症患者,血液中乳酸、C反应蛋白及白细胞介素6等炎症因子的急剧升高和淋巴细胞的急剧减少,以及肺内病变急剧发展的影像学证据,都可以作为重症患者早期诊断的重要指标。
对于COVID-19患者的预后尚待研究。但致病性更强的SARS-CoV导致重症病人更多而病情也更重的SARS,其预后也良好[37]。虽然肺纤维化不可逆也无药可治,但SARS引起的肺纤维化经过一段时间还是可以完全吸收,而患者也可以完全康复[38]。SARS的后遗症,很多是由高剂量类固醇激素治疗所引起,与其他因使用类固醇而引起的并发症无异[38]。
2.2 传播
SARS-CoV-2主要通过呼吸道分泌物排出,经喷嚏、唾液等飞沫传播,也可以通过与患者或病毒携带者的密切接触传染[1,32]。与SARS-CoV一样,在患者的粪便中也检测到SARS-CoV-2,说明病毒可能在消化道复制并且存在,提示存在粪口传播的可能[39]。然而,目前尚无明确证据支持粪口传播在COVID-19疫情中确有重要作用。对它可能在哪些特定条件下发生并发挥重要影响,仍待通过流行病学研究阐明。同样,气溶胶传播可能仅在密闭空间内从事抽痰、气管插管等医疗程序时发生,并不适用于整体或一般情况下的传播。目前没有任何确实证据支持SARS-CoV-2可通过气溶胶传播和母婴传播的猜测。以往研究SARS-CoV在中国香港淘大花园的传播时有观点认为,粪便中的病毒可能通过含有病毒的飞沫形成气溶胶方式的传播,这是无法证实的猜测。对此值得加强警觉,但无需过分担心。
COVID-19的潜伏期为1–14 d,多在一周之内(2–7 d),中位数为4 d。从出现症状到确诊的时间中位数为5 d (2–9 d),从发病到死亡的时间中位数为9.5 d (4.8–13 d),两周以后传染的可能性较小[1]。虽然早期的COVID-19患者包括一个与武汉市华南海鲜批发市场有关的群组[1],但随着该市场及多数地区野生动物交易市场的关闭,野生动物已不再是当前疫情流行的主要传染源。最初认为人际传播有限显属误判,至于人际传播能否持续及病毒经过多轮人际传播后传染性及致病性是否有变化,则仍待认真分析现有流行病学数据加以查明。这是目前需要解答的重要课题,应集中力量以实事求是的精神尽快提出切实的分析和解答。
与SARS-CoV-2相比,SARS-CoV及MERS-CoV经过几轮人际传播后其传染性或传播力有所减弱[39]。人类的MERS-CoV感染最初都由骆驼传给人,最后在人间终结。病毒虽经反复由骆驼传入人类,但始终未能持续有效地展开人际传播[40]。如果SARS-CoV-2与这两种病毒一样,COVID-19在全球的疫情最终将归于平寂。但由于SARS-CoV-2的传播和致病性也呈现出其他4种社区获得性人类冠状病毒的特征,如果SARS-CoV-2的传播力始终没有减弱,我们更应做好继续应对及疫苗研制的准备。
香港大学微生物学系研究团队通过分析一个聚集感染的家庭,在国际上首次证实SARS-CoV-2的人际传播,指出此病毒在家庭内的传播力极强而攻击率达83%,并报道无症病毒携带者的存在[41]。这篇高水平的临床研究论文,对于国家判断疫情及采取果断措施应对发挥了重要作用。此后的研究表明,SARS-CoV-2的无症携带者中有一部分人体内含有较高的病毒载量,但没有或只有轻微及不容易察觉的症状[42]。这些无症状病毒携带者再加上部分轻症患者及在潜伏期最后一两天具有传染性的患者,对于有效控制疫情构成重大挑战。在COVID-19疫情中,无症携带者的数目、比例及传染性亟待查明,应作为重中之重的研究课题之一。
评价一个病毒的传染性或传播力的强弱,其中一个重要的参数叫做基本再生数R0(Basic production number),表示在自然传播下一个患者平均能把疾病传染给多少个人[43]。当患者被隔离的时候,R0可以有效降到最低。相比R0为2–5的SARS-CoV,SARS-CoV-2的传染性与SARS-CoV至少相似或者更强,在前一段的R0已达到4–6[44-45]。有研究预测了中国内地几大城市疫情发展情况,推测会在2020年4月前后到达高峰[46]。由于隔离和防护等措施到位,疫情规模已显著缩小,高峰也提前在2月中旬到达。
2.3 易感人群
各类人群对SARS-CoV-2普遍易感。在过往研究中,SARS和MERS发病的严重程度与患者的年龄、性别及过往健康史有关。对SARS-CoV-2感染者的流行病学分析显示,年长的患者病情通常更严重,死亡率也更高[36]。虽然各个年龄段都存在感染者,确诊病例以30–65岁的患者为主,有近一半患者年龄大于50岁,男性发病率高于女性。疾病发生呈现明显的地理聚集性。
相似的是,SARS及MERS同样是对于50岁以上人群感染率和致死率较高。对于MERS,有抽烟史、高血压、糖尿病和心血管疾病者更容易受感染并死亡[47]。同样,长期病患者感染SARS-CoV-2后也更容易发展为重症。此外,儿童及婴幼儿也有发病,但病情相对较轻,很少年幼患者出现危重。需要注意的是,医护人员和患者家属在治疗和陪护时同患者近距离密切接触,也有较高的感染风险。武汉大学中南医院2020年1月1日至28日连续入院的138例患者中,医务人员的比例高达29%。这提示医护人员的防护尤为重要[35]。联系到这批住院病人有45%接受了甲基泼尼松龙的治疗,很有必要查明传染医护人员的患者是否有接受激素治疗,而激素对于病毒载量、病程及愈后的影响也应一并分析清楚。
3 SARS-CoV-2的进化与溯源
追本溯源,大多数冠状病毒的最初源头都是蝙蝠。已知SARS-CoV和MERS-CoV 最早的祖先就源于蝙蝠[48]。其他的冠状病毒如NL63和229E也来自蝙蝠。NL63和229E经过多年适应,已经在人类中非常普遍,但只是引起普通感冒[49-51]。OC43和HKU1虽然最初也可能来自蝙蝠,但它们应该已适应啮齿类动物,再传给家畜,其中人类的OC43直接来源于牛。如果追溯回去一两百年甚至更长时间,这些病毒刚刚进入人体的时候,也一样会引起全球性大流行,而且其致病性照样会比较高,很可能跟现在SARS-CoV-2的情况相似,但后期毒力慢慢减弱,最终只引起普通感冒。历史上有记载在1890年前后曾发生呼吸道疾病的世界大流行,推断与OC43从牛到人的跨种传播时间恰好吻合[52]。如上所述,近20年出现的SARS-CoV和MERS-CoV从动物到人后经过几代人际传播后传播力都呈减弱之势,说明它们仍未完全适应人体,SARS-CoV-2是否也具有如此传播特征,对疫情控制有关键影响,值得深入探究。
作为多种已知冠状病毒的宿主,蝙蝠拥有独特的免疫系统,为通过突变和重组形成新的冠状病毒创造了条件[48]。进化分析显示,SARS-CoV-2与来自中菊头蝠(中马蹄蝠的一种) 的蝙蝠SARS样冠状病毒(Bat SARS- related coronaviruses)最为相似,核苷酸一致性达到84%,与人类SARS-CoV的核苷酸一致性则达到78%,与MERS病毒的核苷酸一致性达到约50%[20]。中国科学院武汉病毒研究所石正丽研究团队进一步将SARS-CoV-2基因组与实验室早年检测到的蝙蝠冠状病毒的部分基因序列进行比较,发现该病毒与来源于中菊头蝠样本的一株冠状病毒RaTG13最相似,两种病毒核苷酸序列一致性高达96.2%[20]。目前,学界广泛接受的推测是,SARS-CoV-2最原始的宿主为蝙蝠,通过某种动物宿主扩散到人类,并引起疾病的发生。不过我们也不能排除蝙蝠直接把冠状病毒传给人,因为吃蝙蝠而在宰杀蝙蝠的过程中也有直接将病毒传给人类的可能。有人认为229E就是直接从蝙蝠传播给人类的[51]。
病毒从蝙蝠传给人往往需要有一个或多个中间宿主。SARS-CoV在果子狸等野生动物也曾经被发现[53]。有研究发现,人和蝙蝠中的MERS-CoV有高度同源性并能感染骆驼[54-55]。在中东和非洲地区,很多骆驼都携带着MERS病毒并不断传播给人[54-55]。研究人员把MERS病毒接种到骆驼身上,骆驼只是出现较轻症状[54]。鉴于中东地区骆驼对于当地运输的重要作用,人们不可能选择把骆驼全部宰杀,这就是为什么从2012年到现在不断有人类感染MERS病例出现的原因。
大多数种类的蝙蝠栖息于热带和亚热带雨林或岩洞中,远离人类活动区域。蝙蝠病毒需要进入某种半野生状态的哺乳动物(即动物宿主) 体内继续进化,经过一定的突变和重组后传播到人类[48]。流行病学调查显示,此次疫情与武汉市华南海鲜批发市场(存在野生动物交易) 有关。最早41例确诊病例中,27例有华南海鲜批发市场的接触史[1]。同时,在该市场的案板及部分从业人员的手套等环境样本中也查出SARS-CoV-2病毒核酸。由此推测,在野生动物买卖、经营、运输、屠宰、交易等过程中,病毒由动物宿主传到人,进而再出现人际传播。全面查禁野生动物饲养买卖之后,要查出哪种动物受到感染并不容易。如果此动物只是短暂或一过性的中间宿主,如SARS-CoV感染果子狸的情况,也许永远也无法重现华南海鲜批发市场或其他野生动物市场曾经出现的实际情况。最近有研究表明,穿山甲可能是SARS-CoV-2的动物宿主之一,从穿山甲分离的β冠状病毒与目前感染人的毒株序列高度相似,全基因组核苷酸序列一致性约90%[56]。虽然在全基因组核苷酸序列水平上RaTG13与SARS-CoV-2的一致性更高,但比较受体结合域的氨基酸序列,SARS-CoV-2与穿山甲β冠状病毒更接近,与RaTG13的差异却比较大。目前的证据只能说明穿山甲可携带与SARS-CoV-2近似的β冠状病毒,尚不能说明SARS-CoV-2是穿山甲传给人类的。
对于上述3种冠状病毒的关系,目前尚无定论,至少有3种可能。第一,穿山甲也许与果子狸传播SARS-CoV的情况一样。它们从蝙蝠获得SARS-CoV-2样病毒,然后传给人类。有迹象显示感染冠状病毒的穿山甲有病甚至重病,而且人类确实因为喜吃野味而与穿山甲密切接触。这些都支持穿山甲是SARS-CoV-2的一过性中间宿主。如果确实如此,应该在穿山甲中找到与SARS-CoV-2极为相似的病毒,而用SARS-CoV-2实验感染穿山甲也应引起严重疾病。第二,穿山甲与蝙蝠有共同的生境,它们之间的β冠状病毒可能发生重组,形成新的蝙蝠SARS-CoV-2样病毒,再传给人类。也就是说,SARS-CoV-2样病毒虽然在蝙蝠中形成,但穿山甲β冠状病毒为其贡献了重要的基因片段。这种情况下,应该在蝙蝠中找到与SARS-CoV-2极为相似的病毒。第三,穿山甲和蝙蝠都没有与SARS-CoV-2极为相似的病毒,但它们的SARS-CoV-2样病毒在第3种宿主中重组,再传给人类。这时与SARS-CoV-2极为相似的病毒应该在第3种宿主中找到。因此,寻觅SARS-CoV-2动物宿主的工作仍待继续。
比较SARS-CoV、MERS-CoV和SARS-CoV-2基因组核苷酸的突变率,SARS-CoV最高,达到每个细胞每个碱基0.80–2.38×10–3个核苷酸改变,与人免疫缺损病毒(每个细胞每个碱基4.1× 10–3±1.7×10–3个核苷酸改变)已比较接近[57]。但SARS-CoV-2的突变率与MERS-CoV相似,都比较低。直接一点作粗略比较,SARS-CoV不同分离株之间在近30 000个核苷酸中有两三百个不 同[57],而SARS-CoV-2不同分离株之间只有几个核苷酸的差异[58-59],相差一到两个数量级。应该说,SARS-CoV-2不同分离株之间核苷酸序列的差异非常小,变异不大,其特性应该很相似。要弄清这些微小的差异对表型的影响,必须通过实验和流行病学调查。仅凭生物信息学分析,不结合实验病毒学和流行病学,完全无法判定不同的SARS-CoV-2分离株在致病性和传染性方面有任何差异。
SARS-CoV-2的突变率较低,合理的推测是它可能与MERS-CoV一样有稳定的中间储存宿主,已经过一定时间的适应。相比之下,SARS-CoV进入果子狸和人的时间尚短,所以突变率仍然比较高[38]。无论如何,鉴于冠状病毒对各种动物的高感染率和广泛分布,其基因组的遗传多样性高和频繁重组,以及人与动物间接触活动的增加,各种新型冠状病毒可能因频繁的跨种传播和偶尔的动物传人事件而周期性地出现在人类面前。
4 COVID-19的诊断
在疫情初期,判断是否要进行SARS-CoV-2检测的依据是患者的临床症状及旅游与接触史。目前有关SARS-CoV-2特异性诊断的主要方法仍然是用荧光逆转录聚合酶链反应(RT-PCR)方法来检测病毒RNA。随着疫情的发展,各地对于确定核酸检测对象的标准出现不同的考虑。一些国家和地区继续采取严防死守策略,尽可能找出所有感染者特别是轻症和无症感染者。他们把核酸检测的对象扩大到有肺炎或呼吸道感染症状的病人以及所有与确诊病人有关的人士。另一些国家则根据资源、感染者数目及疾病轻重危急程度,采取抓住重症、放开轻症的策略,重点对需要救治和入院的病人进行检测。两者孰优孰劣还是殊途同归,将来可以根据防控效果及社会和经济成本进行综合评估。
SARS-CoV-2的核酸检测是在咽拭子、痰、下呼吸道分泌物、肺泡灌洗液、肛拭子、粪便、血液等标本中检测病毒RNA。核酸检测阳性是确诊受到SARS-CoV-2感染的主要标准。然而,这种方法的弊端就是假阴性率较高,在筛查中难免会有漏网之鱼。这种情况在当年SARS暴发时也碰到过[60],所以单凭核酸检测是不够的。特别是当病毒载量不高时,其检出率比较低,容易出现假阴性。更值得注意的是核酸检测只能说明受到病毒感染,不能对COVID-19病情的严重程度及其发展过程作出评判。如果患者没有痰,加上咽拭子采样不当便极有可能造成假阴性,这时还需要结合胸部影像学来作辅助诊断。
影像学诊断包括CT和胸部X射线检查,可看出肺部是否有炎症。在多数COVID-19患者中表现为双侧斑片状阴影或肺部磨玻璃影,并伴有肺炎及急性肺损伤。部分重症患者可发展为ARDS[35],引发炎症、体液积聚和进行性纤维化,严重影响呼吸。近期对COVID-19的首次尸检的病理学分析表明,死于该病的患者肺中的肺细胞受损,透明膜形成,并出现间质淋巴细胞浸润以及多核合胞细胞,属于典型的ARDS[61]。影像学分析能够有效区分轻症、中症和重症。然而,COVID-19作为病毒性肺炎的一种,其影像学表现与很多其他病毒性肺炎类似,所以病原体的鉴定还是离不开核酸检测。
另外,实验室检查也起到一定作用。比如患者在发病早期外周血白细胞数量正常或减少,以及天冬氨酸转氨酶水平升高;在某些患者的血液中肝酶、肌酶和肌红蛋白的水平增加,血液中红细胞沉降增加等[33]。这些都可以和COVID-19的临床特征一起提供一定的诊断依据。综上,疾病史、临床表现、核酸检查和影像学检查结果的结合对于COVID-19的有效诊断至关重要。特别是后两者,相辅相成,不可互相取代。目前有多个科研团队或公司在夜以继日地针对核酸检查灵敏度作优化和改进,或者开发新的试剂盒作抗体及抗原检测。比如,麻省理工学院张锋团队正在研发的一种基于CRISPR原理的SHERLOCK技术检测SARS-CoV-2的方法,可检测极微量的SARS-CoV-2 RNA片段。据称该技术需时短,操作简单,但仍在试验中,缓不济急。需要指出,中国香港所使用的国际市场上的核酸检测产品灵敏度及特异性都比较高,并未出现假阴性问题。目前利用胶体金法和酶联免疫吸附法检测IgM和IgG抗体的试剂已经研制成功,正待扩大生产规模。高质量抗体检测试剂的广泛使用,可补充核酸检测之不足,也可帮助判定恢复期病人、康复者和无症曾受感染者,使SARS-CoV-2感染和COVID-19的诊断水平再上一个台阶。
国内外在SARS-CoV-2感染和COVID-19的特异性诊断实践中碰到所谓“复阳”问题,也就是核酸检测阴性后在短期内再次检测阳性。有人提出病毒在这些病人中持续残留,或者出现间歇性病毒释放,均无明确依据。最大的可能还是核酸检测的假阴性。COVID-19病程多在两周内,与其他人类冠状病毒感染无异。但具体患者病程长短不一,与病毒清除的快慢、病人本身身体素质及接受的治疗方案都有一定关系。已知使用类固醇激素会使SARS和MERS的病程拖长,病毒释放量加大而时间也加长[62-63]。COVID-19病程拖长是否与激素治疗有关,值得查明。
国内外不少COVID-19病人在症状减退消失后很长时间内肛拭子核酸检测依然保持阳性。类似的情况过去在SARS-CoV感染中也有报道。中国香港和美国的不少SARS病人在痊愈后23–26 d粪便中仍有病毒,最长的达到69 d[64-65]。但并未发现他们出现症状或传染给他人。类似的情况在COVID-19中也完全可能发生。
5 COVID-19的防控
SARS-CoV-2的传播力强而症状不典型的特点,为病毒防控造成了很大困难。SARS大暴发的前车之鉴,让我们终于在认识到武汉疫情的严重性后,果断地实施“封城”以切断传染源。通过延长春节假期、延迟各大中小学校开学时间等措施,有效地避免了人员聚集。科普教育和媒体宣传在防疫过程中也发挥了积极作用,比如广泛地宣传如何正确使用口罩和正确洗手等等。国民对疫情危害和防控的公众意识和社会责任感成为遏制疫情上升的重要因素。同时全面隔离可疑病例和接触者,制定有效的治疗程序(目前以支持治疗为主),并加快对药物的筛选和疫苗的开发,是打赢当前这场无硝烟战争的关键。而从长远来看,加强市场监管,严格限制野生动物的驯化和食用,可有效防止重蹈覆辙。此外,将来痛定思痛,还要认真总结我国在此次疫情应对方面的得失、经验及教训,特别要加强和落实传染病防治法,改革与完善国家与地方疾病预防控制体系,同时理顺科研、医疗和疾病控制等方面的关系,为下一次应对重大传染病疫情作好充分准备。
5.1 COVID-19的治疗
临床上主要为COVID-19患者提供支持性治疗,同时尝试各种之前已使用或提议用于治疗其他冠状病毒(如SARS-CoV和MERS-CoV) 感染的方法。少数患者需要使用重症监护室、进行有创通气治疗等。目前所用的方法主要有吸氧、机械通气、静脉用抗生素和一些抗病毒药物(如奥司他韦)治疗、激素治疗、康复者血浆抗体治疗及中药治疗等。以下逐一对一些重要的治疗措施作出点评。
5.1.1 支持性治疗
虽然SARS-CoV-2也有机会攻击其他器官如胃肠道和肾脏,此病毒主要感染肺部,呼吸困难与衰竭是对患者的主要威胁和死亡原因[38]。因此呼吸支持对于缓解症状和挽救生命至关重要,对疾病的严重程度影响甚大。所谓的呼吸支持疗法包括常规氧气疗法、高流量氧气疗法、无创通气治疗和机械通气治疗。出现ARDS的患者必须尽早接受体外膜氧合器(ECMO) 治疗。ECMO是一种改良的模拟心肺支持的技术,用于治疗危及生命的心脏或呼吸衰竭,可以使肺处于休息状态。此外,对于重症SARS-CoV-2患者,维持电解质平衡、预防和治疗继发感染和败血性休克以及保护重要器官的功能也是必不可少的[38]。例如应密切观察发现心功能或肾功能衰竭等情况,积极作出对症治疗。
5.1.2 抗炎治疗
细胞因子风暴是系统性炎症反应的一种形式,其特征是释放了一系列细胞因子,包括肿瘤坏死因子α、白细胞介素1β、2和6、α干扰素、β干扰素、γ干扰素和MCP-1[66]。有研究报告指出,炎症反应及促炎因子的释放与组织损伤及各种症状密不可分[67]。因此抗炎在病毒病治疗中应有一定的意义。在过往SARS和MERS的治疗经验中,患者免疫系统反应过度便会引起细胞因子风暴[66]。针对细胞因子风暴的免疫抑制疗法包括遏制T细胞引导的免疫应答,阻断γ干扰素、白细胞介素1β和肿瘤坏死因子α的作用,抑制JAK或抑制STAT4等[68-69]。白细胞介素6显著升高的患者可使用针对白细胞介素6受体的人源化单克隆抗体Tocilizumab治疗。此外,通过血液净化治疗清除炎症因子也是可行的方案。前面提过,病毒与免疫是COVID-19中的一对主要矛盾,而治疗COVID-19的两大目标是抗病毒和抗炎。抗病毒是本,抗炎是标,标本要兼治。抗病毒是釜底抽薪,而抗炎则减轻症状,两者不可偏废。具有免疫抑制功能的抗炎药如类固醇可有效抑制炎症反应,对于减轻症状可能有立竿见影的效果。类固醇曾广泛用于SARS治疗,但中国香港等地在2003年后进行的回顾性调查表明使用类固醇弊大于利,不但疗效不彰,而且会显著减慢病毒的清除[62, 70-71]。采用类固醇治疗MERS,同样发现病毒清除显著减缓,而且死亡率高于采用干扰素加利巴韦林治疗的一组[63, 72]。类固醇对模式冠状病毒即小鼠肝炎病毒复制和感染的刺激作用,早在20世纪50年代和60年代就有很多研究[73-74]。它对冠状病毒复制的激活并不意外,因此使用类固醇治疗COVID-19存在重大危险,无异于饮鸩止渴。而且,高剂量的类固醇对SARS和COVID-19的严重肺损伤非但没有益处,还可能引起严重副作用,影响预后[71, 75]。第七版的COVID-19诊疗方案虽然对大剂量使用类固醇延缓病毒清除提出了警示,但仍建议对危重患者在短期内谨慎使用中低剂量类固醇。作为病毒学工作者,我们对此有很大保留。我们认为,类固醇激素在COVID-19病程中能够发挥有益作用的窗口期很短,而且不容易抓住。类固醇只能在机体免疫已完全控制并消除SARS-CoV-2病毒但细胞因子风暴及炎症反应仍处于高峰时,才可以起到抗炎而不是促病毒的效果。短暂注射中低剂量类固醇并同时处方抗病毒药物,也许不会激活潜伏感染而且一般病毒载量已极低的乙型肝炎病毒[76],但能否刺激进行性急性感染的SARS-CoV-2,仍然存在很大疑问。事实上,1969年国际上对小鼠肝炎病毒的研究已表明,类固醇可以刺激病毒载量提高50–1 000倍,而且即使很低剂量的类固醇(每日0.05 mg氢化可的松或0.1 mg甲基泼尼松龙) 也有显著的刺激冠状病毒复制的效果[74]。因此,在目前尚无对SARS-CoV-2的实验研究的情况下,应假定其可以激活而不是相反。类固醇激活SARS-CoV-2复制,其后果很严重。第一是加重病情,第二是延长病程,第三是提高病毒载量释放更多病毒,增加院内感染及对医护人员的威胁。鉴于武汉中南医院报道住院病人中45%都使用了激素[35],而某项中西医结合研究报告中更对三分之二的病人使用激素[77],临床上滥用激素的倾向实在堪忧。我们认为有必要尽快进行实验研究、动物试验和流行病学调查,厘清类固醇激素对SARS-CoV-2复制的影响,为下一版诊疗方案的修订提供可靠的依据。同时,凡使用类固醇都必须同时检测病毒核酸,密切注意其变化,一旦病毒RNA量增加应立刻停药。一定要用类固醇,必须同时加用抗病毒药物,在抗炎的同时保持抗病毒活性不减[76],从而避免刺激病毒复制,这也正是目前治疗COVID-19时需要找到的平衡。
5.1.3 康复者血浆抗体治疗
从经典免疫学角度来看,最有效的方法就是从传染性疾病中康复的患者中收集血液以治疗其他患有相同疾病的患者,或者保护健康个体免于感染该疾病[78]。通常情况下,康复的患者可在相当长时间内在他们的血液中含有相对较高水平的中和抗体,从而有效对抗病原体的再次入侵。抗体是由B淋巴细胞识别病原体或者其他抗原产生的免疫球蛋白,它们识别抗原表面蛋白并直接中和其活性。基于此,有研究从COVID-19中恢复的患者血液中收集血浆并将其注射到重症患者中,他们的症状在24 h内得到改善,同时炎症和病毒载量减少,血液中的氧饱和度提高。不过该方法也有利有弊,例如,抗体有可能会过度刺激免疫反应并引起细胞因子释放综合征,严重时可威胁生命。异体血清除有用的中和抗体外还有其他蛋白质致敏原甚至不明病原。重症患者的病毒载量很高,用此法治疗时对血浆的需求量巨大,很难足够快地开发和生产足够多而且高质量的特异性抗体以对抗全球流行病。输血浆抗体如同输血,采用此类被动免疫疗法需要谨慎行事,一般仅用于救急,只适用于重症病人。因此,从康复的患者中分离B细胞并鉴定编码有效抗体的基因或筛选针对SARS-CoV-2必需蛋白质的有效抗体,可能更为关键、实用并具备可持续性。
5.1.4 中药治疗
中医用于传染病的救治有一定的争议性和挑战性。目前由于缺乏有效且特异性的COVID-19治疗,中医已成为轻症中症或已从重症恢复的患者的主要替代治疗方法之一。有简单初步的研究声称舒风解毒胶囊和莲花清瘟胶囊对COVID-19有效,并有报道患者结合使用中医治疗后治愈率有所提高,但具体机制有待研究[77]。但该项中西医结合研究对三分之二的患者使用类固醇激素,其中约四分之三的患者仅属于普通型。这样的治疗方案存在极大隐忧。令人震惊的是,该项研究中两组病人分别有33.3%及5.9%由普通型转为重症[77],究竟与使用激素是否有关而在普通型COVID-19患者中使用激素的依据又是什么,有关医政药政部门应予调查,认真总结经验教训。COVID-19在轻症中症患者中基本上属于一种自限性疾病,可以完全自愈。只要无害,使用中医并无妨。但在抗炎中药中添加类固醇激素,中国香港、内地和新加坡过去都有发现[79-80]。在新加坡一项研究中曾对多种中药的类固醇成分作出过分析[79]。此外,中药材中也存在一些具有类固醇活性的天然产物[81]。因此,采用中药治疗COVID-19,仍应同时监测病毒载量,防止病毒复制受到不明来源的类固醇样物质的刺激。
5.1.5 抗病毒治疗
目前尚无确认有效并批准使用的抗SARS-CoV-2药物,但有几种药物被建议临床试用。在美国的COVID-19患者中试用核苷酸类似物瑞德西韦(Remdesivir,GS-5734) 静脉给药,似有一定效果[82]。瑞德西韦是一种新型抗病毒药,最初用于治疗由埃博拉和马尔堡病毒引起的疾病。作为RNA依赖的RNA聚合酶(RdRp) 竞争性抑制剂,其三磷酸核苷酸产物Remdesivir-TP可以与RdRp竞争底物ATP,因此可以干扰病毒RNA的合成[83],而RdRp则广泛分布于RNA病毒。最近发现该药也可抑制具有校正功能的NSP14外切核糖核酸酶[83]。在其他人类冠状病毒,NSP14缺失后因突变率暴升而导致毒力减弱甚至不能存活[84]。因此抑制NSP14也可达到抗SARS-CoV-2的效果。在体外和动物模型中,已有证据表明瑞德西韦对 SARS-CoV、MERS-CoV及社区获得性人类冠状病毒均有明显的抑制活 性[85-86]。该药在体外抑制SARS-CoV-2的活性及在武汉初步临床试用于COVID-19的治疗均取得令人鼓舞的结果[87]。需要指出,即使此药确实有效而其毒副作用也相对较轻,仍应注意观察它对肝肾功能的损伤。
除了瑞德西韦,磷酸氯喹(Chloroquine diphosphate)在体外研究中也展示出非常好的抗SARS-CoV-2活性[87]。磷酸氯喹在历史上因其抗疟活性而闻名,它也是研究自噬抑制作用时广泛使用的生物学研究工具,后来用于治疗类风湿性关节炎等。曾有研究表明,磷酸氯喹在酸性溶酶体中积累并增加了溶酶体的pH,抑制了溶酶体水解酶并阻止了自噬体融合和降解[88]。实验结果显示,在Vero E6细胞中,瑞德西韦对SARS-CoV-2的半数有效浓度EC50为0.77 μmol/L,选择指数 (Selective index,SI) 大于129;而磷酸氯喹的EC50为1.13 μmol/L,SI大于88,说明这两种药物在细胞水平上能有效抑制SARS-CoV-2的感染[87],但是依然需要进行更多随机对照的临床试验,以确定在研药物用于治疗COVID-19的安全性和有效性。对于磷酸氯喹的细胞毒性及毒副作用,也要十分注意。
此外,通过老药新用将具有广谱抗病毒活性的利巴韦林(Ribavirin)、洛匹那韦(Lopinavir)/利托那韦(Ritonavir)、α2a干扰素和β干扰素用于COVID-19的治疗,也是应有之义。中国香港采用以上两三种药物联用治疗COVID-19,初步结果也令人满意。利巴韦林主要通过抑制mRNA加帽反应从而抑制病毒mRNA的转录。洛匹那韦/利托那韦是蛋白酶抑制剂,其中利托那韦主要通过抑制细胞色素P450对洛匹那韦的代谢而增强后者的作用[89]。重组α及β干扰素也是广泛应用的抗病毒药物。这些抗病毒药物虽然有效,仍应注意使用的时机和毒副作用。在病毒感染的早期使用干扰素,其抗病毒活性明显。事实上,用于分离SARS-CoV-2的Vero-E6细胞不能产生干扰素[20],反证干扰素在抑制病毒复制和感染方面的作用。但如果在病毒感染的后期使用干扰素,也有可能反而加重细胞因子风暴和炎症[90-91]。除了上面所述例子,还有更多的新药等待开发。针对新药开发的思路,我们在下文再作详细探讨。
临床上奥司他韦和抗生素也广泛试用于COVID-19的治疗。奥司他韦抑制流感病毒复制是通过抑制神经氨酸酶对唾液酸的切割反应而起作用[92]。SARS-CoV-2没有神经氨酸酶也不需要切割唾液酸,因此使用奥司他韦治疗COVID-19并无任何依据,奥司他韦也注定难有确切的抗SARS-CoV-2活性。除非合并感染流感病毒,我们建议慎用奥司他韦治疗COVID-19。同理,除非继发感染细菌,以抗生素治疗COVID-19显属滥用,应当尽量避免。
5.2 冠状病毒的新药筛选
新药研发是一个非常漫长的过程,对突如其来的疫情,按照新药研发的传统规律来开展常常缓不济急,特别是后期的临床试验和评估,耗时很长。最快和最直接的方法就是“老药新用”——充分利用现有的已经具备的研究基础,在已经上市和已经开展临床试验的药物当中进行系统化、大规模的筛选,从已知安全性的药物中去挖掘可以抗新型冠状病毒的药物。针对减少免疫损伤,增加免疫反应和减少ARDS的抗COVID-19新药是药物研究的重要方向。药物筛选之后有待临床进一步验证,即必须在确保安全性的前提下确定药物对COVID-19的有效性。除了瑞德西韦和磷酸氯喹,对于前述几种试用于COVID-19治疗的老药,它们是否确实有效,仍需大量试验及临床验证。
除了“老药新用”,我们还可以在详细了解病毒对宿主的致病机理的基础上,研制针对宿主因子的抗病毒新药。对于针对宿主的抗病毒药物的研发,有以下几个屡试不爽的思路值得借鉴。第一,可高通量地利用小分子化合物库筛选可以抑制病毒活性的药物,并结合体外和动物试验对筛选出来的化合物进行评估。这些小分子化合物,可以作用于病毒生命史中的任意一环,如复制或释放等。第二,Ⅰ型与Ⅲ型干扰素具有抗病毒活性,可用于抗冠状病毒治疗[5]。比如有研究发现Ⅲ型干扰素可以更快更强地发挥抗流感病毒功能同时又避免刺激炎症反应[93]。因此,Ⅲ型干扰素在治疗冠状病毒感染中是否也有类似优点,值得查明。例如冠状病毒的某个蛋白对Ⅰ型干扰素产生及信号转导有抑制作用,因此抑制该蛋白后IFN-β可以更好地发挥作用。如此看来,利用该冠状病毒蛋白的抑制剂与IFN-β联用以取得协同抗病毒效应也就不难理解。这些都需要用大量实验去验证。第三,考虑到SARS-CoV与SARS-CoV-2之间的相关性[6],一些已经研究出来的针对SARS-CoV的药物也可能适用于SARS-CoV-2,包括一些针对SARS-CoV生命史而设计的siRNA分子或者抑制剂[94]。比如SARS-CoV与SARS-CoV-2都是以ACE2为受体,抑制ACE2的表达是否有抗病毒活性,值得验证。前述蛋白酶抑制剂最初用于艾滋病,其他一些用于艾滋病的药物对于抗SARS-CoV-2药物的研发也有启发意义。例如抑制人免疫缺损病毒的细胞受体CD4的人源化单克隆抗体、抑制病毒包膜与细胞膜融合的肽类小分子和可结合并抑制CCR5辅助受体的小分子化合物都获得成功,并成功应用于临床[17-18]。同样的设计思路可能也适用于SARS-CoV-2。
5.3 SARS-CoV-2的疫苗研发
SARS-CoV-2疫苗在以下多种情况下都可能有必要广泛接种或在特殊的高风险人群中接种。第一,SARS-CoV-2的地区性流行和全世界大流行在2020年未能终结,COVID-19继续对人类健康构成重大威胁。第二,SARS-CoV-2成为新的一种地区获得性的人类冠状病毒,其在人际传播的传染性持续保持在较高水平,但致病性并未显著减弱或减弱至足够低的水平。第三,SARS-CoV-2病毒对瑞德西韦等有效药物出现抗药性。无论如何,针对SARS-CoV-2的疫苗研发目前是全球生物医药界的一大研究热点。早前开发SARS-CoV疫苗的基本思路也可以应用于SARS-CoV-2[95-96],常见的疫苗设计方案包括灭活病毒疫苗(Inactivated vaccines)、减毒活病毒疫苗(Live attenuated vaccines)、S蛋白疫苗(S protein-based vaccines)、载体疫苗(Vectored vaccines)、DNA疫苗(DNA vaccines) 和组合疫苗(Combination vaccines) 等[97]。
SARS-CoV灭活疫苗是最简单直接的方案,其有效性已经得到动物实验的支持,也经过初步的人体试验[98]。同样的方案可用于SARS-CoV-2,我们不在乎其简单或复杂,有效就好。然而其主要顾虑在于安全性。其弊端在于需要大量培养病毒,存在没有完全灭活病毒的风险,而且某些病毒蛋白可能在特定人群中引发严重的炎症反应。有单篇报道认为SARS-CoV灭活疫苗接种后出现依赖于抗体的感染增强现象(Antibody-dependent enhancement,ADE),也就是疫苗除诱导中和抗体外,还可诱导不具中和活性但可增强病毒感染的抗体[99]。实际情况如何或者ADE如何发生又有多大影响,值得关注更需要深入探究。理论上ADE也会出现于其他疫苗。
减毒活病毒疫苗最简单的办法是利用基因工程原理对SARS-CoV-2基因组进行人工改造,通过各种方法使病毒的毒力大大减弱但仍保持抗原性和一定的复制水平[100]。例如前述的灭活NSP14的办法,或者将激活炎症反应的ORF3a和E基因删除的方案,都在SARS-CoV中取得一定成功[101]。减毒活疫苗所诱导的人体免疫反应最全面也最接近自然感染,特别是可覆盖黏膜免疫。天花病毒、脊髓灰质炎病毒及麻疹病毒的减毒活疫苗已在大量人群中成功使用。但研发SARS-CoV-2减毒活病毒,仍有一些问题要注意。减毒活病毒可以从接种者的呼吸道和消化道排出,感染未接种者,扩大免疫效果。但如何防止减毒活病毒与野生型SARS-CoV-2发生重组,是一大挑战。通过改造SARS-CoV-2亚基因组上的转录调节区可大大减少重组的发生[102],是可以考虑的方案。如果确有ADE,仍可以通过找出并去除SARS-CoV-2病 毒蛋白中可诱导ADE抗体的免疫表位的办法来解决。
SARS-CoV和SARS-CoV-2的S蛋白含有最主要的中和表位,S蛋白疫苗就是利用针对S蛋白的中和抗体收到中和病毒的效果,阻止病毒进入细胞。尽管S蛋白疫苗的安全性较好,ADE仍是关注的焦点。在动物试验中曾经观察到接种S蛋白疫苗后再感染SARS-CoV,仍会诱发ADE,可加剧感染,也会加剧肝脏损伤[103]。
载体疫苗也是开发SARS-CoV-2的思路之一。例如把SARS-CoV-2的结构蛋白(S、N、M、E等一个或者多个) 放到一个减毒的流感病毒疫苗载体中。同理也可以利用腺病毒或腺伴随病毒(AAV)载体表达S蛋白,研制成载体疫苗[104]。类似的方案还包括将S蛋白的优势免疫表位例如其胞外域植入麻疹减毒活疫苗或者已通过安全性试验的复制缺陷型MVA痘苗病毒载体[105, 106]。
另外还有DNA疫苗、mRNA疫苗和组合疫苗等几种新型疫苗。DNA疫苗顾名思义就是将有SARS-CoV-2的编码基因(S、N、M、E等一个或者多个) 的表达质粒DNA直接注射到肌肉或皮下,表达后积聚的抗原通过抗原提呈细胞的交叉提呈得以激发免疫反应。DNA疫苗容易制备,产品稳定性安全性俱佳,也能同时激发细胞免疫和体液免疫。但其免疫原性低,目前并无成功的DNA疫苗应用于人体。有关SARS-CoV和MERS-CoV的DNA疫苗曾经进行人体试验,初步结果也支持其有效性[107-108]。mRNA疫苗是直接注射表达SARS-CoV-2抗原的mRNA,优点是不可能整合到细胞基因组[109]。与RNA药物一样,mRNA疫苗可以采用脂质纳米颗粒来提高递送效果。组合疫苗是通过先注射DNA疫苗或mRNA疫苗,再用灭活SARS-CoV加强免疫的方法,其刺激抗体产生的效果大为增强,优于单一的DNA疫苗或mRNA疫苗。
此外,在最近的疫情中,发现感染SARS-CoV-2的病人体内的抗体显示出在低血清稀释度下中和病毒的潜力,而且抗SARS-CoV的特异性抗体能与SARS-CoV-2交叉反应[24],推测保护性细胞免疫也可能有交叉。经过近20年对冠状病毒疫苗的研究,发现S蛋白的中和抗体最有效,但仍需要优化并排除有害的免疫反应。SARS-CoV-2疫苗的研发可以使我们在此病毒成为社区获得性人类冠状病毒或再次暴发时及时控制其蔓延。
6 结语与展望
目前,SARS-CoV-2疫情在全球仍然呈现扩大的趋势,已导致多个国家出现活跃传播。SARS-CoV-2的感染数和死亡数已经超越了2003年的SARS-CoV,令经济损失惨重。造成这种局面其部分归因于经济全球化下,交通四通八达令人流物流增加,跨国旅游、工作或学习的人员越来越多。2003年SARS疫情后,我国在预防医学领域科研能力有了大幅提升,此次疫情暴发不到两个月内,在病因确认、诊断试剂开发、病毒特性分析和临床救治策略等方面均较快取得一些进展。同时我们也应清醒地认识到,我国传染病防治体系仍存在多方面的短板和隐患,我们需要正视并采取切实措施解决多年积累的一些深层次问题,使我国疾病控制和卫生健康事业更上一层楼。
疫情来势汹涌,积极开发相应的药物以及疫苗刻不容缓。此外,有关SARS-CoV-2的基础研究,特别是其无症状传播的机理,以及体内的免疫逃逸机制,都有待进一步揭示。在抗击疫情的当下,对SARS-CoV-2的溯源并不是当务之急。然而从长远看仍然存在冠状病毒或其变种卷土重来的风险。找出SARS-CoV-2中间动物宿主并加强防控,才能更好地将这一病毒对人类健康的威胁降到最低。对于病毒的科普教育和宣传也应当继续,这样才能在疫情来时,做到冷静理智应 对——不恐慌、不轻视。鉴于现在已知有多种冠状病毒都有极强的传播性和致病性,我们需要未雨绸缪,更多地开发广谱的抗病毒药物和疫苗,以应对此类冠状病毒和未来更多新发病毒的挑战。
[1] Huang CL, Wang YM, Li XW, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet, 2020, 395(10223): 497–506.
[2] Woo PCY, Lau SKP, Lam CSF, et al. Discovery of seven novel mammalian and avian coronaviruses in the genussupports bat coronaviruses as the gene source ofandand avian coronaviruses as the gene source ofand. J Virol, 2012, 86(7): 3995–4008.
[3] Chan JFW, To KKW, Tse H, et al. Interspecies transmission and emergence of novel viruses: lessons from bats and birds. Trends Microbiol, 2013, 21(10): 544–555.
[4] Su S, Wong G, Shi WF, et al. Epidemiology, genetic recombination, and pathogenesis of coronaviruses. Trends Microbiol, 2016, 24(6): 490–502.
[5] Wong LYR, Lui PY, Jin DY. A molecular arms race between host innate antiviral response and emerging human coronaviruses. Virol Sin, 2016, 31(1): 12–23.
[6] Chan JFW, Kok KH, Zhu Z, et al. Genomic characterization of the 2019 novel human-pathogenic coronavirus isolated from a patient with atypical pneumonia after visiting Wuhan. Emerg Microbes Infect, 2020, 9(1): 221–236.
[7] Weiss SR, Navas-Martin S. Coronavirus pathogenesis and the emerging pathogen severe acute respiratory syndrome coronavirus. Microbiol Mol Biol Rev, 2005, 69(4): 635–664.
[8] Zhu N, Zhang DY, Wang WL, et al. A novel coronavirus from patients with pneumonia in China, 2019. N Engl J Med, 2020, 382(8): 727–733.
[9] Siu KL, Yuen KS, Castano-Rodriguez C, et al. Severe acute respiratory syndrome coronavirus ORF3a protein activates the NLRP3 inflammasome by promoting TRAF3-dependent ubiquitination of ASC. FASEB J, 2019, 33(8): 8865–8877.
[10] Lai MMC, Cavanagh D. The molecular biology of coronavirusesAdv Virus Res, 1997, 48: 1–100.
[11] Snijder EJ, van der Meer Y, Zevenhoven-Dobbe J, et al. Ultrastructure and origin of membrane vesicles associated with the severe acute respiratory syndrome coronavirus replication complex. J Virol, 2006, 80(12): 5927–5940.
[12] Fehr AR, Perlman S. Coronaviruses: an overview of their replication and pathogenesis//Maier H, Bickerton E, Britton P, eds. Coronaviruses. New York, NY: Humana Press, 2015, 1282: 1–23.
[13] Beniac DR, Andonov A, Grudeski E, et al. Architecture of the SARS coronavirus prefusion spike. Nat Struct Mol Biol, 2006, 13(8): 751–752.
[14] Nal B, Chan C, Kien F, et al. Differential maturation and subcellular localization of severe acute respiratory syndrome coronavirus surface proteins S, M and E. J Gen Virol, 2005, 86(5): 1423–1234.
[15] Nieto-Torres JL, DeDiego ML, Verdiá-Báguena C, et al. Severe acute respiratory syndrome coronavirus envelope protein ion channel activity promotes virus fitness and pathogenesis. PLoS Pathog, 2014, 10(5): e1004077.
[16] Cui L, Wang HY, Ji YX, et al. The nucleocapsid protein of coronaviruses acts as a viral suppressor of RNA silencing in mammalian cells. J Virol, 2015, 89(17): 9029–9043.
[17] Schenone M, Dančík V, Wagner BK, et al. Target identification and mechanism of action in chemical biology and drug discovery. Nat Chem Biol, 2013, 9(4): 232–240.
[18] Sawyer N, Watkins AM, Arora PS. Protein domain mimics as modulators of protein-protein interactions. Acc Chem Res, 2017, 50(6): 1313–1322.
[19] Li WH, Moore MJ, Vasilieva N, et al. Angiotensin-converting enzyme 2 is a functional receptor for the SARS coronavirus. Nature, 2003, 426(6965): 450–454.
[20] Zhou P, Yang XL, Wang XG, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature, 2020, 579(7798): 270–273, doi: 10.1038/s41586-020-2012-7.
[21] Wrapp D, Wang NS, Corbett KS, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science, 2020, 367(6483): 1260–1263, doi: 10.1126/science.abb2507.
[22] Hamming I, Timens W, Bulthuis MLC, et al. Tissue distribution of ACE2 protein, the functional receptor for SARS coronavirus. A first step in understanding SARS pathogenesis. J Pathol, 2004, 203(2): 631–637.
[23] Tian XL, Li C, Huang AL, et al. Potent binding of 2019 novel coronavirus spike protein by a SARS coronavirus-specific human monoclonal antibody. Emerg Microbes Infect, 2020, 9(1): 382–385.
[24] Hoffmann M, Kleine-Weber H, Schroeder S, et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor. Cell, 2020, doi: 10.1016/j.cell.2020.02.052.
[25] Walls AC, Tortorici MA, Snijder J, et al. Tectonic conformational changes of a coronavirus spike glycoprotein promote membrane fusion. Proc Natl Acad Sci USA, 2017, 114(42): 11157–11162.
[26] Vigant F, Santos NC, Lee B. Broad-spectrum antivirals against viral fusion. Nat Rev Microbiol, 2015, 13(7): 426–437.
[27] Garg H, Viard M, Jacobs A, et al. Targeting HIV-1 gp41-induced fusion and pathogenesis for anti-viral therapy. Curr Top Med Chem, 2011, 11(24): 2947–2958.
[28] Xu GG, Guo J, Wu YT. Chemokine receptor CCR5 antagonist maraviroc: medicinal chemistry and clinical applications. Curr Top Med Chem, 2014, 14(13): 1504–1514.
[29] Duan SM, Zhao XS, Wen RF, et al. Stability of SARS coronavirus in human specimens and environment and its sensitivity to heating and UV irradiation. Biomed Environ Sci, 2003, 16(3): 246–255.
[30] Geller C, Varbanov M, Duval RE. Human coronaviruses: insights into environmental resistance and its influence on the development of new antiseptic strategies. Viruses, 2012, 4(11): 3044–3068.
[31] Kampf G, Todt D, Pfaender S, et al. Persistence of coronaviruses on inanimate surfaces and their inactivation with biocidal agents. J Hosp Infect, 2020, 104(3): 246–251.
[32] Jiang S, Shi ZL. The first disease X is caused by a highly transmissible acute respiratory syndrome coronavirus. Virol Sin, 2020, doi: 10.1007/s12250-020-00206-5.
[33] Chen NS, Zhou M, Dong X, et al. Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study. Lancet, 2020, 395(10223): 507–513.
[34] Lu RJ, Zhao X, Li J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet, 2020, 395(10224): 565–574.
[35] Wang DW, Hu B, Hu C, et al. Clinical characteristics of 138 hospitalized patients with 2019 novel coronavirus-infected pneumonia in Wuhan, China. JAMA, 2020, doi: 10.1001/jama.2020.1585.
[36] Epidemiology Working Group for NCIP Epidemic Response. The epidemiological characteristics of an outbreak of 2019 novel coronavirus diseases (COVID-19) in China. Chin J Epidemiol, 2020, 41(2): 145–151 (in Chinese). 中国疾病预防控制中心新型冠状病毒肺炎应急响应机制流行病学组. 新型冠状病毒肺炎流行病学特征分析. 中华流行病学杂志, 2020, 41(2): 145–151.
[37] Guan WJ, Ni ZY, Hu Y, et al. Clinical characteristics of coronavirus disease 2019 in China. N Engl J Med, 2020, doi: 10.1056/NEJMoa2002032.
[38] Cheng VCC, Lau SKP, Woo PCY, et al. Severe acute respiratory syndrome coronavirus as an agent of emerging and reemerging infection. Clin Microbiol Rev, 2007, 20(4): 660–694.
[39] Bolles M, Donaldson E, Baric R. SARS-CoV and emergent coronaviruses: viral determinants of interspecies transmission. Curr Opin Virol, 2011, 1(6): 624–634.
[40] Dudas G, Carvalho LM, Rambaut A, et al. MERS-CoV spillover at the camel-human interface. Elife, 2018, 7: e31257.
[41] Chan JFW, Yuan SF, Kok KH, et al. A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster. Lancet, 2020, 395(10223): 514–523.
[42] Bai Y, Yao LS, Wei T, et al. Presumed asymptomatic carrier transmission of COVID-19. JAMA, 2020, doi: 10.1001/jama.2020.2565.
[43] Delamater PL, Street EJ, Leslie TF, et al. Complexity of the basic reproduction number (R0). Emerg Infect Dis, 2019, 25(1): 1–4.
[44] Zhao S, Lin Q, Ran JJ, et al. Preliminary estimation of the basic reproduction number of novel coronavirus (2019-nCoV) in China, from 2019 to 2020: A data-driven analysis in the early phase of the outbreak. Int J Infect Dis, 2020, 92: 214–217.
[45] Tuite AR, Fisman DN. Reporting, epidemic growth, and reproduction numbers for the 2019 novel coronavirus (2019-nCoV) epidemic. Ann Intern Med, 2020, doi: 10.7326/M20-0358.
[46] Li Q, Guan XH, Wu P, et al. Early transmission dynamics in Wuhan, China, of novel coronavirus-infected pneumonia. N Engl J Med, 2020, doi: 10.1056/NEJMoa2001316.
[47] Rahman A, Sarkar A. Risk factors for fatal Middle East respiratory syndrome coronavirus infections in Saudi Arabia: Analysis of the WHO line list, 2013–2018. Am J Public Health, 2019, 109(9): 1288–1293.
[48] Hu B, Ge XY, Wang LF, et al. Bat origin of human coronaviruses. Virol J, 2015, 12: 221.
[49] Donaldson EF, Haskew AN, Gates JE, et al. Metagenomic analysis of the viromes of three North American bat species: viral diversity among different bat species that share a common habitat. J Virol, 2010, 84(24): 13004–13018.
[50] Huynh J, Li S, Yount B, et al. Evidence supporting a zoonotic origin of human coronavirus strain NL63. J Virol, 2012, 86(23): 12816–12825.
[51] Pfefferle S, Oppong S, Drexler JF, et al. Distant relatives of severe acute respiratory syndrome coronavirus and close relatives of human coronavirus 229E in bats, Ghana. Emerg Infect Dis, 2009, 15(9): 1377–1384.
[52] Vijgen L, Keyaerts E, Moës E, et al. Complete genomic sequence of human coronavirus OC43: molecular clock analysis suggests a relatively recent zoonotic coronavirus transmission event. J Virol, 2005, 79(3): 1595–1604.
[53] Guan Y, Zheng BJ, He YQ, et al. Isolation and characterization of viruses related to the SARS coronavirus from animals in southern China. Science, 2003, 302(5643): 276–278.
[54] Cotten M, Lam TT, Watson SJ, et al. Full-genome deep sequencing and phylogenetic analysis of novel human betacoronavirus. Emerg Infect Dis, 2013, 19(5): 736–742.
[55] Annan A, Baldwin HJ, Corman VM, et al. Human betacoronavirus 2c EMC/2012-related viruses in bats, Ghana and Europe. Emerg Infect Dis, 2013, 19(3): 456–459.
[56] Lam TTY, Shum MHH, Zhu HC, et al. Identification of 2019-nCoV related coronaviruses in Malayan pangolins in southern China. BioRxiv, 2020, doi: 10.1101/2020.02.13.945485.
[57] Vega VB, Ruan YJ, Liu JJ, et al. Identifying SARS-CoV-2-related coronaviruses in Malayan pangolins. Nature, 2020, doi: 10.1038/s41586-020-2169-0.
[58] Ceraolo C, Giorgi FM. Genomic variance of the 2019-nCoV coronavirus. J Med Virol, 2020, 92(5): 522-528, doi: 10.1002/jmv.25700.
[59] Benvenuto D, Giovanetti M, Salemi M, et al. The global spread of 2019-nCoV: a molecular evolutionary analysis. Pathog Glob Health, 2020: 1–4, doi:10.1080/20477724.2020.1725339.
[60] Chang SC. Clinical findings, treatment and prognosis in patients with severe acute respiratory syndrome (SARS). J Chin Med Assoc, 2005, 68(3): 106–107.
[61] Xu Z, Shi L, Wang YJ, et al. Pathological findings of COVID-19 associated with acute respiratory distress syndrome. Lancet Respir Med, 2020, doi: 10.1016/S2213-2600(20)30076-X.
[62] Auyeung TW, Lee JSW, Lai WK, et al. The use of corticosteroid as treatment in SARS was associated with adverse outcomes: a retrospective cohort study. J Infect, 2005, 51(2): 98–102.
[63] Arabi YM, Mandourah Y, Al-Hameed F, et al. Corticosteroid therapy for critically ill patients with Middle East respiratory syndrome. Am J Respir Crit Care Med, 2018, 197(6): 757–767.
[64] Cheng PKC, Wong DA, Tong LKL, et al. Viral shedding patterns of coronavirus in patients with probable severe acute respiratory syndrome. Lancet, 2004, 363(9422): 1699–1700.
[65] Isakbaeva ET, Khetsuriani N, Beard RS, et al. SARS-associated coronavirus transmission, United States. Emerg Infect Dis, 2004, 10(2): 225–231.
[66] Channappanavar R, Perlman S. Pathogenic human coronavirus infections: causes and consequences of cytokine storm and immunopathology. Semin Immunopathol, 2017, 39(5): 529–539.
[67] Tisoncik JR, Korth MJ, Simmons CP, et al. Into the eye of the cytokine storm. Microbiol Mol Biol Rev, 2012, 76(1): 16–32.
[68] Behrens EM, Koretzky GA. Review: Cytokine storm syndrome: Looking toward the precision medicine era. Arthritis Rheumatol, 2017, 69(6): 1135–1143.
[69] Kedzierski L, Linossi EM, Kolesnik TB, et al. Suppressor of cytokine signaling 4 (SOCS4) protects against severe cytokine storm and enhances viral clearance during influenza infection. PLoS Pathog, 2014, 10(5): e1004134.
[70] Booth CM, Matukas LM, Tomlinson GA, et al. Clinical features and short-term outcomes of 144 patients with SARS in the greater Toronto area. JAMA, 2003, 289: 2801–2809.
[71] Lee N, Allen Chan KC, Hui DS, et al. Effects of early corticosteroid treatment on plasma SARS-associated coronavirus RNA concentrations in adult patients. J Clin Virol, 2004, 31: 304–309.
[72] Alfaraj SH, Al-Tawfiq JA, Assiri AY, et al. Clinical predictors of mortality of Middle East Respiratory Syndrome Coronavirus (MERS-CoV) infection: A cohort study. Travel Med Infect Dis, 2019, 29: 48–50.
[73] Gallily R, Warwick A, Bang FB. Effect of cortisone of genetic resistance to mouse hepatitis virusand. Proc Natl Acad Sci USA, 1964, 51: 1158–1164.
[74] Datta DV, Isselbacher KJ. Effects of corticosteroids on mouse hepatitis virus infection. Gut, 1969, 10(7): 522–529.
[75] Russell CD, Millar JE, Baillie JK. Clinical evidence does not support corticosteroid treatment for 2019-nCoV lung injury. Lancet, 2020, 395(10223): 473–475.
[76] Shouval D, Shibolet O. Immunosuppression and HBV reactivation. Semin Liver Dis, 2013, 33(2): 167–177.
[77] Xia WG, An CQ, Zheng CJ, et al. Clinical observation on 34 patients with novel coronavirus pneumonia (COVID-19) treated with intergrated traditional Chinese and western medicine. J Tradit Chin Med, 2020, 61(5): 375–382 (in Chinese).夏文广, 安长青, 郑婵娟, 等. 中西医结合治疗新型冠状病毒肺炎34例临床研究. 中医杂志, 2020, 61(5): 375–382.
[78] Ko JH, Seok H, Cho SY, et al. Challenges of convalescent plasma infusion therapy in Middle East respiratory coronavirus infection: a single centre experience. Antivir Ther, 2018, 23(7): 617–622.
[79] Fung FY, Linn YC. Steroids in traditional Chinese medicine: what is the evidence? Singapore Med J, 2017, 58(3): 115–120.
[80] Hon KLE, Lee VW, Leung TF. Stop tarnishing steroid and Chinese medicine. World J Pediatr, 2016, 12(2): 133–134.
[81] Navarro VJ, Khan I, Björnsson E, et al. Liver injury from herbal and dietary supplements. Hepatology, 2017, 65(1): 363–373.
[82] Holshue ML, DeBolt C, Lindquist S, et al. First case of 2019 novel coronavirus in the United States. N Engl J Med, 2020, 382(10): 929–936.
[83] Agostini ML, Andres EL, Sims AC, et al. Coronavirus susceptibility to the antiviral Remdesivir (GS-5734) is mediated by the viral polymerase and the proofreading exoribonuclease. mBio, 2018, 9(2): e00221–18.
[84] Graepel KW, Agostini ML, Lu XT, et al. Fitness barriers limit reversion of a proofreading-deficient coronavirus. J Virol, 2019, 93(20): e00711–19.
[85] Sheahan TP, Sims AC, Leist SR, et al. Comparative therapeutic efficacy of remdesivir and combination lopinavir, ritonavir, and interferon beta against MERS-CoV. Nat Commun, 2020, 11: 222.
[86] Sheahan TP, Sims AC, Graham RL, et al. Broad-spectrum antiviral GS-5734 inhibits both epidemic and zoonotic coronaviruses. Sci Transl Med, 2017, 9(396): eaal3653.
[87] Wang ML, Cao RY, Zhang LK, et al. Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV). Cell Res, 2020, 30(3): 269–271.
[88] Kimura T, Takabatake Y, Takahashi A, et al. Chloroquine in cancer therapy: a double-edged sword of autophagy. Cancer Res, 2013, 73(1): 3–7.
[89] Cvetkovic RS, Goa KL. Lopinavir/ritonavir: a review of its use in the management of HIV infection. Drugs, 2003, 63(8): 769–802.
[90] Cheng L, Ma JP, Li JY, et al. Blocking type I interferon signaling enhances T cell recovery and reduces HIV-1 reservoirs. J Clin Invest, 2017, 127(1): 269–279.
[91] Fung SY, Yuen KS, Ye ZW, et al. A tug-of-war between severe acute respiratory syndrome coronavirus 2 and host antiviral defence: lessons from other pathogenic viruses. Emerg Microbes Infect, 2020, 9(1): 558–570, doi: 10.1080/22221751.2020.1736644.
[92] Beard KR, Brendish NJ, Clark TW. Treatment of influenza with neuraminidase inhibitors. Curr Opin Infect Dis, 2018, 31(6): 514–519.
[93] Galani IE, Triantafyllia V, Eleminiadou EE, et al. Interferon-λ mediates non-redundant front-line antiviral protection against influenza virus infection without compromising host fitness. Immunity, 2017, 46(5): 875–890.e6.
[94] Du LY, He YX, Zhou YS, et al. The spike protein of SARS-CoV-a target for vaccine and therapeutic development. Nat Rev Microbiol, 2009, 7(3): 226–236.
[95] Jiang S, He YX, Liu SW. SARS vaccine development. Emerg Infect Dis, 2005, 11(7): 1016–1020.
[96] Enjuanes L, Zuñiga S, Castaño-Rodriguez C, et al. Molecular basis of coronavirus virulence and vaccine development. Adv Virus Res, 2016, 96: 245–286.
[97] Afrough B, Dowall S, Hewson R. Emerging viruses and current strategies for vaccine intervention. Clin Exp Immunol, 2019, 196(2): 157–166.
[98] Lin JT, Zhang JS, Su N, et al. Safety and immunogenicity from a phase I trial of inactivated severe acute respiratory syndrome coronavirus vaccine. Antivir Ther, 2007, 12(7): 1107–1113.
[99] Luo F, Liao FL, Wang H, et al. Evaluation of antibody-dependent enhancement of SARS-CoV infection in Rhesus macaques immunized with an inactivated SARS-CoV vaccine. Virol Sin, 2018, 33(2): 201–204.
[100] Minor PD. Live attenuated vaccines: Historical successes and current challenges. Virology, 2015, 479–480: 379–392.
[101] Regla-Nava JA, Nieto-Torres JL, Jimenez-Guardeño JM, et al. Severe acute respiratory syndrome coronaviruses with mutations in the E protein are attenuated and promising vaccine candidates. J Virol, 2015, 89(7): 3870–3887.
[102] Yount B, Roberts RS, Lindesmith L, et al. Rewiring the severe acute respiratory syndrome coronavirus (SARS-CoV) transcription circuit: engineering a recombination-resistant genome. Proc Natl Acad Sci USA, 2006, 103(33): 12546–12551.
[103] Wang SF, Tseng SP, Yen CH, et al. Antibody-dependent SARS coronavirus infection is mediated by antibodies against spike proteins. Biochem Biophys Res Commun, 2014, 451(2): 208–214.
[104] Du LY, Zhao GY, Lin YP, et al. Priming with rAAV encoding RBD of SARS-CoV S protein and boosting with RBD-specific peptides for T cell epitopes elevated humoral and cellular immune responses against SARS-CoV infection. Vaccine, 2008, 26(13): 1644–1651.
[105] Escriou N, Callendret B, Lorin V, et al. Protection from SARS coronavirus conferred by live measles vaccine expressing the spike glycoprotein. Virology, 2014, 452–453: 32–41.
[106] Veit S, Jany S, Fux R, et al. CD8+T cells responding to the Middle East respiratory syndrome coronavirus nucleocapsid protein delivered by vaccinia virus MVA in mice. Viruses, 2018, 10(12): 718.
[107] Martin JE, Louder MK, Holman LA, et al. A SARS DNA vaccine induces neutralizing antibody and cellular immune responses in healthy adults in a Phase I clinical trial. Vaccine, 2008, 26(50): 6338–6343.
[108] Modjarrad K, Roberts CC, Mills KT, et al. Safety and immunogenicity of an anti-Middle East respiratory syndrome coronavirus DNA vaccine: a phase 1, open-label, single-arm, dose-escalation trial. Lancet Infect Dis, 2019, 19(9): 1013–1022.
[109] Pardi N, Hogan MJ, Porter FW, et al. mRNA vaccines - a new era in vaccinology. Nat Rev Drug Discov, 2018, 17(4): 261–279.
Diagnosis, treatment, control and prevention of SARS-CoV-2 and coronavirus disease 2019: back to the future
Zi-Wei Ye, and Dong-Yan Jin
Department of Microbiology and School of Biomedical Sciences, Li Ka Shing Faculty of Medicine, the University of Hong Kong, Hong Kong, China
The ongoing outbreak of the coronavirus disease 2019 (COVID-19) as named by the World Health Organization has millions of confirmed cases around the world and has claimed hundreds of thousands of lives. The virus was named SARS-CoV-2 in February by International Committee on Taxonomy of Viruses. COVID-19 presents as fever, dry cough, dyspnea, headache and pneumonia. In a small subset of severe cases, the disease quickly progresses to respiratory failure and even death. Since the 21st century, there have been three major outbreaks caused by human coronaviruses, including the severe acute respiratory syndrome (SARS) that broke out in 2003, the Middle East respiratory syndrome (MERS) in 2012, and the recent pandemic of COVID-19. Since 2003, significant progress has been made in the study of SARS-CoV and MERS-CoV concerning their natural origins, pathogenesis, antiviral development and vaccine design. Since SARS-CoV-2 and SARS-CoV are closely related, previous findings on SARS-CoV are highly relevant to a better understanding as well as diagnosis, treatment, prevention and control of SARS-CoV-2. In this review, we highlight recent progresses in the field; compare the biological characteristics of SARS-CoV and SARS-CoV-2; summarize the urgently-needed diagnostic, treatment, prevention and control options; and provide future perspectives for the outcome of the outbreak and research questions to be answered, including some of the difficulties in vaccine development. Hopefully, our comments and suggestions would prove useful for the control of the SARS-CoV-2 epidemic in China and the world.
coronavirus disease 2019 (COVID-19), severe acute respiratory syndrome coronavirus (SARS-CoV), SARS-CoV-2, Middle East respiratory syndrome coronavirus, virological diagnosis, coronavirus vaccines, antiviral therapy
金冬雁 香港大学霍广文伉俪基金精准医学教授,任职于医学院生物医学学院,兼任研究学院副院长。1993年获选为中国首届中青年医学科技之星,2001年获国家自然科学二等奖,2014年获裘槎优秀科研奖,2020年当选美国微生物科学院院士。多年从事分子病毒学研究,曾在2008年和2014年分别发现SARS冠状病毒和MERS冠状病毒编码的干扰素拮抗蛋白。在病毒与细胞相互作用及固有抗病毒免疫方面的多项发现均被认为是该领域的突破,为防治病毒感染提供了新思路。

March 12, 2020;
March 27, 2020
Supported by:Hong Kong Health and Medical Research Fund (No. HKM-15-M01), Hong Kong Research Grants Council (No. T11-707/15-R).
Dong-Yan Jin. Tel: +852-39179491; Fax: +852-28551254; E-mail: dyjin@hku.hk
香港医疗卫生研究基金(No. HKM-15-M01),香港研究资助局(No. T11-707/15-R) 资助。
10.13345/j.cjb.200115
叶子葳, 金冬雁. 2019新型冠状病毒及其诊疗与防控:回顾与展望. 生物工程学报, 2020, 36(4): 571–592.
Ye ZW, Jin DY. Diagnosis, treatment, control and prevention of SARS-CoV-2 and coronavirus disease 2019: back to the future. Chin J Biotech, 2020, 36(4): 571–592.
(本文责编 陈宏宇)

