基于二代测序的甘蓝型油菜白花基因候选区间定位及连锁标记验证
陈雪,王瑞,井付钰,张胜森,贾乐东,段谋正,吴宇
基于二代测序的甘蓝型油菜白花基因候选区间定位及连锁标记验证
陈雪,王瑞,井付钰,张胜森,贾乐东,段谋正,吴宇
(西南大学农学与生物科技学院,重庆 400715)
【目的】近几年随着观光农业的兴起,花色的选育和改良已成为甘蓝型油菜种质资源鉴定和材料创制的重要研究方向。以甘蓝型油菜黄白花分离F2群体为研究对象,通过二代测序技术,对白花性状基因候选区间定位,开发与白花性状连锁的分子标记,为定位白花候选基因和选育白花新材料提供新思路。【方法】以甘蓝型油菜DH纯系黄花Y05和甘蓝型油菜纯系白花W01杂交,观察F1和F2群体的花色分离,分析白花性状遗传模式。在F2群体中选取30株纯白花和30株纯黄花构建DNA叶片子代池和RNA花瓣子代池,对亲本和DNA叶片子代池进行30×重测序,对RNA花瓣子代池进行5×测序。以法国甘蓝型油菜Darmor-bzh、中双11、Darmor、Tapidor为参考序列,重测序QTL-seq分析流程计算2个DNA子代池的SNP-index和delta(SNP-index)。利用R包画出SNP-index和delta(SNP-index)滑窗分析图,鉴定候选区间。转录组MMAPPR分析流程以法国甘蓝型油菜Darmor-bzh为参考序列,计算SNP频率,ED4(Loess fit)检测峰值和鉴定候选区间。利用MISA进行重复序列鉴定,使用Prime3在候选区间进行SSR引物设计,在F2群体中采用聚丙烯酰胺凝胶电泳方法对SSR引物进行筛选。【结果】甘蓝型油菜黄花与白花杂交F2群体中,白花和黄花性状分离比符合3﹕1,暗示白花性状受1对显性主效基因控制。全基因组重测序区间定位结果显示,白花性状基因候选区间在Darmor-bzh C03染色体52—55 Mb。同时以甘蓝型油菜中双11、Darmor、Tapidor分别为参考序列,均鉴定出白花基因候选区间在C03染色体上的一致性和稳定性。转录组测序定位白花性状基因位于Darmor-bzh C03染色体54—55 Mb。转录组测序和重测序定位染色体结果高度一致。在此区间内MISA和Primer3结合设计SSR引物,聚丙烯酰胺凝胶电泳筛选到6个与白花性状紧密连锁共分离的SSR标记。6个SSR标记区间范围在760 kb(52.81—53.57 Mb)。此候选区间与甘蓝、白菜共线性分析,对应白菜A02染色体56.76—57.40 Mb区间,对应甘蓝C03染色体10.99—11.28 Mb区间。【结论】甘蓝型油菜白花性状由1对显性主效基因控制。白花性状基因候选区间在法国甘蓝型油菜Darmor-bzh C03染色体52—55 Mb区间内。此区间760 kb范围内筛选出6个与白花性状基因紧密连锁共分离的SSR标记。
甘蓝型油菜;白花;测序;候选区间;SSR
0 引言
【研究意义】甘蓝型油菜属十字花科(Cruciferace)芸薹属()植物,是中国重要油料作物[1],可作为食用植物油、蛋白质饲料和能源的原料作物。近几年,随着油菜不同花色品种示范与推广,带动了乡村观光旅游,促进了农民增收。因此,加快选育创制不同遗传背景的油菜花色新品种具有重要意义。【前人研究进展】油菜白花花瓣不仅有观赏和装饰价值,还可以转育不育系和恢复系,成为鉴定杂交种纯度的指示性状。油菜的白花性状很早就有报道。PERSON等[2]发现一种白菜白色花突变体,证明白色是由单个显性基因控制的。种间杂交也会产生白色花。CHEN等[3]、HENEEN等[4]、ZHANG等[5]利用白菜和甘蓝人工杂交,获得甘蓝型油菜白花品系。通过多年对油菜白花遗传模式和遗传效应研究,发现白花性状为显性并且没有细胞质效应[6-7]。JAMBHULKAR等[8]在埃塞俄比亚芥中发现白花性状是由1对不完全显性基因控制。芥菜型油菜白花性状是由2对基因互作影响[9-10]。多位学者研究甘蓝型油菜白花由1对核基因控制,白花对黄花为显性[11-13]。分子标记辅助选择是转育质量性状的重要技术手段。LIU等[13]鉴定出白花性状由5个QTL控制。HAN等[14]通过InDel标记将甘蓝白花性状定位于C03染色体上。HUANG等[12]利用AFLP和SSR得到与白花基因连锁的2个标记,距离为3.0和3.2 cM。ZHANG等[11]构建回交和DH群体,用AFLP和SSR标记把甘蓝型油菜白花定位到C03染色体上,再与甘蓝参考组序列比对,获得白花候选基因CCD4。近年来,基于二代测序的BSA分析技术为质量性状或主效基因快速准确定位提供了强大工具。目前,已在水稻[15-17]、小麦[16]、大豆[18]、番茄[19]、黄瓜[20-22]等作物中用于对质量性状或主效基因进行遗传定位研究,快速筛选靶基因获得紧密连锁分子标记。在利用BSA重测序定位油菜花色方面,YAO等[23]将甘蓝型油菜橙色花性状基因定位于C09染色体151 kb区间,在此区间开发了连锁SSR和InDel标记。ZHANG等[24]利用BSA重测序将芥菜型白花性状定位于B04染色体,区间为2.45 Mb,并开发出SSR连锁标记。【本研究切入点】迄今为止,已有多位作者运用传统分子标记技术将白花相关基因定位到C03染色体,并开发了紧密连锁的分子标记。但利用二代测序技术快速精准定位甘蓝型油菜白花基因候选区间仍鲜见报道。【拟解决的关键问题】本研究通过甘蓝型油菜黄花DH系和白花DH系杂交,对获得的F2代2个极端子代池开展全基因组重测序和转录组测序,定位白花基因候选区间。在区间内设计SSR引物,用SSR连锁标记验证候选区间定位的准确性,为精细定位白花候选基因和分子标记辅助选育甘蓝型油菜白花新材料奠定基础。
1 材料与方法
1.1 材料
西南大学油菜生物学团队将甘蓝型油菜和羽衣甘蓝远缘杂交,在分离群体中,获得白花突变体,再与甘蓝型油菜多代回交获得甘蓝型白花油菜,同时小孢子加倍获得甘蓝型白花纯系DH材料W01。DH纯系黄花材料Y05是通过小孢子加倍选育的隐性纯系临保系。2016年用DH纯系黄花Y05与纯系白花W01杂交获得F1,次年获得F2。从F2分离群体中选取极端白花单株和极端黄花单株,构建白花子代池和黄花子代池,用于白花基因候选区间定位。
1.2 田间试验和性状调查
2017年3月花期,F1单株自交获得F2。2017年9月25日对亲本和F2进行小区育苗,10月将单株移栽到西南大学歇马油菜基地试验田,行距为0.2 m,株距为0.2 m。2017年12月苗期对F2群体213个单株插牌编号。2018年3月初花期和盛花期对F2群体213个单株分别记录花色。
1.3 子代池构建
2017年12月苗期,对亲本Y05和W01以及F2群体每个单株按插牌编号取幼嫩叶0.2 g。2018年3月初花期和盛花期按苗期插牌编号记录单株花色。选取极端纯白花30株和极端纯黄花30株,对应到苗期编号取幼嫩叶。利用OMEGA HP Plant DNA试剂盒对2个亲本和极端黄、白花的幼嫩叶提取DNA。将纯白花幼嫩叶和纯黄花幼嫩叶各30株的DNA等量混合,构建白花DNA子代池和黄花DNA子代池。在F2群体处于初花期时,选取纯白花和纯黄花各30株,取每株刚张开的花瓣0.15 g,利用EZ-10 Total RNA Mini-Preps Kits试剂盒提取RNA。将极端纯白花花瓣和极端纯黄花花瓣各30株的RNA等量混合,构建白花RNA子代池和黄花RNA子代池。2个DNA子代池和2个亲本DNA建库类型为DNA-350 bp,以illumina HiSeq PE150方法测序,测序深度为30×。2个RNA子代池建库类型为DNA-300 bp,以illumina HiSeq PE125方法测序,测序深度为5×。
1.4 数据处理
对DNA子代池和2个亲本30×重测序的原始数据去除接头和低质量序列,得到分析数据。启动QTL-seq shell流程[25],FASTX-TOOLKIT软件过滤低质量reads;将过滤后的亲本reads与法国甘蓝型油菜参考组Darmor-bzh Brassica_napus.v4.1.fa(http://www. genoscope.cns.fr/ brassicanapus/)比对,并替换SNP,构建亲本参考组,再将亲本reads重新与新构建的亲本参考基因组比对,发现错配造成的SNP,用于后续的排除和过滤。将2个DNA子代池质控过滤后的reads分别与亲本参考基因组比对。使用Coval Refine对比对结果进行过滤,Coval Call检测变异位点,排除由于错配导致的SNP位点,计算2个DNA子代池的SNP-index以及2个子代池之间的delta(SNP-index)。利用R包画出SNP-index和delta(SNP-index)滑窗分析图,鉴定候选区间。
RNA子代池测序数据需要以下预处理:利用bwa软件将白花RNA子代池和黄花RNA子代池测序数据与法国甘蓝型油菜参考基因组Darmor-bzh比对,对得出的sam文件进行sort排序,去除PCR重复,建立索引文件。GATK软件再重新比对获得白花子代池和黄花子代池bam文件,启动MMAPPR分析流程[26],计算SNP频率,Loess fit of ED4检测峰值和鉴定候选区间。
1.5 引物设计与电泳
利用MISA(http://pgrc.ipk-gatersleben.de/misa/)进行重复序列鉴定并使用Prime3在候选区间进行SSR引物设计,引物序列由上海生工生物工程技术服务有限公司合成。随机选取F2群体极端黄花和极端白花各11株幼嫩叶DNA为模板,进行PCR扩增。PCR体系为2.2 μL模板DNA、0.25 μL 2.5 mmol·L-1dNTP、前后引物各0.36 μL、0.31 μL Taq酶(2.5 U·μL-1)、1.9 μL 10×PCR buffer(含Mg2+)。PCR程序为94℃ 5 min;94℃ 30 s,52℃—60℃ 30 s,72℃ 30 s,共35个循环;72℃ 5min;4℃保存。PCR扩增产物经8%变性聚丙烯酰胺凝胶电泳分离60min,用银染法进行显影。
2 结果
2.1 黄花Y05和白花W01组合后代表型观察和遗传分离
甘蓝型油菜黄花DH纯系Y05与白花纯系W01杂交,F1均为白花,F2呈明显主效基因分布特点,白花对黄花为显性性状。F2群体中纯白花和纯黄花分离明显(图1)。
对2年田间试验数据进行卡方测验,结果表明,黄白花性状分离比符合3﹕1的分离规律(表1),暗示白花性状受1对显性主效基因控制。
2.2 重测序DNA子代池数据分析
分别用已发表的法国甘蓝型油菜[27]Brassica_ napus.v4.1.fa(http://www.genoscope.cns.fr/ brassicanapus/)、甘蓝型油菜中双11[28](http://ocri-genomics.org/Brassia_ napus_genome_ZS11/)、2个澳大利亚甘蓝型油菜[29](Darmor、Tapidor(https://www.ncbi.nlm.nih.gov/bioproject/ 342383/;http://appliedbioinformatics.com.au/index.php/ Darmor_Tapidor/)为参考基因组序列。以2 Mb为窗口,50 kb步长对delta(SNP-index)在19条染色体上作图。以95%和99%作为置信区间,置信水平以上窗口作为候选区间(图2)。区间定位结果显示,白花性状基因候选区间在Darmor-bzh C03染色体52— 55 Mb,在ZS11 C03染色体62Mb左右,在Tapidor C03染色体48 Mb左右,在Darmor C03染色体61 Mb附近。以这4个甘蓝型油菜基因组序列为参考组,均鉴定出白花基因候选区间在C03染色体上的一致性和稳定性。

A:黄花Y05;B:F2黄花;C:白花W01;D:F2白花
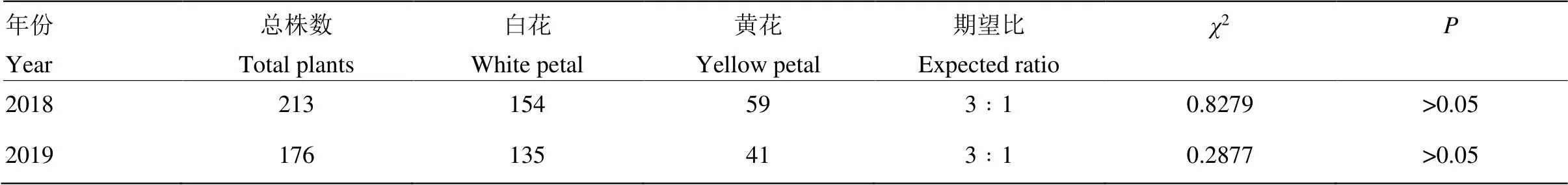
表1 黄花Y05与白花W01杂交F2花色分离比例
2.3 转录组子代池数据分析
MMAPPR[26]分析白花花瓣和黄花花瓣2个转录组子代池时,需要更改参考组Darmor-bzh基因序列染色体名称以符合流程要求。将chr.A01—chr.A10、chr.C01—chr.C09更改为chr.1—chr.19。MMAPPR方法以2个子代池SNP频率为基础计算出ED4(Loess fit)(图3),以0.6为阈值,白花性状基因定位于chr.13(C03)染色体54—55 Mb。转录组测序和重测序定位染色体结果高度一致,仅定位区间有细小差别,可能是转录组子代池比对分析时,以法国甘蓝型油菜Darmor-bzh为参考组,未利用亲本信息,然后直接计算子代池SNP频率所致。
2.4 SSR引物设计和聚丙烯酰胺电泳
依据法国甘蓝型油菜Darmor-bzh参考序列,在C03染色体52—55 Mb区间运用MISA和Primer3结合设计SSR引物(表2)。筛选到6个能明显区分F2极端黄花单株和极端白花单株的SSR标记(图4)。6个标记大约在760 kb范围之内。这6对SSR引物进行单株验证均未检测到交换株,推测控制白花性状候选基因与这些SSR标记紧密连锁共分离。
2.5 白花候选区间基因线性比对
6个SSR连锁标记范围确定的区间(52.81—53.57 Mb)内有80个注释基因。利用https://gsthub.com/ tanghaibao/jcvi/Mcscan-(Python-version) python分析此候选区间基因与甘蓝、白菜物种之间的共线性(图5)。白菜A02染色体56.76—57.40 Mb区间有26个同源基因,甘蓝C03染色体10.99—11.28 Mb区间有63个同源基因。甘蓝与白菜种间杂交可人工合成甘蓝型油菜,种间杂交合成的甘蓝型油菜易出现白花性状,共线性分析揭示了白花性状相关同源基因由二倍体到四倍体物种的进化关系,暗示甘蓝型油菜白花基因来源于甘蓝或白菜。
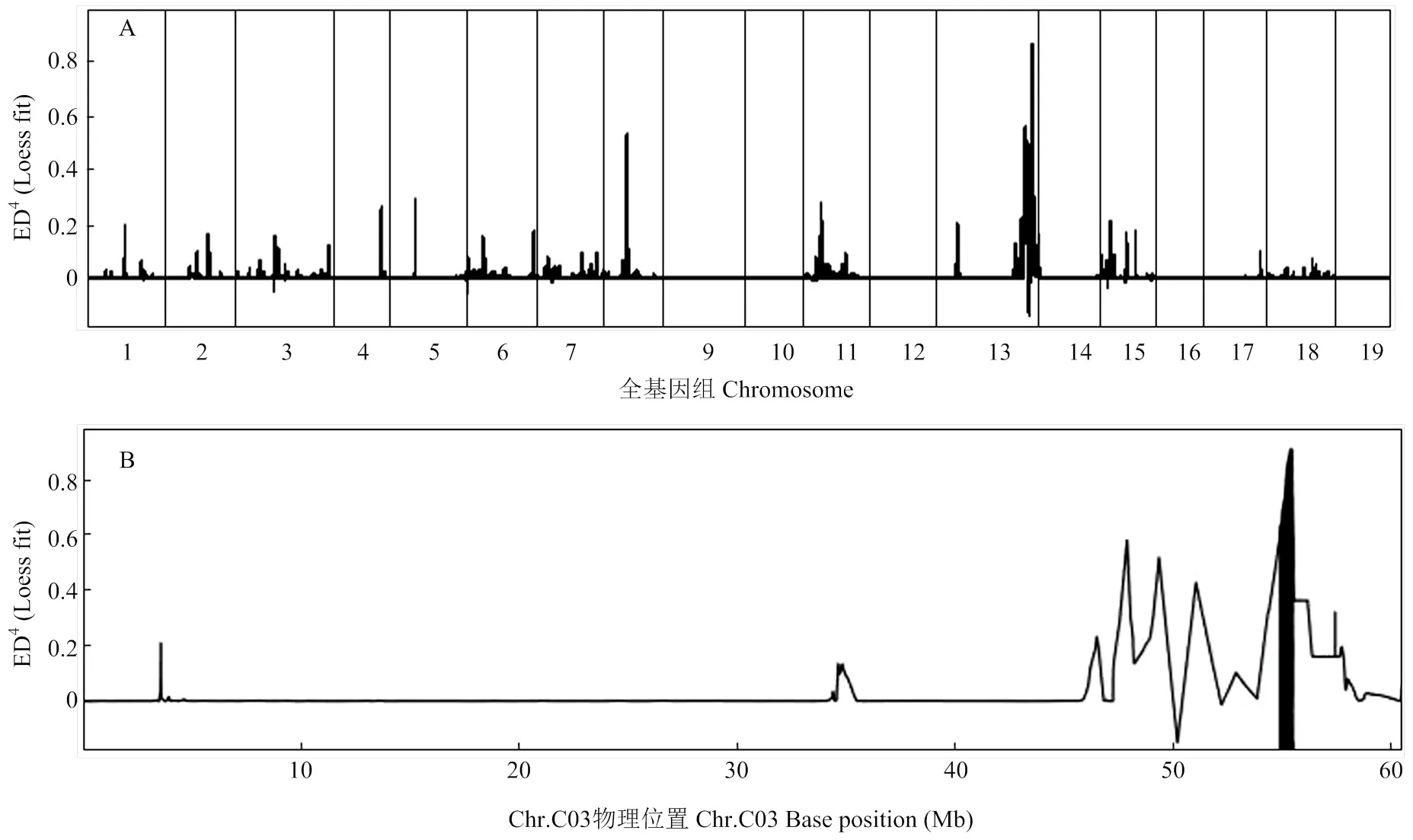
全基因组(A)和染色体Chr.13(C03)(B)上SNP频率对应的ED4(Loess fit)曲线

表2 SSR引物序列

A:SSR149;B:SSR154;C:SSR157;D:SSR161;E:SSR180;F:SSR222;M:20 bp ladder;1:亲本Y05;2:亲本W01;3—13:F2群体11个黄花单株;14—24:F2群体11个白花单株
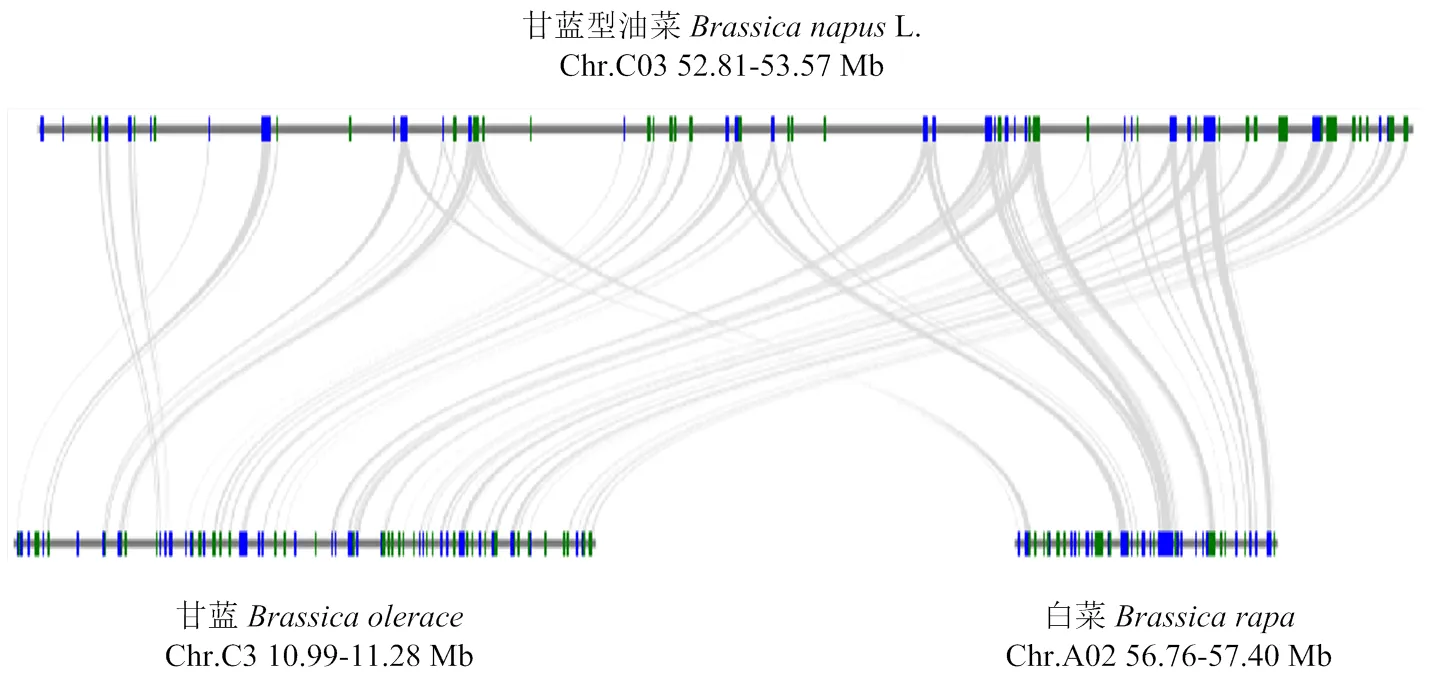
图5 甘蓝型油菜与甘蓝、白菜的白花基因候选区间线性比对
3 讨论
3.1 基于二代测序正向遗传学油菜白花性状定位
以往对芸薹属白花性状的研究主要集中在孟德尔遗传模式、遗传图谱构建和连锁分子标记开发以及辅助选择。二代测序方法的迅速发展促进了正向遗传学性状定位。目前,仅有ZHANG等[24]构建了芥菜型白花性状的回交群体,对分离后代黄白花子代池重测序,把白花性状定位在B04染色体,区间约2.45 Mb。在此区间上设计SSR引物,用隐性单株群体把候选区间缩小至0.25 cM。而对甘蓝型油菜白花性状,利用二代测序方法快速精准定位甘蓝型油菜白花基因候选区间仍鲜见报道。XIAO等[30]构建了甘蓝型油菜黄花和白花的182株DH群体,利用黄白花各10株构建混池方法,在全染色体筛选SSR引物,找到5个与白花性状连锁的SSR标记,定位于C03染色体上。LIU等[31]再利用这5个与白花性状连锁最近的2个SSR标记序列与甘蓝参考组比对,在甘蓝对应的区间内又开发了9个SSR连锁标记。ZHANG等[11]构建了白花和黄花的BC5F2群体,选取1120株黄花隐性单株,继续使用这14个SSR标记进行精细定位。将与白花性状基因最近的两端SSR标记BoGMS3990和BoGMS3988序列与甘蓝参考组和甘蓝注释文件比对,推测出此区间包含24个基因,其中一个为。比对到甘蓝参考组C03染色体48.6Mb附近。由于调控类胡萝卜素合成,暗示参与甘蓝型油菜白花代谢调控。以上所述甘蓝型油菜白花候选区间定位方法极为繁琐并且成本昂贵。本研究结合使用QTL-seq和MMAPPR分析流程,以最新发布的法国甘蓝型油菜Darmor-bzh序列为参考组[29],仅用甘蓝型油菜黄×白F2小群体,二代方法重测序和转录组测序相互验证快速鉴定出甘蓝型油菜白花性状候选基因在C03染色体,物理区间为52—55 Mb。与ZHANG等[11]传统方法相比,不需要构建复杂的定位群体,不依赖遗传图谱和大量分子标记,周期短效率高。白花性状基因候选物理区间52—55Mb再通过与甘蓝型油菜注释文件区间比对,发现此区间3个MYB蛋白基因(、和)和3个WD40重复蛋白基因(、和)。MYB转录因子和WD40转录因子均是通过调控类黄酮代谢途径,进而影响到色素的合成。因而,本研究获得的甘蓝型油菜白花候选基因物理区间和区间内有关色素基因与前人研究结果完全不同。
3.2 参考基因组选择及亲本信息利用
参考基因序列组装质量越好,信息越全,注释文件信息也相对齐全,对于后续基因定位和候选基因注释都会更加准确,可以锁定候选区间并估计候选区域的大小。由于法国甘蓝型油菜Darmor-bzh序列组装质量和注释文件版本不断更新,有学术团队维护和发布最新版本,甘蓝型油菜重测序和转录组分析流程中的参考组序列一般优先选用法国甘蓝型油菜Darmor-bzh。但Darmor-bzh与本文中所测材料遗传背景差异大,用亲本序列替换后构建新参考组再进行比对,所获结果就更加准确。QTL-seq分析2个子代池和亲本重测序数据,亲本reads与参考组Darmor-bzh序列进行替换,构建新的参考组提高比对率。以新参考组为参照比对2个子代池测序数据,进行SNP变异分析计算SNP-index和delta(SNP-index)。因而,本文设计SSR引物以QTL-seq流程利用Darmor-bzh参考组分析获得的候选区间为依据。Darmor-bzh参考组52—55 Mb区间内,MISA软件微卫星和复合微卫星识别,再使用Primer3进行SSR引物设计。选取2个子代池相减最高频率的2 Mb区间内的20对SSR引物,用F2群体的极端白花单株和极端黄花单株验证,获得6个与白花性状紧密连锁共分离的SSR标记,这6个SSR标记在760 kb范围之内,其中SSR 222标记与MYB蛋白基因物理距离小于16 kb。因此,下一步的工作需要扩大F2种植群体,利用6个共分离SSR标记同时开发新SSR和InDel连锁标记对白花性状基因进行精细定位,缩小候选基因范围,为甘蓝型油菜白花性状基因图位克隆奠定工作基础。
4 结论
甘蓝型油菜白花性状基因在C03染色体52—55 Mb区间。在此区间760 kb范围内筛选出6个与白花性状基因紧密连锁共分离的SSR标记。
[1] 王汉中, 殷艳. 我国油料产业形势分析与发展对策建议. 中国油料作物学报, 2014, 36(3): 414-421.
WANG H Z, YIN Y. Analysis and strategy for oil crop industry in China., 2014, 36(3): 414-421. (in Chinese)
[2] PEARSON O H. A Dominant white flower color inL.., 1929, 63: 561-565.
[3] CHEN B, HENEEN W, JONSSON R. Independent inheritance of erucic acid content and flower colour in the C-genome ofL.., 1988, 100: 147-149.
[4] HENEEN W, CHEN B, CHENG B, JONSSON A, SIMONSEN V, JORGENSEN R, DAVIK J. Characterization of the A and C genomes ofand., 1995, 123: 251-267.
[5] ZHANG B, LU C M, KAKIHARA F, KATO M. Effect of genome composition and cytoplasm on petal color in resynthesized amphidiploids and sesquidiploids derived from crosses betweenand., 2002, 121: 297-300.
[6] LEE S, LEE S C, BYUN D H, LEE D Y, PARK J Y, LEE J H, LEE H O, SUNG S H, YANG T J. Association of molecular markers derived from the BrCRTISO1 gene with prolycopene-enriched orange-colored leaves in.,2014, 127: 179-191.
[7] RAHMAN M H. Inheritance of petal colour and its independent segregation from seed colour in., 2001, 120: 197-200.
[8] JAMBHULKAR S, RAUT R. Inheritance of flower colour and leaf waxiness inA. Br., 1995, 17: 66-67.
[9] RAWAT D S, ANAND I J. Inheritance of flower colour in mustard mutant., 1986, 56: 206-208.
[10] SINGH K H, CHAUHAN J S. Genetics of flower colour in Indian mustard (L. Czern $ Coss)., 2011, 71: 377-378.
[11] ZHANG B, LIU C, WANG Y, YAO X, WANG F, WU J, KING G J, LIU K. Disruption of a CAROTENOID CLEAVAGE DIOXYGENASE 4 gene convents flower colour from white to yellow in., 2015, 206: 1513-1526.
[12] HUANGE Z, BAN Y Y, BAO R, ZHANG X X, XU A X, DING J. Inheritance and gene mapping of the white flower inL.., 2014, 42(2): 111-117.
[13] LIU X P, TU J X, CHEN B Y, FU T D. Identification of the linkage relationship between the flower colour and the content of erucic acid in the resynthesizedL.., 2004, 31: 357-362.
[14] HAN F Q, YANG C, FANG Z Y, YANG L M, ZHUANG M, LV H H, LIU Y M, LI Z S, LIU B, YU H L, LIU X P, ZHANGH Y Y. Inheritance and InDel markers closely linked to petal color gene (cpc-1) in., 2015, 35: 160.
[15] MITHRA S V A, KAR M K, MOHAPATRA T, ROBIN S, SARLA N, SESHASHAYEE M, SINGH K, SINGH N K, SHARMA R P. DBT propelled national effort in creating mutant resource for functional genomics in rice., 2016, 110(4): 543-548.
[16] HENRY I M, NAGALAKSHMI U, LIEBERMAN M C, NGO K J, KRASILEVA K V, VASQUEZ-GROSS H, AKHUNOVA A, AKUNOV E, DUBCOVSKY J, TAI T H, COMAI L. Efficient genome-wide detection and cataloging of EMS-induced mutations using exome capture and next-generation sequencing., 2014, 26(4): 1382-1397.
[17] WEI F J, DROC G, GUIDERDONI E, HSING Y I C. International consortium of rice mutagenesis: resources and beyond., 2013, 6(1): 39.
[18] TSUDA M, KAGA A, ANAI T, SHIMIZU T, SAYAMAT, TAKAGI K, MACHITA K, WATANABE S, NISHIMURA M, YAMADA N, MORI S, SASAKI H, KANAMORI H, KATAYOSEY, ISHIMOTO M. Construction of a high-density mutant library in soybean and development of a mutant retrieval method using amplicon sequencing., 2015, 16: 1014.
[19] JUST D, GARCIA V, FERNANDEZ L,BRES C, MAUXION J P, PETIT J, JORLY J, ASSALI J, BOURNONVILLE C, FERRAND C, BALDET P, LEMAIRE-CHAMLEY M, MORI K, OKABE Y, ARIIZUMI T, ASAMIZU E, EZURA H, ROTHAN C. Micro-Tom mutants for functional analysis of target genes and discovery of new alleles in tomato., 2013, 30(3): 225-231.
[20] LIN T, WANG S H,ZHONG Y, GAO D L, CUI Q Z, CHEN H M, ZHANG Z H, SHEN H L, WENG Y Q, HUANG S W. A truncated F-box protein confers the dwarfism in cucumber., 2016, 43(4): 223-226.
[21] LUN Y Y, WANG X, ZHANG C Z, YANG L, GAO D L, CHEN H M, HUANG S W. A CsYcf54 variant conferring light green coloration in cucumber., 2016, 208(3): 509-517.
[22] ZHOU Q, WANG S H, HU B W, CHEN H M, ZHANG Z H, HUANG S W. An ACCUMULATION AND REPLICATION OF CHLOROPLASTS 5 gene mutation confers light green peel in cucumber., 2015, 57(11): 936-942.
[23] YAO Y M, LI K X, LIU H D, DUNCAN R W, GUO S M, XIAO L, DU D Z. Whole-genome re-sequencing and fine mapping of an orange petal color gene () in springL. to a 151-kb region., 2017, 213: 165.
[24] ZHANG X X, LI R H, NIU S L, CHEN L, GAO J, WEN J, YI B, MA C Z, TH J X, FU T D, SHEN J X. Fine-mapping and candidate gene analysis of thewhite-flowered mutant Bjpc2 using the whole-genome resequencing., 2017, 293(2): 359-370.
[25] TAKAGI H, ABE A, YOSHIDA K, KOSUGI S, NATSUME S, MITSUOKA C, UEMURA A, UTSUSHI H, TAMIRU M, TAKUMO S, INNAN H, CANO L M, KAMOUN S, TERAUCHI R. QTL-seq: rapid mapping of quantitative trait loci in rice by whole genome resequencing of DNA from two bulked populations., 2013, 74(1): 174-183.
[26] JONATHON T H, BRADLEY L D, BRENT W B, BUSHRA G, YI C S, H J Y. MMAPPR: Mutation mapping analysis pipeline for pooled RNA-seq., 2013, 23: 687-697.
[27] CHALHOUB B, DENOEUD F, LIU S, PARKIN A P, TANG H, WANG X, CHIQUET J. Early allopolyploid evolution in the post- Neolithicoilseed genome., 2014, 345: 950-953.
[28] SUN F M, FAN G Y, HU Q, ZHOU Y M, GUAN M, TONG C B, LI J N, DU D Z, QI C K, JIANG L C,LIU W Q, HUANG S M, CHEN W B, YU J Y, MEI D S, MEN J L, ZENG P, SHI J Q, LIU K D, WANG X, WANG X F, LONG Y, LIANG X M, HU Z Y, HUANG G D, DONG C H, ZHANG H, LI J, ZHANG Y L, LI L W, SHI C C, WANG J H, MING-YUEN L S, GUAN C, XU X, LIU S Y, LIU X, CHALHOUB B, HUA W, WANG H Z. The high-quality genome ofcultivar ‘ZS11’ reveals the introgression history in seni-winter morphotype., 2017, 92: 452-468.
[29] BAYER P E, HURGOBIN B, GOLICZ A A, CHAN C K, YUAN Y X, LEE H T, RENTON M,MENG J L, LI R Y, LONG Y, ZOU J, BANCROFF L, CHALHOUB B, KING G J, BATLEY J,EDWARDS D. Assembly and comparison of two closely relatedgenomes., 2017, 15: 1602-1610.
[30] XIAO S, XU J, LI Y, ZHANG L, SHI S, SHI S, WU J, LIU K.Generation and mapping of SCAR and CAPS markers linked to the seed coat color gene inusing a genome-walking technique., 2007, 50(7): 611-618.
[31] LIU S Y, LIU Y M, YANG X H, TONG C B, EDWARDS D, PARKIN IA, ZHAO M X, MA J X, YU J Y, HUANG S M, WANG X Y, WANG Y J, LU K, FANG Z Y, BANCROFT L, YANG T, HU Q, WANG X F, YUE Z, LI H J, YANG L F, WU Q, WANG W X, KING G J, PIRES J, LU C X, WU Z Y, SAMPATH P, WANG Z, GUO H, PAN S K, YANG L M, MIN J M, ZHANG D, JIN D C, LI W S, BELCRAM H, TU J X, GUAN M, QI C K, DU D Z, LI J N, JIANG L C, BATELY J, SHARPE A G, PARK B, RUPERAO P, CHENG F, WAMINAL N E, HUANG Y, DONG C H, WANG L, LI J P, HU Z Y, LI Z Y, LI X, ZHANG J F, XIAO L, ZHOU Y M, LIU Z S, LIU X Q, QIN R, TANG X, LIU W B, WANG Y P, ZHANG Y Y, LEE J H, KIM H H, DENOEUD F, XU X, LIANG X M, HUA W, WANG X W, WANG J, CHALHOUB B, PATERSON A H. Thegenome reveals the asymmetrical evolution of polyploid genomes., 2014, 5: 3930.
Location and Linkage Markers for Candidate Interval of the White Petal Gene inL. by Next Generation Sequencing
CHEN Xue, WANG Rui, JING FuYu, ZHANG ShengSen, JIA LeDong, DUAN MouZheng, WU Yu
(College of Agronomy and Biotechnology, Southwest University, Chongqing 400715)
【Objective】Since the petal colour can be used for ornamental and landscaping purposes, the petal color has been one of the major goals of breeding and genetic research inL.. In this paper, Genetic analysis, candidate interval identification, linkage markers and synteny analysis were applied to elucidate the genetic control of the white petal inL.. 【Method】 To Map the white petal locus, an inbred line Y05, which has yellow flowers, was crossed with an inbred line W01, which has white flowers. The F1plants were self-crossed to develop F2mapping population. For BSA, parental and two pools with 30 yellow petal lines and 30 white petal lines of F2were constructed by mixing an equal amount of DNA or RNA respectively. 30× or 5× depth of genome-sequencing was conducted. Darmor-bzh, Zhongshuang11(ZS11), Darmor and Tapidor as the reference genome were aligned to sequence data from the 2 bulks and parents using QTL-seq workflow. The sliding window method with a window size of 2Mb and a step size of 50kb was used to present the SNP indexes of the whole genome. The difference between the SNP indexes of the two pools was calculated as the delta (SNP- index). Candidate regions for petal color were identified from the chromosomes with 95% confidence intervals. Mutation Mapping Analysis Pipeline for Pooled RNA-seq (MMAPPR) without parental strain information and requiring Darmor-bzh reference genome calculated allelic frequency by Euclidean distance followed by Loess regression analysis, and identified the region where the mutation lies, and generated a list of putative coding region mutations in the linked genomic segment. The SSR primers were designed by using MISA and Prime 3 for repeated sequence identification, and the SSR primers were screened by polyacrylamide gel electrophoresis in the F2population. 【Result】The segregation of white petal and yellow petal among F2population fitted the Mendelian segregation ratio of 3:1. This indicates that the white petal trait was controlled by a major gene and that white petal was dominant over yellow petal. The results of the candidate interval using whole-genome re-sequencing showed that a candidate interval (52-55 Mb) exceeding the threshold value was identified for the petal color on chromosome C03 when Darmor-bzh was used as reference genome. While ZS11, Darmor and Tapidor were aligned to sequence data, candidate intervals for white petal were all identified on chromosome C03. Linked region peaks (54-55 Mb) identified by MMAPPR for the petal color was on chromosome C03 of Darmor-bzh. Six SSR markers that were located in the interval (760 kb) were closely linked to the white flower gene. Synteny analysis showed that the interval 760 kb (52.81-53.57 Mb) was corresponding to chromosome A02 (56.76-57.40 Mb) ofand chromosome C03 (10.99-11.28 Mb) of【Conclusion】The white petal was controlled by a major gene which was dominant over yellow petal. Six SSR markers closely linked to the white petal gene were selected. A candidate interval for white petal gene was identified on chromosome C03 (52-55 Mb). The present study may facilitate cloning of the white petal gene as well as marker assisted selection.
L.; white petal; sequencing; candidate interval; SSR
2019-08-21;
2019-10-30
国家重点研发计划“七大农作物育种”(2016YFD0101300)
陈雪,Tel:15683993928;E-mail:cx_526@163.com。通信000作者王瑞,Tel:13883344308;E-mail:ruiwang71@163.com
(责任编辑 李莉)

