不同水分条件下格陵兰岛冻土活性甲烷氧化菌群落分异规律①
刘 蓓,Bo Elberling,贾仲君
不同水分条件下格陵兰岛冻土活性甲烷氧化菌群落分异规律①
刘 蓓1,2,Bo Elberling3,贾仲君2*
(1 中国科学院大学中丹学院,北京 100190;2土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所),南京 210008;3 Center for Permafrost (CENPERM), Department of Geosciences and Natural Resource Management, University of Copenhagen, Copenhagen K 1350)
揭全球气候变化导致丹麦格陵兰岛形成了旱地和间歇淹水的土壤景观,采用稳定性同位素核酸探针技术和高通量测序16S rRNA及基因的分析方法,开展了格陵兰岛旱地和间歇淹水土壤微宇宙培养试验,探究不同水分条件下冻土的甲烷氧化潜力及活性好氧甲烷氧化菌群落演替规律。结果表明:与旱地土壤相比,淹水土壤氧化高浓度甲烷的速率呈现降低趋势,分别为12.38和 12.17 μg/(g·d),但后者对甲烷碳同化利用效率显著高于前者,土壤13C-有机碳原子百分比从自然丰度1.08%,分别增加至1.64% 和1.99%。超高速密度梯度离心分析13C-DNA发现甲烷氧化菌群落发生演替,旱地土壤中甲烷氧化菌16S rRNA基因丰度仅为0.04%,而在间歇淹水土壤中为23.78%,增幅高达557倍;类型II甲烷氧化菌则从33.76% 增至44.38%。然而,类型I甲烷氧化菌的丰度明显降低,从旱地土壤10.15% 显著降低为间歇淹水0.14%;进一步通过基因高通量测序分析,也得到了类似的结果,特别是类型I甲烷氧化菌RPCs从旱地土壤15.61% 显著降低至间歇淹水土壤的0.13%。这些结果表明:尽管格陵兰冻土中经典的类型II甲烷氧化菌主导了旱地土壤和间歇性淹水土壤好氧甲烷氧化过程,但水分可能是甲烷氧化菌群落演替的重要环境驱动力,水分增加导致活性的类型I种群丰度降低,同时显著刺激了新型甲烷氧化菌的大量生长并可能在间歇淹水土壤中发挥了重要作用。
好氧甲烷氧化菌;活动层;稳定性同位素示踪DNA;冻土;土壤含水量
丹麦格陵兰岛是温室气体排放与全球变化研究的发源地和敏感区。近年来开展的对极地冰芯古气候重建研究发现温室气体大幅升高,引起了世界各国政府和学术界对全球变暖的高度重视[1]。例如,大气中甲烷浓度从工业革命前的0.773 µl/L增加至约1.774 µl/L,被认为是全球气温升高的原因之一[2]。土壤甲烷排放是温室气体的重要源,包括了甲烷产生、氧化消耗以及大气传输3个过程。微生物氧化是大气甲烷的重要汇,其甲烷氧化能力显著影响全球甲烷排放总量[3]。据估算,淹水环境中产生的甲烷,大约43% ~ 90% 被微生物通过好氧甲烷氧化过程氧化生成二氧化碳或者合成生物物质[5-6],而土壤所氧化的甲烷占全球甲烷净排放总量的7% ~ 10%[7]。因此,好氧甲烷氧化菌是全球变化生物学研究的重要内容。
格陵兰岛地势高处常年易受风力侵蚀影响,其积雪层厚度以及分布状况稳定性较差,特别是随着近年来全球升温变化加剧,夏季气温的逐渐上升,表层积雪融化速度加快,最早在七月下旬积雪即可完全融化,融水迅速流向地势低洼处,导致高处土壤处于相对干旱状态,致使表层植被在生长后期无充足的水分来源。而岛上地势低处洼地土壤积雪融化速度缓慢,使得该区域植被在整个生长期内都处于积雪融化阶段,保证了持续不断的水源供给,从而全年处于间歇性淹水状态[13]。格陵兰岛高地旱地土壤和洼地间歇淹水土壤多年来处于不同的土壤水分环境,其中由于水分状况不同导致的生态分异不仅表现在不同的土壤植被方面,也有可能影响土壤甲烷排放过程和甲烷氧化过程。氧气是好氧甲烷氧化过程的重要限制因素,大气甲烷氧化过程也主要发生在好氧旱地的表层土壤[7]。土壤水分则很大程度上影响好氧甲烷氧化菌所需氧气的扩散速率,从而影响土壤的甲烷氧化过程。当土壤经历周期性淹水时,缺氧/厌氧环境有利于产甲烷古菌生长并产生大量甲烷,从而刺激好氧甲烷氧化菌的生长和繁殖。然而土壤水分对北极冻土活跃层甲烷氧化潜力,以及其中参与甲烷氧化过程的活性好氧甲烷氧化菌群落结构的影响却鲜有研究。
根据形态以及代谢途径等特征,甲烷氧化菌可分为type I和type II两类:type I主要包括,,,,和等属,而type II则包括,,和等属;同时,type II倾向于在胁迫环境中生长,如低浓度甲烷、低浓度氧气以及氮素匮乏环境等,而type I更倾向于富营养环境中生长,如高浓度甲烷和氧气,特别是其生长过程依赖于充足的氮源[8-10]。type I和type II甲烷氧化菌都被发现存在于北极区域的土壤中[11-12]。长期不同的水分胁迫下,格陵兰岛旱地和间歇淹水两种土壤环境中甲烷氧化菌的适应机制尚未见报道。据此,本研究采集了旱地和间歇淹水典型土壤,通过加入CH4428.57 mg/kg soil 的高浓度13C标记甲烷在室内培养35 d后,利用稳定性同位素示踪技术[14-15]和高通量测序技术研究土壤中活性好氧甲烷氧化菌组成分异,探讨土壤水分对土壤甲烷氧化微生物过程的影响。
1 材料与方法
1.1 土壤样品概况
样本采集于格陵兰岛冻土活动层(74°30′N, 20°30′W),其中包括旱地土壤和间歇淹水土壤。2012年7月采集了0 ~ 10 cm的表层土壤,研磨过筛(2 mm)并保存。旱地采样区域植被为,土壤含水饱和度范围为60% ~ 80%; 间歇淹水区域植被为,土壤含水饱和度在65% ~ 90% 之间[13]。旱地土壤样本基本理化性质如下:pH 6.4,全碳含量12.47 g/kg,全氮含量1.09 g/kg,最大持水量62.5%;间歇淹水土壤样本基本理化性质如下:pH 6.0,全碳含量7.50 g/kg,全氮含量0.58 g/kg,最大持水量73.7%。
1.2 微宇宙培养中土壤的甲烷好氧氧化过程
稳定性同位素核酸探针技术也被称为DNA/ RNA-based stable isotope probing (DNA/RNA-SIP)。DNA-SIP示踪土壤好氧甲烷氧化菌的室内培养流程具体如下:首先称取10 g风干土壤于120 ml培养瓶中,加入适量的无菌去离子水,将土壤水分调至最大持水量的40%,在28 ℃避光预培养2 d。然后将水分调至最大持水量的60%,并用橡胶塞加以铝环密封。对于13CH4–标记处理,通过向密封培养瓶内加入5% 的13CH4进行培养,而12CH4–对照组则加入5% 的12CH4进行培养。此外,还设有空气培养对照组,该组未加入任何外源甲烷,目的在于观察土壤对大气浓度甲烷的氧化情况。每个处理分别设置3个重复。培养35 d后对培养瓶中的剩余甲烷浓度进行测定后,打开瓶盖进行破坏性土壤样品采集。其中0.5 g土壤用于测定土壤有机碳中的13C同位素丰度;其余土壤样本保存于 –20 ℃用于提取DNA以及其他后续分析。
1.3 土壤核酸DNA提取以及稳定性同位素标记的13C-DNA分离鉴定
培养结束后,针对13CH4、12CH4、空气培养3个处理的土壤样品共计9个重复,分别称取0.5 g 新鲜土样,使用FastDNAâSpin Kit for Soil(MP Bio)试剂盒,按照说明书的操作进行土壤总DNA的提取。最后将提取得到的土壤总DNA溶解于100 μl Eluent Buffer 中,通过紫外分光光度计(NanoDropâND-1000)测定DNA浓度,其中一部分DNA进行16S rRNA和基因高通量测序;一部分DNA进行超高速密度梯度离心分离和鉴定13C-DNA[16-17]。具体步骤的简要操作如下:将总量为2.0 μg的土壤总DNA与氯化铯溶液混合均匀于6.0 ml的超高速离心管中,确保离心管中溶液初始密度为1.725 g/ml;然后将该超高速离心试管置于贝克曼超高速离心机中,在20 ℃条件下,以190 000´g的超高速密度梯度离心44 h;通过自动分离装置从6.0 ml的超高速离心管中获得15个不同浮力密度的DNA溶液,经过PEG和乙醇等系列的纯化清洗过程,最终将其溶解于30 μl无菌水中进行保存。针对离心所得到的DNA分层溶液,利用Real-time quantitative PCR (qPCR) 实时荧光定量PCR技术对不同浮力密度DNA中的基因进行定量[18]。实时荧光定量qPCR的引物为A189F/ mb661R,其20 μl的反应体系包括:10 μl的SYBR Premix Ex Taq (Takara),引物各0.5 μl,1 μl的DNA模版和8 μl的无菌水。qPCR扩增条件为 95 ℃,2 min;39´(95 ℃,10 s; 55 ℃,30 s; 72 ℃,30 s);72 ℃,5 min。通过比较13CH4-标记、12CH4-对照处理不同浮力密度DNA中基因绝对数量,即可明确鉴定含有13C-DNA的标记重层,进一步通过对该重层的13C-DNA进行16S rRNA和基因高通量测序,即可明确土壤中活性好氧甲烷氧化菌的群落结构,包括其分类组成、绝对数量和相对丰度。
1.4 16S rRNA和pmoA基因高通量测序
针对培养后的13CH4–标记、12CH4–对照组的土壤总DNA,以及DNA-SIP重层13C-DNA样本进行高通量16S rRNA和基因测序。16S rRNA基因的PCR扩增体系为50 μl,包括:25 μl的Premix TaqTM (TaKaRa),通用引物515F/907R[19]各1 μl,2 μl的DNA模版和21 μl 的无菌水。该反应体系的扩增条件为: 94 ℃,5 min; 32´( 94 ℃,30 s ;55 ℃,30 s ;72 ℃,45 s);72 ℃,5 min。基因采用的扩增引物为A189F/mb661R[18],反应体系包括:25 μl的Premix TaqTM (TaKaRa),A189F/Mb661R引物各1 μl,2 μl的DNA模版和无菌水至50 μl反应体系。基因扩增的反应条件: 95 ℃,2 min ;39´(95 ℃,10 s;55 ℃, 30 s;72 ℃,30 s);72 ℃,5 min。获得16S rRNA和基因扩增产物后,利用Agarose Gel DNA Purification kit 试剂盒(TaKaRa)将PCR产物切胶纯化,然后对不同样品的纯化产物进行等摩尔数混合,利用Illumina 公司 MiSeq 测序仪完成高通量测序。利用QIIME软件(1.9.1)开展16S rRNA和基因高通量测序数据处理,详细操作及原理参见http://qiime.org[18]。简而言之,首先通过“”将正反序列进行拼接,而样本的特异barcodes 将利用“”进行去除;“”用于对序列进行质控和去重复。所有16S rRNA基因序列利用“”命令,而基因序列采用的是“”,但两者均按照相似度97% 进行操作分类单元(OTU)进行分类。
1.5 数据分析
所有数据采用Excel 2016、Origin 2016和SPSS 16.0 进行处理分析,处理之间的差异采用Duncan多级检验进行分析,<0.05 表示显著差异。SIP重层13C-DNA中活性好氧甲烷氧化菌的鉴定过程如下:将所得的甲烷氧化菌序列在NCBI 的GenBank数据库中进行Blast比对分析,从而获得高度相近的同源序列;然后将样品序列与数据库中的同源序列一同采用MEGA软件[21](7.0)进行分析,利用Neight-Joining 法分别建立16S rRNA和基因的系统发育树。
2 结果与分析
2.1 冻土活动层好氧甲烷氧化活性
采用5% 的初始甲烷浓度培养35 d之后,无论13CH4–标记或12CH4–对照处理,旱地土壤和间歇淹水土壤培养瓶中的甲烷几乎被完全消耗,表明两种土壤均有较强的好氧甲烷氧化潜力。同时,空气对照处理中,未有甲烷氧化过程,甲烷浓度维持在大气浓度水平,大约2 µl/L,表明高浓度甲烷培养刺激了微生物氧化过程。根据甲烷浓度随时间的降低趋势,发现好氧甲烷氧化速率具有明显差异,如图1A所示,旱地土壤的甲烷氧化速率显著高于间歇性淹水土壤,分别为12.38和12.17 μg/(g·d)。然而,值得注意的是,淹水土壤微生物的甲烷同化量显著高于旱地土壤,土壤有机碳13C-atom从本底自然丰度1.08%,分别增加至1.99% 和1.64%(图1B),表明淹水土壤中好氧甲烷氧化菌的碳同化能力更强。同时,空气培养,或者5% 高浓度12CH4培养条件下,土壤有机碳的13C均与背景自然丰度1.08% 相同,表明标记处理中的土壤有机碳13C增加来自于13CH4氧化。
2.2 16S rRNA基因高通量测序好氧甲烷氧化菌丰度变化情况
如图1C所示,高通量测序土壤总DNA的微生物16S rRNA基因表明,土壤好氧甲烷氧化微生物随着甲烷氧化过程显著增加。旱地土壤甲烷氧化过程中,好氧甲烷氧化菌从0.10% 增加至6.71%,增幅高达67倍。而淹水土壤则从0.32% 增加至8.17%,增幅达25倍。两种土壤均检测到甲烷氧化菌的5个属,其中3个属于type I甲烷氧化菌,2个属于type II 甲烷氧化菌。旱地土壤中,与空气对照相比,5%13CH4培养后,type I和type II甲烷氧化菌的相对丰度从空气对照0.01% 和0.09%,分别增加到2.29% 和4.42%。而间歇淹水土壤中,则从0.01% 和0.31%,分别增加到2.42% 和5.75%。旱地土壤中,type I甲烷氧化菌和type II甲烷氧化菌的增长趋势相较于空气对照非常明显。例如,在从0.01% 增至2.09%,增幅高达209倍;则从0.08% 增至4.33%,增幅达54倍。而在间歇淹水土壤中,type I甲烷氧化菌则以增加最为显著,其相对丰度在空气对照组和13CH4–标记组中分别为0.004% 和2.39%,增幅高达733倍;type II甲烷氧化菌丰度分别为0.27% 和5.70%,增幅21倍;表明这些微生物在高浓度甲烷氧化过程中可能发挥了重要作用。
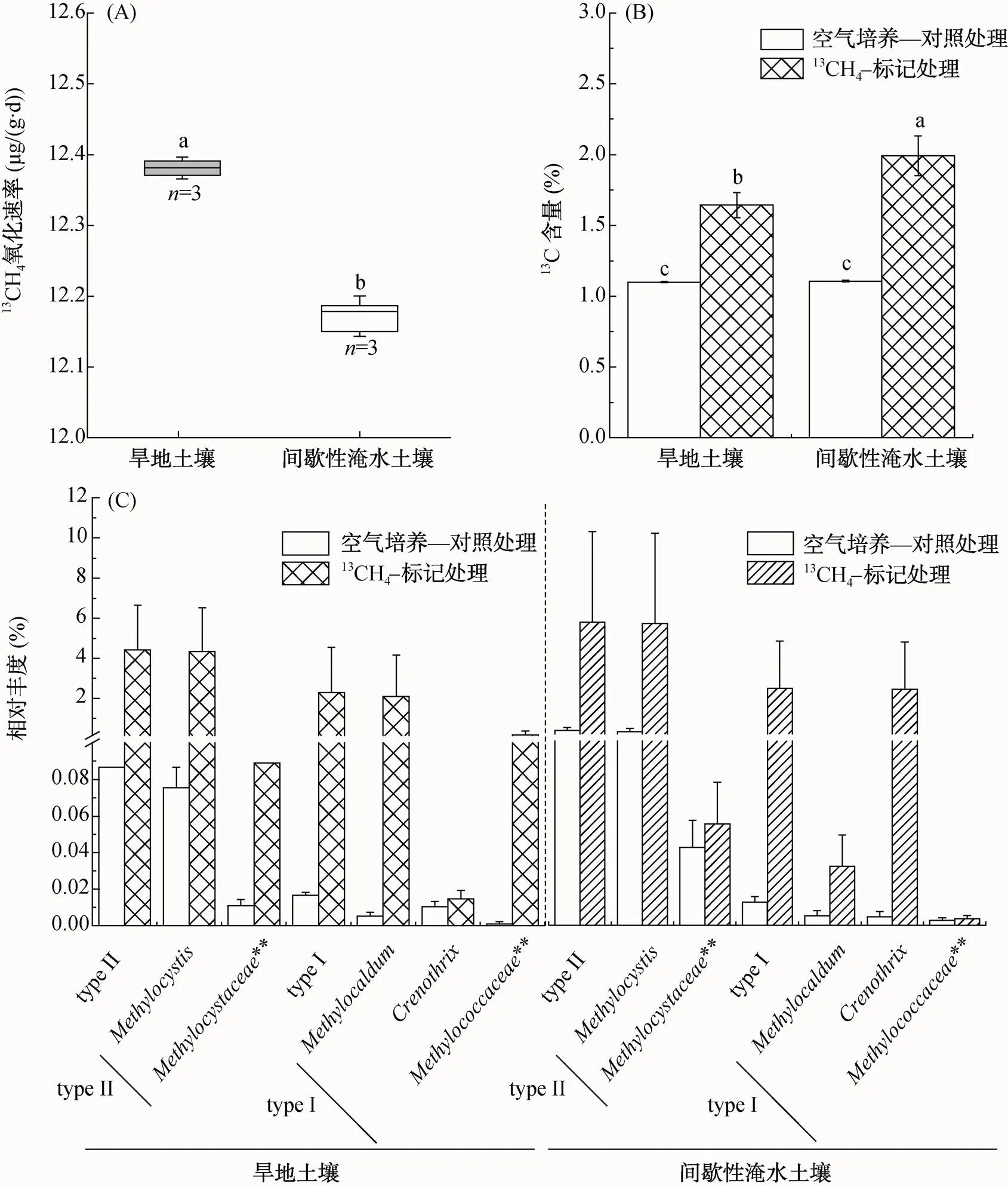
(图中小写字母不同表示处理间差异达P<0.05显著水平)
2.3 稳定性同位素示踪土壤活性好氧甲烷氧化菌类群
超高速密度梯度离心获得不同浮力密度的DNA后,对其中的甲烷氧化菌基因进行定量分析,13CH4–标记组的重浮力密度DNA中发现了大量基因扩增产物,而在相同位置的12CH4–对照组中并没有检测到明显的基因,表明甲烷氧化菌同化了大量的甲烷来源碳并合成了核酸DNA,在超高速密度梯度离心过程中,这些甲烷氧化菌的13C-DNA与没有标记的土壤DNA分离并集中在重浮力密度区域(图2A)。
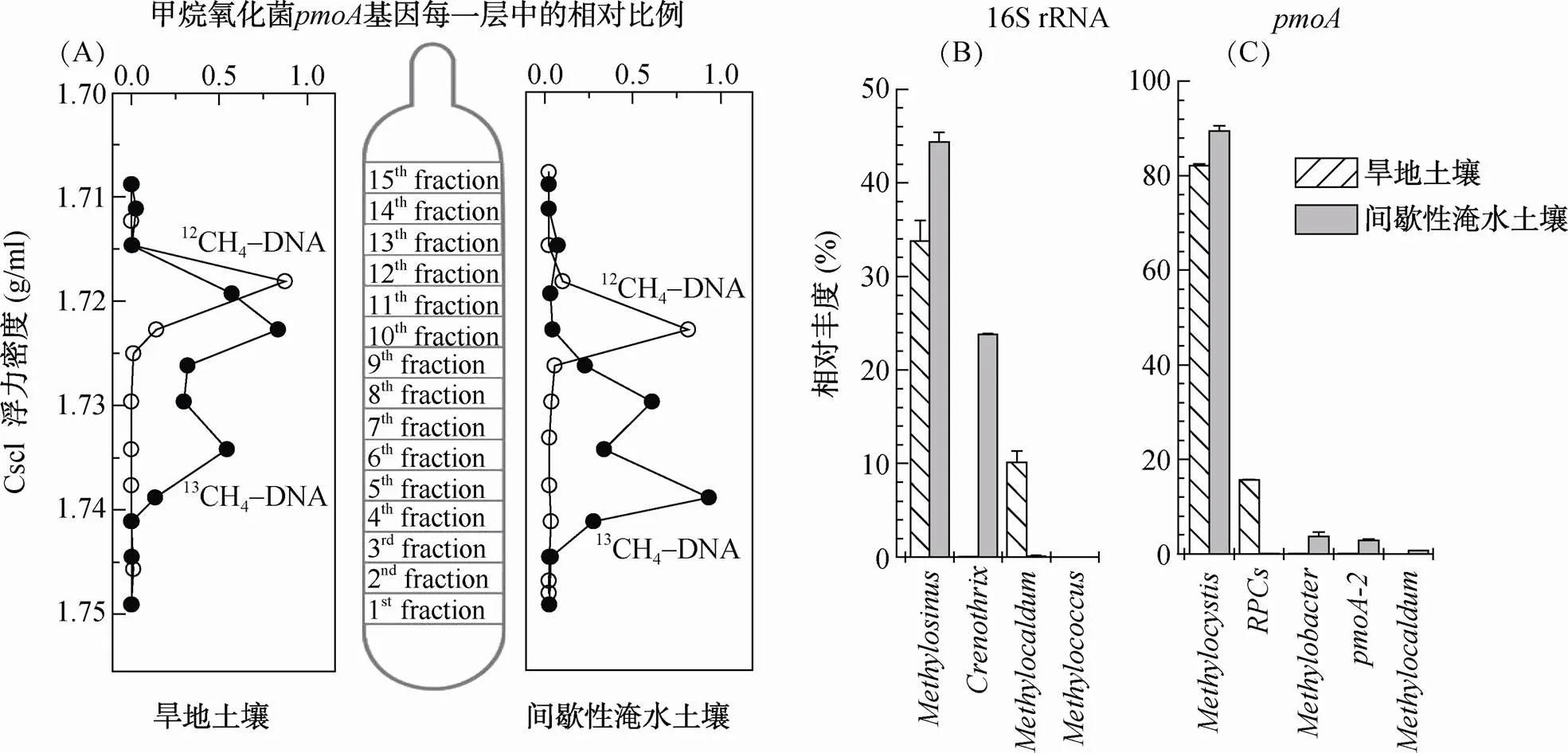
图2 甲烷氧化菌特异功能基因pmoA在不同浮力密度DNA中的相对比例(A),稳定性同位素13C-DNA重层16S rRNA(B)和pmoA(C)基因测序甲烷氧化菌的相对丰度
对13C-DNA进行16S rRNA基因高通量测序发现,甲烷氧化菌在13C-DNA中发生了高度富集,在旱地和间歇淹水土壤中的占比分别高达43.96% 和68.3%;其中type II为优势甲烷氧化菌,占比分别为33.76% 和44.38%(图2B)。然而,值得注意的是,数量上不占优势的type I甲烷氧化菌发生了明显的分异,例如,旱地土壤中的活性好氧甲烷氧化菌中type I甲烷氧化菌主要为(10.15%),而则是间歇淹水土壤中type I甲烷氧化菌的主要类群,占比达23.78%。
进一步针对13C-DNA进行基因高通量测序,如图2C所示,旱地和间歇淹水土壤中占据主导地位的活性甲烷氧化菌均为type II甲烷氧化菌,其丰度分别为82.09% 和89.39%。在旱地土壤中居次要地位的活性甲烷氧化菌为(15.61%)。因基因尚无普适性的通用引物,而本研究中的A189F/mb661R特异性较强,不能扩增新型甲烷氧化菌功能基因,导致在间歇淹水土壤基因结果中未能发现相关序列,使得居次要地位的活性甲烷氧化菌为(3.67%)和(2.82%)。
对13C-DNA进行16S rRNA和基因的系统发育树分析发现(图3),和在系统发育树上具有较高的同源性,均表明/是主导冻土活动层高浓度甲烷氧化过程的菌群。而旱地土壤的高浓度甲烷氧化过程中,16S rRNA基因和基因测序结果同样表明居于次要地位的和在系统发育树上具有较高的同源性。由此分析可知,对13C-DNA进行16S rRNA和基因高通量测序的结果具有一致性。
这些结果表明,高浓度甲烷氧化过程中,基于13C-16S rRNA基因的分析发现,旱地和间歇淹水性土壤中优势的活性好氧甲烷氧化菌皆为type II类型,但type I甲烷氧化菌发生明显演替。旱地和淹水土壤中type I型活性甲烷氧化菌分别为和。基于13C-基因的高通量测序也得到了类似结果,type II甲烷氧化菌为优势类群,但type I甲烷氧化菌发生了明显分异。
3 讨论
本研究发现长期不同水分影响条件下,type I活性甲烷好氧氧化菌群落发生明显演替并可能对土壤活性有机碳累积产生显著影响。利用13CH4稳定性核酸探针示踪技术,旱地和间歇淹水在5% 高浓度甲烷培养35 d后,土壤有机碳13C-atom含量均发生不同程度的显著增加,表明好氧甲烷氧化微生物利用了外源碳合成了自身细胞物质。尽管旱地土壤在培养期间的甲烷氧化速率略高,但是其有机碳同位素含量却低于间歇淹水土壤,表明前者的活性甲烷氧化菌碳同化效率低于后者。而在两种土壤中,通过高通量测序土壤总DNA的16S rRNA基因发现,显著增加的好氧甲烷氧化菌均为。大量的研究表明,大气甲烷氧化菌/属具有双甲烷单加氧酶系统,能够适应甲烷浓度剧烈变化的土壤环境,从而解释了其在旱地土壤和间歇淹水土壤中占据主导地位的现象。尽管在两种土壤的甲烷氧化过程中,type I甲烷氧化菌在相对丰度上仅占次要地位,但是,土壤水分对土壤甲烷好氧氧化过程中的type I甲烷氧化菌的影响非常显著。土壤水分的增加促使旱地土壤中的type I甲烷氧化菌(2.09%)逐渐被淹水土壤中的(2.39%)所取代。

图3 稳定性同位素13C-DNA重层中的甲烷氧化菌16S rRNA基因序列(A)和甲烷氧化菌pmoA基因(B)系统发育树
稳定性同位素13C-DNA的高通量分析也得到类似结果。土壤总DNA进行超高速密度梯度离心后,旱地土和间歇淹水土壤的13C标记处理重浮力密度中均出现了明显的基因信号峰,表明土壤中活性甲烷氧化菌利用13C-CH4合成了细胞物质。根据13C/12C的基因信号峰偏移程度可以发现间歇淹水土壤的碳同化程度要高于旱地土壤,该现象也与土壤培养后的有机碳13C-atom含量结果相吻合,可推测间歇淹水过程定向选择了碳同化能力较强的甲烷氧化菌类群。针对13C-DNA的16S rRNA基因高通量则为活性甲烷好氧氧化菌演替提供了有力证据,发现在间歇淹水土壤中的重要作用。已经有研究发现,在水生环境中是氧化甲烷的主要微生物[22]。经典的type I甲烷氧化菌生存策略倾向于-选择,type II甲烷氧化菌具有-选择策略;但不同于经典的type I好氧甲烷氧化菌,其生长缓慢,生理生态特征更类似于-选择策略,其碳同化途径更加高效,使其更容易适应多水缺氧的环境胁迫[10]。由此可推测,的存在很有可能就是导致间歇性淹水土壤碳同化能力高于旱地土壤的原因。值得注意的是,由于引物偏好性,高通量测序13C-DNA中的pmoA基因未能发现相关序列。尽管如此,在间歇淹水土壤中居次要地位的活性甲烷氧化菌为和,与在旱地土壤中居次要地位的/也具有明显差异。
格陵兰岛被认为是全球气候变化最为敏感的区域之一,一直以来得到了科学家和世界各国政府的高度重视。我们早期的研究发现在原位环境下南极区域冻土中土壤含水饱和度对大气浓度甲烷氧化速率以及甲烷氧化菌群落结构具有重要影响[23],而本研究通过对北极原位冻土进行微宇宙培养试验,深入分析不同水分状况的土壤在氧化高浓度甲烷的过程中占据主导地位的甲烷氧化菌类型,揭示了水分作为主要环境驱动因素在甲烷氧化菌群落演替中发挥的作用,为全面理解格陵兰岛在气候变化下土壤微生物生态的响应机制提供数据支撑。
4 结论
采用稳定性同位素技术示踪土壤甲烷氧化过程,以甲烷氧化菌特有的功能基因和16S rRNA基因为研究对象,结合高通量测序技术,准确鉴别了处于长期不同水分环境的土壤好氧甲烷氧化活性的微生物类群。在微生物分类属水平上,占据次要地位的type I甲烷氧化菌群落在土壤水分增加的刺激下发生了从到的演替过程,从而导致间歇淹水土壤同化甲烷碳的能力增强。但是由于技术手段限制,在基因高通量测序结果中并没有发现相关序列,未来仍需发展适合的扩增引物,从而进入深入研究其在甲烷氧化过程中发挥的重要作用。
[1] Spahni R, Chappellaz J, Stocker T F, et al. Atmospheric methane and nitrous oxide of the late pleistocene from Antarctic Ice Cores[J]. Science, 2005, 310(5752): 1317–1321.
[2] IPCC Climate Change. The physical science basis[M]. Cambridge: Cambridge University Press, 2007.
[3] IPCC Climate Change. The physical science basis[M]. Cambridge: Cambridge University Press, 2013.
[4] 马静, 徐华, 蔡祖聪. 施肥对稻田甲烷排放的影响[J]. 土壤, 2010, 42(2): 153–163.
[5] Roslev P, King G M. Aerobic and anaerobic starvation metabolism in methanotrophic bacteria[J]. Applied Environmental Microbiology, 1995, 61: 1563–1570.
[6] Le Mer J, Roger P. Production, oxidation, emission and consumption of methane by soils: A review[J]. European Journal of Soil Biology, 2001, 37: 25–50.
[7] Kirschke S, Bousquet P, Ciais P, et al. Three decades of global methane sources and sinks[J]. Nature Geoscience, 2013, 6: 813–823.
[8] Hanson R S, Hanson T E. Methanotrophic bacteria[J]. Microbiology Reviews, 1996, 60: 439–471.
[9] Amaral J A, Knowles R. Growth of methanotrophs in methane and oxygen counter gradients[J]. FEMS Microbiology Letters, 1995, 126: 215–220.
[10] Graham D W, Chaudary J A, Hanson R S, et al. Factors affecting competition between type I and type II methanotrophs in continuous-flow reactors[J]. Microbial Ecology, 1993, 25: 1–17.
[11] Liebner S, Wagner D. Abundance, distribution and potential activity of methane oxidizing bacteria in permafrost soils from the Lena Delta, Siberia[J]. Environmental Microbiology, 2007, 9: 107–117.
[12] Wagner D, Liebner S. Global warming and carbon dynamics in permafrost soils: Methane production and oxidation[J]. Permafrost Soils, Soil Biology, 2009, 16: 219–236.
[13] Elbering B, Tamstorf M P, Michelsen A, et al. Soil and plant community-characteristics and dynamics at Zeckenberg[J]. Advances in Ecological Research, 2008, 40: 223–248.
[14] Radajewski S, Ineson P, Parekh N R, et al. Stable-isotope probing as a tool in microbial ecology[J]. Nature, 2000, 403(6770): 646–649.
[15] Manefield M, Whiteley A S, Griffiths R I, et al. RNA stable isotope probing, a novel means of linking microbial community function to phylogeny[J]. Applied and Environmental Microbiology, 2002, 68(11): 5367–5373.
[16] 贾仲君. 稳定性同位素核酸探针技术DNA-SIP原理与应用[J]. 微生物学报, 2011, 51(12): 1585–1594.
[17] 郑燕, 贾仲君. 基于核酸 DNA/RNA 同位素示踪技术的水稻土甲烷氧化微生物研究[J]. 土壤学报, 2016, 53(2): 490–501.
[18] 郑燕, 贾仲君. 新一代高通量测序与稳定性同位素示踪DNA/RNA 技术研究稻田红壤甲烷氧化的微生物过程[J]. 微生物学报, 2013, 53(2): 173–184.
[19] Stubner S. Enumeration of 16S rDNA of desulfotomaculum lineage 1 in rice field soil by real-time PCR with SybrGreen TM detection[J]. Journal of Microbiological Methods, 2002, 50(2): 155–164.
[20] Caporaso J G, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335–336.
[21] Kumar S, Stecher G, Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870–1874.
[22] Oswald K, Jon G S, Sten L, et al. Crenothrix are major methane consumers in stratified lakes[J]. The ISME Journal, 2017, 11: 2124–2140.
[23] Wang P, D`Imperio L, Liu B, et al. Sea animal activity controls CO2, CH4and N2O emission hotspots on south Georgia, sub-Antarctica[J]. Soil Biology and Biochemistry, 2019, 132: 174–186.
The Emergence of Novel Methane Oxidizers in Greenland Permafrost Soil Under Periodically Water Saturated Conditions
LIU Bei1,2, BO Elberling3, JIA Zhongjun2*
(1 Sino-Danish Center for Education and Research, University of Chinese Academy of Sciences, Beijing 100190, China; 2 State Key Laboratory ofSoil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China; 3Center for Permafrost (CENPERM), Department of Geosciences and Natural Resource Management, University of Copenhagen, Copenhagen K 1350, Denmark)
This study investigated the impact of soil moisture on methane oxidation potential and the species of active methanotrophs in two contrasting soil types: permanent aerobic upland and periodically water saturated land in Zackenberg, Greenland. Each soil sample was incubated in a 120 ml bottles with 5%13C-methane or with air in case of the control treatment. Stable isotope probing was used to label the active methanotrophs, while high throughput sequencing of the 16S rRNA andgenes was performed to analyze how the active methanotrophs change in abundance and composition in response to the different soil moisture conditions. Both soil samples showed high activity of aerobic methane oxidation, but the periodically water saturated land developed the higher potential of carbon assimilation. The difference between the species with subprime status in the two types of soil samples could explain the variance of soil methane oxidation and carbon assimilation. The results indicated that the periodical water saturation promote methane oxidation by altering the composition of the subprime active methanotrophs (and) rather than the dominant ones (/) in permafrost soil.
Aerobic methanotrophs; Active layer; DNA-Stable isotope probing; Permafrost; Water moisture
Q938
A
10.13758/j.cnki.tr.2020.01.013
刘蓓, Bo Elberling, 贾仲君. 不同水分条件下格陵兰岛冻土活性甲烷氧化菌群落分异规律. 土壤, 2020, 52(1): 90–96.
国家自然科学基金委重点项目(41530857, 91751204)资助。
刘蓓(1997—),女,江西吉安人,硕士研究生,主要从事土壤微生物研究。E-mail: bei.liu@thuenen.de

