北极王湾夏季海水中DMSP降解基因的丰度及分布调查
刘会军 曾胤新 陆志波 俞勇
研究论文
北极王湾夏季海水中DMSP降解基因的丰度及分布调查
刘会军1曾胤新2陆志波1俞勇2
(1同济大学环境科学与工程学院, 上海 200092;2自然资源部中国极地研究中心, 上海 200136)
作为海洋中最丰富的有机含硫化合物之一, 二甲基巯基丙酸内盐(DMSP)不仅在浮游植物细胞内具有重要的生理生化及生态功能, 同时也是海洋微生物的重要营养物, 其分解产物二甲基硫(DMS)还是海洋中最重要的还原态挥发性生源有机硫化物, 因此DMSP的代谢在全球硫循环中扮演着重要角色。细菌可通过脱甲基与裂解这两条途径降解海水中的DMSP, 从而决定硫元素是以含硫蛋白形式进入微生物食物网还是以DMS形式进入大气。截至目前, 在高纬度北极海域开展的有关细菌降解DMSP的相关研究报道很少。本文采用荧光定量PCR技术, 首次对北极王湾夏季(2015及2016年)海水中涉及细菌降解DMSP的两条主要代谢途径中的脱甲基酶基因及裂解酶基因的丰度及分布进行了检测。结果显示, 2015—2016年海水中DMSP降解酶基因(、)与细菌16S rRNA基因的丰度比值的平均值分别为0.25%±0.31%、0.32%±0.58%。2015年水样中, 除K5站位的基因之外, 表层水中及的丰度相对值从湾口至湾内均呈递增趋势。但2016年的结果却显示出很大差异: 湾口附近基因丰度相对值总体高于湾内; 表层水中的基因丰度相对值变化趋势不明显, 而深层水中基因丰度相对值从湾口到湾内总体呈递减趋势。本次研究的结果初步表明, 含有DMSP降解基因的浮游细菌在王湾夏季水体中丰度很低, 而且参与不同代谢途径的DMSP降解菌在海水中的时空分布存在很大变化。对于王湾水域中细菌参与DMSP分解代谢及其在当地硫元素循环中的生态地位的进一步认识, 尚待后续研究工作的深入开展。
荧光定量PCR技术 北极王湾基因基因
0 引言
二甲基巯基丙酸内盐(DMSP)为海洋生态系统中普遍存在的一种有机硫化合物, 每年由海洋浮游植物产生的DMSP超过10亿吨[1]。Challenger等[2]在1948年最先从海洋红藻中分离出DMSP, 其降解产生的丙烯酸能够起到威慑捕食者的作用, 从而保护浮游植物免受原生动物跟浮游动物的伤害。DMSP在藻类细胞中含量较高, 具有渗透调节剂、抗冷冻保护剂及抗氧化剂的功能[3], 初步估计海洋中有多达10%的初级生产者可以合成DMSP[4]。DMSP也是海洋细菌重要的碳、硫来源。
微生物对海水中的DMSP的降解主要有两条途径: 一条途径是通过脱甲基化反应, 生成甲硫醇(MeSH), 最后以含硫蛋白形式进入微生物食物网[5-6], 约有70%的DMSP通过这种途径被降解[7]; 另一途径则是在裂解酶的作用下生成丙烯酸盐(或3-羟基丙酸辅酶A)和二甲基硫(DMS)[8]。每年释放的DMS多达3亿吨, 其中约10%来自于海水, 构成了大气中硫的最大自然排放源[9-10]。进入大气的DMS可经过一系列反应, 实现硫元素的海陆循环[11-12]。据统计, 约有三分之一的表层海洋细菌具有DMSP脱甲基酶及其同系物, 涉及脱甲基通路中第一步的脱甲基酶基因最先在玫瑰杆菌属中被发现, 随后经研究表明该基因广泛存在于海洋细菌中[4,13]。在海洋细菌中发现的DMSP裂解酶, 大部分也是来源于玫瑰杆菌及其近缘菌[14-15]。目前在细菌中已发现了7种DMSP裂解酶基因, 分别为、、、、、和[10,16,17]。全球海洋采样考察(GOS)调查发现,和基因是海洋宏基因组中含量最丰富的DMSP裂解酶基因[18-19]。DMSP脱甲基酶基因大量存在于表层海水中, 而DMSP裂解酶基因、和在表层海水中丰度较低[4,15,20]。目前关于DMSP降解菌的研究工作多集中在中低纬度海域, 而高纬度地区的相关研究较少。已有研究表明, 北极王湾海水中存在、和基因, 而且均与玫瑰杆菌支系有关[12]。
王湾(Kongsfjorden)地处高纬(12ºE, 79ºN), 位于北极斯瓦尔巴群岛(Svalbard)西北侧, 长26 km, 宽6—14 km, 内湾相对较浅, 水深不足100 m。该峡湾不但受北大西洋暖水团和北极冷水团的影响, 还受到了陆地冰川融水的终年干扰[21]。由于王湾地理位置独特, 已成为国际上重要的环境研究监测站点。目前全球变暖趋势明显, 王湾地区冰川融化和大西洋水比重的增加等导致了该地区水温盐度不断发生变化, 这对栖息于该地区的生物群落造成了一定影响[22]。
为了解王湾水体中DMSP降解菌的丰度及分布情况, 本研究首次采用荧光定量PCR(QPCR)方法, 对2015年和2016年夏季水体中的基因和基因的相对丰度进行了调查。结果初步表明, 王湾水体中存在参与DMSP脱甲基途径及裂解途径的海洋细菌, 但与中低纬度海域相比, 相关功能细菌的丰度极低, 并且存在较大的时空差异。
1 材料与方法
1.1 菌株
参考菌株DSS-3购自德国微生物与细胞保藏中心(DSMZ)。
1.2 样品采集
分别于2015年和2016年黄河站度夏期间, 沿王湾湾口(K1站)至湾内(K5站)采集表层及深层水样(图1)。其中2015年样品来自K1、K3、K5这3个站位, 2016年样品来自K1、K2、K3、K4、K5这5个站位。将500 mL海水样品中的微生物收集到0.2 μm孔隙的滤膜上。滤膜放入2 mL无菌离心管中, 加入1.5 mL裂解缓冲液后, 置–80°C储存, 运回国内实验室做进一步分析。
1.3 环境参数的测定
温度、盐度、含氧量、荧光值、密度等数据由现场采样时配备的SAIV CTD SD-204获取; 使用Whatman GF/F膜(47 mm)过滤2 L海水, 然后将滤膜用铝箔包裹避光并于–80°C冷冻保存, 携带回国后采用Turner 10-AU荧光计测定Chl; 同时取60 mL过滤后的海水冷冻保存, 携带回国进行营养盐分析, 其中NO2-N, PO4-P采用SkalarSan++营养盐自动分析仪测定[23]。
1.4 样品DNA提取
将含有菌体的滤膜放在无菌培养皿中, 参考董培艳[24]的方法提取样品DNA。
1.5 荧光定量PCR(QPCR)
本次研究采用绝对定量方法, 利用已知标准曲线推算待测样品中目标基因的含量[25]。分别以基因表征参与DMSP脱甲基途径的细菌、基因表征参与DMSP裂解途径的细菌、16S rRNA基因表征总细菌; 同时, 分别计算DMSP降解基因(或)拷贝数与16S rRNA基因拷贝数的比值, 得到两种DMSP降解基因在水体中的相对丰度值[26-28]。
1.5.1 标准品制备
参考菌株DSS-3含有及基因[16,29], 因此可作为阳性对照用于标准曲线的制备。课题组采用DNA提取试剂盒(OMEGA公司)提取菌株DSS-3的 DNA作为模板, 并分别采用引物对27F和1492R[26]、dmdA160F和591R[16]、dddP_277F和dddP_1112R[30]扩增该菌的16S rRNA基因(1498 bp)、基因(459 bp)以及基因(836 bp)。相应目标基因的片段经割胶回收纯化、连接至pMD18-T载体、转化至DH5 α感受态细胞中, 最后涂布于含氨苄青霉素的LB平板上, 并对其中的阳性克隆进行测序验证。经测序验证为目标基因后, 采用质粒提取试剂盒(天根生化科技有限公司)分别提取含16S rRNA基因、基因、基因的质粒, 用于标准曲线制备。

图1 北极王湾采样点
Fig.1. Locations of sampling sites in Kongsfjorden, Arctic
1.5.2 标准曲线制备及待测样品的目标基因检测
采用Invitrogen Qubit 3.0荧光定量仪测定含16S rRNA基因、基因、基因的质粒浓度, 然后根据公式[31-32](6.02×1023)×(质粒浓度ng·μL–1×10–9)/(DNA length×660)分别计算出含16S rRNA基因、基因、基因的质粒拷贝数: 6.31×109copy·μL–1、3.53×1010copy·μL–1、3.9×109copy·μL–1。将含有目的基因的DNA液分别做10倍系列稀释(每种基因均做5个稀释梯度)后, 用于后续目的基因标准曲线的制作。每次测试时均做2个平行样品。
针对16S rRNA、和三种基因, 分别选用引物BACT1369F和PROK1492R[33]、dmdA160F和dmdA323R(5’-ACCGGATCRTTCA GCAT-3’)、dddP982F(5’-CGGCACGCGCATGAGC ATAT-3’)和dddP_1112R, 采用实时荧光定量 PCR 仪(美国ABI公司7500型)进行QPCR实验反应体系为20 μL, 包括10μL 2× SYBR premix Ex Taq, 0.4μL 50× ROX reference dye, 各为0.4μL、10 mM的引物(0.2 mM), 6.8 μL无菌水, 以及2 μL模板DNA。
16S rRNA基因QPCR反应程序: 95°C预变性5 min; (95°C, 30 s→50°C, 30 s→72°C, 30 s)× 40 个循环; 95°C延伸1 min; 50°C, 1 min。基因QPCR 的复性温度为62°C,基因QPCR的复性温度为55°C, 其余反应条件同16S rRNA基因。
反应结束后, 以阳性模板拷贝数的自然对数为横坐标、以PCR扩增反应过程中的荧光信号Ct值为纵坐标, 分别绘制16S rRNA基因、基因及基因丰度的标准曲线。待测样品中目标基因的拷贝数量可根据相应的标准曲线读取出来。
1.6 数据分析
使用OriginPro 8.5软件[34]制作基因丰度的柱状图, 以不同采样点作为横坐标, 以/16S rRNA或/16S rRNA的基因拷贝数比值作为纵坐标。使用SPSS 22软件[35-36]对不同站位基因丰度值与环境参数之间的关系进行分析。所得皮尔森相关系数(Pearson product-moment correlation coefficient)的值介于1和–1之间, 1代表两个变量呈完全正相关, 0代表两者不相关, –1则代表两个变量呈完全负相关。
2 结果
2.1 2015年王湾DMSP降解基因的分布情况
2015年王湾K1、K3、K5站位样品中的16S rRNA基因、基因和基因的QPCR标准曲线的2值分别为0.995、0.998和0.993。结果显示(表1), 浮游DMSP降解基因在王湾水体中的丰度相对值极低。DMSP脱甲基酶基因的平均丰度相对值为0.46%±0.41%, 裂解酶基因的平均丰度相对值为0.83%±0.82%(未包括样品K5A)。让人意外的是, 站位K5的表层水体(K5A)中基因与16S rRNA 基因的拷贝数比值超过1。
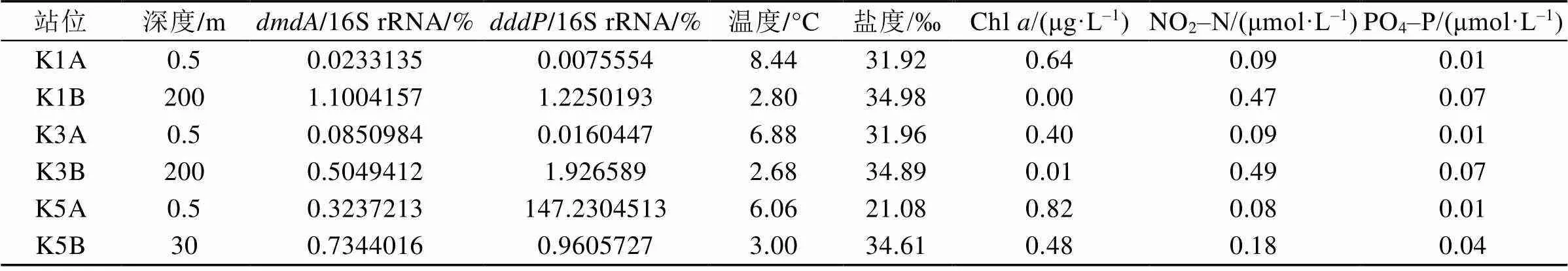
表1 2015年北极王湾夏季海水中DMSP降解基因丰度相对值及环境参数
K1A、K3A、K5A为表层水体; K1B、K3B、K5B为深层水体
从DMSP降解基因的分布来看(图2), 表层海水中的DMSP脱甲基酶基因和裂解酶基因从湾外(K1站)到湾内(K5站)的丰度相对值都呈递增趋势, 而深层海水无明显变化趋势。此外, 在各个采样点, 深层海水中DMSP降解基因的丰度相对值普遍高于表层海水, 而且的丰度相对值又高于(K1A和K3A除外)。

图2 2015年王湾夏季海水中DMSP降解基因dmdA与dddP的分布
Fig.2. Geographical distribution ofandof DMSP degrading genes in the seawater from Kongsfjorden in the summer of 2015
基于SPSS 22软件得到的相关性分析结果(表2)显示, 表层水体中基因丰度相对值与海水盐度、NO2-N有显著的负相关关系(=–1.000,< 0.01)。
2.2 2016年王湾DMSP降解基因的分布情况
2016年王湾5个站位样品中的16S rRNA基因、基因和基因QPCR标准曲线的2值依次为0.981、0.995和0.984。结果(表3)显示, DMSP脱甲基酶基因的平均丰度相对值为0.12%±0.13%, 裂解酶基因为0.07%±0.11%, 两种基因丰度的数量级与2015年结果相似, 表明王湾水域这两类DMSP降解基因含量极少。
2016年北极王湾夏季海水中, 从湾外到湾内(K3站位例外)表层海水和深层海水中的基因整体上都呈递减趋势(图3)。与2015年相似, 2016年夏季各站点深层海水中DMSP降解基因的丰度相对值普遍高于表层海水。不同的是,基因的丰度相对值大多高于。但在湾口K1站位深层海水(样品K1B)中, 检测到异常高的基因丰度相对值。
相关性分析结果(表4)显示, 在深层水体中的基因丰度相对值与海水温度(=–0.966,<0.01)、荧光值(=–0.962,<0.01)呈显著负相关, 与海水盐度(=0.911,<0.05)、密度(=0.929,< 0.05)呈显著正相关。
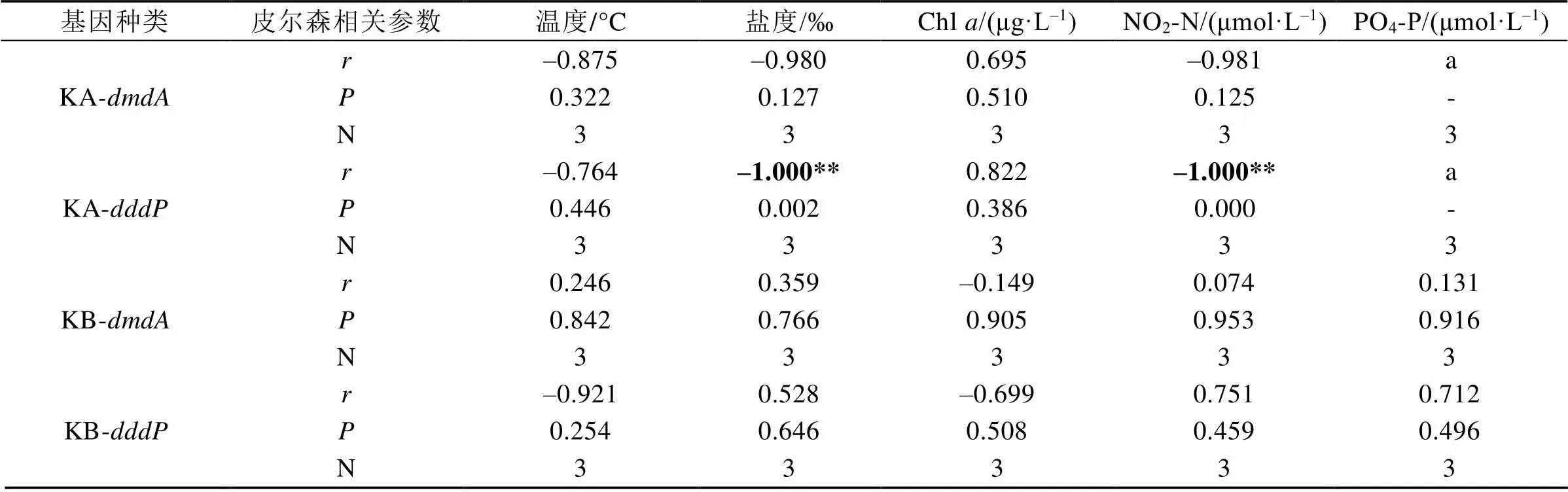
表2 2015年北极王湾夏季水样中dmdA、dddP基因与环境参数的相关性
KA: 表层水体; KB: 深层水体; r: 皮尔森(Pearson) 相关系数; P: 显著性(双尾); N: 样本个数; *表示相关性在0.05层上显著(双尾); **表示相关性在0.01层上显著(双尾); a: 无法计算, 因为至少其中一个变数为常数; -: 无数值
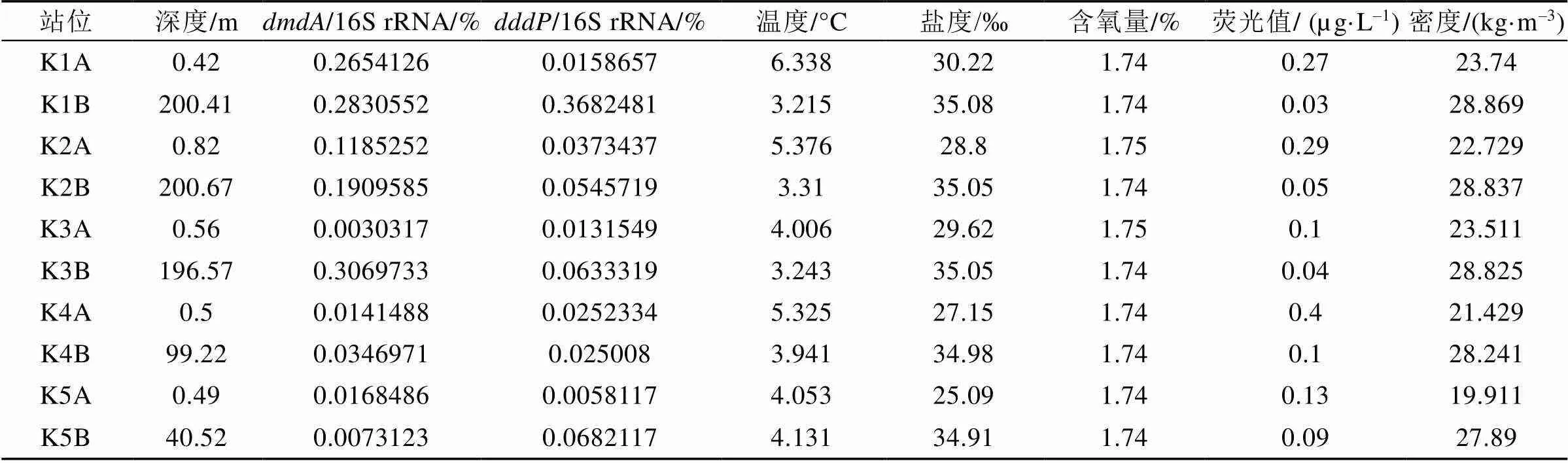
表3 2016年北极王湾夏季海水中DMSP降解基因丰度相对值及环境参数
K1A、K2A、K3A、K4A、K5A为表层水体; K1B、K2B、K3B、K4B、K5B为深层水体
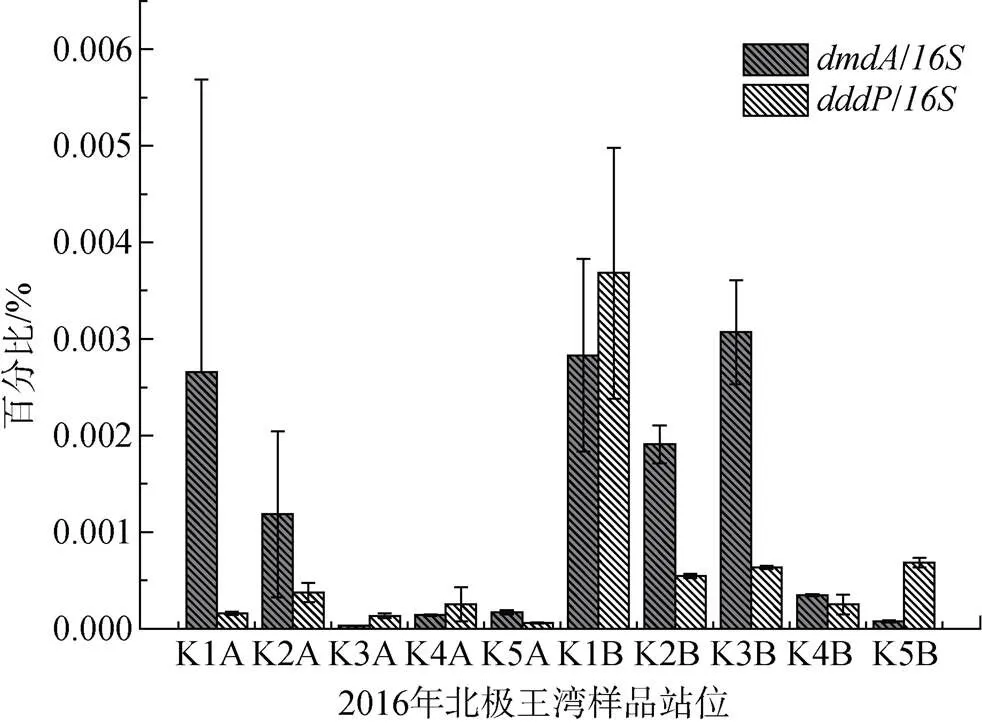
图3 2016年王湾夏季海水中DMSP降解基因dmdA与dddP的分布
Fig.3. Geographical distribution ofandof DMSP degrading genes in the seawater from Kongsfjorden in the summer of 2016
3 讨论
在王湾夏季水样中, 无论是基因还是基因, 总体而言深层水体中的丰度高于表层水体。这可能反映了陆地淡水输入对水体中DMSP降解细菌的影响。夏季期间王湾表层水体受冰川融水等陆源性淡水输入的影响明显。淡水的输入可以导致表层海水中浮游微生物群落结构及其生态功能发生变化, 例如, 带来陆源性微生物、影响海洋微生物生理代谢活动、导致湾内浮游动植物减少等[37]。而浮游植物与微生物关系密切, 如浮游植物能提供细菌所需的碳源等营养物质, 而细菌可为浮游植物提供维生素。相对于表层水体, 王湾深层水体主要是受到外来洋流的影响(靠近陆地冰川的K5站位除外), 大西洋变异水会对200 m以深的水柱造成重要影响[38]。

表4 2016年北极王湾夏季水样中dmdA、dddP基因与环境参数的相关性
KA: 表层水体; KB: 深层水体; r: 皮尔森(Pearson)相关系数; P: 显著性(双尾); N: 样本个数; *表示相关性在0.05层上显著(双尾); **表示相关性在0.01层上显著(双尾); c: 无法计算, 因为至少其中一个变数为常数; -: 无数值
在2015年夏季湾内K5站位的表层水体中,基因的丰度相对值异常高。在不考虑采样因素外(2015年样品为他人代采), 课题组推测可能原因包括: 本次研究测定的16S rRNA基因只涵盖细菌等原核生物, 而基因在真核生物中有发现, 如子囊菌[14,39-41]。因此QPCR所检测到的基因有可能部分来自真核生物。此外,基因可能在真菌中的拷贝数较多。同时, K5站位靠近陆地冰川, 夏季冰川融水的输入会给该站点的环境带来显著影响, 进而又影响到浮游植物及浮游细菌的生长代谢等一系列生理生态状况。该异常原因尚待进一步研究工作的开展。
比较2015年与2016年的结果, 可以看出王湾夏季海水中DMSP降解基因的分布具有较大的时空差异: 2015年表层海水中DMSP脱甲基酶基因从湾外到湾内, 其丰度呈递增趋势, 而在2016年却呈现递减趋势。在2015年样品中,的丰度普遍高于, 说明水体中DMSP的分解代谢是以裂解途径占优势, 而在2016年样品中基因的丰度普遍高于, 说明水体中DMSP的分解代谢是以脱甲基途径占优势。相关性分析结果显示, 不同年份、不同水深的样品中DMSP降解基因的丰度相对值与同种环境因子之间的相关性差异明显, 表明王湾夏季期间影响DMSP降解基因丰度及其分布的原因较为复杂。对中国东海DMSP降解菌的研究中表明[42],基因丰度与海水中的叶绿素含量呈正相关, 在本文中2015年北极王湾表层水样中基因丰度与叶绿素虽然呈正相关(=0.822), 但相关性并不显著。对2013年夏季东海和黄海水域的研究结果显示[43], 温度、盐度与DMSP降解菌丰度的相关性并不显著, 而本文中除2016年王湾深层水体中的基因与温度显著负相关外, 两年数据中的DMSP降解菌丰度与温度相关性均不显著, 结果较为相似。但与赵丽军[43]的盐度结果不同, 2015年王湾表层水体的基因与盐度显著负相关, 2016年深层水体的基因与盐度显著正相关。营养盐是影响海洋浮游植物DMSP合成的重要因素[44], 当营养盐充足时会促进浮游植物生长、加快代谢合成更多的DMSP。而焦念志等[45]的研究结果显示, 海水中DMSP和氮盐是两段式关系, 氮盐浓度低于阈值时两者为正相关, 高于阈值时两者为负相关, 该阈值因海区、浮游植物种类和季节等条件不同发生变化。本文2015年王湾表层水体中的基因与营养盐中的氮盐NO2-N呈显著负相关, 但由于缺乏海水DMSP数据, 因此尚不明确其具体关系。
在热带和亚热带等太平洋地区的表层海水中, DMSP降解基因和的相对丰度为33%±12%[26]。而在我国东海地区表层海水中,基因相对丰度为19.53%±6.70%, 底层海水中为16.00%±8.73%[42]。这些地区的DMSP降解基因丰度均远高于北极王湾。对2011年夏季样品中DMSP降解基因多样性的分析结果显示, 王湾中及基因均与来自玫瑰杆菌支系(clade)细菌的相关基因具有亲缘关系[16]。但基于高通量测序技术的浮游细菌群落组成分析表明, 2015年王湾水样中以亚硫酸杆菌属()为代表的玫瑰杆菌支系的相对丰度超过4%(数据未发表)。结合本次研究结果, 说明王湾海水中只有部分玫瑰杆菌支系细菌含有DMSP降解基因。不同海域中的细菌16S rRNA拷贝数的差异可能是原因之一。有相关研究发现, 表层海水中细菌平均含有1.8个拷贝数的16S rRNA, 在开放海域是1.3个拷贝数, 而在靠近海岸的地区则为2.8个拷贝数[46]。其次, 本次研究中所用扩增引物有别于文献中采用的DMSP降解基因扩增引物对, 有可能因为特异性太高导致覆盖范围不够广、遗漏掉了其他亦含有或基因的细菌[47]。同时, 本次研究的样品中DMSP降解基因丰度的确就是低也存在可能性。这些因素的叠加, 有可能就导致本次结果中DMSP降解基因的相对丰度远远低于温带、亚热带及热带海域的数据。具体原因还不明确, 尚待更多实验的开展去探究。
本次研究通过QPCR方法首次对北极王湾夏季期间海水中DMSP降解基因和的相对丰度及分布进行了研究, 为认识北极高纬度海域中细菌参与海洋DMSP分解代谢及硫的生物地化循环提供了初步结果。在后续的研究工作中, 还需要考虑如下因素, 以便能对王湾海域细菌介导DMSP/DMS代谢并参与硫元素生物地化循环过程有更深入的认识: (1)包括海水DMSP/DMS浓度在内的更多环境参数的获取; (2)浮游植物、动物以及微生物的群落组成及相互关联; (3)具有DMSP代谢能力的模式菌株的获取与研究。
致谢感谢自然资源部第二海洋研究所季仲强高级工程师提供王湾海水化学参数。感谢自然资源部中国极地研究中心曹叔楠博士采集2015年夏季水样。
1 JOHNSTON A W, GREEN R T, TODD J D. Enzymatic breakage of dimethylsulfoniopropionate: a signature molecule for life at sea[J]. Current Opinion in Chemical Biology, 2016, 31: 58-65.
2 CHALLENGER F, SIMPSON M I. A precursor of the dimethyl sulphide evolved by Polysiphonia fastigiata; dimethyl-beta-propiothetine (dimethyl-beta-carboxythylsulphonium hydroxide) and its salts[J]. The Biochemical Journal, 1947, 41(3): xl.
3 BULLOCK H A, LUO H W, WHITMAN W B. Evolution of dimethylsulfoniopropionate metabolism in marine phytoplankton and bacteria[J]. Frontiers in Microbiology, 2017, 8: 637. doi:10.3389/fmicb.2017.00637.
4 HOWARD E C, HENRIKSEN J R, BUCHAN A, et al. Bacterial taxa that limit sulfur flux from the ocean[J]. Science, 2006, 314(5799): 649-652.
5 KIENE R P, LINN L J, GONZÁLEZ J, et al. Dimethylsulfoniopropionate and methanethiol are important precursors of methionine and protein-sulfur in marine bacterioplankton[J]. Applied and Environmental Microbiology, 1999, 65(10): 4549-4558.
6 SIMÓ R. Production of atmospheric sulfur by oceanic plankton: biogeochemical, ecological and evolutionary links[J]. Trends in Ecology & Evolution, 2001, 16(6): 287-294.
7 KIENE R P, LINN L J. The fate of Dissolved dimethylsulfoniopropionate (DMSP) in seawater: tracer studies using 35S-DMSP[J]. Geochimica et Cosmochimica Acta, 2000, 64(16): 2797-2810.
8 KIENE R P. Production of methanethiol from dimethylsulfoniopropionate in marine surface waters[J]. Marine Chemistry, 1996, 54(1/2): 69-83.
9 KIENE R P, LINN L J, BRUTON J A. New and important roles for DMSP in marine microbial communities[J]. Journal of Sea Research, 2000, 43(3/4): 209-224.
10 CURSON A R J, TODD J D, SULLIVAN M J, et al. Catabolism of dimethylsulphoniopropionate: microorganisms, enzymes and genes[J]. Nature Reviews Microbiology, 2011, 9(12): 849-859.
11 CHARLSON R J, LOVELOCK J E, ANDREAE M O, et al. Oceanic phytoplankton, atmospheric sulphur, cloud albedo and climate[J]. Nature, 1987, 326(6114): 655-661.
12 张晓华, 刘骥, 柳敬丽, 等. DMSP的生物合成与裂解及其在硫循环中的作用[J]. 中国科学基金, 2018, 32(5): 471-478.
13 FRADE P R, SCHWANINGER V, GLASL B, et al. Dimethylsulfoniopropionate in corals and its interrelations with bacterial assemblages in coral surface mucus[J]. Environmental Chemistry, 2016, 13(2): 252.
14 WANG P, CHEN X L, LI C Y, et al. Structural and molecular basis for the novel catalytic mechanism and evolution of DddP, an abundant peptidase-like bacterial Dimethylsulfoniopropionate lyase: a new enzyme from an old fold[J]. Molecular Microbiology, 2015, 98(2): 289-301.
15 HOWARD E C, SUN S L, BIERS E J, et al. Abundant and diverse bacteria involved in DMSP degradation in marine surface waters[J]. Environmental Microbiology, 2008, 10(9): 2397-2410.
16 ZENG Y X, QIAO Z Y, YU Y, et al. Diversity of bacterial dimethylsulfoniopropionate degradation genes in surface seawater of Arctic Kongsfjorden[J]. Scientific Reports, 2016, 6: 33031.
17 SUN J, TODD J D, THRASH J C, et al. The abundant marine bacterium Pelagibacter simultaneously catabolizes dimethylsulfoniopropionate to the gases dimethyl sulfide and methanethiol[J]. Nature Microbiology, 2016, 1(8): 16065.
18 RUSCH D B, HALPERN A L, SUTTON G, et al. The Sorcerer II Global Ocean Sampling Expedition: Northwest Atlantic through Eastern Tropical Pacific[J]. PLoS Biology, 2007, 5(3): e77.
19 TODD J D, CURSON A R J, KIRKWOOD M, et al. DddQ, a novel, cupin-containing, dimethylsulfoniopropionate lyase in marine roseobacters and in uncultured marine bacteria[J]. Environmental Microbiology, 2011, 13(2): 427-438.
20 TODD J D, CURSON A R J, DUPONT C L, et al. ThedddPgene, encoding a novel enzyme that converts dimethylsulfoniopropionate into dimethyl sulfide, is widespread in ocean metagenomes and marine bacteria and also occurs in some Ascomycete fungi[J]. Environmental Microbiology, 2009, 11(6): 1376-1385.
21 SVENDSEN H, BESZCZYNSKA-MØLLER A, HAGEN J O, et al. The physical environment of Kongsfjorden–Krossfjorden, an Arctic fjord system in Svalbard[J]. Polar Research, 2002, 21(1): 133-166.
22 BEUCHEL F, GULLIKSEN B, CARROLL M L. Long-term patterns of rocky bottom macrobenthic community structure in an Arctic fjord (Kongsfjorden, Svalbard) in relation to climate variability (1980–2003)[J]. Journal of Marine Systems, 2006, 63(1/2): 35-48.
23 高生泉, 陈建芳, 李宏亮, 等. 2008年夏季白令海营养盐的分布及其结构状况[J]. 海洋学报(中文版), 2011, 33(2): 157-165.
24 董培艳. 北极王湾海域浮游细菌及攻瑰杆菌支系丰度及多样性调查[D]. 厦门: 厦门大学, 2013.
25 安钢力. 实时荧光定量PCR技术的原理及其应用[J].中国现代教育装备, 2018, (21): 19-21.
26 CUI Y S, SUZUKI S, OMORI Y, et al. Abundance and distribution of dimethylsulfoniopropionate degradation genes and the corresponding bacterial community structure at dimethyl sulfide hot spots in the tropical and subtropical Pacific Ocean[J]. Applied and Environmental Microbiology, 2015, 81(12): 4184-4194.
27 赵传鹏, 浦跃朴, 尹立红, 等. 实时荧光定量PCR法检测环境假单胞菌属细菌丰度[J]. 东南大学学报(自然科学版), 2006, 36(1): 143-146.
28 MCCANN C M, CHRISTGEN B, ROBERTS J A, et al. Understanding drivers of antibiotic resistance genes in High Arctic soil ecosystems[J]. Environment International, 2019, 125: 497-504.
29 GONZALEZ J M. Silicibacter pomeroyi sp. nov. and Roseovarius nubinhibens sp. nov., dimethylsulfoniopropionate-demethylating bacteria from marine environments[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(5): 1261-1269.
30 PENG M J, XIE Q Y, HU H, et al. Phylogenetic diversity of thedddPgene for dimethylsulfoniopropionate-dependent dimethyl sulfide synthesis in mangrove soils[J]. Canadian Journal of Microbiology, 2012, 58(4): 523-530.
31 FEY A, EICHLER S, FLAVIER S, et al. Establishment of a real-time PCR-based approach for accurate quantification of bacterial RNA targets in water, using salmonella as a model organism[J]. Applied and Environmental Microbiology, 2004, 70(6): 3618-3623.
32 YAN W K, MA H M, SHI G T, et al. Independent shifts of abundant and rare bacterial populations across east Antarctica glacial foreland[J]. Frontiers in Microbiology, 2017, 8: 1534. doi:10.3389/fmicb.2017.01534.
33 SUZUKI M T, TAYLOR L T, DELONG E F. Quantitative analysis of small-subunit rRNA genes in mixed microbial populations via 5'-nuclease assays[J]. Applied and Environmental Microbiology, 2000, 66(11): 4605-4614.
34 孙逸敏. 利用SPSS软件分析变量间的相关性[J]. 新疆教育学院学报, 2007, 23(2): 120-123.
35 STEVENSON K J . Review of originpro 8.5[J]. J Am Chem Soc, 2015, 133(14): 5621-5621.
36 SRIDHAR K, CHARLES A L.antioxidant activity of Kyoho grape extracts in DPPH and ABTS assays: Estimation methods for EC50 using advanced statistical programs[J]. Food Chemistry, 2019, 275: 41-49.
37 HOP H, PEARSON T, HEGSETH E N, et al. The marine ecosystem of Kongsfjorden, Svalbard[J]. Polar Research, 2002, 21(1): 167-208.
38 季仲强, 高生泉, 金海燕, 等. 北极王湾2010年夏季水体营养盐分布及影响因素[J]. 海洋学报(中文版), 2014, 36(10): 80-89.
39 TODD J D, CURSON A R J, DUPONT C L, et al. ThedddPgene, encoding a novel enzyme that converts dimethylsulfoniopropionate into dimethyl sulfide, is widespread in ocean metagenomes and marine bacteria and also occurs in some Ascomycete fungi[J]. Environmental Microbiology, 2009, 11(6): 1376-1385.
40 KIRKWOOD M, TODD J D, RYPIEN K L, et al. The opportunistic coral pathogen Aspergillus sydowii contains dddP and makes dimethyl sulfide from dimethylsulfoniopropionate[J]. The ISME Journal, 2010, 4(1): 147-150.
41 CURSON A R J, SULLIVAN M J, TODD J D, et al. DddY, a periplasmic dimethylsulfoniopropionate lyase found in taxonomically diverse species of Proteobacteria[J]. The ISME Journal, 2011, 5(7): 1191-1200.
42 LIU J L, LIU J, ZHANG S H, et al. Novel insights into bacterial dimethylsulfoniopropionate catabolism in the East China sea[J]. Frontiers in Microbiology, 2018, 9: 3206. doi:10.3389/fmicb.2018.03206.
43 赵丽军. 东海和黄海海水中DMSP降解菌的分布、多样性和降解特性的研究[D]. 青岛: 中国海洋大学, 2015.
44 SUNDA W G, HARDISON R, KIENE R P, et al. The effect of nitrogen limitation on cellular DMSP and DMS release in marine phytoplankton: climate feedback implications[J]. Aquatic Sciences, 2007, 69(3): 341-351.
45 焦念志, 柳承璋, 陈念红. 东海二甲基硫丙酸的分布及其制约因素的初步研究[J]. 海洋与湖沼, 1999, 30(5): 525-531.
46 BIERS E J, SUN S, HOWARD E C. Prokaryotic genomes and diversity in surface ocean waters: interrogating the global ocean sampling metagenome[J]. Applied and Environmental Microbiology, 2009, 75(7): 2221-2229.
47 VARALJAY V A, HOWARD E C, SUN S, et al. Deep sequencing of a dimethylsulfoniopropionate-degrading gene (dmdA) by using PCR primer pairs designed on the basis of marine metagenomic data[J]. Applied and Environmental Microbiology, 2010, 76(2): 609-617.
ABUNDANCE AND DISTRIBUTION OF DIMETHYLSULFONIOPROPIONATE-DEGRADING GENES IN KONGSFJORDEN IN THE ARCTIC IN SUMMER
Liu Huijun1, Zeng Yinxin2, Lu Zhibo1, Yu Yong2
(1College of Environmental Science and Engineering, Tongji University, Shanghai 200092, China;2Ministry of Natural Resources, Polar Research Institute of China, Shanghai 200136, China)
As one of the most abundant organic sulfur compounds in the oceans, dimethylsulfoniopropionate (DMSP) has important physiological and ecological functions in phytoplankton cells and is also an important nutrient for marine microorganisms. Dimethyl sulfide (DMS) is produced during DMSP decomposition, and is also the most important source of volatile organic sulfide in a reduced state in the oceans. Therefore, the metabolism of DMSP plays an important role in the global sulfur cycle. Bacteria can degrade DMSP in seawater through demethylation and cleavage pathways, thus determining whether sulfur elements enter the microbial food web in the form of sulfur-containing proteins or enter the atmosphere as DMS. To date, there have been few reports on bacterial degradation of DMSP in high-latitude Arctic waters. Demethylase geneand lyase geneare involved in the two major pathways of DMSP degradation. We examined abundance and distribution of these two genes in the waters of Kongsfjorden in the Arctic in summer 2015 and 2016 using quantitative polymerase chain reaction (qPCR). Average abundance ratios of DMSP-degrading enzyme genes (and) to 16S rRNA genes in seawater were 0.25%±0.31%, 0.32%±0.58%, respectively. Measurements along transects from the outer to the inner fjord indicate that relative abundances ofandin surface waters were lower in the outer fjord and increased towards the inner fjord in 2015, with the exception of thegene at the K5 site. However, in 2016, there was no obvious trend of relative abundance ofgene in surface waters in the fjord; relative abundance ofgene in deep waters was higher in the outer fjord and decreased towards the inner fjord. Abundance of bacterioplankton containing DMSP degradation genes was very low in Kongsfjorden in summer. There were large spatial and temporal variations in the distribution of DMSP-degrading bacteria that are involved in different DMSP catabolism pathways. Further studies are needed to improve our understanding of bacterial DMSP degradation in Kongsfjorden and the ecological role of these bacteria in the local sulfur cycle.
quantitative fluorescence PCR, Arctic Kongsfjorden,,
2019年5月收到来稿, 2019年8月收到修改稿
国家自然科学基金(41476171)资助
刘会军, 女, 1994年生。硕士, 主要从事极地微生物学研究。E-mail: liuhuijun@tongji.edu.cn
俞勇, E-mail: yuyong@pric.org.cn
10. 研究论文13679/j.jdyj.20190028

