转基因检测中大豆蛋白DNA提取方法筛选及其lectin基因降解分析
杜 艳,陈复生*,陈 晨,刘 营
(河南工业大学粮油食品学院,河南 郑州 450001)
大豆(Glycine max (L.) Merr)通称黄豆,起源于中国,种植面积广泛,距今已有五千多年的栽培历史,是现今人们的主要食品之一。大豆蛋白中含有大量的必需氨基酸,不含胆固醇,具有非常高的营养价值[1],逐渐走向人们的生活饮食中。当前市售的大豆蛋白主要有大豆分离蛋白(soybean protein isolate,SPI)和大豆浓缩蛋白(soybean protein concentrate,SPC)[2]。
近年来,我国的大豆消耗量巨大,以至于国产大豆供不应求,无法满足人们及饲料行业日益增长的需要。因此,大量的大豆依赖进口。数据显示,2017ü2018年度中国大豆消费量为1.1亿 t,而自产量仅1 500万 t左右,大豆进口量高达9 700万 t,这其中大部分为来自美国和巴西的转基因大豆[3]。
转基因作物自出现以来,争议不断。为规范转基因作物在国际市场上的流通,世界各国纷纷建立转基因标识制度[4],我国农业部颁布的《农业转基因生物标识管理办法》要求“凡列入标识管理目录并用于销售的农业转基因生物,应进行标识”。然而转基因成分含量甚微且可能在产品加工过程中发生降解或损失,使得定性和定量检测难度增大。
为检测饲料和食品中的转基因成分,国家农业部发布第1485号公告[5],描述了适用于富含多糖的植物及其粗加工测试样品DNA提取和纯化的溴化十六烷基三甲铵(hexadecyl trimethyl ammonium bromide,CTAB)法,适用于植物深加工样品DNA提取和纯化的改良CTAB法,以及适用于蛋白含量较高的植物及其粗加工测试样品DNA提取和纯化的十二烷基硫酸钠(sodium dodecyl sulfate,SDS)等DNA提取方法。此外,GB/T 19495.3ü 2004《转基因产品检测 核酸提取纯化方法》[6]描述了用于DNA提取的酚-三氯甲烷法、聚乙烯吡咯烷酮法、CTAB法和硅土法。国家农业部2259号公告[7-8]和其他标准[9-14]均针对不同种类的转基因植物及其产品,提出了转基因成分定性聚合酶链式反应(polymerase chain reaction,PCR)、实时荧光定量PCR、数字PCR以及基因芯片等检测方法。
DNA的提取是转基因检测的关键步骤[15]。豆类食品成分复杂,常含有多糖、酚类、蛋白等PCR抑制剂,食品加工过程中的机械作用、热效应以及化学处理等也会对DNA的含量和完整性造成影响,为DNA提取带来难度[16]。而DNA模板的浓度和纯度决定着PCR的成败,从而对检测结果产生影响。DNA模板浓度或纯度过低,可能由于含量低于PCR检测限或PCR抑制剂的存在,降低PCR的灵敏度,造成假阴性结果。
研究人员对多种DNA提取方法进行了比较,并提出对于不同的食品基质应选用不同的提取方法。Kamiya等[17]表示SDS法有助于大豆及大豆粕中DNA的提取。Wang Xiumin等[18]分别采用CTAB法、SDS法和盐酸胍试剂盒对山东、江苏、广东、河北、广西五省的54 种大豆种子和大豆粕进行DNA提取,从成本、DNA质量浓度和纯度以及反应时间4 个层面进行分析,发现SDS法每次反应约消费0.13 美元,成本较CTAB法(0.24 美元)更低,DNA产量高(164~1 750 ng/mg),A260nm/A280nm在1.8~2.0之间,符合PCR要求,且提取时间适中,比另2 种方法更适用于大豆及豆粕中DNA提取。Turkec等[4]随机选取了47 种市售豆制品,研究CTAB法和Foodproof试剂盒、Dneasy Plant Mini Kit(Dneasy试剂盒)、Wizard Genomic DNA Purification Kit(Wizard试剂盒)、GeneSpin试剂盒的适用范围,并未发现某一种方法可通用于所有样品DNA的提取,大豆种子、饼干、巧克力、婴儿配方奶粉宜使用CTAB法,豆粉、豆芽、豆奶宜使用GeneSpin试剂盒,豆粕、豆酱、面包和肉宜使用Foodproof试剂盒。
此外,刘欣等[19]分别使用DuPont试剂盒、Bayer试剂盒、Qiagen试剂盒、Monsanto试剂盒和Tiangen试剂盒提取大豆籽粒DNA,结果表明Monsanto试剂盒和Bayer试剂盒均由于PCR抑制剂的存在而影响后续检测,Qiagen试剂盒、DuPont试剂盒与Tiangen试剂盒得到的DNA质量浓度高、纯度好,完整度高,为大豆转基因检测中DNA提取的适宜方法。喻志学等[20]对比了CTAB法和改良SDS法对转基因和非转基因大豆DNA的提取效果,认为SDS法更优。然而,大豆蛋白作为一种重要的动物饲料和食品添加剂[21],针对其DNA提取方法的对比讨论相对较少。
因此,本研究选择DNA提取常用的Macherey-Nagel Nucleo Spin Kit(Nucleo试剂盒)、Wizard试剂盒、Dneasy试剂盒、Ezup柱式植物基因组DNA抽提试剂盒(Ezup试剂盒)、Dzup基因组DNA抽提试剂盒(Dzup试剂盒)和SDS法6 种方法,对比其对7 种中国市售大豆蛋白(4 种SPI和3 种SPC)和大豆籽粒中DNA的提取效果,旨在提高转基因大豆及其蛋白制品检测的灵敏度和准确性,为转基因检测方法的健全和转基因标识制度的完善奠定基础。最后,使用优选出的方法提取市售SPI和SPC中的DNA,并对其中大豆内源lectin基因不同长度的片段进行扩增,探究大豆蛋白中lectin基因的降解情况。
1 材料与方法
1.1 材料与试剂
4 种市售SPI分别来自广东(SPI 1)、四川(SPI 2)、陕西(SPI 3)和临沂(SPI 4),3 种SPC分别来自上海(SPC 1)、四川(SPC 2)和河南(SPC 3),3 种大豆籽粒分别来自东北(大豆籽粒1)、河南(大豆籽粒2)和巴西(大豆籽粒3)。
Wizard试剂盒(目录号:A1120) 美国Promega公司;Nucleo试剂盒(目录号:740945) 德国Macherey-Nagel公司;Dneasy试剂盒(目录号:69104) 德国Qiagen公司;Ezup试剂盒(目录号:B518261)、Dzup试剂盒(目录号:B518203)、SDS(分子生物级)、琼脂糖M、4S Green Plus核酸染色剂 生工生物工程(上海)股份有限公司;蛋白酶K、核糖核酸酶A(ribonuclease A from bovine pancreas,RNase A)、2hPremix Taq 日本TaKaRa公司;6hLoading Buffer 北京Solarbio科技有限公司;λ DNA/Hind III Marker、DNA Marker I、Marker D2000 DNA北京Tiangen生化科技有限公司;荧光定量PCR扩增试剂PowerUp SYBR Green Master Mix 美国Thermo Fisher公司;其他化学试剂均为分析纯。
1.2 仪器与设备
FW-100高速万能粉碎机 北京市光明医疗仪器有限公司;BSA224S-CW型分析天平 德国Sartorius公司;Sorvall BIOFUGE Stratos全能台式高速离心机、Nanodrop 2000微量紫外分光光度计、QuantStudio 3实时荧光PCR扩增仪 美国Thermo Fisher公司;HH-4J数显恒温水浴锅 上海维诚仪器有限公司;D1008掌中宝离心机 北京大龙兴创实验仪器有限公司;T100 Thermal Cycler PCR仪、UV light tray Gel Doc XR+凝胶成像系统美国Bio-Rad公司。
1.3 方法
1.3.1 DNA提取方法
1.3.1.1 Nucleo试剂盒
取0.1 g磨碎的样品于2 mL无菌离心管,根据试剂盒说明书进行后续的DNA提取,最后用50 μL TE缓冲液洗脱,-20 ℃保存,备用。
1.3.1.2 Wizard试剂盒
取0.1 g磨碎的样品于2 mL无菌离心管,根据试剂盒说明书进行后续的DNA提取,最后用50 μL TE缓冲液溶解DNA,-20 ℃保存,备用。
1.3.1.3 Dneasy试剂盒
取0.1 g磨碎的样品于2 mL无菌离心管,加入800 μL AP1缓冲液和4 μL RNase A,根据试剂盒说明书进行后续的DNA提取,最后用50 μL TE缓冲液洗脱,-20 ℃保存,备用。
1.3.1.4 Ezup试剂盒
取0.1 g磨碎的样品于2 mL无菌离心管,加入800 μL PCB缓冲液(预热至65 ℃),振荡混匀,置于65 ℃水浴保温25 min,加入20 μL RNase A,室温放置2~5 min,加同体积混合溶液(氯仿-异戊醇24∶1,V/V),12 000hg离心5 min,移上清液于新的离心管,根据试剂盒说明书进行后续的DNA提取,最后用50 μL TE缓冲液洗脱,-20 ℃保存,备用。
1.3.1.5 Dzup试剂盒
取0.1 g磨碎的样品于2 mL无菌离心管,加入400 μL Dzup溶液,振荡混匀,后续操作按照试剂盒说明书进行,最后用50 μL TE缓冲液溶解DNA,-20 ℃保存,备用。
1.3.1.6 SDS法
在Tian Fang等[22]方法基础上稍作修改。取0.1 g磨碎的样品于2 mL无菌离心管,加入1 mL 65 ℃预热的SDS溶液,于65 ℃保温混匀1 h,间或混匀,再加入10 μL RNase A,摇匀后于37 ℃保温30 min。之后14 000hg离心10 min,移上清液于新的离心管中,加入0.7 倍的混合溶液(氯仿-异戊醇24∶1,V/V),于12 000hg离心10 min,将上清液移入到新的2 mL离心管。再加0.7 倍的混合溶液(氯仿-异戊醇24∶1,V/V),12 000hg离心10 min,将上清液移入到新的2 mL离心管。加0.6 倍的异丙醇,混匀后于-20 ℃保存1 h,12 000hg离心10 min,弃去上清液,向沉淀中加入500 μL无水乙醇,将沉淀弹至溶解后,于12 000hg离心5 min,弃去上清液。使用500 μL无水乙醇重复洗涤沉淀1~2 次,于12 000hg离心5 min后,弃去上清液,并将沉淀晾干,然后用50 μL TE缓冲液溶解,-20 ℃保存,备用。
1.3.2 DNA质量浓度和纯度检测
用Nanodrop 2000微量紫外分光光度计测定各DNA样品的浓度和纯度。
1.3.3 DNA分子完整性检测
用0.5 hT B E溶液配制1%的琼脂糖凝胶,以0.1 μL/mL的比例,向琼脂糖凝胶中加入4S Green Plus核酸染色剂。取10 μL DNA溶液与2 μL 6hLoading Buffer混合后点样。于80 V恒压条件下电泳1 h。使用UV light tray Gel Doc XR+凝胶成像系统对DNA扩增结果进行观察、拍照和分析[23]。
1.3.4 DNA用于实时荧光PCR的灵敏度检测
选取大豆内源lectin基因81 bp的片段进行实时荧光PCR扩增,反应体系总体积为20 μL,包括PowerUp SYBR Green Master Mix 10 μL,上下游引物(表1)各0.3 μmol/L,模板DNA 40~50 ng,剩余体积用无菌水补齐。实时荧光PCR条件如表2所示。

表1 lectin基因实时荧光PCR及普通定性PCR的扩增引物序列及长度Table 1 Primers used in real-time PCR and qualitative PCR ampli fi cation of lectin gene
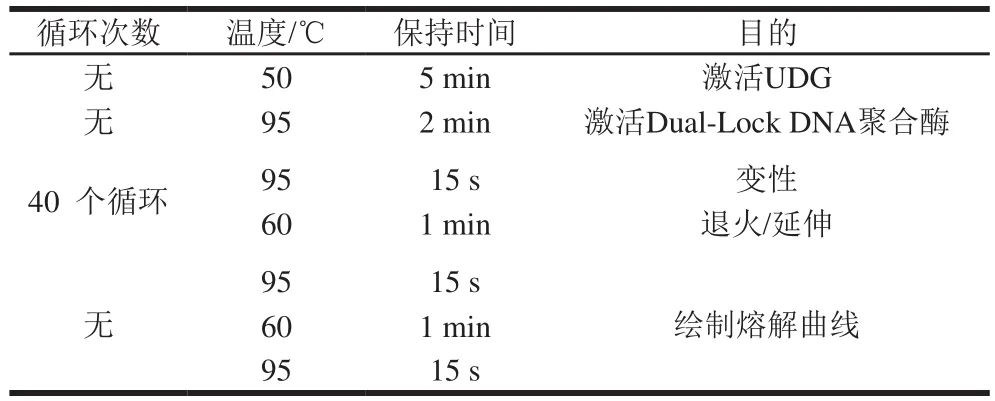
表2 lectin基因实时荧光PCR条件Table 2 Conditions for real-time PCR ampli fi cation of the lectin gene
1.3.5 不同长度lectin基因片段的定性PCR检测
以Dneasy试剂盒提取的大豆蛋白DNA作为模板,分别扩增不同长度的大豆内源lectin基因片段,探究大豆蛋白中lectin基因的降解情况。定性PCR体系的总体积为25 μL,包括2hPremixTaq10 μL,上下游引物(表1)各0.4 μmol/L, 模板DNA 40~50 ng,剩余体积用无菌水补齐。定性PCR条件如表3所示。

表3 lectin基因普通定性PCR条件Table 3 Conditions for qualitative PCR amplification of the lectin gene
1.4 数据统计与处理
所有实验均重复4 次,所有测试均进行4 次以上,采用SPSS 20.0软件进行方差分析,实验数值之间以Duncan法进行差异显著性分析(P<0.05,差异显著),采用Origin 8.5软件对实验结果作图。
2 结果与分析
2.1 6 种DNA提取方法得到的DNA质量浓度和纯度
应用6 种常用的DNA提取方法提取0.1 g大豆蛋白或大豆中的DNA,DNA质量浓度和纯度结果如表4所示。所有方法均可有效提取所有样品中的DNA,但DNA质量浓度和纯度差异显著,有必要针对不同样品种类进行具体分析。根据表4,将所有的样品分为SPI、SPC和大豆籽粒3 类,分别求得6 种方法提取这3 类样品得到的DNA质量浓度和DNA纯度(A260nm/A280nm和A260nm/A230nm)平均值,结果如图1、2所示。

表4 6 种方法提取所得的DNA质量浓度、纯度、Ct值结果对比Table 4 Comparison of DNA yield, purity, and real-time PCR results by six extraction methods
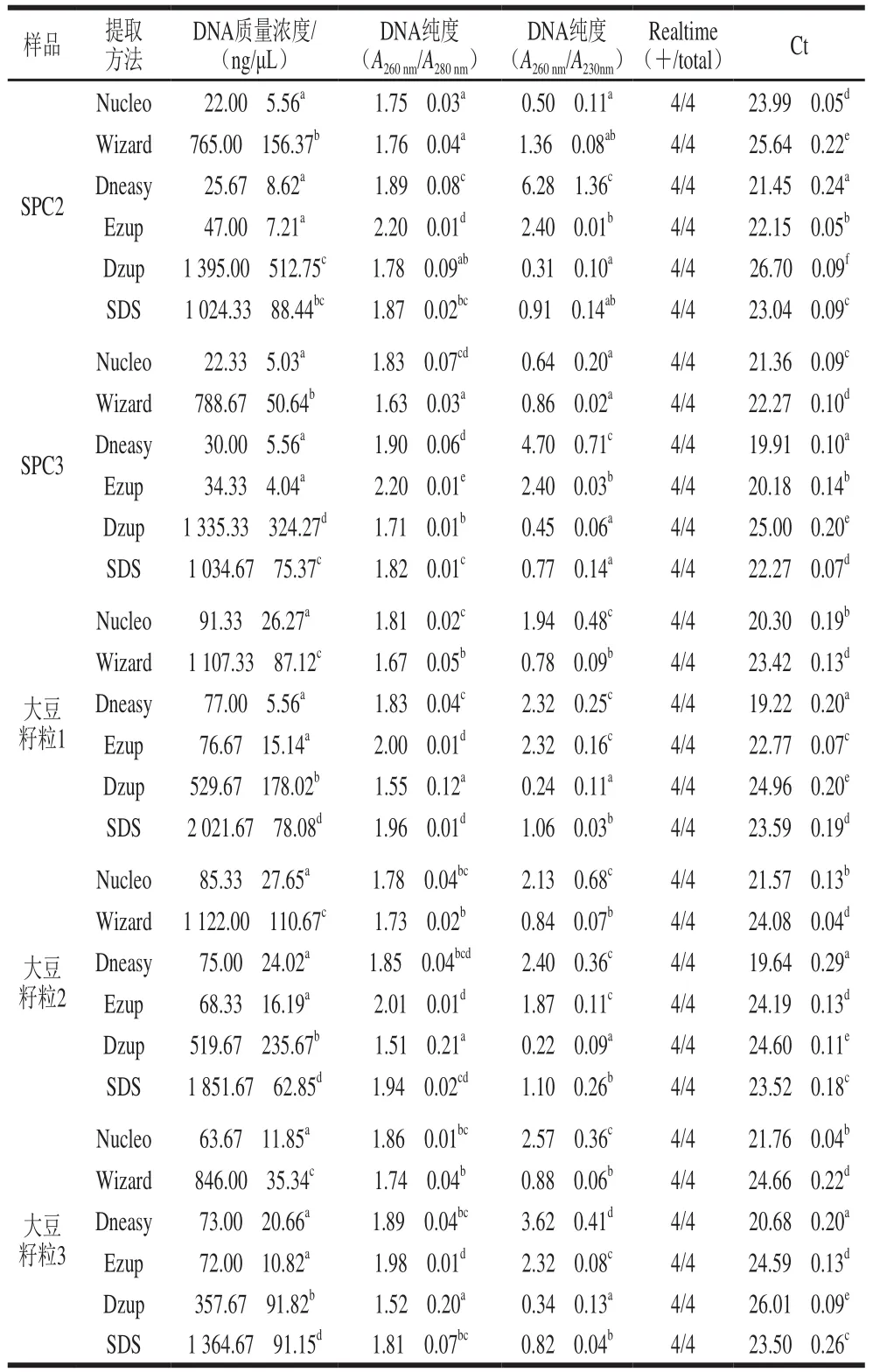
续表4
由图1可知,对于SPI,不同方法提取得到的DNA质量浓度从高到低依次为Dzup、SDS、Wizard、Nucleo和Dneasy、Ezup;对于SPC,该顺序依次为Dzup、SDS、Wizard和Ezup、Dneasy、Nucleo。大豆籽粒成分复杂,不同提取方法得到的DNA质量浓度与SPI和SPC相比,无明显的规律,如SDS法提取得到的大豆籽粒DNA质量浓度均高于SPI和SPC,Dzup试剂盒提取得到的大豆籽粒DNA质量浓度均低于SPI和SPC,而Wizard试剂盒提取得到的大豆籽粒DNA质量浓度界于SPI和SPC之间。这可能与不同的DNA提取方法对大豆细胞裂解程度不同,从而导致DNA释放差异。
DNA提取的基本原则是释放样品中的DNA,随后纯化DNA,去除PCR抑制剂[6]。Nucleo、Dneasy和Ezup试剂盒技术原理属于膜过滤法,分别用盐酸胍、AP1缓冲液和PCB缓冲液裂解样品,通过硅胶吸附柱将释放出的DNA吸附[28],再采用化学试剂溶解清除DNA上附着的蛋白质、多酚、多糖等物质,最后使用能够溶解和稳定贮存DNA的TE缓冲液将纯化的DNA从硅胶吸附柱上洗脱下来。Wizard试剂盒技术原理属于CTAB法,是利用阳离子去污剂CTAB可与蛋白质和多糖形成聚合物,并从低离子强度溶液中沉淀核酸与酸性多糖的能力,通过有机溶剂抽提,进一步除去蛋白、多糖、酚类等杂质,在乙醇的沉淀作用下,达到分离核酸的目的。Dzup试剂盒技术原理属于盐酸胍法,通过高pH值条件下的胍盐等离液剂和变性剂将植物组织裂解,并水解RNA,再经过乙醇沉淀得到基因组DNA。SDS法是基于阴离子去污剂SDS在高温下能裂解细胞,使染色体解离、蛋白变性,同时与蛋白和多糖形成聚合物,释放核酸。整体而言,本研究中的Nucleo、Dneasy、Ezup试剂盒提取的DNA质量浓度均低于Dzup、Wizard试剂盒和SDS法提取的DNA质量浓度。这表明膜过滤法在DNA纯化过程中,可能造成部分DNA的损失,从而降低产品DNA质量浓度。
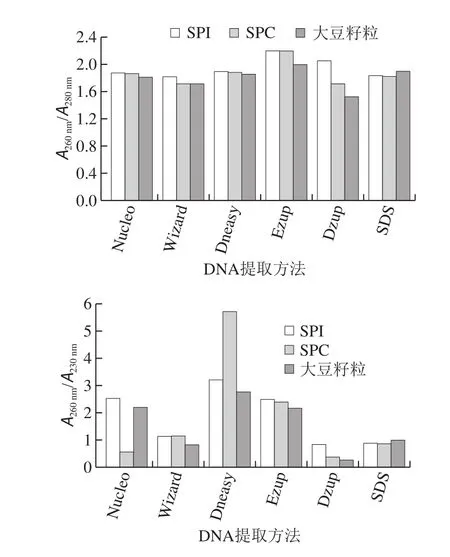
图2 6 种方法提取SPI、SPC和大豆籽粒中的DNA纯度Fig. 2 DNA purities from SPI, SPC, and soybean seeds by six extraction methods
DNA的纯度主要包含两个方面[29]:通过DNA溶液在260 nm和280 nm波长处的吸光度比值(A260nm/A280nm)的大小判断DNA样品受蛋白质、酚类的污染情况,通过DNA溶液在260 nm和230 nm波长处的吸光度比值(A260nm/A230nm)判断多糖、盐等小分子的污染情况[30]。6 种方法提取SPI、SPC和大豆籽粒中的DNA纯度对比结果如图2所示。
由图2可知,对于SPI,Nucleo试剂盒、Dneasy试剂盒得到的样品A260nm/A280nm值在1.7~2.0之间,A260nm/A230nm>2.0,表明样品纯度高;Wizard试剂盒和SDS法提取DNA的A260nm/A280nm值也界于1.7~2.0,但A260nm/A230nm<2.0,表明这两种方法对蛋白和RNA等的去除效果较好,但对盐等小分子的去除效果较差;Ezup试剂盒提取的DNA A260nm/A280nm和A260nm/A230nm均在2.0以上,表明样品中有RNA污染;Dzup试剂盒得到的DNA纯度最低,A260nm/A280nm>2.0,A260nm/A230nm<2.0,表明样品中不仅存在RNA,也存在盐等杂质。
对于SPC,仅Dneasy试剂盒提取的DNA纯度最高,A260nm/A280nm为1.9f0.1,A260nm/A230nm为5.7f1.7;Nucleo试剂盒、Wizard试剂盒、Dzup试剂盒和SDS法得到的DNA样品,A260nm/A280nm值界于1.7~2.0,但A260nm/A230nm<2.0,其中Dzup试剂盒A260nm/A230nm值最小,仅为0.4,表明采用这4 种方法提取的DNA中残存多糖、盐和其他小分子等杂质;Ezup试剂盒得到DNA的A260nm/A280nm和A260nm/A230nm均在2.0以上,表明样品受RNA污染。
对于大豆籽粒,所有方法得到DNA的A260nm/A280nm均处于1.7~2.0的范围内,A260nm/A230nm值从高到低依次为Dneasy试剂盒、Nucleo试剂盒、Ezup试剂盒、SDS法、Wizard试剂盒、Dzup试剂盒,DNA样品的纯度随之递减。
造成DNA纯度不同的原因与不同方法纯化DNA的原理不同有关。Nucleo试剂盒、Dneasy试剂盒、Ezup试剂盒是基于硅胶吸附柱将DNA吸附,再通过化学试剂除杂,从而提高样品纯度。而Wizard试剂盒、Dzup试剂盒、SDS法不借助物理吸附,仅通过蛋白酶、RNA酶及其他化学试剂,除去样品中的蛋白和RNA,接着根据相似相溶原理将样品中的非DNA类物质溶解,并通过离心去除。尽管膜过滤法得到的DNA样品浓度偏低,但其纯度较高。在浓度不影响后续检测的情况下,DNA纯度对转基因检测十分重要。DNA纯度越高,PCR抑制剂存在越少,越有利于提高后续定性、定量PCR的重复性、稳定性和精确性,降低出现假阴性或假阳性结果的概率[15]。
2.2 6 种DNA提取方法的成本和时间
如图3所示,DNeasy试剂盒提取DNA的过程耗时最短,约2 h即可完成,但成本最高,提取一个样品DNA需要41 元;SDS法成本最低,一次提取折合不到1 元钱,但耗时最长,完成一轮操作往往需要12 h以上。Wizard试剂盒和Ezup试剂盒的时间成本和经济成本比较适中。

图3 6 种DNA提取方法的成本(A)和时间(B)对比Fig. 3 Differences in cost (A) and time (B) between six DNA extraction methods
2.3 6 种DNA提取方法得到的DNA完整性

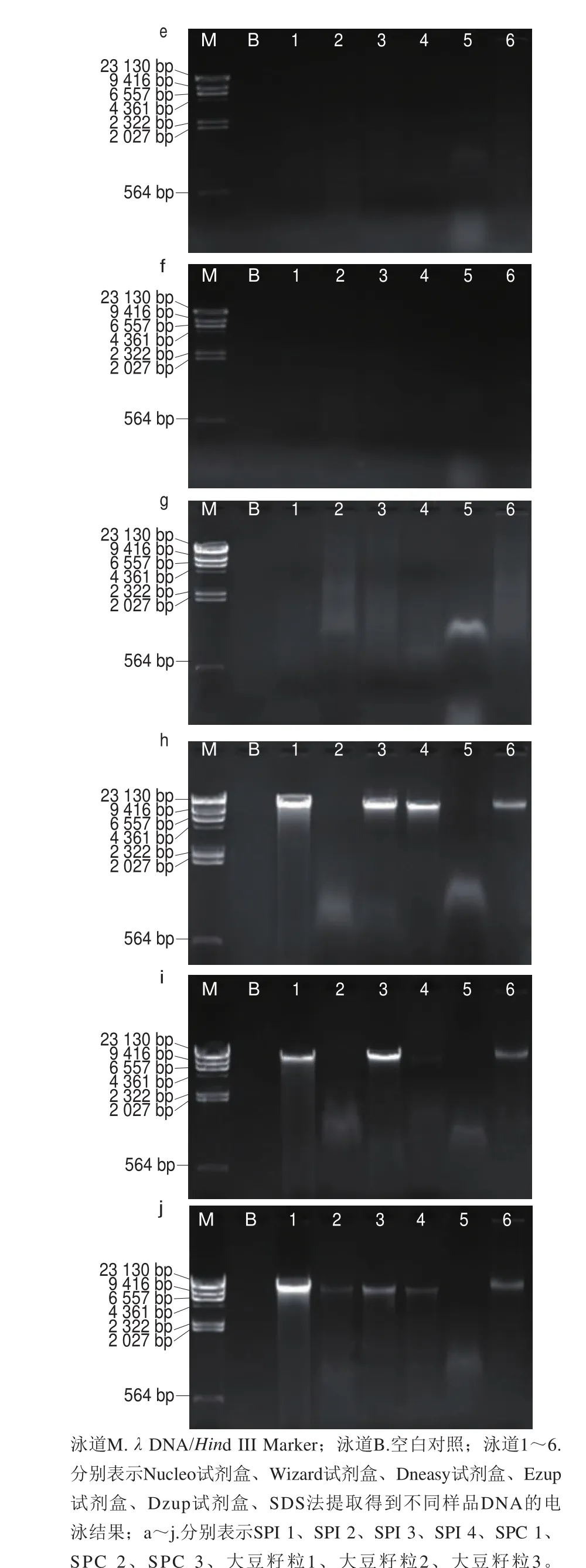
图4 6 种方法提取SPI、SPC和大豆籽粒的DNA凝胶电泳结果Fig. 4 Agarose gel electrophoresis analysis of DNA samples extracted from SPI, SPC and soybean seeds by six extraction methods
由图4a~g可以看出,SPI和SPC的基因组DNA分布于整个泳道或亮度很弱,几乎检测不到,表明以上样品的DNA存在降解,也印证了大豆蛋白制备工艺参数可能会对大豆DNA产生降解作用。大豆蛋白中DNA的降解可能与蛋白制备工艺有关。国内外生产SPI大多采用碱溶酸沉法,是将脱脂大豆粉用稀碱液浸提后,离心除去不溶性的多糖或其他杂质后再将浸出液的pH值调至4.5左右,使蛋白质处于等电状态沉淀下来[31]。在制备过程中主要经历碱性、酸性环境的化学作用、热作用和离心等物理作用。SPC的制备主要采用溶剂浸提法,逐步除去大豆粉中的非蛋白质类可溶性物质,并将剩下的不溶物干燥而得[2]。在此过程中DNA主要受到热作用和有机溶剂的化学作用,最常用的溶剂为乙醇。
前人研究表明,pH值[32]、温度[33]和化学试剂[34]会影响DNA的稳定性。在偏碱性条件下,基因的稳定性较高,受温度影响不大,而在极端pH值条件下双链DNA会变性成为单链DNA,甚至发生脱嘌呤反应,当pH值为4.0时,辅以温度65 ℃处理90 min,DNA降解率达到99%[35]。热处理过程中,DNA会发生物理变性,100 ℃以上可引起显著的链断裂,脱嘌呤或脱氨基反应,二级结构发生不可逆损失[32]。Lu Zhengfei等[36]在水醇浸提洋甘菊提取物时,发现不论水醇比例为多少,洋甘菊提取物中的DNA均无法被完全除去,DNA的片段长度大多在100~400 bp之间。
图4h~j为未经加工处理的大豆籽粒DNA,可见,Nucleo试剂盒、Dneasy试剂盒、Ezup试剂盒、SDS法均可提取出片段大小在23 kb左右的DNA,完整性较高,较适用于大豆DNA降解规律的研究。
2.4 大豆内源lectin基因的实时荧光PCR分析[37]
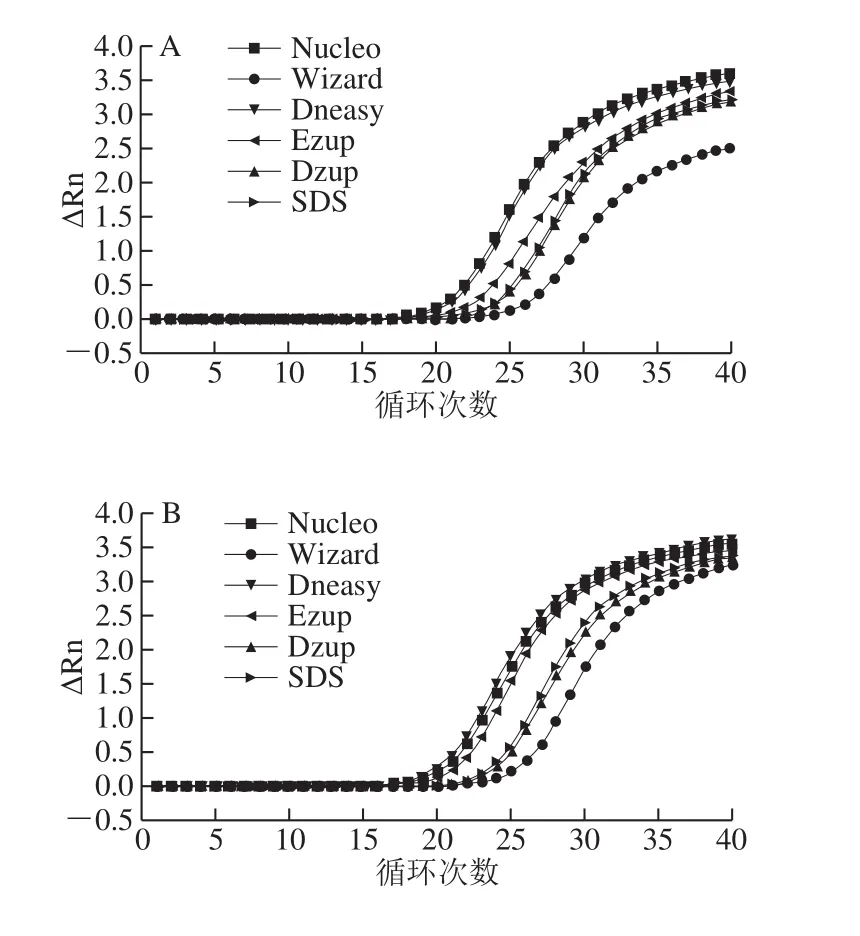
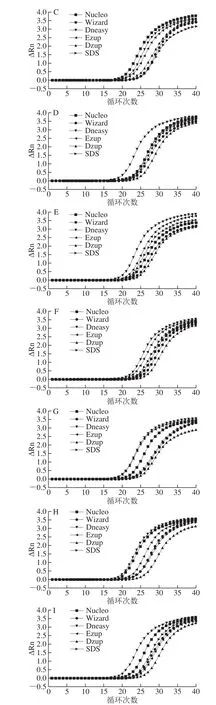
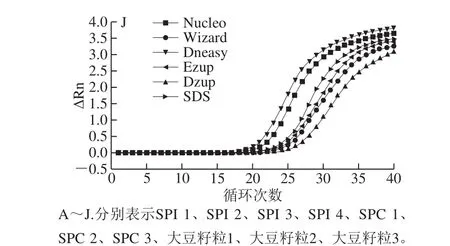
图5 6 种方法对lectin基因的扩增结果Fig. 5 Fluorescent PCR amplification curves of the lectin gene using DNA samples extracted by six extraction methods
6 种方法对10 种样品中大豆内源lectin基因81 bp的实时荧光PCR检测结果如图5所示,所有方法提取出的DNA均可以得到典型的扩增曲线,4 次平行实验的扩增结果均呈阳性(表4)[13],但Ct值大小差异明显。Fernandes等[38]也有类似结论:不同的DNA提取方法得到的DNA样品在进行荧光PCR时,Ct值大小可能不同。其中,Ct值越小,表明样品中的PCR抑制剂越少[39]。对于SPI,Nucleo试剂盒与Dneasy试剂盒的Ct值(19.9~22.7)均显著小于其他4 种方法,Dzup试剂盒和SDS法的Ct值相对偏高,Wizard试剂盒、Ezup试剂盒较为适中。对SPC以及大豆籽粒,Dneasy试剂盒的Ct值(19.2~21.5)均显著小于其他5 种方法,而Dzup试剂盒的Ct值(24.6~26.7)均显著大于其他5 种方法。该现象与DNA纯度结果基本一致,表明Dneasy试剂盒得到的DNA纯度较高,对实时荧光PCR检测具有较高的灵敏度,而Dzup试剂盒纯度较低,可能存在实时荧光PCR抑制剂,在实际检测中可能会影响结果的准确性。
2.5 SPI和SPC中lectin基因片段降解情况
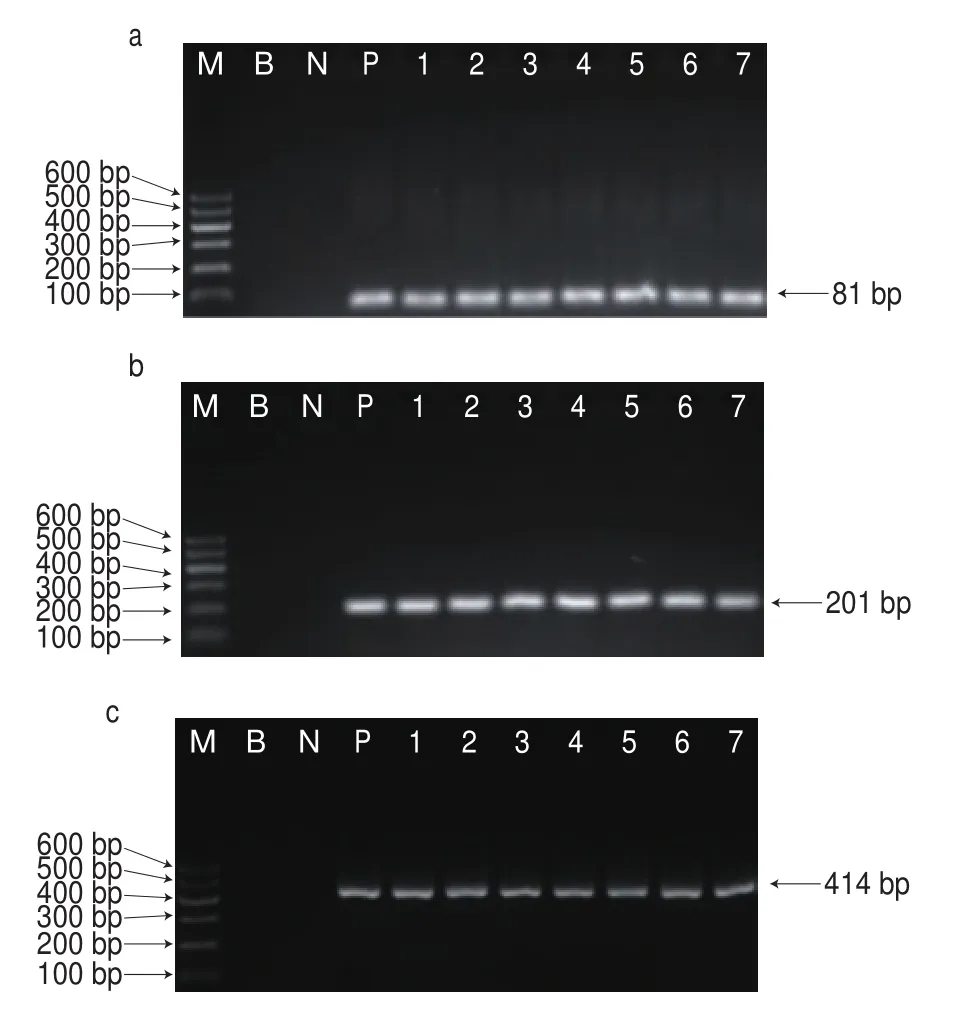

图6 SPI和SPC样品不同长度lectin基因片段的扩增电泳图Fig. 6 Agarose gel electrophoresis of PCR amplified products of the lectin gene with different lengths in SPI and SPC samples
以Dneasy试剂盒提取得到的大豆蛋白样品DNA作为模板,对不同长度的大豆内源lectin基因进行扩增,电泳结果如图6所示。所有样品中均可以明显扩增出lectin基因81、201、414 bp的片段,扩增条带清晰、明亮。lectin基因836 bp的片段也广泛存在于SPI和SPC样品DNA中,但SPI 3和SPC 1~3的条带亮度较低。在7 种大豆蛋白样品中,同样存在lectin基因1 487 bp的片段,但条带非常弱,几乎观察不到,这表明在蛋白制备过程中lectin基因可能发生了降解,长片段逐渐消失。通过长片段与短片段的扩增条带亮度对比还可知,基因片段长度越高,越不稳定,与短片段相比更容易发生降解。除此之外,在扩增片段长度相同的情况下,样品不同得到的条带亮度不同,说明不同产地的蛋白样品可能经受了不同的制备过程,在此期间DNA所受到的降解程度和降解规律有所不同。
3 结 论
本实验以不同产地的SPI、SPC和大豆籽粒为原料,研究6 种常用的DNA提取方法——Nucleo试剂盒、Wizard试剂盒、Dneasy试剂盒、Ezup试剂盒、Dzup试剂盒、SDS法对DNA的提取效果,筛选出最适合SPI、SPC和大豆籽粒中DNA提取的方法,以根据检测工作的实际需求,有针对性地选择合适的方法,提高工作效率和结果可靠性。
通过对比可知,Dneasy试剂盒提取DNA的浓度较低,但纯度最高,操作过程耗时最短,成本较高,DNA产品的完整性保持较好,实时荧光PCR检测过程中灵敏度高,在现场快检和对结果灵敏度、准确度要求较高的检测中较为适用。同样地,Nucleo试剂盒得到的DNA质量浓度也较低,但纯度较高,能够很好地保持DNA完整度,操作时间短,成本适中,对SPI的检测灵敏度高,而在SPC的检测方面灵敏度略低于Dneasy试剂盒。Ezup试剂盒和SDS法提取的DNA同样可以保持较高的完整性,SDS法价格最低,但纯度较差。Wizard试剂盒提取的DNA完整性相对较差,但由于该方法的实时荧光PCR灵敏度以及价格和操作时间均较低,也可以作为备选方法之一。Dzup试剂盒无论从DNA完整性还是浓度等方面均无法与以上5 种方法相提并论,因此不推荐作为大豆DNA的提取方法。
除此之外,本研究还发现SPI和SPC中DNA含量不同,SPI中的DNA含量整体高于SPC,推测其原因可能与DNA的降解有关。通过对4 种市售SPI和3 种SPC中不同长度的内源lectin基因扩增结果,印证了这一推测:在大豆蛋白制备过程中lectin基因836 bp和1 487 bp存在降解,条带亮度与81、201、414 bp相比明显较暗,也表明长片段比短片段更容易发生降解,且制备工艺不同,降解程度也有所不同。
因此,下一步可以在筛选出最适提取方法的基础上,对大豆蛋白制备过程中DNA的降解规律进行研究,为转基因大豆制品的检测和生产调控提供科学依据。

