鸟嘌呤四链体在生化分析中的应用进展
刘卓靓 陶呈安 王建方

摘 要 鸟嘌呤四链体(G-四链体)是一种特殊的核酸二级结构,它可与高铁血红素结合,形成具有过氧化物酶活性的核酶; 也可增强特殊结构染料的荧光强度。G-四链体作为功能核酸中的一种,具有性质稳定、特异性好、功能多样等特点,被广泛应用于各种生化分析中。本文对近年来G-四链体在生化分析中的研究和应用进展进行了评述,对其应用前景进行了展望。
关键词 G-四链体; 功能核酸; 核酶; 生化分析; 评述
1 引 言
随着现代医学的发展,特定核酸、蛋白、酶等的检测,已成为某些疾病的直接或间接诊断指标[1~3]。而传统的检测方法,如高效液相色谱法、聚合酶链式反应(PCR)、免疫印迹法、电泳法[4~6]都涉及专业人员操作以及大型仪器的使用,存在检测时间长、操作复杂、检测灵敏度不高等问题。而比色法及荧光法[7,8]作为仪器分析中的经典方法,重复性好、操作简单,易于试剂盒的开发。比色法或荧光法中如何高效稳定输出信号的报告分子是分析方法的研究热点。功能核酸鸟嘌呤四链体(G-四链体)的特殊理化性质,如与高铁血红素结合生成具有过氧化物酶活性的核酶、增强特殊结构染料的荧光强度等,可灵活地实现信号的稳定输出。利用G-四链体作为报告分子,与各种功能核酸或核酸信号放大方法联用,可实现多种目标分子高特异性、高灵敏的检测,为多种生化分析提供快速、简便的检测方法。
本文首先介绍了G-四链体的分类和功能,以及利用其与金属离子作用引起的结构变化而导致性质的变化,结合特定染料或药物分子的性质,与其它功能核酸联用等,在小分子、蛋白质、酶、金属离子、目标DNA或RNA检测、细胞成像及癌症治疗等方面应用的研究进展进行了评述。
2 G-四链体简介
2.1 G-四链体的分类
鸟嘌呤平面是由4个鸟嘌呤通过氢键构成的方形平面,G-四链体由几个鸟嘌呤平面堆叠而成,其结构由处于平面之间的金属离子形成稳定金字塔结构 [9]。中心稳定离子为一价或者二价金属离子,其中K+是最常见的金属离子,金属离子的大小决定G-四链体结构的稳定性[10,11]。
根据形成G-四链体的DNA或RNA链的数目分类,由一条链形成四链体结构称为分子内G-四链体,多条链构成的称为分子间G-四链体。根据链的走向可分为3类:当四条链的方向均相同时,为平行结构; 当两条链方向相同,另两条链与其方向相反时,为反平行结构; 其它情况称为混合平行结构(图1)[12,13]。G-四链体的结构可通过圆二色谱测定。所有的G-四链体结构在210 nm处都有一个正的特征吸收峰。平行结构的G-四链体在240 nm处有一个负吸收峰,在260 nm附近有一个正吸收峰; 反平行结构的G-四链体在260 nm处有一个负吸收峰,在290 nm处有一个正吸收峰[14]。
G-四链体的结构主要取决于DNA或RNA的序列、核酸链的浓度和稳定离子的种类[15]。以稳定离子种类为例,Kong等[16]发现同样的序列在K+或Na+条件下形成不同的结构,他们系统研究了以T为间隔的碱基序列(5′-G3TiG3TjG3TkG3-3′)。当T的总数目≤5时,在100 mmol/L的K+和Na+条件下均形成平行结构; 当T的总数目为6~7时,在K+存在下依然形成平行结构G-四链体; 而Na+稳定时折叠成混合平行结构。 Li等[17]发现,PW17在K+存在下形成平行结构G-四链体,而Pb2+条件下为反平行结构。
2.2 G-四链体的性质
2.2.1 G-四链体构成的模拟酶 具有酶催化活性的DNA或RNA稱为核酶(DNAzyme/RNAzyme)[18]。目前已发现具有切割或连接DNA/RNA、模拟过氧化物酶等活性的核酶[19~21]。G-四链体与辅因子高铁血红素(Hemin)结合形成具有过氧化物酶活性的核酶,可催化多种底物的氧化,包括ABTS、TMB、AmplexRed、鲁米诺等。这种核酶是Travascio等[22]在筛选可特异结合N-甲基化卟啉IX的核酸适配体时发现的,而后进一步优化得到18个碱基的核酸适配体PS2.M。PS2.M形成的G-四链体与Hemin构成的核酶,其催化活性是Hemin单独存在时的250倍[23]。此类核酶的催化反应机理与辣根过氧化物酶类似,G-四链体模拟辣根过氧化物酶中的组氨酸残基为Hemin中的Fe提供一个轴向配体和一个疏水环境,有利于OO的异裂。G-四链体为Hemin提供一个平面结构,有利于氢的交换[24~26]。目前,对于此类核酶的活性调控主要有3种方法:(1)是改变G-四链体本身的序列,从而改变其拓扑结构来影响其性质; (2)是改变稳定中心离子,从而改变G-四链体的构型而改变Hemin的结合情况; (3)是在G-四链体的序列末端增加腺嘌呤,可将其活性提高至原有活性的4倍[24]。另外,还可加入ATP来稳定催化过程中产生的自由基,提高催化效率[27]。
2.2.2 G-四链体增强荧光的染料 G-四链体可结合一些特定的荧光染料,极大地增强染料自身的荧光强度, 例如卟啉衍生物、酞菁染料、结晶紫、三苯甲烷等染料。荧光增强的原理是G-四链体可为染料分子提供一个疏水环境,防止荧光分子的相互靠近导致的自淬灭现象。
结晶紫(Crystal violet, CV)是一种三苯甲烷染料,堆积在两个鸟嘌呤平面之间,可区分平行和反平行G-四链体。反平行G-四链体的Loop环可结合CV使其不受溶剂的影响,使得荧光大大增强。而平行G-四链体侧链的Loop环不能结合CV,使得CV的荧光增强明显弱于结合反平行G-四链体的CV[16]。
硫磺素T(Thioflavin T, ThT)是一种水溶性染料,可特异性地识别并结合G-四链体,结合后其荧光强度可增大2500倍[28]。
(Z)-4-(3,5-difluoro-4-hydroxybenzylidene)-1,2-dimethyl-1H-imidazol-5(4H)-one(DFHBI)是一种可与RNA G-四链体结合并对其进行特异性识别的染料分子,结合后可模拟绿色荧光蛋白的荧光,用于细胞中的RNA成像[29]。在此基础上,Feng等[30]报道了6种与红色荧光蛋白生色团类似的染料分子,与G-四链体结合后发出的荧光光谱范围几乎覆盖红色荧光蛋白的发射光谱,并且有较高的荧光量子产率,以此可模拟红色荧光蛋白,实现细胞内成像。
2.2.3 G-四链体降低电信号的电化学活性分子
亚甲基蓝(Methylene blue,MB)是一种带正电的芳香结构,是一种常用的电化学信号分子。Zhang等[31]研究发现,由于G平面的末端π-π堆叠效应,MB可竞争Hemin结合到G-四链体上,显著减少扩散到电极表面的MB,从而使得电流下降。
3 G-四链体在生化分析中的应用
3.1 小分子与蛋白质的检测
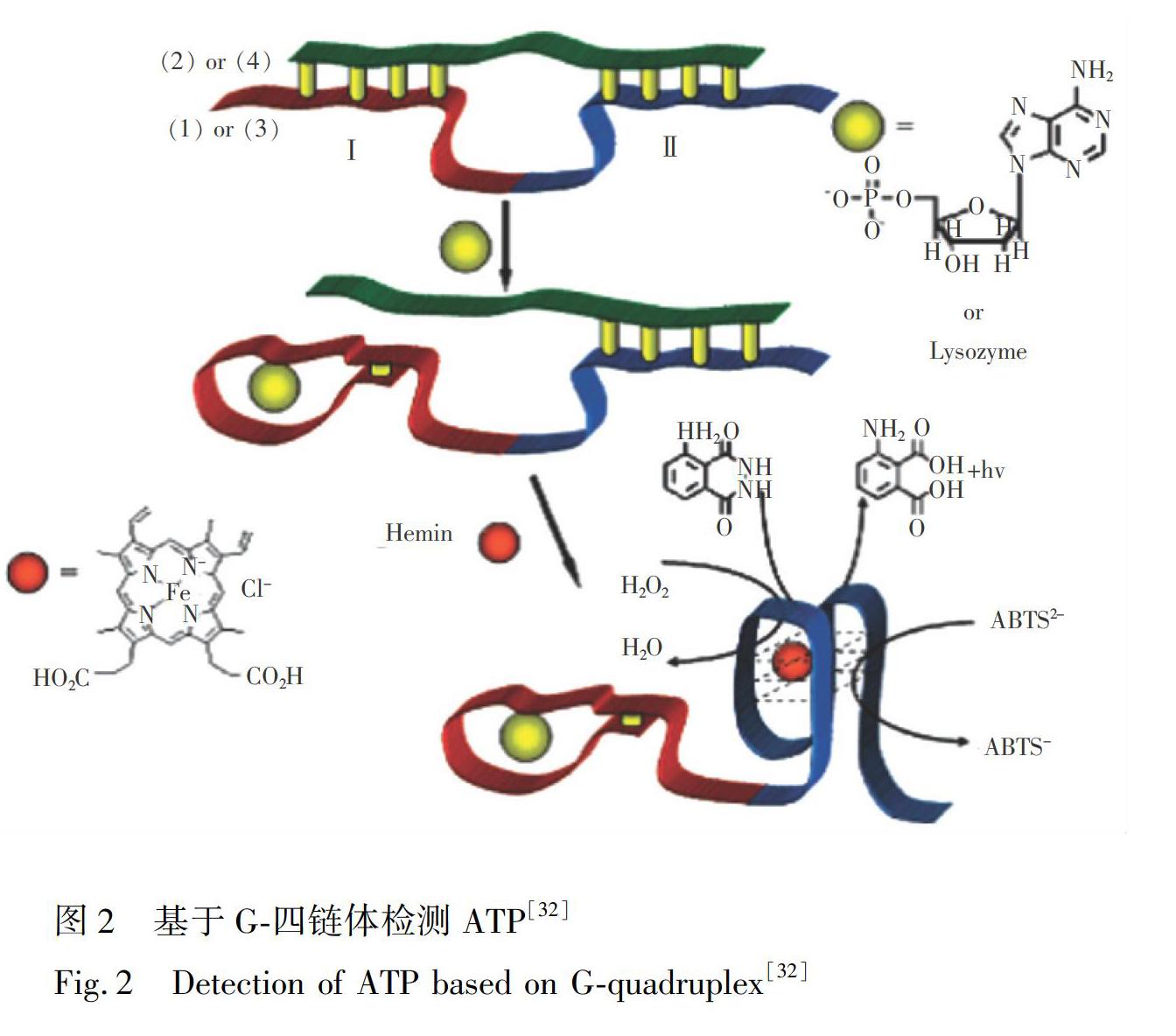
核酸适配体是经体外筛选,可高特异性和高亲和力地识别并结合目标分子的单链DNA或RNA。以G-四链体为信号分子检测小分子和蛋白质的传感分析方法大多与核酸适配体单元联用。如Willner研究组[32]将ATP的核酸适配体连接上一段G-四链体序列作为识别探针,另一条DNA与之部分杂交形成中间留有环状结构的双链结构。当ATP存在时,核酸适配体与ATP结合,识别探针从双链上解离下来,释放出的G-四链体与Hemin形成DNAzyme,催化底物反应(图 2)。利用类似的设计,可实现可卡因、溶菌酶、细胞因子[33~35]等的检测。在此类设计中,要求仔细筛选G-四链体和核酸适配体中被双链结构封闭的碱基数目,使得在有目标分子条件下G-四链体可从双链解离产生信号,而无目标分子时,G-四链体不会自动解离产生较高的背景信号。值得注意的,凝血酶有两种长度分别为15及29个碱基的核酸适配体,且都可形成G-四链体结构。Li等[36]利用凝血酶的核酸适配体结合凝血酶后,可促进核酸适配体形成的G-四链体,并更好地结合Hemin以提高DNAzyme的催化活性,从而实现凝血酶的检测。
3.2 酶活性的分析
酶是生命过程中不可或缺的一部分,各种疾病的发生都与酶活性的异常相关,因此酶活性的检测是生化分析中的重要内容。以G-四链体为信号分子分析酶活性的报道中,检测对象大多是以核酸为底物的酶。以下介绍几种在疾病诊断、分子生物学中有重要意义的酶的检测方法。
3.2.1 T4多聚核苷酸激酶(T4 polynucleotide kinase,T4 PNK) T4 PNK是一种双功能酶,可移除3′磷酸基团以恢复3′羟基基团,也可实现5′磷酸化。Liu等[37]报道了一种基于G-四链体-DNAyzme比色检测T4 PNK的方法。在该体系中,设计了两条探针,一条是3′磷酸化的引物,一条是封闭有G-四链体序列的发夹探针。当T4 PNK存在时,引物的3′磷酸被去磷酸化,生成3′羟基,此时聚合酶可将引物延伸打开发夹,释放G-四链体序列。该方法可检测低至0.01 U/mL的T4 PNK。基于T4 PNK5′磷酸化活性,裴仁军研究组[38]设计了一种裂分的G-四链体 DNAzyme检测体系,实现最低浓度0.014 U/mL的T4 PNK的检测。
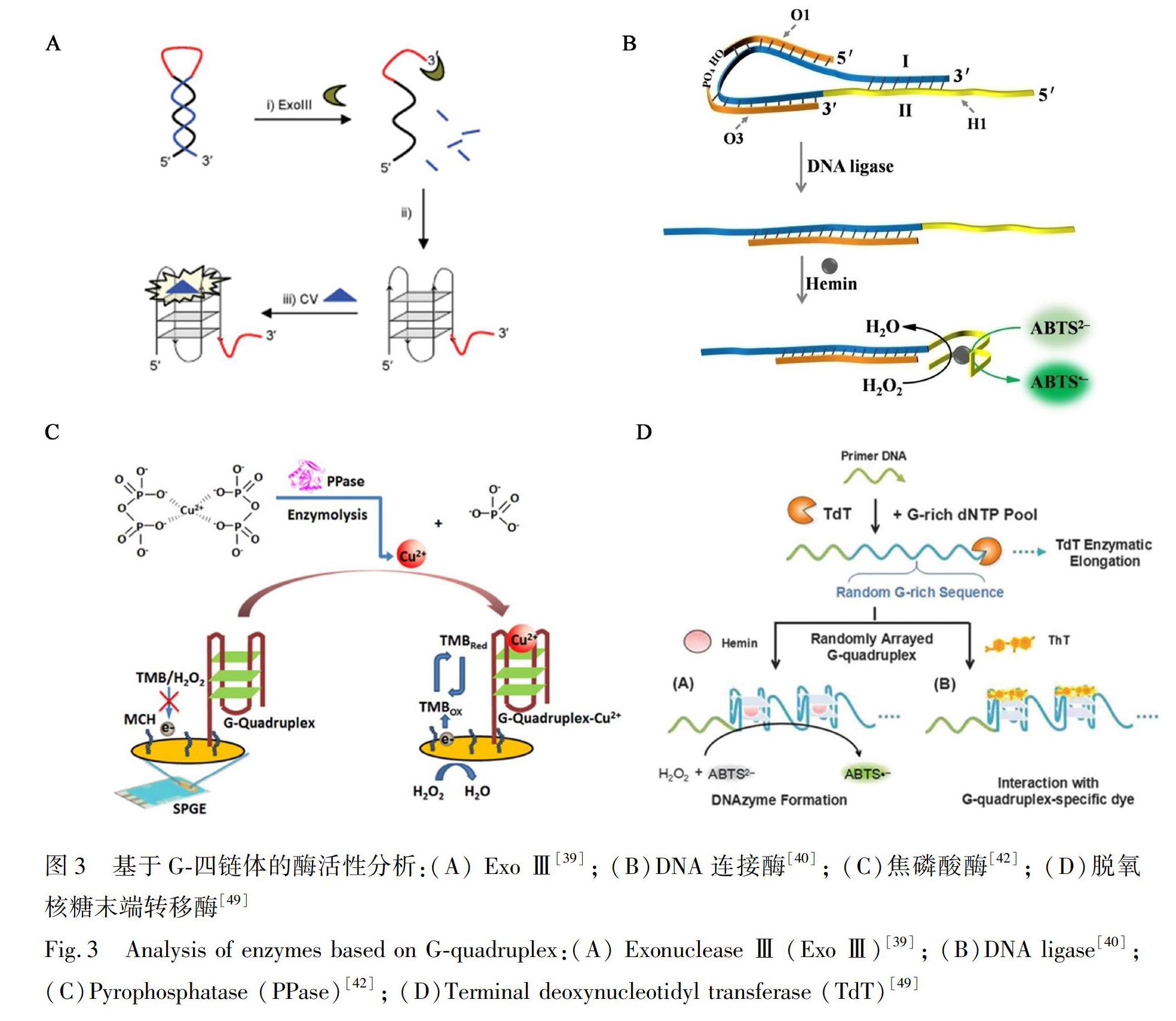
3.2.2 核酸外切酶Ⅲ(Exonuclease Ⅲ,Exo Ⅲ) Exo Ⅲ具有从3′-5′方向切割DNA的活性,在DNA复制等生理过程中有重要意义。Leung等[39]设计了发夹茎部含有G-四链体结构的探针用于分析ExoⅢ的活性。核酸内切酶Ⅲ从发夹茎部的3′端切断,最后剩下的单链部分即为G-四链体,與CV结合后大大增加其荧光,以此来检测核酸内切酶Ⅲ的活性(图3A)。
3.2.3 DNA连接酶(DNA ligase) DNA连接酶能催化DNA分子内或分子间毗邻的5′磷酸基团和3′羟基基团形成磷酸二酯键, 将两段相邻的DNA拼合成一条完整的DNA链。He等[40]利用DNA连接前后对于发夹结构的改变,开发了基于G-四链体检测DNA连接酶的方法(图3B),最低可检测0.2 U/mL的DNA连接酶。
3.2.4 DNA甲基化酶(DNA methyltransferase) DNA甲基化酶能识别特定序列中的胞嘧啶或腺嘌呤,并将甲基共价连接到该碱基上,可调控基因表达。Li等[41]利用DNA甲基化酶识别发夹结构中的甲基化位点并对其甲基化,然后特异性切割酶DpnI识别被甲基化的碱基对其切断,将G-四链体短片段释放出来,可实现6 U/mL DNA甲基化酶的检测。为进一步降低检出限,引入聚合酶形成分子机器,通过识别甲基化后的片段DNA,进行多次的聚合及切割,可检测更低浓度的甲基化酶,检出限为0.25 U/mL。
3.2.5 焦磷酸酶(Pyrophosphatase, PPase) PPase是作用于双磷酸键上的酸酐水解酶, 可催化一分子焦磷酸盐转化为两分子磷酸盐离子。Wang等[42]根据Cu2+与焦磷酸配位形成Cu-Ppi复合物,而焦磷酸的水解产物磷酸不结合Cu2+,PPase作用后Cu2+从复合物上释放出来,作为稳定G-四链体结构的中心离子,催化TMB与H2O2反应,实现了PPase的检测,检出限为0.0006 U/mL(图3C)。
3.2.6 碱性磷酸酶(Alkaline phosphatase,ALP) ALP可催化核酸分子脱去磷酸基团,从而转变为羟基末端。Liu等[43]利用其能产生自由羟基末端的性质开发了一种以G-四链体为信号分子的电化学传感器。ALP将杂交在电极上的DNA的磷酸基团除去形成羟基,再加入脱氧核糖末端转移酶延伸得到长链的T序列,此时加入一段末端为polyA的G-四链体探针即能杂交到长链T上,G-四链体与Hemin形成DNAzyme催化电极表面Thi的氧化, 实现信号的输出。此方法可实现最低浓度为0.00003 U/mL的ALP的检测。
3.2.7 尿嘧啶-DNA糖基化酶(Uracil - DNA Glycocasylase, UDG) UDG能特异性识别并除去DNA单链或双链的尿嘧啶残基,在细胞增殖过程中保持基因稳定性。Hu等[44]设计了一种免标记检测UDG的新方法。该方法通过去除双链DNA中的其中一条单链的4个尿嘧啶,形成AP位点使得双链结构解离,释放出的单链可折叠成G-四链体,增强甲基化卟啉(NMM)的荧光,实现UDG的检测,检出限为0.05 U/mL。另一种检测UDG的方法涉及DNA修复酶切割酶的使用。利用UDG对U进行糖基化后,内切酶VI即可识别并切断糖基化后的尿嘧啶产生3′羟基端,再经过聚合酶和限制性内切酶反复地聚合与切割,实现信号放大。此方法可实现最低浓度0.0001 U/mL UDG的检测[45]。
3.2.8 聚合酶(Polymerase) 聚合酶能依据模板序列将dNTP聚合到引物上, 形成与模板互补的单链,对维持基因的稳定性有重要作用。Leung等[46]设计了一个带有悬垂端的发夹分子,当有聚合酶存在时,较短的发夹一段作为延伸引物,另一段悬垂端作为模板进行扩增。对产物进行变性后,聚合产物链在K+存在下形成G-四链体,结合并增强Ir配合物的荧光,实现对聚合酶的检测。
3.2.9 端粒酶(Telomerase) 端粒酶是一种核糖蛋白酶,能将端粒重复序列5′-GGGTTA-3′连接到染色体末端, 从而抑制癌细胞的无限增殖。Xiao等[47]利用端粒酶聚合生成的重复片段将发夹打开,释放出完整的G-四链体,G-四链体与Hemin形成DNAzyme, 催化ABTS显色, 以此来检测端粒酶的活性。Li等[48] 开发了一个更简单的方法,设计了一条含有3组3个鸟嘌呤重复单元的探针。此探针在结合Hemin后只有极低的催化活性。但当端粒酶存在时,端粒酶能在探针末端添加3个鸟嘌呤的重复单元,此时探针可形成G-四链体,高效催化ABTS的氧化,实现端粒酶的检测。
3.2.10 脱氧核糖末端转移酶(Terminal deoxynucleotidyl transferase, TdT) TdT是一种特殊的DNA聚合酶,不需要模板,可催化DNA3′羟基端延伸,是一种被广泛使用的工具酶。Nie的研究组[49,50]报道了在富含dGTP的底物池中,TdT合成的DNA长链可形成多个连续无固定序列的G-四链体,利用生成的G-四链体对ThT的荧光增强(图3D)和CV的电化学信号放大作用,实现了TdT活性分析以及实时监测。
3.3 金属离子的检测
目前,金属离子污染问题日趋严重,开发高效检测金属离子的方法,成为研究者关注的热点。以G-四链体为信号分子的检测方法可简单、高效、便捷地检测金属离子,按其检测原理可分为三类。
3.3.1 特异性稳定G-四链体的金属离子 利用中心离子稳定的G-四链体构型不同而实现检测。Li等[17]报道了一种基于G-四链体结构变化检测Pb2+的方法。PS2.M在K+存在时折叠成平行结构的G-四链体,与Hemin结合后显示较高的过氧化物酶活性。 在Pb2+存在时, PS2.M折叠成反平行的G-四链体,不利于与Hemin的结合,过氧化物酶活性下降。根据K+与Pb2+稳定的G-四链体的催化活性的差异,可实现二者的检测(图4A)。利用此原理,还可实现Tb3+[51] 、Ba2+[52]等的检测。此外,还有一类特殊的例子,如利用Cu+能高效催化叠氮基团与炔基的Click反应,使得两条裂分的DNA连接后能形成G-四链体结构,加入取代链后可将完整的G-四链体序列置换下来,实现CV的荧光增强,对Cu2+进行检测,最低检测浓度可达到65 nmol/L[53]。
3.3.2 与碱基作用的特定金属离子 利用特定金属离子与碱基的作用实现金属离子检测,如T-Hg2+-T, C-Ag+-C结构的形成。其中一类是基于金属离子与碱基作用后抑制G-四链体的形成(Turn-off模式),另一类是促进G-四链体的形成(Turn-on模式)。Willner研究组报道了一种基于T-Hg2+-T的分子机器检测Hg2+(图4B)[54]。Li等[55]报道了基于C-Ag+-C作用下,拉近两条裂分的G-四链体序列,构成完整G-四链体检测Ag+,检出限为2.5 nmol/L。
3.3.3 具有切割活性DNAzyme对应的金属离子 自1994年第一例具有金属离子切割活性的DNAzyme被报道以来,越来越多的金属离子切割活性酶被发现。基于切割活性DNAzyme与G-四链体结构实现金属离子检测的方法,基本思路是利用发夹结构或者DNA的双链结构封闭G-四链体信号区域,当有目标金属离子存在时,对发夹结构或者双链结构中的特定位置切割,通过释放G-四链体序列,检测目标离子。2009年, Yin等[56]报道了基于Cu2+切割型的DNAzyme检测Cu2+的方法。当有目标离子Cu2+存在时,探针链自我切割, 释放出自由的G-四链体部分,G-四链体部分与Hemin结合形成DNAzyme催化底物,实现Cu2+的检测。通过这种方法可灵敏检测Zn2+、UO2+2、Mg2+、Pb2+(图4C)[57~59],由于切割性核酶的特异性,使得该方法能特异性地实现金属离子的检测。
3.4 目标DNA/RNA检测
许多疾病的发生与一些特定DNA或者RNA的异常表达有关,如p53、miRNA141,对这些DNA、RNA早期高灵敏检测可实现疾病的早诊断、早治疗。Weizmann等[60]报道了通过目标DNA打开发夹结构释放G-四链体产生信号的方法检测目标DNA; Deng等[61]利用目標DNA拉近裂分的G-四链体结构作为信号分子检测DNA(图5Ⅰ)。
对低丰度DNA、RNA,可通过信号放大方法实现检测。信号放大的方式包括与其它纳米材料联用,或者引入核酸为底物的信号放大技术。以金纳米颗粒联合G-四链体放大检测目标DNA为例,修饰在金电极上的捕获探针与目标DNA杂交,此时加入修饰识别探针的金纳米颗粒,此识别探针中间含有与目标互补序列以及G-四链体序列。由于金纳米颗粒上修饰多个含有G-四链体的识别探针,实现信号的放大(图5Ⅱ)[62]。以核酸为底物的信号放大方法主要有滚环扩增(Rolling circle amplification, RCA)、链置换扩增(Strand displacement amplification, SDA)、核酸内切酶辅助的放大(Nicking endonuclease signal amplification, NESA)、外切酶Ⅲ辅助信号放大方法等。这些放大方法以G-四链体为信号分子可实现超低浓度的核酸检测[63~65]。Wang等[66]设计了一个含有3段与G-四链体互补以及与目标DNA互补序列的环状DNA,以其为模板,以目标DNA为引物,进行滚环扩增。扩增产物中含有多个G-四链体结构,加入Hemin形成多个DNAzyme,催化底物鲁米诺发光,实现目标DNA的检测。前述的信号放大方法都需要酶的参与,而随着DNA纳米技术的发展,一种无酶链取代技术杂交链式反应(Hybridization chain reaction, HCR)被报道[67]。HCR中设计两个发夹结构,通过触发链(一般为目标DNA)打开其中一个发夹,被打开的发夹结构打开另一个发夹结构,两者交替打开,形成一条长链的双链DNA。
Shimron等[68]对两个发夹结构的序列做了修改,使得在形成长的双链DNA结构时,每个发夹末端有一段裂分G-四链体序列,相邻两个裂分G-四链体片段即可组成一个完整的G-四链体结构,作为信号输出分子。
3.5 纳米结构的形成与调控
平行的G-四链体结构可在特定金属离子存在下形成长的、连续的纳米结构,称为G纳米线。这类纳米结构有良好的机械稳定性、抗热、抗DNase I水解,以及独特的光学和电化学性质,在结构纳米技术中有潜在的应用价值[69]。Ren等[70]利用G纳米线形成后其瑞利散射显著增强,联合Exo Ⅲ放大方法, 实现了Hg2+的灵敏检测。He等[71]发现,G纳米线表面负载CoIIPc后, 可高效、高选择性地电催化CO2的还原。
利用G-四链体结构形成前后其结构的变化而引起链长度的变化,可作为纳米结构的有效调控手段。Sannohe等[72]在两段双链结构中设置裂分的两段G-四链体结构,当K+存在时,G-四链体形成,拉近两段双链,形成以G-四链体为节点的X型纳米结构。G-四链体也可作为纳米材料的距离调控手段。Sathya等[73]将ATP核酸适体的两端分别连接碳点和银纳米颗粒,当没有目标物ATP时,DNA呈松散状态,碳点与银纳米颗粒相距较远,没有作用;而当目标物存在时,核酸适体结合其形成G-四链体结构,处于DNA两端的碳点和银纳米颗粒距离被拉近, 形成有效的FRET对。
3.6 成像分析与癌症治疗
G-四链体可增强一些特异性染料的荧光,利用其特异性以及高倍数的荧光信号放大作用进行成像分析。G-四链体可特异性地结合TMpyP4,TMpyP4具有优良的光热性质,可作为癌症治疗中的光动力治疗分子。因此,这些特异性分子是G-四链体用于成像分析和癌症治疗的基础。
细胞凋亡在细胞和机体生长调节过程中具有重要作用,细胞凋亡与多种疾病(如衰退性疾病和癌症)的发展有关。传统评价细胞凋亡的方法包括DNA ladder实验和TUNEL实验。虽然这些方法可有效地指示细胞凋亡,但是具有消耗时间长、工作量大、多步的分离和清洗、低灵敏度、假阳性、高花费等缺点。为了解决这个问题,Nie研究组[74]基于TdT酶激活G-四链体合成,在单细胞水平对凋亡细胞进行免标记的原位生物成像(图6A)。最近,该团队及合作者针对常见的丙型肝炎病毒,以病毒RNA基因组序列中的G-四链体为靶点,设计合成荧光探针ThT-NE,建立了病毒RNA基因组活细胞荧光成像新方法[75]。该方法可实现非修饰或不需遗传改造的活细胞中病毒RNA基因组分布信息获取,监测整个病毒增殖过程,为病毒生物学、医学诊断提供了新工具。
核仁素是涉及RNA转录、DNA复制和bcl-2稳定的多功能蛋白,很多癌症细胞会高表达核仁素。AS1411是核仁素的核酸适配体,也可形成G-四链体结构。Shieh等[76]利用AS1411可特异结合并增强TMpyP4的荧光,实现对肿瘤细胞的识别与光动力治疗(图6B)。Liu等[77]在AS1411的两端分别设计富含G-C的双链结构,双链结构可作为阿霉素的载体进行运送,因此可通过G-四链体实现两种药物的输送,用于克服肿瘤细胞抗药性的治疗(图6C)。而AS1411的识别作用只限于某些特定种类的肿瘤细胞,为了增加G-四链体作为药物载体以及成像分子适用范围,Tan研究组利用裂分的核酸适配体与G-四链体构建的双功能核酸分子用于细胞成像与药物输送[78] ,以及合成同时具有核酸适配体和G-四链体序列的长链DNA探针实现肿瘤细胞的光动力治疗以及近红外成像[79]。以上研究中设计的治疗探针一般只包含1个G-四链体结构,其输送的光动力治疗分子数目有限。Nie研究组[80]开发了一种基于TdT产生的G-四链体用于细胞成像和光动力治疗的方法。以A549细胞的核酸适配体作为引物分子,TdT聚合生成含有多个G-四链体的长链DNA,加入染料ThT后,可实现目标细胞的成像检测。而TMPyP4与G-四链体结构的亲和力更强,可竞争ThT结合到G-四链体结构上,引入多个TMPyP4分子,可提高光动力治疗的效率(图6D)。前面所述的工作是通过增加G-四链体的数目从而增加光敏剂数量来提高治疗效率。近日,Cheng等[81]开发了一种新型的光敏剂分子(TMPipEOPP)4+·4I,相比于传统卟啉,实现了50 nm的红移以及7.4倍的光吸收效率,极大地增强了单线态氧的产生以及光动力治疗的效率。将 (TMPipEOPP)4+·4I與G-四链体结合作为光敏剂,并与MnO2结合,可实现高效的体内及体外光动力治疗,并有望应用于临床治疗。
4 總结与展望
G-四链体作为一种功能核酸,具有性质稳定、特异性好、功能多样等特点,被广泛用于各种生化分析中。目前基于G-四链体开发的分析检测方法包括比色法、荧光法、电化学、电化学发光等多种形式。由于G-四链体结构的特殊性, 其被用于细胞成像以及癌症治疗等方面的研究。G-四链体未来主要的发展趋势可能有以下方面:(1)提高模拟过氧化物酶的活性。目前的研究结果表明G-四链体构成的核酶虽然可通过前文方法提高活性,但是对比蛋白酶的活性还是有很大差距。因此,提高核酶活性是重要的研究内容。(2)特异性识别的红外染料的合成。随着成像技术的发展,普通荧光成像面临着高背景、易被光漂白等应用方面的限制。开发组织穿透能力高,结合G-四链体后能显著增强荧光的染料,可拓展G-四链体在细胞成像方面的应用。(3)便携式传感器的开发与应用。目前,基于G-四链体的分析传感方法多局限于实验室应用, 开发便携式(如试纸类、数显类)直读型便携式商用传感器将是未来的研究趋势。
References
1 Divne A M, Allen M. Forensic Sci. Int., 2005, 154(2-3): 111-121
2 Wu H, Liu Y, Wang H, Wu J, Zhu F, Zou P. Biosens. Bioelectron., 2015,66: 277-282
3 Duan R, Zuo X, Wang S, Quan X, Chen D, Chen Z, Jiang L, Fan C, Xia F. J. Am. Chem. Soc., 2013,135(12): 4604-4607
4 Seok Y, Byun J Y, Mun H, Kim M G. Microchim. Acta, 2014,181(15-16): 1965-1971
5 Young S J, Wook E B, Nahm M H. Infect. Chemother., 2013,45(4): 351-366
6 Ammann A A. Anal. Bioanal. Chem., 2002,372(3):448-452
7 Stojaovic M N, Landry D W. J. Am. Chem. Soc., 2002,124(33): 9678-9679
8 Cen Y, Yang Y, Yu R Q, Chen T, Chu X. Nanoscale, 2016,8(15): 8202-8209
9 Sen D, Gilbert W. Nature,1988,334(6180): 364-366
10 Schultze P, Hud N, Smith F, Feigon J. Nucleic Acids Res.,1999,27(15):3018-3028
11 Guschlbauer W, Chantot J F, Thiele D. J. Biomol. Struct. Dyn.,1990,8(3):491-511
12 Burge S, Parkinson G N, Hazel P, Todd A K, Neidle S. Nucleic Acids Res., 2006,34(19):5402-5415
13 Patel D J, Phan A T N, Kuryavyi V. Nucleic Acids Res., 2007,35(22):7429-7455
14 Kypr J, Kejnovská I, Renciuk D, Vorlickova M. Nucleic Acids Res., 2009,37(6):1713-1725
15 Phan A T. FEBS J., 2010,277(5): 1107-1117
16 Kong D M, Ma Y E, Guo J H, Yang W, Shen H X. Anal. Chem., 2009,81(7):2678-2684
17 Li T, Wang E, Dong S. J. Am. Chem. Soc., 2009,131(42): 15082-15083
18 Kruger K. Cell,1982,31(1):147-157
19 Carmi N, Shultz L A, Breaker R R. Chem.Biol.,1996,3(12):1039-1046
20 Li Y, Breaker R R. P. Natl. Acad. Sci. USA,1999,96(6):2746-2751
21 Li Y, Liu Y, Breaker R R. Biochemistry, 2000,39(11):3106-3114
22 Travascio P, Li Y, Sen D. Chem. Biol.,1998,5(9):505-517
23 Willner I, Shlyahovsky B, Zayats M, Willner B. Chem. Soc. Rev., 2008,37(6):1153-1165
24 Poulos T L, Kraut J. J. Biol. Chem.,1980,255(17):8199-8205
50 Hu Y, Shen Q, Li W, Liu Z, Nie Z, Yao S. Biosens. Bioelectron., 2015,63: 331-338
51 Chen Q, Zuo J, Chen J, Tong P, Mo X, Zhang L, Li J. Biosens. Bioelectron., 2015,72:326-331
52 Xu L, Chen Y, Zhang R, Gao T, Zhang Y, Shen X, Pei R. J. Fluoresc., 2017,27(2): 569-574
53 Shen Q, Zhou L, Yuan Y, Huang Y, Xiang B, Chen C, Nie Z, Yao S. Biosens. Bioelectron., 2014,55: 187-194
54 Li D, Wieckowska A, Willner I. Angew. Chem. Int. Ed., 2008,47(21):3927-3931
55 Li T, Shi L, Wang E, Dong S. Chem-Eur. J., 2009,15(14):3347-3350
56 Yin B C, Ye B C, Tan W, Wang H, Xie C C. J. Am. Chem. Soc., 2009,131(41):14624-14625
57 Moshe M, Elbaz J, Willner I. Nano Lett., 2009,9(3): 1196-1200
58 Elbaz J, Moshe M, Shlyahovsky B, Willner I. Chem-Eur. J., 2009,15(14):3411-3418
59 Elbaz J, Shlyahovsky B, Willner I. Chem. Commun., 2008,13: 1569-1571
60 Weizmann Y, Cheglakov Z, Willner I. J. Am. Chem. Soc., 2008,130(51):17224-17225
61 Deng M, Zhang D, Zhou Y, Zhou X. J. Am. Chem. Soc., 2008,130(39):13095-13102
62 Niazov T, Pavlov V, Xiao Y, Gill R, Willner I. Nano Lett., 2004,4(9): 1683-1687
63 Yuan L, Tu W, Bao J, Dai Z. Anal. Chem., 2015,87(1): 686-692
64 Gao Y, Li B. Anal. Chem., 2013,85(23): 11494-11500
65 Mittal S, Thakur S, Mantha A K, Kaur H. Biosens. Bioelectron., 2019,124: 233-243
66 Wang F, Lu C H, Liu X, Freage L, Willner I. Anal. Chem., 2014,86(3): 1614-1621
67 Choi H M, Chang J Y, Trinh L A, Padilla J E, Fraser S E, Pierce N A. Nat. Biotechnol., 2010,28(11): 1208
68 Shimron S, Wang F, Orbach R, Willner I. Anal. Chem., 2012,84(2): 1042-1048
69 Hua Y, Changenet B P, Improta R, Vaya I, Gustavsson T, Kotlyar A B, Zikich D, Sket P, Plavec J, Markovitsi D. J. Phys. Chem. C, 2012,116:14682-14689
70 Ren W, Zhang Y, Chen H, Gao Z, Li N, Luo H. Anal. Chem., 2016,88(5):1385-1390
71 He L, Sun X, Zhang H, Shao F. Angew. Chem. Int. Ed., 2018,57(38):12453-12457.
72 Sannohe Y, Endo M, Katsuda Y, Hidaka K, Sugiyama H. J. Am. Chem. Soc., 2010,132(46):16311-16313
73 Sathya S, Velu R, Maria C D, Bhaskar M M. Anal. Bioanal. Chem., 2019,411: 1319-1330
74 Liu Z, Luo X, Li Z, Huang Y, Nie Z, Wang H H, Yao S. Anal. Chem., 2017,89(3):1892-1899
75 Luo X, Xue B, Feng G, Zhang J, Lin B, Zeng P, Li H, Yi H, Zhang X L, Zhu H, Nie Z. J. Am. Chem. Soc., 2019,141(13):5182-5191
76 Shieh Y A, Yang S J, Wei M F, Shieh M J. ACS Nano, 2010,4(3): 1433-1442
77 Liu J, Wei T, Zhao J, Huang Y, Deng H, Kumar A, Wang C, Liang Z, Ma X, Liang X J. Biomaterials, 2016,91: 44-56
78 Wang K, You M, Chen Y, Han D, Zhu Z, Huang J, Williams K, Yang C Y, Tan W. Angew. Chem. Int. Ed., 2011,50(27):6098-6101
79 Yuan Q, Wu Y, Wang J, Lu D Q, Zhao Z L, Liu T, Zhang X B, Tan W H. Angew. Chem. Int. Ed., 2013,52: 13965-13969
80 Shi T H, Wang M L, Li H, Wang M, Luo X Y, Huang Y, Wang H H, Nie Z, Yao S Z. Sci. Rep., 2018,8(1):5551
81 Cheng M, Cui Y X, Wang J, Zhang J, Zhu L N, Kong D M. ACS Appl. Mater. Interfaces, 2019,11:13158-13167
Progress on Applications of G-quadruplex in Biochemical Analysis
LIU Zhuo-Liang1,2, TAO Cheng-An1, WANG Jian-Fang*1
1(Department of Biology and Chemistry, College of Liberal Arts and Sciences,
National University of Defense Technology, Changsha 410073, China)
2(State Key Laboratory of Chemo/Biosensing and Chemometrics,
College of Chemistry and Chemical Engineering, Hunan University, Changsha 410082, China)
Abstract G-quadruplex is a special secondary structure of nucleic acid. G-quadruplex can bind with hemin to form DNAzyme with peroxidase activity, and also it can enhance the fluorescence intensity of some dyes. As one of the functional nucleic acids, G-quadruplex is widely used in various biochemical analyses due to its high stability, specificity and flexibility. The application of G-quadruplex in biochemical analysis and its application prospects are briefly introduced.
Keywords G-quadruplex; Functional nucleic acid; DNAzyme/RNAzyme; Biochemical analysis; Review
(Received 13 July 2019; accepted 13 November 2019)
This work was supported by the National Natural Science Foundation of China (No. 21705163), the Natural Science Foundation of Hunan Province, China (No. 2019JJ50737), the Research Project of the National University of Defense Technology (No. ZK18-03-39) and the Open Subject of State Key Laboratory of Chemo/Biosensing and Chemometrics (No. 2017011).

