黄瓜幼苗下胚轴长度GWAS分析及候选基因挖掘
蔡和序,薄凯亮,周琪,苗晗,董邵云,顾兴芳,张圣平
黄瓜幼苗下胚轴长度GWAS分析及候选基因挖掘
蔡和序,薄凯亮,周琪,苗晗,董邵云,顾兴芳,张圣平
(中国农业科学院蔬菜花卉研究所/农业部园艺作物生物学与种质创制重点实验室,北京 100081)
【】挖掘与黄瓜幼苗下胚轴长度显著相关的SNP位点及候选基因,为揭示下胚轴长度的遗传基础和分子机制提供理论依据,为短下胚轴分子标记辅助选择育种奠定基础。以95份黄瓜核心种质为试验材料,分别于2016年春季、2017年春季、2017年秋季和2018年春季在中国农业科学院南口试验基地塑料大棚进行种植,在两叶一心期调查黄瓜幼苗的下胚轴长度;利用Structure 2.3.4软件分析群体结构,Haploview 软件分析连锁不平衡的衰减;基于最优模型对下胚轴长度进行全基因组关联分析(GWAS),依据关联SNP位点的LD区间序列,预测与下胚轴长度相关的重要关联候选基因,并利用荧光定量PCR对预测基因进行表达模式分析。共检测到8个显著关联的位点(),分别位于1、2、3、4、5、6号染色体,其中,等5个位点被重复检测到两次以上。通过分析关联SNP位点的LD区间序列,获得8个与黄瓜下胚轴长度有关的候选基因,其中既有光形态建成、泛素化、激素信号通路等调控基因,也有调控网络下游参与细胞生长发育,调节细胞大小,直接调控黄瓜下胚轴长度的基因。多基因在不同黄瓜材料中的有机分布,形成了具有不同下胚轴长度的黄瓜种质。基因表达分析显示在短下胚轴材料中高表达。在长下胚轴材料中高表达。检测到等8个与黄瓜下胚轴长度密切关联的SNP位点,挖掘到等8个调控下胚轴长度的候选基因。
黄瓜;下胚轴长度;全基因组关联分析;候选基因
0 引言
【研究意义】黄瓜作为一种重要的蔬菜作物,在全球范围内广泛栽培。目前黄瓜种植中大量采用工厂化穴盘育苗,单位面积幼苗数量越多即密度越大收益越高,带来的问题是容易造成幼苗下胚轴徒长。另外,育苗过程中如果遭遇高温、高湿、弱光等不良环境,也会导致幼苗下胚轴徒长,影响后期丰产性。因此,挖掘黄瓜下胚轴长度的调控位点和基因,开发准确实用的分子标记用于辅助培育短下胚轴品种,对于缓解下胚轴徒长和保证丰产具有重要意义。【前人研究进展】双子叶植物的下胚轴是连接两个胚胎叶(子叶)和初生根(胚根)的胚胎干[1]。下胚轴具有相对简单的结构,是一种非常可塑的器官,受到已知调节细胞伸长的外部和内部因素的强烈影响,例如光照[2-7]、温度[8]和激素[9-11],在高温高湿条件下,下胚轴徒长尤为严重。Bo等[12]从半野生西双版纳黄瓜中克隆了短下胚轴的基因SHORT HYPOCOTYL1(),该基因编码SMARCA3染色质重塑因子,它通过与CsHY5进行互作间接调控细胞伸长相关基因的表达,最终影响黄瓜下胚轴的伸长。苗晗等[13]通过下胚轴长度QTL定位检测到5个下胚轴长度相关QTL,在Chr.5上标记SSR15818和SSR06003之间检测到3个QTL,总贡献率高达61%;Chr.6上重复检出两个相邻位点和。全基因组关联分析(GWAS)是利用自然群体探测物种的遗传变异,进而挖掘与复杂农艺性状相关遗传位点的研究方法,具有不需要构建作图群体和一次性可同时检测多个等位基因位点的优势,近年来在拟南芥[14-15]、玉米[16]、水稻[17]等作物中均有广泛研究。【本研究切入点】随着黄瓜基因组测序的完成,利用高通量数据对黄瓜复杂性状开展GWAS研究越来越便利。QTL定位的方法仅能对在分离群体的亲本材料间存在差异的基因效应进行分析,无法在全基因组范围广泛挖掘参与下胚轴调控的基因。目前关于黄瓜下胚轴的GWAS研究尚未见报道。【拟解决的关键问题】采用GWAS分析鉴定与黄瓜下胚轴长度显著关联的SNP,确定控制性状变异的候选基因。
1 材料与方法
1.1 试验材料
试验材料为95份黄瓜核心种质资源,来源于中国农业科学院蔬菜花卉研究所。本核心种质是利用在染色体上均匀分布的23对高多态性的SSR标记,从 3 342份来源于世界各地的黄瓜资源中筛选出来的,具有广泛的代表性[18]。所有材料均为自然群体,材料编号及来源见电子附表1。
1.2 种植处理
分别于2016年春季、2017年春季、2017年秋季、2018年春季4个时期,将试验材料在28℃下催芽,出芽后播种于32孔穴盘,于中国农业科学院北京南口试验基地的温室育苗。每份材料设置3个重复,每个重复5株,随机区组排列。
1.3 下胚轴长度调查
黄瓜幼苗长至两叶一心时,用直尺测量下胚轴长度,记录原始数据后取平均值。表型数据分析采用SPSS20.0。
1.4 重测序分析与基因分型
整个核心种质群体均完成了全基因组的重测序[18],测序数据见黄瓜基因组网站(http://cucurbitgenomics. org)。过滤掉最小等位基因频率(MAF)<0.05的变异位点;只保留二态的变异位点。再根据次等位基因频率(minor allele frequency,MAF)>0.05 和完整度(即非N的SNP位点数占总SNP位点的比例)>0.8对SNP进行筛选,最终得到均匀分布于黄瓜7条染色体上的53 921个高质量且位置唯一的SNP 矩阵用于后续分析。
1.5 群体结构分析与连锁不平衡分析
群体结构由Structure 2.3.4软件基于贝叶斯数学模型进行分析,并参照QI等[18]确定最终亚群数目。本试验为了简化群体结构复杂性对GWAS结果的影响,K值取为3。用过滤后SNP矩阵进行全基因组连锁不平衡(linkage disequilibrium,LD)分析[19],分析采用软件Haploview进行[20]。
1.6 全基因组关联分析
将苗期调查的表型数据和过滤后构建的高质量SNP矩阵,利用基于R的GAPITR软件包[21],通过混合线性模型(mixed linear model,MLM)进行关联分析。利用ggplot2软件绘制Quantile-Quantile散点图(QQ plot)[22],并在此基础上利用QQman绘制曼哈顿图[23],显示关联分析检测到的与目标性状显著相关的标记位点。对95份黄瓜的苗期下胚轴性状进行全基因组关联分析。
1.7 候选基因预测
根据GWAS结果获得的SNP位点,根据序列位置将其定位到黄瓜基因组参考物理图谱上。基于群体连锁不平衡分析的LD区间,结合黄瓜基因组网站(http://cucurbitgenomics.org/)基因的功能注释信息,根据注释信息找出与性状相关的候选基因。
1.8 候选基因的表达分析
为了验证候选基因的准确性,在黄瓜核心种质中选取4个长下胚轴材料(CG9、CG11、CG106、CG112)和4个短下胚轴材料(CG19、CG44、CG54、CG98),在播种后子叶展平、第一片真叶展平、一叶一心、第二片真叶展平、两叶一心5个时期采集下胚轴。使用日本TaKaRa公司的RNA提取试剂盒(9767)提取总DNA,用1%琼脂糖凝胶电泳检测提取得到的RNA质量。用TaKaRa公司的反转录试剂盒将提取到的RNA反转录成cDNA。用Nanodrop仪器检测cDNA的浓度及纯度,用ddH2O将cDNA稀释到50 ng∙μL-1,用于荧光定量分析。根据预试验结果选择黄瓜作为内参基因;使用2-ΔΔCt计算基因的相对表达量。反应体系混合物的配置如表1,PCR反应程序如表2。

表1 荧光定量反应体系

表2 PCR反应程序
2 结果
2.1 表型统计分析
在2016年春季(16S)、2017年春季(17S)、2017年秋季(17A)和2018年春季(18S)4次不同环境下分别对95份黄瓜种质的下胚轴长度进行调查,下胚轴长度差异明显,统计分析结果见表3。4个批次中材料重复性较好,表现比较一致。长下胚轴材料如CG9、CG11、CG106、CG112等在不同批次中均下胚轴过长,表现为徒长的特性;而短下胚轴材料如CG19、CG44、CG54、CG98等在不同批次中下胚轴长度均较短。4个批次变异系数分别为32.32%、33.27%、26.28%和30.17%,下胚轴长度频次分布图具有显著的正态分布特征(图1),并且4个批次之间黄瓜下胚轴长度均显著相关(表4),表明下胚轴长度性状为典型的数量性状,采用GWAS分析方法可有效进行该性状的基因定位。

表3 黄瓜下胚轴长度统计分析
16S:2016年春季;17S:2017年春季;17A:2017年秋季;18S:2018年春季。下同
16S: spring of 2016; 17S: spring of 2017; 17A: autumn of 2017; 18S: spring of 2018. The same as below
2.2 群体结构与连锁不平衡分析
用过滤后的SNP矩阵进行全基因组连锁不平衡(linkage disequilibrium,LD)分析,当2从0.736衰减到0.368时,所对应的物理距离为17.3 kb(图2)。因此,黄瓜的全基因组平均 LD为17.3 kb(当2值衰减到其最大值一半时对应的物理长度,即为全基因组平均LD)。

表4 4个批次黄瓜下胚轴长度的相关性分析
** 在0.01 水平(双侧)上显著相关
** Significantly correlated at 0.01 level (bilateral)
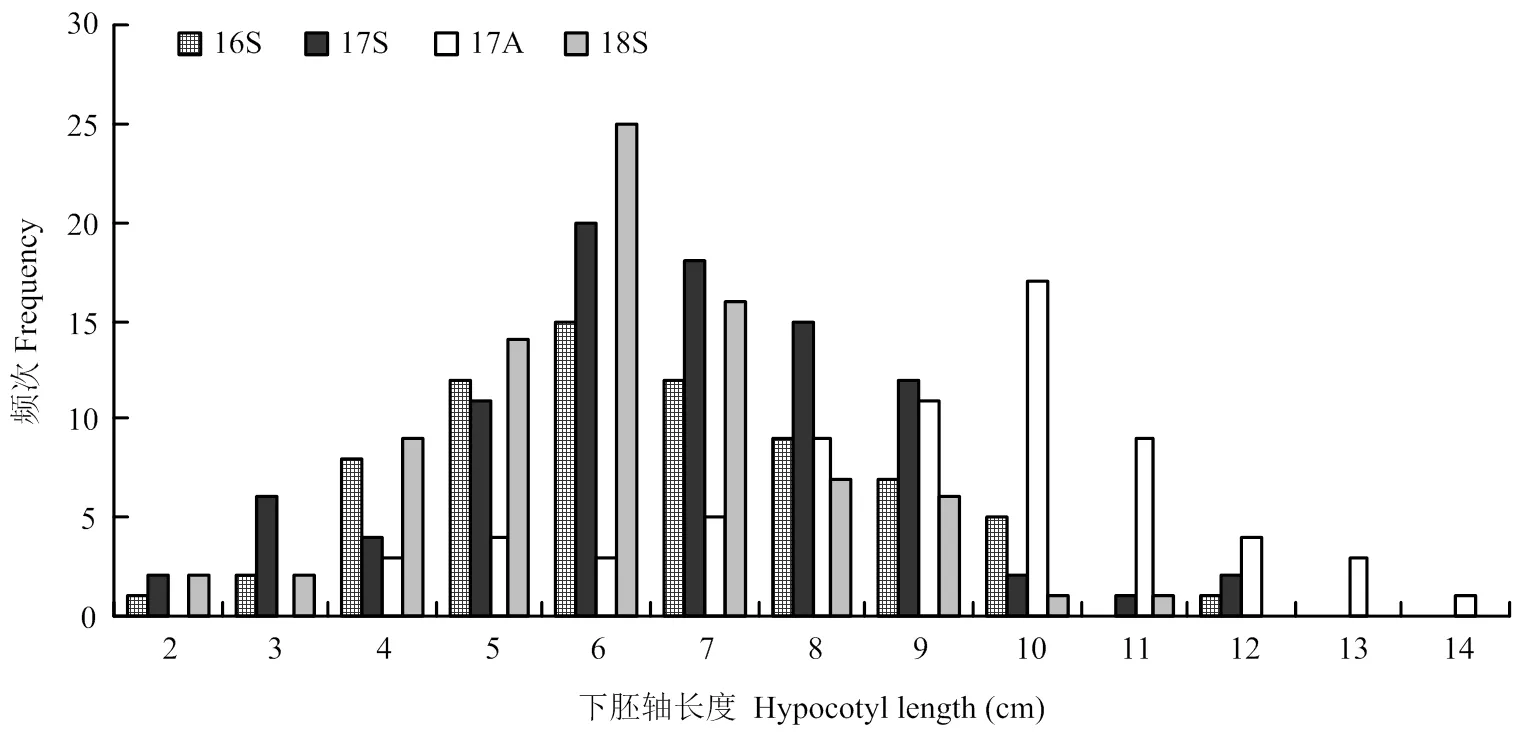
图1 黄瓜下胚轴长度的频次分布
2.3 全基因组关联分析
对黄瓜核心种质苗期下胚轴长度进行全基因组关联分析(图3),根据全基因组连锁不平衡结果,划分4个批次检测出的位点所对应的LD区间,对重叠区间进行合并,合并后共8个区间,检测到8个关联位点,分别位于第1、2、3、4、5、6号染色体,这些位点可解释的表型变异率为13.67%— 31.87%。被检测到3次被检测到两次(电子附表2)。说明是在不同季节稳定存在的关联位点。
2.4 候选基因挖掘
利用已公布的黄瓜基因组测序结果(9930v2),将与下胚轴长度显著关联的SNP标记定位到黄瓜基因组上,进而在显著关联的SNP位点侧翼序列40 kb范围内提取基因。根据黄瓜基因组的注释信息,筛选出候选区段内与下胚轴长度相关的同源基因。本研究共筛选出、、、等8个与下胚轴长度相关的候选基因(表5)。其中,、参与植物细胞形态形成,可能直接参与下胚轴的伸长,、可能通过影响光形态发生来调控下胚轴伸长,可能通过蛋白质泛素化来调控下胚轴伸长,、则参与了RNA的加工,可能通过转录翻译的调控来控制下胚轴伸长,则可能通过参与生长素转运,影响细胞的生长,最终调控下胚轴的伸长。在这些候选基因中,、、、和对应的位点被重复检测到。

图2 95份黄瓜全基因组连锁不平衡衰减

表5 候选基因及注释信息
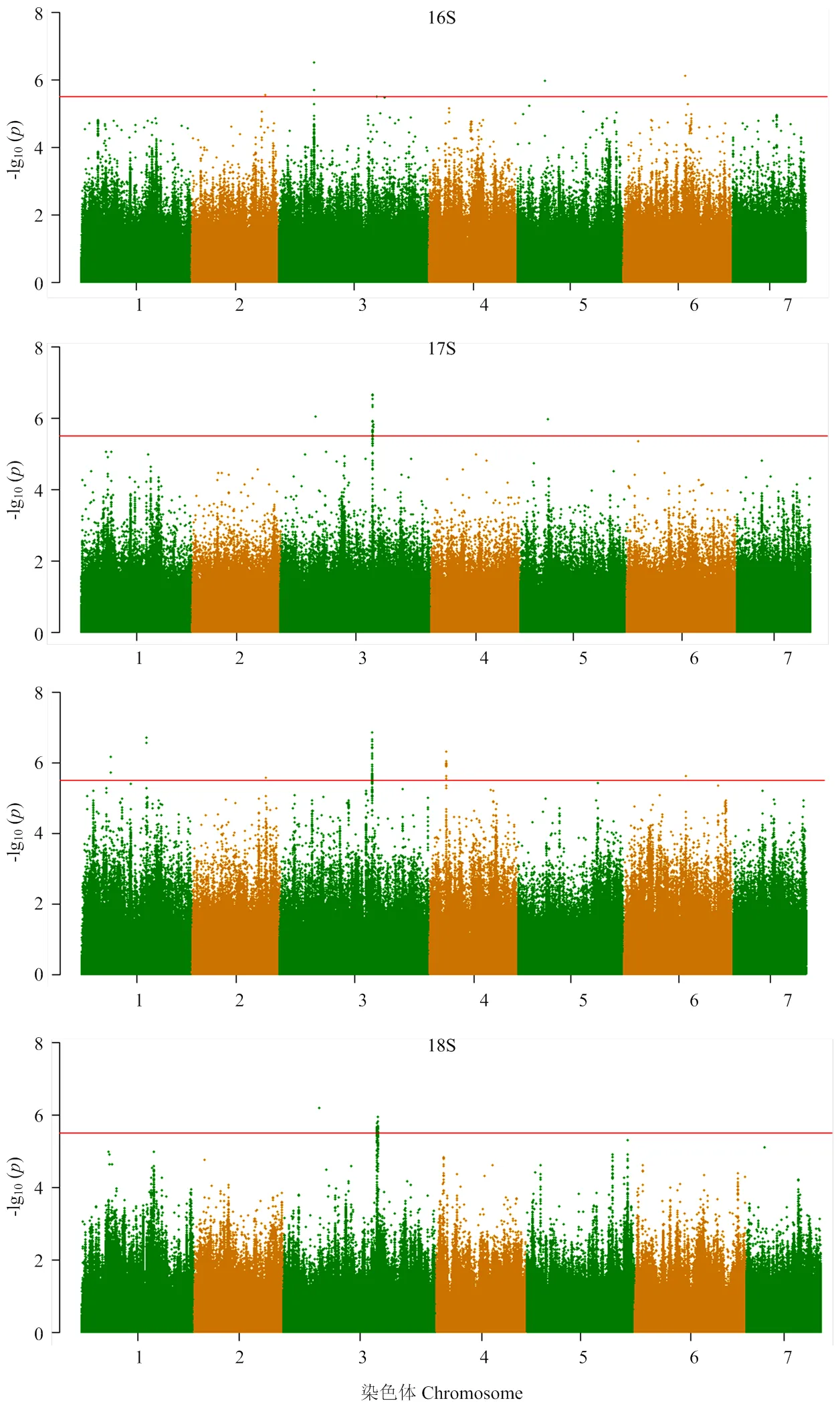
图3 95份黄瓜核心种质幼苗下胚轴长度的全基因组关联分析曼哈顿图
2.5 候选基因的表达模式分析
为了验证候选基因的准确性,在黄瓜核心种质中选取在4个批次中表现一致的4个长下胚轴材料(CG9、CG11、CG106、CG112)和4个短下胚轴材料(CG19、CG44、CG54、CG98),在播种后子叶展平、第一片真叶展平、一叶一心、第二片真叶展平、两叶一心5个时期采集下胚轴进行RNA提取,反转录后发现其比值均在1.8—2.0,表明提取的RNA质量合格。在琼脂糖凝胶电泳的胶图上有两条清晰可见的条带,说明RNA有较好的完整性(图4)。用候选基因和内参的特异性引物分别对其进行荧光定量PCR,其溶解曲线为单峰,说明引物只扩增出单一条带,具有特异性。

图4 总RNA电泳检测
表型调查的数据分析表明,不同材料间下胚轴长度有显著差异(表6,图5)。CG9和CG112下胚轴长度最长,过度徒长;CG11和CG106下胚轴长度次之,徒长较重;CG19和CG44轻微徒长;CG54和CG98没有徒长,幼苗健壮。
荧光定量结果(图6)显示,()在CG54和CG98中表达量显著提高;()在CG19、CG44、CG54和CG98表达量显著提高;()在CG19和CG98表达量显著提高;()在CG9和CG11表达量显著提高;()在CG9、CG11、CG106和CG112中表达量显著提高;()在CG19和CG54中表达量显著提高;()仅在CG11和CG106中表达量显著降低;()在CG9、CG11和CG106表达量显著降低。总体来看,()、()、()、()、()在短下胚轴材料中高表达。()、()在长下胚轴材料中高表达。

两叶一心期CG54(左)与CG112(右)
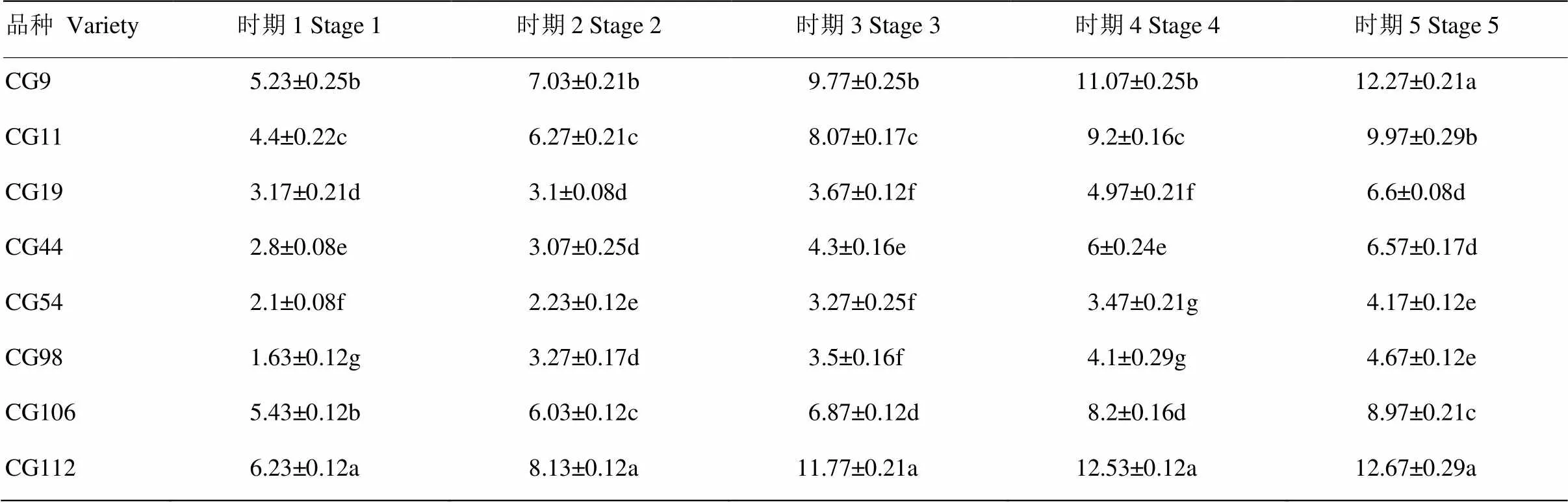
表6 qPCR材料苗期下胚轴长度
时期1:子叶展平;时期2:第一片真叶展平;时期3:一叶一心;时期4:第二片真叶展平;时期5:两叶一心;表中数据单位均为(cm);不同小写字母表示不同品种间差异达0.05显著水平(Duncan’s 法)
Stage 1: the cotyledon stage; Stage 2: the one true leaf stage; Stage 3: the one leaf and one bud stage; Stage 4: the two true leaves stage; Stage 5:the two true leaves and one bud stage; All data units in the table are (cm); Different lowercase letters indicate significant difference between different breeds at 0.05 level (Duncan's method)

CG9、CG11、CG106、CG112均为长下胚轴材料;CG19、CG44、CG54、CG98均为短下胚轴材料;每个候选基因中选取表达量最低的材料,其相对表达量设为1
3 讨论
3.1 黄瓜下胚轴长度的变异分析
下胚轴长度是影响黄瓜种苗的重要因素之一,长度过长会影响种苗的运输、定植后的成活率以及后期单株产量。因此,利用GWAS分析挖掘黄瓜下胚轴长度基因对于黄瓜短下胚轴育种具有重要的价值。本研究选取了来自于东亚、欧洲、美国、西双版纳、印度的黄瓜材料,是从世界各地3 000多份种质中筛选出来的,代表黄瓜75%以上的遗传变异[18],构建了一个具有丰富下胚轴长度变异的自然群体材料作为试验样本,保证了表型数据的多样性。另外,根据黄瓜实际生产中的条件,在温室中对核心种质进行4次下胚轴长度鉴定,幼苗长势和下胚轴长度均表现出明显差异。筛选出长下胚轴材料CG9、CG11、CG106及CG112,短下胚轴材料CG19、CG44、CG54、CG98等。同时,也有少部分下胚轴长度处于中间的材料在不同季节处理中表现不稳定,可能与处理条件、天气状况及温室内幼苗生物排布方式等有关。
3.2 黄瓜下胚轴长度的QTL定位
本研究通过对95份黄瓜核心种质苗期下胚轴长度的全基因组关联分析,共检测出8个显著关联的位点,这表明黄瓜的胚轴长度为微效多基因控制。在我国当前的黄瓜保护地生产中,大量采用南瓜作砧木,通过嫁接一定程度上避免了下胚轴徒长的影响。已有研究大部分探究外部环境的生理生化指标对黄瓜下胚轴徒长的影响,例如光强、光质、温度和激素等[24-26],下胚轴遗传相关的内容较少,鲜有与之相关的位点和基因。
苗晗等[13]通过QTL定位检测到5个下胚轴长度相关QTL,在Chr.5上标记SSR15818和SSR06003之间检测到3个QTL,总贡献率高达61%;Chr.6上重复检出两个相邻位点和。本研究在5号和6号染色体各两次重复检测到1个位点和,但是物理位置相差较大,不是同一个位点。()是生长素信号通路因子,参与生长素转运调节,可能通过生长素途径调节细胞的生长发育,进而调控下胚轴的长度。
3.3 黄瓜下胚轴长度候选基因挖掘
光形态发生是一个关键的植物发育过程。Bo等[12]从半野生西双版纳黄瓜中克隆了短下胚轴的基因SHORT HYPOCOTYL1(),该基因编码SMARCA3染色质重塑因子,它通过与CsHY5进行互作间接调控细胞伸长相关基因的表达,最终影响黄瓜下胚轴的伸长。位于3号染色体,本研究在16年春季和17年春季两次均检测到与该基因紧密连锁的SNP(),相距仅50 kb,基本可以确定位点为。荧光定量结果显示该基因在以西双版纳为代表的短下胚轴材料中表达量显著下调。在拟南芥中,已证实转录因子ELONGATED HYPOCOTYL 5()与组蛋白脱乙酰酶HDA15直接互作,降低组蛋白H4乙酰化水平,参与光形态发生以抑制下胚轴细胞伸长[27-28]。本研究候选基因之一()是HY5在黄瓜中的同源基因,在16年春季和17年秋季被两次重复检测到,且在短下胚轴材料中高表达,该基因极有可能通过参与光形态发生调控黄瓜下胚轴的长度。
在17年春季、17年秋季和18年春季3次检测到了.2,候选基因作为催化亚基和辅助性蛋白质参与组蛋白等多种RNA的加工和降解[29]。基因的表达模式分析显示该基因在长下胚轴材料中显著高表达,很有可能作为各种信号通路的下游功能蛋白,影响细胞的生长发育,进而控制下胚轴长度。SCAR 3(,)在拟南芥中则通过调控肌动蛋白细胞骨架的方向来调节细胞的大小[30],主要表现在表皮毛和叶片的细胞中。在黄瓜中极有可能通过影响下胚轴细胞的大小来调控下胚轴的长度。
秋季温室通常是高温高湿的育苗环境,下胚轴徒长情况尤为严重,因此大量学者研究了高温下黄瓜幼苗下胚轴长度遗传情况[6]。高温情况下黄瓜幼苗下胚轴长度均呈连续变异和正态分布,符合多基因控制的数量性状遗传规律;黄瓜幼苗在高温下的下胚轴长度符合2对加性-显性-上位性主基因+加性-显性多基因遗传模型[8]。本研究在17年秋恰好遇到了高温高湿环境,绝大部分核心种质下胚轴略有徒长。仅17年秋检测到的(在拟南芥中的同源基因参与蛋白质泛素化的途径,与逆境下的反应有关。因此,该位点可能主要与高温条件下黄瓜下胚轴的伸长有关。
对候选基因的表达分析发现,多次重复检测到且表达量与表型一致的基因大多数是直接调控细胞的生长发育,进而可能直接调控下胚轴长度的基因。而其他基因多是光形态建成、泛素化、生长素信号通路等转录因子,推测应该是感知环境因素的调控基因,只在特定的环境诱导下表达,调控下游细胞发育相关基因,进而调控下胚轴长度。长下胚轴组与短下胚轴组相比,基因表达量趋势与表型大致相同,但也有一两个材料不相符,鉴于下胚轴长度是数量性状,可能是缺失该位点导致,说明这些基因不同程度地参与了下胚轴长度的调控。多基因在不同黄瓜材料中的有机分布,决定了不同的下胚轴长度,形成了具有不同下胚轴长度的黄瓜种质。
4 结论
采用MLM模型分析在4个季节中共检测到8个与黄瓜下胚轴长度显著关联的SNP位点,分别位于1、2、3、4、5、6号染色体。其中两次以上重复检测到的位点共5个。通过分析关联SNP位点的LD区间序列,找到8个与黄瓜下胚轴长度有关的候选基因。调控黄瓜下胚轴长度的候选基因功能多样,既有光形态建成、泛素化、激素信号通路等调控基因,也有调控网络下游的功能基因,参与细胞生长发育,调节细胞大小,直接调控黄瓜下胚轴的长度。
[1] LIN Y, SCHIEFELBEIN J. Embryonic control of epidermal cell patterning in the root and hypocotyl of., 2001, 128(19): 3697-3705.
[2] 金正爱, 司龙亭, 李丹丹, 高兴. 弱光下黄瓜幼苗叶片下胚轴长的遗传分析. 江苏农业科学, 2009(3): 158-161.
JIN Z A, SI L T, LI D D, GAO X. Genetic analysis of hypocotyl length in cucumber seedling leaves under weak light., 2009(3): 158-161.(in Chinese)
[3] 李丹丹, 司龙亭, 罗晓梅, 李涛. 弱光胁迫下黄瓜苗期下胚轴性状的遗传分析. 西北农林科技大学学报(自然科学版), 2009, 37(11): 113-119.
LI D D, SI L T, LUO X M, LI T. Genetic analysis of hypocotyl traits in cucumber seedling under low light stress., 2009, 37(11): 113-119. (in Chinese)
[4] 张冠英, 司龙亭, 李丹丹. 弱光胁迫下黄瓜幼苗下胚轴性状QTL分析. 园艺学报, 2011, 38(2): 295-302.
ZHANG G Y, SI L T, LI D D. QTL analysis for hypocotyl traits of cucumber seedlings under low light stress., 2011, 38(2): 295-302. (in Chinese)
[5] 邹士成, 李丹丹, 孙博华, 张成凤, 于思琦. 黄瓜幼苗下胚轴响应弱光胁迫的研究. 安徽农学通报, 2015, 21(19): 29-30.
ZOU S C, LI D D, SUN B H, ZHANG C F, YU S Q. Response of hypocotyl of cucumber seedlings to light stress., 2015, 21(19): 29-30. (in Chinese)
[6] SONG J L, CAO K, HAO Y W, SONG S W, SU W, LIU H C. Hypocotyl elongation is regulated by supplemental blue and red light in cucumber seedling., 2019, 707: 117-125.
[7] 肖苏琪, 王冰华, 曲梅, 高丽红. 冬春季节育苗温室补光光强对黄瓜幼苗质量的影响. 中国蔬菜, 2018(10): 40-45.
XIAO S Q, WANG B H, QU M, GAO L H. Effect of supplementary light intensity on quality of winter-spring cucumber seedling in solar greenhouse., 2018(10): 40-45. (in Chinese)
[8] 张子默, 卢俊成, 齐晓花, 许学文, 陈学好, 徐强. 高温下黄瓜幼苗下胚轴长度遗传效应的研究. 分子植物育种, 2019, 17(4): 1326-1332.
ZHANG Z M, LU J C, QI X H, XU X W, CHEN X H, XU Q. Study on genetic effects of hypocotyl length in cucumber seedlings under high temperature., 2019, 17(4): 1326-1332. (in Chinese)
[9] 董春娟, 曹宁, 王玲玲, 张焕欣, 王红飞, 台连丽, 尚庆茂. 黄瓜子叶源生长素对下胚轴不定根发生的调控作用. 园艺学报, 2016, 43(10): 1929-1940.
DONG C J, CAO N, WANG L L, ZHANG H X, WANG H F, TAI L L, SHANG Q M. Regulatory roles of cotyledon-generated auxin in adventitious root formation on the hypocotyls of cucumber seedling., 2016, 43(10): 1929-1940. (in Chinese)
[10] LOPEZ-JUEZ E, KOBAYASHI M, SAKURAI A, KAMIYA Y, KENDRICK R E. Phytochrome, gibberellins, and hypocotyl growth (a study using the cucumber (L.) long hypocotyl mutant)., 1995, 107(1): 131-140.
[11] DAN H, IMASEKI H, WASTENEYS G O, KAZAMA H. Ethylene stimulates endoreduplication but inhibits cytokinesis in cucumber hypocotyl epidermis., 2003, 133(4): 1726-1731.
[12] BO K L, WANG H, PAN Y P, BEHERA T K, PANDEY S, WEN C L, WANG Y H, SIMON P W, LI Y H, CHEN J F, WENG Y Q. SHORT HYPOCOTYL 1 encodes a SMARCA3-like chromatin remodeling factor regulating elongation., 2016, 172: 1273-1292.
[13] 苗晗, 顾兴芳, 张圣平, 张忠华, 黄三文, 王烨, 方智远. 黄瓜苗期主要农艺性状相关QTL定位分析. 园艺学报, 2012, 39(5): 879-887.
MIAO H, GU X F, ZHANG S P, ZHANG Z H, HUANG S W, WANG Y, FANG Z Y. Mapping QTLs for seedling-associated traits in cucumber., 2012, 39(5): 879-887. (in Chinese)
[14] ATWELL S, HUANG Y S, VILHJALMSSON B J, WILLEMS G, HORTON M, LI Y, MENG D, PLATT A, TARONE A M, HU T T, JIANG R, MULIYATI N W, ZHANG X, AMER M A, BAXTER I, BRACHI B, CHORY J, DEAN C, DEBIEU M, de MEAUX J, ECKER J R, FAURE N, KNISKERN J M, JONES J D, MICHAEL T, NEMRI A, ROUX F, SALT D E, TANG C, TODESCO M, TRAW M B, WEIGEL D, MARJORAM P, BOREVITZ J O, BERGELSON J, NORDBORG M. Genome-wide association study of 107 phenotypes ininbred lines., 2010, 465(7298): 627-631.
[15] BRACHI B, MORRIS G P, BOREVITZ J O. Genome-wide association studies in plants: The missing heritability is in the field., 2011, 12(10): 232.
[16] WANG M, YAN J B, ZHAO J R, SONG W, ZHANG X B, XIAO Y N, ZHENG Y L. Genome-wide association study (GWAS) of resistance to head smut in maize., 2012, 196: 125-131.
[17] HUANG X H, WEI X H, SANG T, ZHAO Q A, FENG Q, ZHAO Y, LI C L, ZHU C R, LU T T, ZHANG Z W, LI M, FAN D L, GUO Y L, WANG A H, WANG L, DENG L W, LI W J, LU Y Q, WENG Q J, LIU K Y, HUANG T, ZHOU T Y, JING Y F, LI W, LIN Z, BUCKLER E S, QIAN Q, ZHANG Q F, LI J Y, HAN B. Genome-wide association studies of 14 agronomic traits in rice landraces., 2010, 42(11): 961-967.
[18] QI J J, LIU X, SHEN D, MIAO H, XIE B Y, LI X X, ZENG P, WANG S H, SHANG Y, GU X F, Du Y C, LI Y, LIN T, YUAN J H, YANG X Y, CHEN J F, CHEN H M, XIONG X Y, HUANG K, FEI Z J, MAO L Y, TIAN L, STADLER T, RENNER S S, KAMOUN S, LUCAS W J, ZHANG Z H, HUANG S W. A genomic variation map provides insights into the genetic basis of cucumber domestication and diversity., 2013, 45(12): 1510-1515.
[19] YANG J A, LEE S H, GODDARD M E, VISSCHER P M. A tool for genome-wide complex trait analysis., 2011, 88(1): 76-82.
[20] BARRETT J C, FRY B, MALLER J, DALY M J. Haploview: Analysis and visualization of LD and haplotype maps., 2005, 21(2): 263-265.
[21] ZHANG Z W, ERSOZ E, LAI C Q, TODHUNTER R J, TIWARI H K, GORE M A, BRADBURY P J, YU J M, ARNETT D K, ORDOVAS J M, BUCKLER E S. Mixed linear model approach adapted for genome-wide association studies., 2010, 42(4): 355-360.
[22] BRADBURY P J, ZHANG Z W, KROON D E, CASSTEVENS T M, RAMDOSS Y, BUCKLER E S. TASSEL: Software for association mapping of complex traits in diverse samples., 2007, 23(19): 2633-2635.
[23] ZHANG Z W, ERSOZ E, LAI C Q, TODHUNTER R J, TIWARI H K, GORE M A, BRADBURY P J, YU J M, ARNETT D K, ORDOVAS J M, BUCKLER E S. Mixed linear model approach adapted for genome-wide association studies., 2010, 42(4): 355-360.
[24] DAN H, IMASEKI H, WASTENEYS G O, KAZAMA H. Ethylene stimulates endoreduplication but inhibits cytokinesis in cucumber hypocotyl epidermis1., 2003, 133(4): 1726-1731.
[25] ZHAO Q X, YUAN S, WANG X, ZHANG Y L, ZHU H, LU C M. Restoration of mature etiolated cucumber hypocotyl cell wall susceptibility to expansin by pretreatment with fungal pectinases and egta., 2008, 147(4): 1874-1885.
[26] KOEDA S, SATO K, SAITO H, NAGANO A J, YASUGI M, KUDOH H, TANAKA Y. Mutation in the putative ketoacyl-ACP reductase CaKR1 induces loss of pungency in., 2019, 132(1): 65-80.
[27] ZHAO L M, PENG T, CHEN C Y, JI R J, GU D C, LI T T, ZHANG D D, TU Y S, WU K Q, LIU X C. HY5 interacts with the histone deacetylase HDA15 to repress hypocotyl cell elongation in photomorphogenesis., 2019, 180(3): 1450-1466.
[28] CHENGE-ESPINOSA M, CORDOBA E, ROMERO-GUIDO C, TOLEDO-ORTIZ G, LEON P. Shedding light on the methylerythritol phosphate (MEP)-pathway: Long hypocotyl 5 (HY5)/phytochrome- interacting factors (PIFs) transcription factors modulating key limiting steps., 2018, 96(4): 828-841.
[29] RAIJMAKERS R, NOORDMAN Y E, van VENROOIJ W J, PRUIJN G J M. Protein-protein interactions of hCsl4p with other human exosome subunits., 2002, 315(4): 809-818.
[30] BREMBU T, WINGE P, SEEM M, BONES A M. NAPP and PIRP encode subunits of a putative wave regulatory protein complex involved in plant cell morphogenesis., 2004, 16(9): 2335-2349.
GWAS Analysis of Hypocotyl Length and Candidate Gene Mining in Cucumber Seedlings
CAI HeXu, BO KaiLiang, ZHOU Qi, MIAO Han, DONG ShaoYun, GU XingFang, ZHANG ShengPing
(Institute of Vegetables and Flowers, Chinese Academy of Agricultural Sciences, Beijing 100081)
【】The aim of this study was to identify SNP loci and candidate genes significantly correlated with cucumber hypocotyl length trait, which could provide a theoretical basis for revealing the genetic basis and molecular mechanism of cucumber hypocotyl length trait, and lay a foundation for marker-assisted selection breeding of cucumber hypocotyl length trait.【】The natural population including 95 cucumber germplasm was employed in this study, and seedlings were grown in the plastic house in Nankou Experimental Field of Chinese Academy of Agricultural Sciences in spring 2016, spring 2017, autumn 2017 and spring 2018, respectively. The hypocotyl length was measured at the two true leaves stage. Structure 2.3.4 software was used to analyze the population structure, and Haploview software was used to analyze the attenuation of linkage imbalance. Then, the whole genome association analysis of hypocotyl length was carried out based on the optimal model. The important candidate genes related to hypocotyl length were predicted according to the LD interval sequence of the associated SNP loci, and the expression pattern of candidate genes were performed by fluorescence quantitative PCR. 【】A total of 8 loci, includingandwere detected on Chr. 1, 2, 3, 4 and 5, respectively. Five of them,and, were detected repeatedly in two or more different environments. By analyzing the LD interval sequences of the associated SNP loci, eight candidate genes,and, were predicted, which were related to cucumber hypocotyl length. Some of the candidate genes involved in regulating plant photomorphogenesis, ubiquitination, and hormone signaling pathway. And some of them were downstream genes regulating cell growth, development and cell size, thus they directly regulated hypocotyl length. Thus, the varied distribution of above genes in different cucumber materials resulted in the different hypocotyl length cucumber germplasm. The organic distribution of polygenes in different cucumber materials formed cucumber germline with different Hypocotyl length. Gene expression analysis showed that,,,andwere highly expressed in short hypocotyl materials andandwere highly expressed in long hypocotyl materials.【】Eight SNP loci linked with hypocotyl length,and, were detected in this study. Eight candidate genes regulating hypocotyl length were predicted, including,,,,,,and.
cucumber; hypocotyl length; genome-wide association study; candidate gene
10.3864/j.issn.0578-1752.2020.01.012

2019-07-24;
2019-09-16
国家自然科学基金(31701929)、中央级公益性科研院所基本科研业务费专项(Y2017PT52)
蔡和序,E-mail:82101172229@caas.cn。薄凯亮,E-mail:bokailiang@caas.cn。蔡和序和薄凯亮为同等贡献作者。通信作者张圣平,E-mail:zhangshengping@caas.cn。通信作者顾兴芳,E-mail:guxingfang@caas.cn
(责任编辑 赵伶俐)

