人肝细胞癌中HNF4α近端启动子区生物信息学分析
曹 辉,许 钟,陈燕平,张玲玲,鲁 亮
肝核转录因子4(hepatocyte nuclear factor 4,HNF4)是细胞核激素受体家族中的核心成员之一,在调控肝细胞分化和维持其生物学功能上具有重要作用,其中HNF4α与肝脏特异基因的表达密切相关[1]。研究[2-3]表明HNF4α在肝细胞癌变中扮演着抑癌基因的角色,在肝硬化、肝癌中其表达明显下调。作为肝细胞中的一个重要的转录因子,HNF4α与超过40%肝基因的启动子区相结合[4],其重要性可见一斑,由此不难理解近年来肝脏疾病研究中HNF4α受到国内外学者的重视[5-7]。该研究拟应用生物信息学方法对HNF4α基因启动子区进行序列分析,预测该基因近端启动子区的转录因子结合位点, 进一步分析其在肝细胞癌中下调有关的转录因子,为研究其转录调控机制、探索肝癌治疗新思路提供理论基础。
1 材料与方法
1.1 基因启动子区序列的获得从美国国家生物技术信息中心在线核苷酸数据库(Gene Bank)获得HNF4α基因序列,基于HNF4α的基因序列ID,下载NCBI网站上外链接GeneCopoeia Inc.所提供的启动子序列。
1.2 启动子区转录因子结合位点的预测分析通过在线启动子区域预测分析软件PromotorScan(http://www-bimas.cit.nih.gov/molbio/proscan/)分析启动子序列,通过转录因子数据库TRANSFAC中的在线转录因子预测软件包括Patch、P-Match、AliBaba2(http://www.gene-regulation.com/pub/programs.html)预测HNF4α启动子序列顺式作用元件,分析其可能转录因子结合位点。
1.3 肝癌组织中相关转录因子的筛选应用在线肝脏基因数据库liveratlas(http://liveratlas.hupo.org.cn/)[8]筛查肝癌组织中表达下调的基因,与预测的转录因子进行对比,获得目标转录因子数据集。
1.4 相关性分析通过TCGA数据库(https://cancergenome.nih.gov/)提取相关转录因子与HNF4α的肝癌组织芯片表达结果,进行相关性分析。
1.5 统计学处理采用GraphPad Prism 5软件进行数据分析,相关性分析计算相关系数r,以P<0.05为差异有统计学意义。
2 结果
2.1 人HNF4α基因基本信息及启动子序列人HNF4α基因ID:3 172,定位于20号染色体正链(20q13.12),全长77 045 bp,其mRNA ID包括:NM_000457.4、NM_001030003.2、NM_001030004.2、NM_001258355.1、NM_001287182.1、NM_001287 183.1、NM_001287184.1、NM_175914.4、NM_178849.2和NM_178850.2。
通过ACEVIEW(https://www.ncbi.nlm.nih.gov/IEB/Research/Acembly/)了解其转录本情况,选择NM_000457.4,对应的基因启动子克隆产品ID为HPRM20338,HPRM30433。
提取两者序列,应用NCBI Blast比对,HPRM30433序列包含HPRM20338转录起始位点(TSS)上游的全部序列,故取HPRM30433序列来进一步分析,其全长1 471 bp,位于转录起始位点-1 371 bp至+99 bp之间。
2.2 人HNF4α基因启动子区转录因子结合位点预测分析结果应用PromotorScan分析启动子序列,分析结果共35个转录因子结合位点,其中Sp1结合位点共16个、AP-2共6个。去重后涵盖AP-2、APRT-mouse_US、EARLY-SEQ1、GCF、HSV-tk-2nd_distal_si、JCV_repeated_sequenc、LF-A2、PuF、SDR_RS、Sp1、T-Ag、UCE.2共12个转录因子。
应用Patch在线软件分析,参数设置set of sites选择“vertebrates”,Lower score boundary设为“90”,其它采用默认设置,预测结果共1 567个转录因子结合位点,手工筛查物种为人的转录因子结合位点共785个,经汇总去重,共涵盖了161个转录因子。
应用P-Match在线软件分析,当Cut-offs使用参数为“to minimize the sum of both error rates”时,预测到17个转录因子结合位点(图1),去除重复共涵盖12个转录因子:AP-4、ARP-1、CDP CR3、COUP-TF、CREB、c-Rel、E47、HNF-4、NF-kappaB、NRF-2、Olf-1、ZID;当使用参数为“to minimize false negative matches”,共预测到324个转录因子结合位点,经汇总去重,共涵盖27个转录因子:AP-4、AREB6、ARP-1、BSAP、CDP、CDP CR3、COUP-TF、CREB、c-Rel、E47、Elk-1、Evi-1、HFH-1、HLF、HNF-4、myogenin/NF-1、NF-E2、NF-kappaB、NF-kappaB(p50)、NF-kappaB(p65)、Nkx2-5、NRF-2、Olf-1、RREB-1、STATx、YY1、ZID。

图1 P-Match预测转录因子结合位点结果图
应用AliBaba2在线软件分析,取在线软件默认设置参数,预测结果共164个转录因子结合位点,经汇总去重,共涵盖了61个转录因子。
以上软件预测结果汇总后去除重复,结果共涵盖了225个转录因子。
2.3 筛查肝癌中人HNF4α启动子区可能转录因子应用在线基因数据库liveratlas检索,选择“Disease”,以“hepatocellular carcinoma”为检索词检索,依次点击“HuLDi00052”和“more...”,提取肝细胞癌中表达下调的基因共2 538个,与上述预测的225个转录因子进行对比,取交集获得目标转录因子。结果预测的转录因子中共有17个在肝细胞癌中表达下调:Egr-1、GATA-3、HLF、IRF-1、MAZ、MyoD、Pax-5、POU1F1a、RelA、RREB-1、RXR-alpha、SMAD-3、Sp1、TBP、TCF-4、USF2、WT1。
2.4 表达相关性分析从TCGA数据库中提取以上17个转录因子及HNF4α在癌旁组织及肝细胞癌中表达的数据,用GraphPad Prism 5软件进行相关性分析及绘图,结果显示HLF、RREB1、RXRA等三个转录因子与HNF4α的表达成正相关,计算的Pearson相关系数r分别为0.553 4、0.407 9、0.424 7,P<0.000 1,差异有统计学意义(图2~4)。
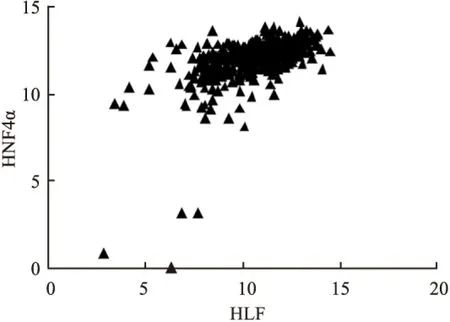
图2 转录因子HLF与HNF4α的相关性分析
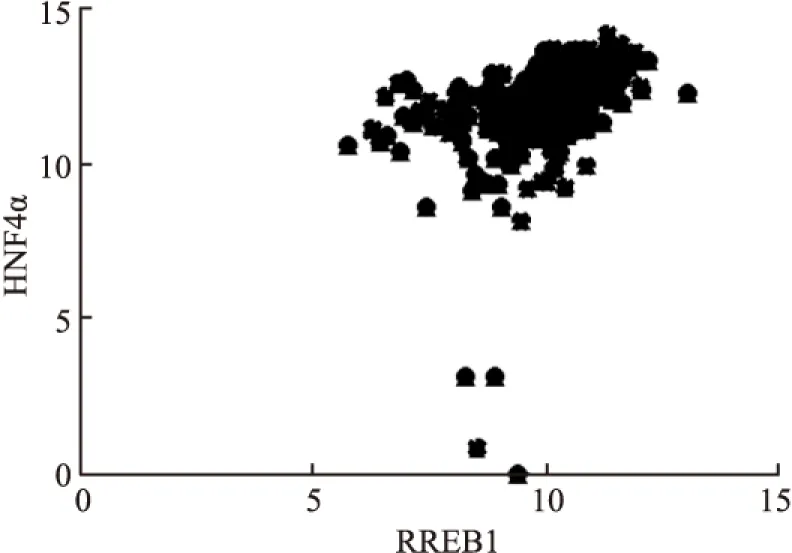
图3 转录因子RREB1与HNF4α的相关性分析
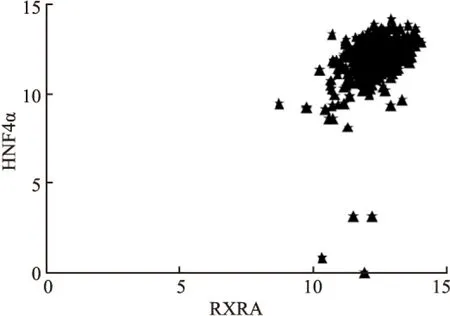
图4 转录因子RXRA与HNF4α的相关性分析
3 讨论
研究抑癌基因表达调控机制既有助于揭开恶性肿瘤发生发展的分子机制,也可为探索癌症治疗新策略提供新的思路。肝癌是我国和世界上最常见的恶性肿瘤之一,基于临床样本检测的研究越来越丰富[9],多种芯片的数据也通过在线数据库公开报道。对这些数据进行深度分析,结合启动子区序列转录因子的生物信息学分析,可为抑癌基因转录调控研究提供更精准的方向。
HNF4α是肝癌发生中受关注的抑癌基因之一,相关的信号通路研究表明JNK/c-Jun、Notch和MAPK等信号通路可抑制HNF4α的功能或抑制其表达[10-12],PI3K/AKT信号通路可以促进其表达[13]。而对HNF4α转录调控机制的进一步解析,可在分子水平上更充分的阐明其表达调控机制,有助于深入认识肝细胞癌变的机制。
HNF4α在肝癌发生中表达受抑制,与之相对应,调控其表达的转录因子在肝癌中的表达通常是下调的。本研究应用北京蛋白质组研究中心/蛋白质组国家重点实验室建立的在线肝脏基因数据库liveratlas[8],基于公开发表的芯片等数据提取到在肝细胞癌中表达下调的基因共2 538个。在NCBI的基因数据库中获得HNF4α的启动子序列后,应用多种启动子序列分析软件来预测其顺式作用元件,合并多个软件预测结果以减少遗漏的可能,预测结果共对应225个转录因子。通过两组数据的交集,有效缩小了在肝癌中研究HNF4α的转录调控蛋白的范围。
随后,通过TCGA数据库,详细地分析了潜在转录因子与HNF4α表达的相关性,很幸运地从17个潜在目标中找到3个转录因子与HNF4α表达成正相关, 使进一步的实验研究变得更加可行。通过文献检索,未见HLF、RREB1与HNF4α之间相互作用的报道;Tomaru et al[14]通过Matrix RNAi(结合siRNA敲除和QRT-PCR)在HepG2细胞系中的研究结果表明,RXRA可调控HNF4α的表达,为本研究的分析结果提供了佐证,下一步将在此基础上,通过小鼠肝癌模型等体内实验来验证。
深度的数据发掘,可以借助于综合利用公开的数据库资源、结合生物信息学分析、充分应用计算机和网络资源来实现其价值。尽管预测分析结果的真
实情况有待体内外实验的评价,但通过对生物信息的获取与加工、分析与阐释,可有效缩小在肝癌发生中HNF4α转录调控机制探索的研究范围,节约成本和资源,提高研究效率,是一行之有效的途径,值得推广应用。

