基于SNP标记的西瓜种质资源遗传多样性分析
易丽聪 王运强 焦春海 姚明华 龚钰 王舒景 戴照义
摘 要:种质资源遗传背景狭窄是当前西瓜育种面临的一大瓶颈。利用449个SNP标记对64份西瓜材料进行遗传多样性分析,结果表明,平均多态性信息含量(PIC)为0.229,平均Neis多样性指数(H) 为0.28,平均Shannon 多样性指数(I)为 0.43,平均期望杂合度(He)为0.282;64份材料的最优群体结构数为4,各亚群材料的地理来源与遗传关系没有显著相关性;材料之间的平均遗传距离为0.175。农艺性状统计分析表明,4个亚群在首雌花节位、坐果节位和果皮厚度3个性状之间存在显著差异。本研究将为西瓜种质资源创制、杂交育种的亲本选择和培育优异、适应性强的西瓜新品种提供理论依据。
关键词:西瓜;种质资源;遗传多样性;SNP标记
中图分类号:S651 文献标志码:A 文章编号:1673-2871(2020)12-008-06
Abstract: Watermelon is an important fruit in China, the narrow genetic background of germplasm resources is a bottleneck problem in watermelon breeding, 449 SNP markers were used to analyze the genetic diversity of 64 watermelon germplasms in this research. The results showed that, the average polymorphism information content (PIC) was 0.229, the average Nei's diversity index (H) was 0.28, the average Shannon's information index (I) was 0.43, and the average expected heterozygosity (He) was 0.282. The optimal subgroup number was 4, however, no significant correlation was found between the subpopulation and geographic origin. The average genetic distance between the 64 watermelon accessions was 0.175. According to the agronomic traits statistical analysis, the traits of the first female flower node, fruiting node and rind thickness were observed significant differences among the four subgroups. The results of this study will provide theoretical guidance for the creation of watermelon germplasm resources, the selection of parents in cross breeding and the breeding of new watermelon varieties with excellent and strong adaptability.
Key words: Watermelon; Germplasm; Genetic diversity; SNP markers
西瓜原產于非洲,在我国有上千年的栽培驯化历史,不同生态区驯化产生了一系列形态特征差异明显、各具特色的地方品种,如华北地区的‘三白瓜、兰州的‘大花皮和江浙地区的‘马铃瓜等[1]。近年来,西瓜的种植面积和范围迅速扩大,成为重要的经济作物。目前,我国西瓜总生产面积达150万hm2,面积、产量和销量均居世界第一(FAOSTAT)。但是,长期的人工选择和驯化也带来许多问题,最突出的是育种材料遗传多样性狭窄[2-3],从而导致品种同质化严重,培育突破性新品种日益困难。
明确育种材料的遗传背景有助于杂交育种中亲本的选择,进而提高培育杂种优势明显的新品种的效率。20世纪80年代,分子标记技术的出现迅速推动了对植物各性状调控基因的定位、克隆和分子机制的解析[4-6]。在许多作物中与抗病、雄性不育、生育期等重要性状相关的分子标记已经应用[7-10]。利用分子标记进行材料的遗传背景鉴定和种质资源核酸指纹构建也在大部分植物中展开[11-13]。
西瓜是较早完成基因组测序的植物之一[14],北京市农林科学院蔬菜研究中心基于对西瓜属7个种的414份资源的基因组重测序共鉴定出19 725 853个SNPs和6 675 290个InDels(Insertion-Deletion)标记[15]。该结果为西瓜种质资源鉴定、核酸指纹图谱构建和基因定位提供了丰富的分子标记。在此基础上,笔者挑选了覆盖西瓜11条染色体的449个SNP位点,利用高通量SNP分型对64份西瓜自交系材料进行遗传背景鉴定,解析其遗传多样性和群体结构,为西瓜杂交育种的亲本选配和资源利用提供理论依据。
1 材料与方法
1.1 材料
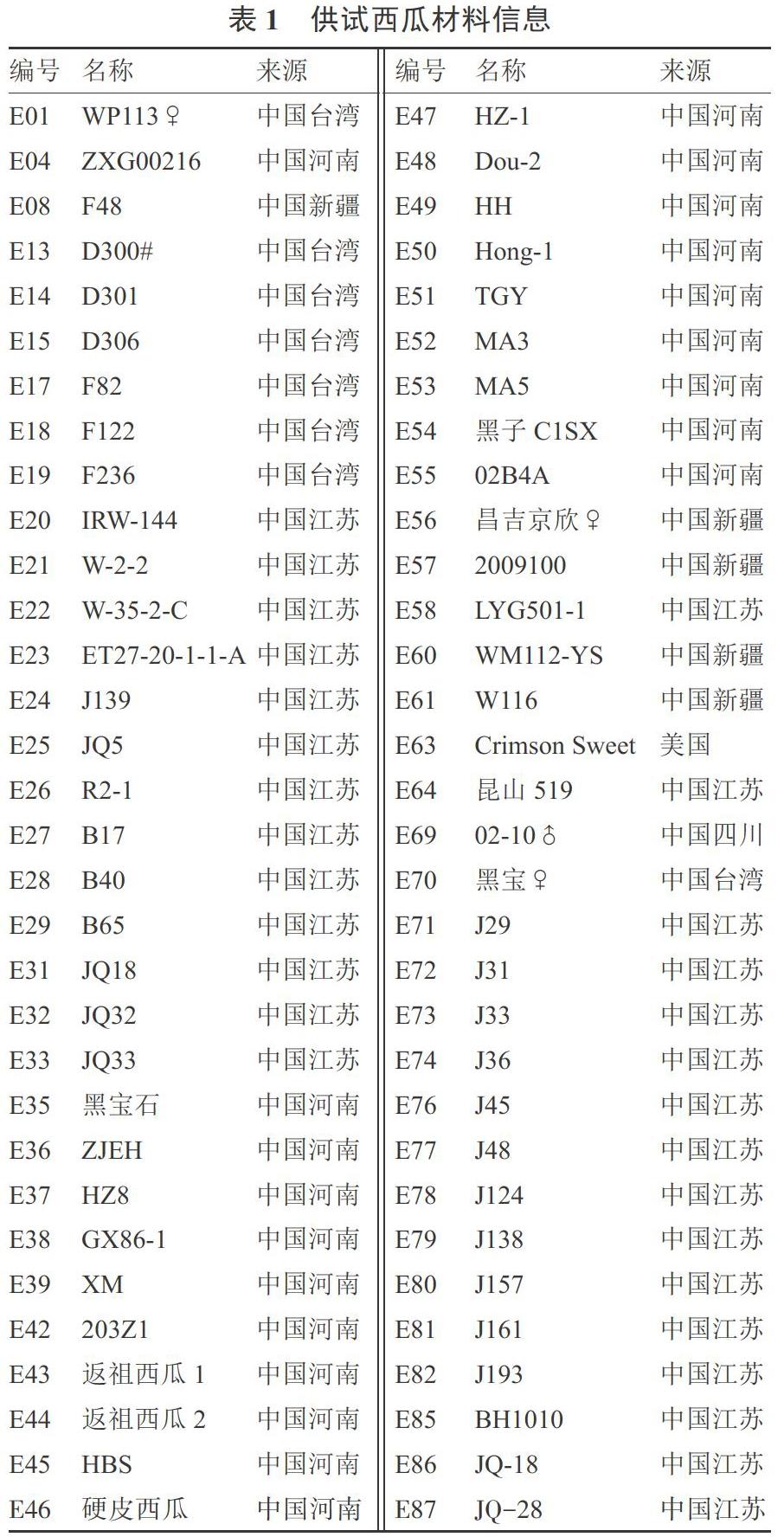
64份西瓜自交系材料为湖北省农业科学院经济作物研究所收集并保存,材料编号、名称和来源见表1。
1.2 表型性状调查
64份自交系材料的表型数据引用戴照义等[16],这些材料于2013 年春季种植在湖北省农科院蔬菜基地,每份材料定植20株,坐瓜方式采用人工辅助授粉。性状描述、数据采集和记录参照《西瓜种质资源描述规范和数据标准》[17],每份材料取10株状态良好、生长一致的植株进行性状采集。可溶性固形物含量由手持式折光仪测定。
1.3 分子标记和基因型分析
SNP位点信息来源于公开发表的文献[15]和葫芦科作物基因组数据库 (CuGenDB)(ftp://cucurbitgenomics.org/pub/cucurbit/reseq/watermelon/v2/)。选取在西瓜11条染色体上均匀分布的449个SNP位点进行测检和分析。
2019年11月对64份西瓜资源进行浸种催芽(55 ℃温汤浸种,32 ℃温箱催芽),取1~2 cm长胚根,采用CTAB法提取总DNA,该试验在蔬菜种质创新与遗传改良湖北省重点实验室完成。随后将DNA送至武汉基诺赛克科技有限公司进行后续检测和分析,主要步骤如下:首先用Qubit 3.0检测DNA质量和浓度;再通过多重PCR捕获目标SNP位点;最后利用Illumina 2500平台进行SNP分型。
1.4 数据分析
使用Cervus 3.0.7 软件计算多态性信息含量(PIC)。使用POPGENE 1.32 软件计算Nei's基因多样性指数(H)、Shannon 信息指数(I)和期望杂合度(He)。使用STRUCTURE 2.3.4软件[18]对64份材料进行亚群划分,burnin period 和MCMC iterations 参数分别为 10 000和100 000,亚群数目(K)设置为1~10个,每个K值独立运算10次,根据ΔK法确定最佳亚群数。根据最大遗传比例进行亚群归类。使用PLINK 1.9[19]软件进行主成分分析(PCA)。利用MEGA 7.0.14 [20]软件计算材料的遗传距离,构建基于遗传距离的UPGMA聚类树和基于SNP的系统发育树。系统发育树采用NJ(Neighbor-Joining)法,Bootstrap值设置为1 000。采用单因素方差分析(one-way ANOVA)和Tukeys多重比较统计性状差异。
2 结果与分析
2.1 遗传多样性
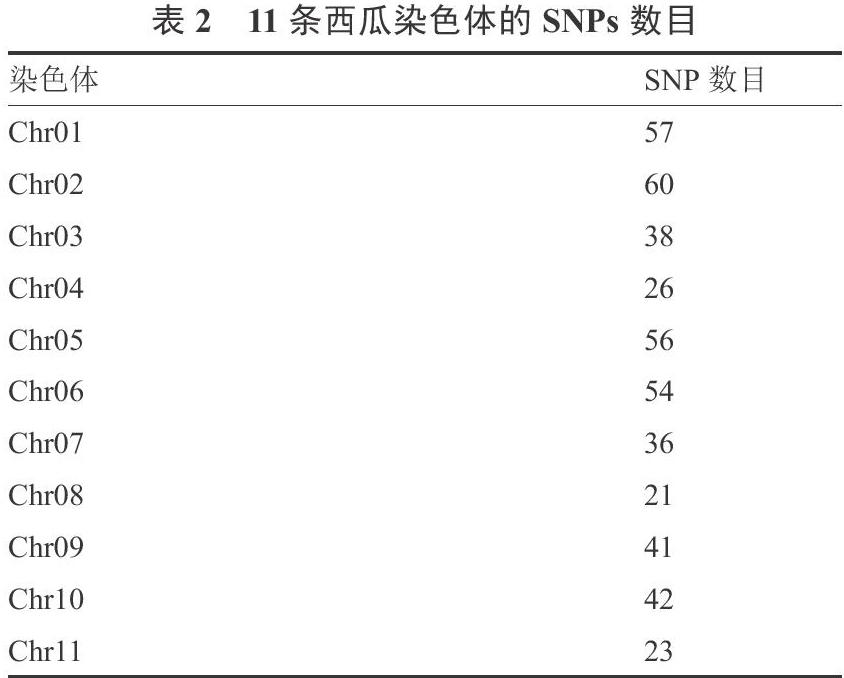
利用高通量SNP分型的方法对64份普通栽培西瓜材料进行检测,共检测到449个具有多态性的有效SNP位點,每条染色体的SNP位点见表2。所有位点均包含2个等位基因,位点PIC值在0.015~0.375之间,平均0.229。Neis 基因多样性指数(H)在 0.016~0.500之间,平均0.28。平均Shannon 信息指数(I)为 0.43。期望杂合度(He)在0.016~0.504之间,平均0.282。
2.2 群体结构分析
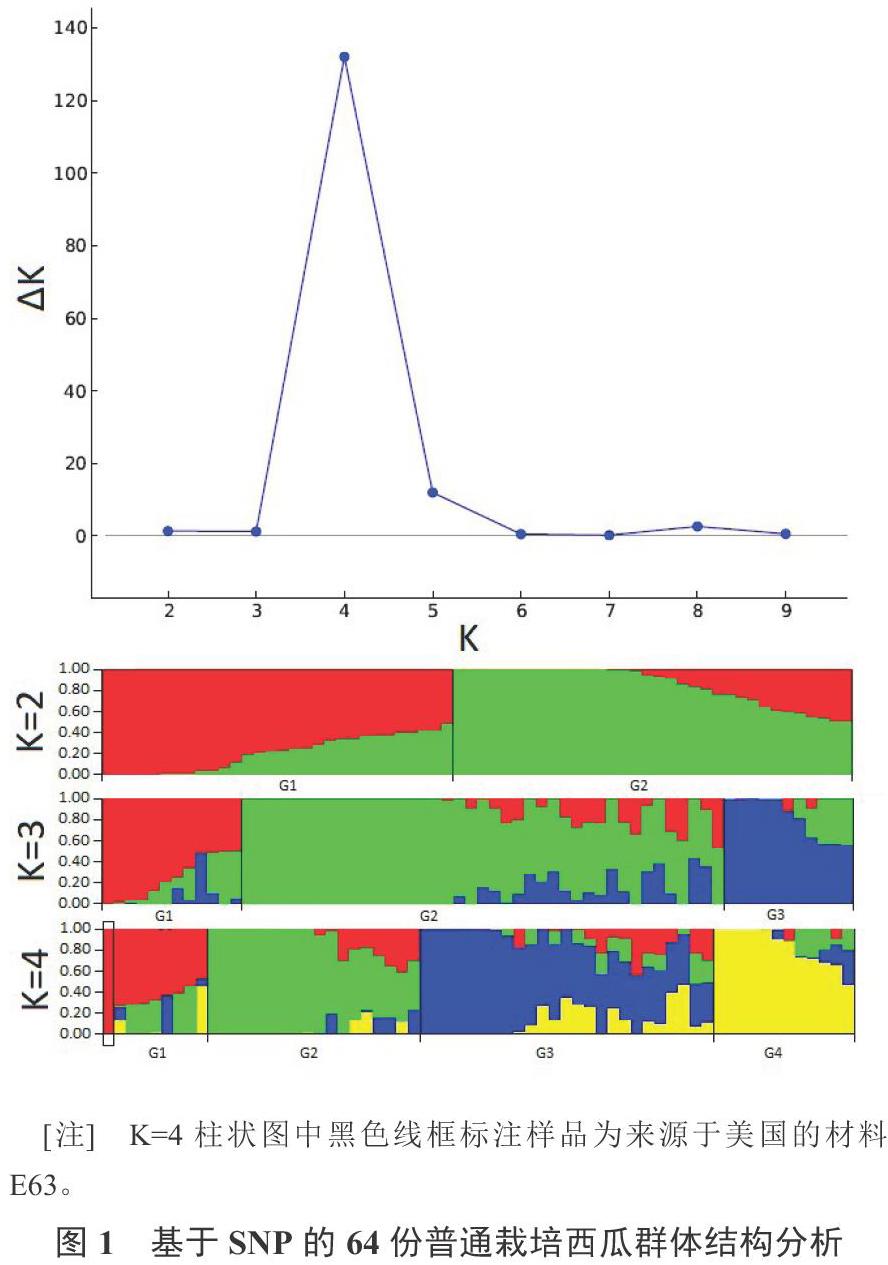
根据449个SNP位点的基因型数据进行群体结构分析,ΔK法确定64份材料的最佳亚群数为4(G1、G2、G3和G4)(图1),4个亚群分别包含9份、18份、25份和12份材料。来源于美国的材料E63(‘Crimson Sweet)划分到G1亚群(G1群组比例为0.998)。在所有亚群中,G3亚群材料的遗传背景最为混杂。64份材料主要来源于江苏(29份)、河南(20份)、台湾(8份)和新疆(5份),对亚群和材料的来源地进行独立性测验,结果表明,亚群的划分与材料来源无显著相关性。
2.3 PCA分析
对64份西瓜材料进行主成分分析,前2个主成分分别解释16.96% 和13.91%的变异(图2)。PCA分析与群体结构分析结果的一致性较好,其中G3亚群的材料不能完全区分,部分散落在G1和G4亚群中,但G1、G2和G4亚群之间可以完全区分开。
2.4 聚类分析
64份材料之间的遗传距离在0.000~0.393之间,平均0.175。材料E33(‘JQ33)和E63(‘Crimson Sweet)遗传距离最远,为0.393,分别属于G4亚群和G1亚群。相互之间遗传距离为0的材料较多,在G2、G3和G4亚群内部均有相似度极高的材料。根据遗传距离构建UPGMA聚类树,64份材料被划分为7类(图3-A):其中G2和G4亚群的聚类效果最好,分别聚集在Ⅶ类和Ⅳ类;G1亚群材料可进一步划分为3类(Ⅰ-Ⅲ);G3亚群大部分材料聚类在一起(Ⅵ类),有少部分分散在其他类中。根据SNP数据构建系统发育树,64份材料可以划分为7个大的分枝(图3-B),其中G2和G4亚群的材料分别聚类在分枝Ⅰ和分枝Ⅶ,G1和G3亚群的少部分材料聚类在一起或混在其他亚群中。基于遗传距离的UPGMA聚类和基于SNP的系统发育树一致性较好。
2.5 亚群性状变异分析
利用单因素方差分析和多重比较检测不同亚群部分农艺性状的差异,包括首雌花节位、坐果节位、果皮厚度、中心可溶性固形物含量和种子千粒重。结果表明,4个亚群的中心可溶性固形物含量和种子千粒重没有显著性差异,但在首雌花节位、坐果节位和果皮厚度上存在显著差异(图4)。G1、G2、G3和G4亚群的平均首雌花节位分别为10.2±1.9节、9.6±1.2节、7.8±1.3节和8.2±0.9节,G3和G4亚群首雌花节位极显著低于G1和G2。G1、G2、G3和G4亚群的平均坐果节位分别为18.2±5.3节、13.1±2.6节、18.1±3.6节和17.9±1.9节,G2亚群的平均坐果节位极显著低于其他3个亚群。4个亚群的平均果皮厚度分别为1.01±0.29、1.01±0.17、0.78±0.21和0.72±0.30 cm,G4亚群的平均果皮厚度显著小于G1和G2亚群,同时G3亚群的平均果皮厚度极显著小于G2亚群。
3 讨论与结论
遗传多样性丰富的种质资源是创造优异新品种的基础。西瓜作为外来引入物种,早期育种可用的资源并不多,导致现有大部分西瓜品种遗传背景狭窄,差异化程度低。分子标记技术是鉴定和评价西瓜种质资源遗传多样性的有效手段,对于挖掘和利用优异西瓜种质、拓宽育种材料遗传背景和提高育种效率有重要意义。
西瓜基因组序列和重测序数据的公布,极大地促进了西瓜种质分子鉴定和分子辅助育种的进程[14-15]。张海英等[21]利用23对SSR标记对西瓜属全部7个种的1 197份资源进行遗传多样性评估,发现1 197份资源的平均Neis 基因多样性指数为0.56,Shannon 信息指数平均为 0.99,期望杂合度值平均为0.56。郭禄芹等[22]利用SSR标记对来源于美国、中国 、南非、印度、加纳和尼日利亚、土耳其、日本等国家的167份西瓜资源进行研究,表明167份西瓜的平均Shannon 信息指数和平均期望杂合度分别为0.94和0.52。笔者基于SNP标记对63份西瓜国内来源和1份美国来源(表1)的自交系材料进行遗传多样性分析,其平均Neis 基因多样性指数、平均Shannon 信息指数和平均期望杂合度分别为0.28、0.43和0.282。通过比较发现,本研究得出的材料间遗传多样性明显偏低,主要原因是本研究选用的材料几乎全为国内育成的商业品种,而前人则是利用来源更为广泛的资源,包括近缘种、野生种、半野生种、国外品种和地方品种等。因此,进一步收集种类丰富的国内外地方品种进行遗传多样性评价对西瓜资源的开发利用更有理论指导意义。此外,上述结果表明,国内西瓜品种改良还有很大空间,通过引入国外品种和充分利用来源更为广泛的地方种、野生种质等创制新的育种材料将有效提高我国西瓜品种的多样性。
分類学上将栽培西瓜划分为4个生态型:东亚型、美国型、新疆-俄罗斯型和中国华北型[1]。本研究的群体结构分析表明,64份西瓜的最优亚群数为4(图1),其中G3亚群有较大部分资源的遗传背景较为混杂。通过分析各亚群材料的来源地,发现亚群的划分与地区来源无显著相关性(p = 0.339),说明国内育种资源交流较为频繁。本研究中唯一的国外资源E63(‘Crimson sweet,美国)被划分到G1亚群,其G1亚群比例为0.998,该亚群其他8份资源的G1群组比例在0.731~0.470之间,说明该亚群材料主要为美国生态型血统。由于缺乏明确的其他生态型材料,故而不能确定其他3个亚群材料的血统组成。对64份材料的5个农艺性状的统计分析发现,不同亚群材料的中心可溶性固形物含量和种子千粒重没有显著差异,但首雌花节位、坐果节位和果皮厚度存在显著性差异。该结果体现了高糖、较小籽粒等与品质相关的性状是共同的育种目标,但由于各地区的气候差异,熟性和耐裂性等与适应性相关的性状有所不同。
综上所述,本研究利用分布于西瓜全基因组的449个SNP标记对64份栽培西瓜进行遗传多样性分析,结果表明,大部分国内资源的遗传背景相似,且不同地区之间存在相似度极高的材料。因此,在未来的西瓜杂交育种和改良中应在了解和掌握亲本材料性状特征和遗传背景的基础上,引入遗传多样性更为丰富的野生资源、国外品种和地方品种,适当拓宽亲本间的遗传距离。
参考文献
[1] 中国农业科学院郑州果树研究所,中国园艺学会西甜瓜专业委员会,中国园艺学会西甜瓜协会,等.中国西瓜甜瓜[M].北京:中国农业出版社,2000:20-21.
[2] WU S,WANG X,REDDY U,et al.Genome of ‘Charleston Gray,the principal American watermelon cultivar,and genetic characterization of 1365 accessions in the U.S.National Plant Germplasm System watermelon collection[J].Plant Biotechnology Journal,2019,17(12):2246-2258.
[3] ZHANG H Y,WANG H,GUO S G,et al.Identification and validation of a core set of microsatellite markers for genetic diversity analysis in watermelon,Citrullus lanatus (Thunb.)Matsum.& Nakai[J].Euphytica,2012,186(2):329-342.
[4] INGHELANDT D,MELCHINGER A E,LEBRETON C,et al.Population structure and genetic diversity in a commercial maize breeding program assessed with SSR and SNP markers[J].Theoretical and Applied Genetics,2010,120(7):1289-1299.
[5] GAO Q,YUE G D,LI W Q,et al.Recent progress using high-throughput sequencing technologies in plant molecular breeding[J].Journal of Integrative Plant Biology,2012,54(4):215-227.
[6] CHEN H D,XIE W B,HE H,et al.A high-density SNP genotyping array for rice biology and molecular breeding[J].Molecular Plant,2014,7(3):541-553.
[7] 朱玉君,樊叶杨,王惠梅,等.应用分子标记辅助选择培育兼抗稻瘟病和白叶枯病的水稻恢复系[J].分子植物育种,2014,12(1):17-24.
[8] 王飞名,孔德艳,刘国兰,等.分子标记辅助选择改良‘黄华占的褐飞虱抗性与抗旱性[J].上海农业学报,2020,36(3):9-14.
[9] 李怡斐,蒋晓英,张世才,等.加工型辣椒细胞质雄性不育育性基因分子标记及辅助育种[J].分子植物育种,2016,14(4):946-952.
[10] 宋伟,苏爱国,邢锦丰,等.京724玉米自交系S型细胞质雄性不育系分子标记辅助选育研究[J].玉米科学,2016,24(1):33-36.
[11] GARCIA-MAS J,OLIVER M,GOMEZ-PANIAGUA H,et al.Comparing AFLP,RAPD and RFLP markers for measuring genetic diversity in melon[J].Theoretical and Applied Genetics,2000,101(5/6):860-864.
[12] 刘军,许美荣,赵志伟,等.丝瓜种质资源遗传多样性的SSR与SRAP分析[J].中国瓜菜,2010,23(2):1-4.
[13] NINJUDENWAR N,AWUKU F J,DIERS B,et al.Genetic diversity,population structure and key phenotypic traits driving variation within soybean (Glycine max) collection in Ghana[J].Plant Breeding,2019,138(5):577-587.
[14] GUO S G,ZHANG J G,SUN H H,et al.The draft genome of watermelon (Citrullus lanatus)and resequencing of 20 diverse accessions[J].Nature genetics,2013,45(1):51-58.
[15] GUO S G,ZHAO S J,SUN H H,et al.Resequencing of 414 cultivated and wild watermelon accessions identifies selection for fruit quality traits[J].Nature Genetics,2019,51(11):1616-1623.
[16] 戴照义,王运强,刘志雄,等.西瓜种质资源的遗传多样性分析[J].中国瓜菜,2015,28(6):5-9.
[17] 马双武,刘君璞,王吉明,等.西瓜种质资源描述规范和数据标准[M].北京:中国农业出版社,2005:9-53.
[18] PRITCHARD J K,STEPHENS M,DONNELLY P.Inference of population structure using multilocus genotype data[J].Genetics,2000,155(2):945-959.
[19] SHAUN P,BENJAMIN N,KATHE T B,et al.PLINK:a tool set for whole-genome association and population-based linkage analyses[J].The American Journal of Human Genetics,2007,81(3):559-575.
[20] KUMAR S,STECHER G,TAMURA K.MEGA7:Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J].Molecular biology and evolution,2016,33(7):1870-1874.
[21] ZHANG H Y,FAN J G,GUO S G,et al.Genetic diversity,population structure, and formation of a core collection of 1197 Citrullus accessions[J].HortScience,2016,51(1):23-29.
[22] 郭祿芹,赵世豪,朱华玉,等.167份西瓜种质材料的遗传多样性分析[J].中国瓜菜,2018,31(1):5-11.

