亚洲棉全基因组KNOX基因家族鉴定及组织表达分析
孙润润,王园园,郭新磊,秦腾飞,张金宝,金翠萍,董涛,王清连*
(1.河南科技学院,现代生物育种河南省协同创新中心,河南新乡453003;2.中国农业科学院蔬菜花卉研究所,北京100081)
1894年, 威廉·贝特森提出同源异型一词,用来定义生物中一个正常身体组成部分因发生不正常排列而变成另一身体部位的现象[1-2]。科学家们最早在果蝇中发现了同源异型现象,一只正常果蝇触角突变成了一对足,进一步研究发现在果蝇第3 条染色体存在多个同源异型盒基因[3-5],随后证实该基因广泛存在于动植物和真菌中[6-10]。 同源异型盒基因家族(Homeobox gene family)编码产生含有同源异型盒结构域的转录因子。同源异型盒结构域是由1 段长度约180 bp、 高度保守的DNA 序列编码产生,60 个氨基酸组成3 个螺旋区域,其中第1 和第2 个螺旋区结合形成环,后两个螺旋区结合形成螺旋-转角-螺旋组成[11]。
同源异型盒基因参与调控动植物生长发育过程。在植物中,根据同源异型盒基因的结构特征,将其分为 14 类:HD-ZIPⅠ、HD-ZIPⅡ、HD-ZIPⅢ、HD-ZIP Ⅳ 、PLINC、WOX、DDT、PHD、NDX、LD、PINTOX、SAWADEE、KNOX 和 BEL,其中 KNOX和BEL 家族基因编码产生包含63 个氨基酸残基的非典型同源异形盒结构域, 这63 个氨基酸组成的3 个螺旋区域中,第1 和第2 个螺旋之间存在3个额外的氨基酸(P-Y-P),所以称这两个家族为同源异型盒蛋白超家族[12-13]。
KNOX(KNOTTED1-like homeobox genes)基因家族在植物中广泛存在, 它具有KNOX1、KNOX2、ELK 和 HOX 4 个典型结构域[9,14]。 研究表明,KNOX通常可以分为2 个亚家族:ClassⅠ和ClassⅡ,2 个亚家族基因在植物生长发育中的调控作用不同。 在被子植物中,ClassⅠ亚家族基因主要在顶端分生组织中表达, 是形成和维持分生组织的重要基因[15-16]。ClassⅡ亚家族基因表达部位比较广泛,在根、茎、叶、花等器官中均有表达, 它主要参与调控植物器官分化[17],此外还参与次级细胞壁的生物合成[18-19]。
玉米Knotted-1(Kn1)基因是植物中第1 个被发现的同源异型盒基因[20],随后KNOX基因在多种植物中被鉴定。例如,在拟南芥和水稻中,分别鉴定到8 个和13 个KNOX基因[9];在大豆中鉴定到27 个[21];在番茄中鉴定到 8 个[22];在毛竹中鉴定到12 个[23];在普通烟草中鉴定到18 个,在二倍体祖先绒毛烟草和林烟草中分别鉴定出 5 个和 8 个KNOX基因[24]。
棉花是世界上最重要的纤维作物之一,KNOX基因在棉花中调控作用的研究较少。本研究以二倍体棉花石系亚1 号为研究材料,在亚洲棉全基因组序列的基础上, 以KNOX 家族蛋白质结构域为搜索条件,鉴定亚洲棉中KNOX基因,对 KNOX 转录因子的理化性质、进化关系、基因结构和基因复制进行分析,并研究KNOX基因的表达模式,为进一步探索棉花KNOX基因的调控作用奠定基础。
1 材料与方法
1.1 亚洲棉KNOX家族基因的鉴定
亚洲棉基因组数据 (Gossypium arboreum,BGI_V1.0) 下载自 Cottongen 数据库(https://www.cottongen.org/)。 从 Pfam 数据库(http://pfam.xfam.org/)下载KNOX 蛋白结构域的隐马尔可夫模型文件(PF03790,PF03791)[25]。 以 PF03790 和 PF03791文件为探针,利用HMMER 3.0 软件搜索亚洲棉全基因组数据[26]。 将获得的蛋白序列分别提交到Pfam、NCBI 保守结构域数据库(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)和 SMART(http://smart.embl-heidelberg.de) 验证蛋白质结构域[27-28],去除不含KNOX 保守结构域的序列。 依据基因所在染色体及在染色体上的起始位置进行对获得的KNOX 家族基因进行命名。 将KNOX 家族蛋白序列递交到EXPASY ProtParam 数据库中(http://web.expasy.org/protparam/),分析蛋白质长度、相对分子质量和等电点。
1.2 染色体定位与基因复制
Mapinspect 软件用来绘制基因在染色体上的分布图,将基因所在染色体和在染色体上的起始位置信息提交到Mapinspect 软件中, 即可将基因定位于染色体上。 MCScanX 软件用来查找染色体间的共线性区域[29-30]。
1.3 进化分析
利用MEGA 7.1 软件构建系统进化树。 运用ClustalW 的比对方法对KNOX 家族蛋白序列进行多重比对。选择邻接法(Neighbor-joining)法构建系统进化树, 具体参数设置如下: 采用自展法检验(Bootstrap method),校正参数为 1 000,模型为泊松校验(Poisson correction),数据缺失处理为比对删除(Pairwise deletion)[26]。
MEME(http://meme-suite.org/tools/meme)工具用来查找KNOX 蛋白质的保守基序。 参数设置为:输出基序数量为10, 根据KNOX 蛋白质结构域的长度,选择基序的最大长度为51。
1.4 基因结构示意图绘制
根据亚洲棉全基因组测序注释文件信息,利用GSDS(http://gsds.cbi.pku.edu.cn/)基因结构显示系统绘制基因结构示意图[31]。
1.5 基因表达谱构建
从 ccNET 数据库(http://structuralbiology.cau.edu.cn/gossypium/)中下载亚洲棉KNOX基因转录组数据[32],利用公式 log2(FPKM)对数据进行标准化处理,将处理后数据导入Excel,绘制KNOX基因表达量柱形图。
2 结果与分析
2.1 亚洲棉KNOX基因的鉴定与命名
经HMMER 搜索,共获得26 个候选蛋白质序列, 用 Pfam、SMART、NCBI 数据库验证蛋白质结构域,去除不含基本结构域的蛋白质序列,共鉴定到23 个GaKNOX 家族成员。根据基因所在染色体及 在 染 色 体 上 的 起 始 位 置 命 名 为GaKNOX1-GaKNOX23(表1)。在GaKNOX家族基因编码的蛋白质中,GaKNOX6 基因编码197 个氨基酸, 是编码氨基酸数目最少的基因,GaKNOX1编码氨基酸数目最多,为483 个氨基酸。 GaKNOX蛋白质分子量在21.82 kDa~53.38 kDa, 等电点在4.66~8.53,除 GaKNOX7 外,其它 GaKNOX 蛋白质的等电点均小于7, 说明大部分GaKNOX基因编码产生酸性蛋白质。
2.2 亚洲棉KNOX基因的染色体定位
为了解GaKNOX基因在染色体上的分布情况, 利用MapInspect 软件绘制染色体定位图 (图1)。 23 个GaKNOX基因定位在 11 条染色体上,其中 1、6、11、12 和 13 号 染 色 体 上 均 含 有 1 个GaKNOX基因, 分别是GaKNOX1、GaKNOX10、GaKNOX21、GaKNOX22 和GaKNOX23;3、4 和 7号染色体均含有 2 个GaKNOX基因;5、8 和 10 号染色体上GaKNOX基因的数目最多, 均含有4 个。利用MCScanX 查找GaKNOX基因的共线性,发现GaKNOX15 和GaKNOX16 为串联重复。
2.3 亚洲棉KNOX蛋白质结构域分析
多重序列比对证实大部分GaKNOX 蛋白质具有的 4 个典型结构域 (表1, 图2), 即 KNOX1、KNOX2、ELK 和 HOX, 但 也 存 在 个 别 例 外 ,GaKNOX6 和 GaKNOX15 仅 含 有 KNOX1 和KNOX2 结 构 域;GaKNOX20 和 GaKNOX23 缺 失ELK 结构域;GaKNOX13 缺失 HOX 结构域。 完整的蛋白结构是发挥功能的基础,结构域的缺失可能导致功能的丧失。

表1 亚洲棉KNOX 基因家族信息

图1 亚洲棉KNOX 基因的染色体定位及复制分析

图2 亚洲棉KNOX 蛋白质多序列对比分析
2.4 亚洲棉KNOX基因结构分析
亚洲棉KNOX基因结构示意图结果显示 (表1,图3),GaKNOX17 含有 8 个外显子,是含有外显子最多的基因;GaKNOX1 和GaKNOX23 有 6 个外 显 子 ;GaKNOX18 和GaKNOX22 有 4 个 外 显子;GaKNOX6 和GaKNOX15 含有 3 个外显子,为外显子最少的基因; 其余GaKNOX基因均含有5个外显子。
2.5 KNOX蛋白质进化分析
亚洲棉KNOX 家族成员的进化关系结果显示,GaKNOX 可以分为 2 组,即 ClassⅠ和 ClassⅡ,两组成员之间基因结构和基序分布较为相似 (图3)。ClassⅠ包含 13 个成员,可以分为两个亚组,ClassⅠ A 含 有 GaKNOX15、GaKNOX16、GaKNOX17、GaKNOX7、GaKNOX22、GaKNOX14 和 GaKNOX-18,ClassⅠB 含有 GaKNOX3、GaKNOX5、GaKNOX-21、GaKNOX10、GaKNOX6 和 GaKNOX20。 ClassⅡ 包 含 GaKNOX2、GaKNOX11、GaKNOX12、GaKNOX8、GaKNOX9、GaKNOX19、GaKNOX23、GaKNOX4、GaKNOX1 和 GaKNOX13 共 10 个 蛋白,分为 ClassⅡA 和 ClassⅡB 两个亚组。
GaKNOX 家族蛋白包含 10 个 motif, 所有成员均含有motif 3 (图3)。 ClassⅠ所有成员均含有motif 5,部分成员含有 motif 1、motif 2、motif 7、motif 8 和 motif 10 中的 1 个或几个。ClassⅡ组所有成员均包含motif 1、motif 4 和 motif 7,部分具有motif 8 和 motif 9, 部分具有 motif 2, 部分具有 motif 6。由GaKNOX 蛋白质结构域长度及分布分析可以看出,motif 3 与 KNOX1 结构域相对应;motif 5 和进化树分支Class I 组的KNOX2 结构域相对应;motif 4 和 motif 7 与进化树分支 Class Ⅱ组的KNOX2 结构域相对应;motif 2 和 HOX 结构域相对应。
利用拟南芥、 水稻和亚洲棉的KNOX 蛋白质序列构建进化树(图 4),在 3 个物种中,KNOX 基因家族分成ClassⅠ和ClassⅡ2 组, 每一组又分为两个亚组。 ClassⅠA 中 GaKNOX15、GaKNOX16、GaKNOX17 和 GaKNOX7 与拟南芥的 AtKNAT1相 似 性 较 高 ; GaKNOX22 、 GaKNOX14 和GaKNOX18 与拟南芥AtSTM 处于同一分支。ClassⅠB 中 GaKNOX10、GaKNOX6 和 GaKNOX20 同拟南芥 AtKNAT2 和 AtKNAT6 相似性较高,而GaKNOX3、GaKNOX5 和 GaKNOX21 与 水 稻 中OSH6 和 OSH71 处于同一分支。 ClassⅡA 中的GaKNOX 同拟南芥中的 AtKNAT7、 水稻中的HOS66 同源性较高。 ClassⅡB 中的 GaKNOX 与拟南芥的 AtKNAT3、AtKNAT4 和 AtKNAT5 同源性较高。

图3 亚洲棉KNOX 基因结构及编码蛋白的结构域
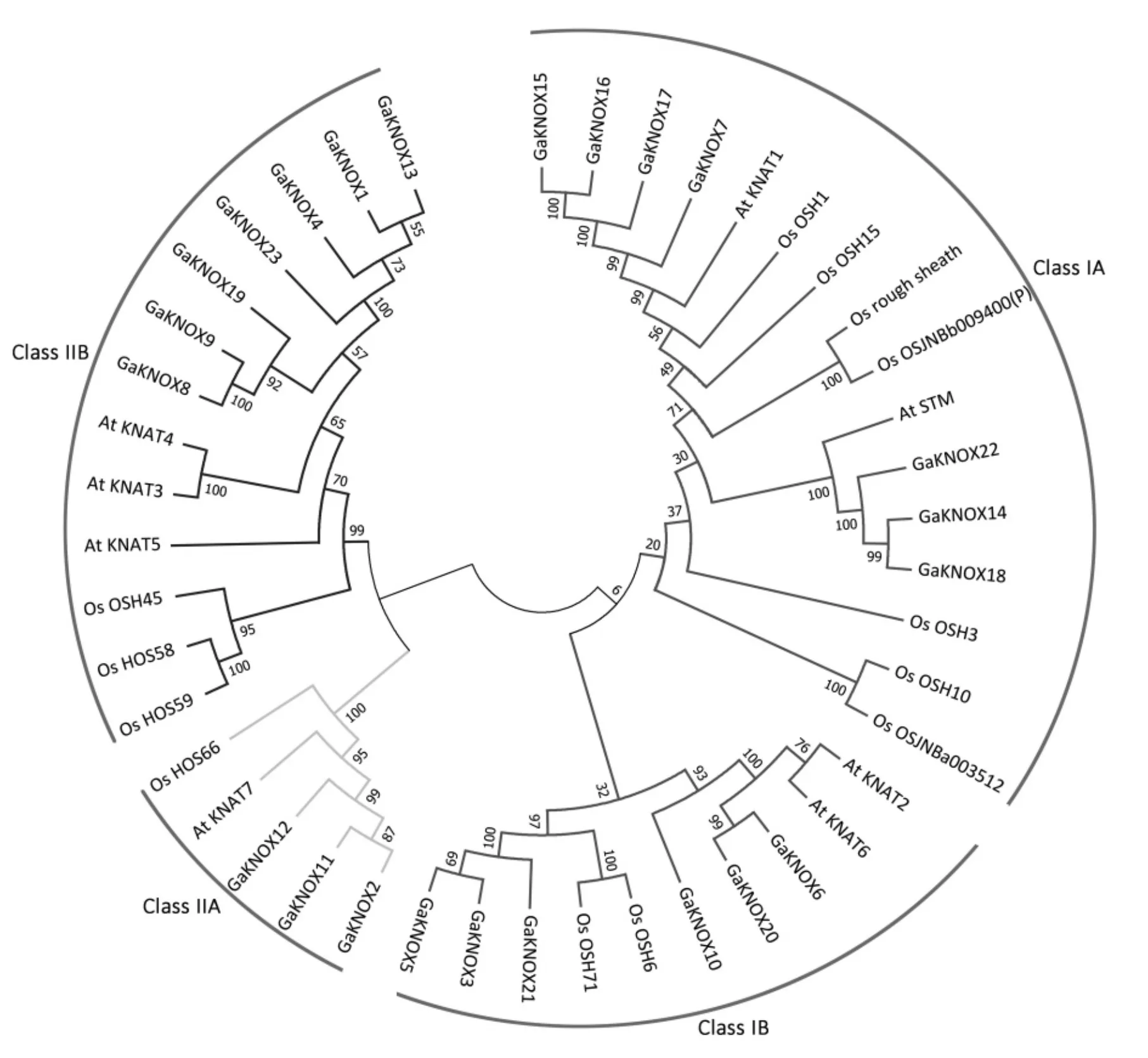
图4 亚洲棉、拟南芥和水稻中KNOX 蛋白质进化树
2.6 亚洲棉KNOX基因表达分析

图5 亚洲棉KNOX 基因在不同组织中的表达模式
根据CCNET 数据库下载的转录组数据构建GaKNOX基因表达图 (图5)。GaKNOX8、GaKNOX9 和GaKNOX15 在所有测定的组织中表达 量 均 较 低 ; 而GaKNOX1、GaKNOX2、GaKNOX11、GaKNOX19 和GaKNOX23 在所有器官中均高表达。GaKNOX12 在开花后20 d 的纤维和种子中的表达量较其他测定组织高。GaKNOX7、GaKNOX16 和GaKNOX17 在根和茎中高表达。GaKNOX13 在营养器官中的表达量高于在生殖器官中的表达。GaKNOX21 在叶和茎中的表达量较高。GaKNOX5 在茎中的表达量最高,开花后10 d的纤维和种子中次之。
3 讨论与结论
目前关于棉花KNOX基因的报道较少, 仅有Gong 等[33]关于KNL1 在纤维发育过程中调控作用的报道。GhKNL1 是 KNOX 基因 ClassⅡ类的成员,在棉纤维发育初期表达量较低,随着纤维发育表达量逐渐升高,在次生壁合成期达到最高,证明了GhKNL1 参与调控棉纤维发育过程。聂晓莹等[34]通过构建GhKNL1 的RNA 干扰载体,分析转基因棉花表型, 发现GhKNL1 在棉纤维发育过程发挥负调控作用, 抑制GhKNL1 基因表达影响次生壁发育相关基因的表达,进而影响次生壁加厚。 关于亚洲棉中KNOX家族基因的研究未见报道。
本研究在亚洲棉全基因组数据中共鉴定到23个GaKNOX基因,数目比拟南芥、水稻、玉米和番茄中的KNOX基因多[9,22],且各成员的保守结构域存在差别,如GaKNOX13 缺失HOX 结构域。 拟南芥的KNATM 也缺少HOX 结构域,参与调控叶片叶近端- 远端形态[35],推测GaKNOX13 在棉花中可能也有相似的功能。 GaKNOX20 和GaKNOX23缺失ELK 结构域。GaKNOX6 基因仅编码197 个氨基酸,GaKNOX15 基因编码234 个氨基酸,序列分析显示这2 个基因编码的蛋白质同时缺失ELK结构域和HOX 结构域。 植物家族基因进化过程中不具有典型结构域的家族基因可能是假基因,编码不完整蛋白质,但假基因可能进化成新基因[36-37],不具有典型结构域的KNOX基因的功能如何还有待深入研究。
亚洲棉KNOX基因转录组数据结果显示,除GaKNOX8 和GaKNOX9 在所有组织中表达量均较低外,ClassⅡ类GaKNOX基因几乎在测定的所有组织中表达水平都高于ClassⅠ类GaKNOX基因,而大多数ClassⅠ类GaKNOX基因在7 种不同组织中的表达模式较相似, 在茎中的表达量较高,在其他组织中的表达水平均很低,这种表达模式与已报道的其他物种一致, 表明植物KNOX 蛋白在功能上可能具有高度的保守性[38]。 值得一提的是,ClassⅡ类基因GaKNOX12 在开花后20 d 的纤维和胚珠中的表达量最高,说明其可能参与调控棉纤维发育过程中的次生壁形成阶段,关于该基因的功能有待于进一步研究[39-40]。GaKNOX基因在不同组织中的表达模式说明它可能广泛参与了亚洲棉的生长发育过程, 本研究为进一步探讨KNOX基因在棉花中的调控机制奠定基础。
——紫 苏

