基于600K SNP 芯片技术对宁海黄鸡和广西黄鸡繁殖性状的全基因组关联分析
曹海月,谭雨革,董信阳,毛海光,马有智,卢 磊,姜俊保,尹兆正*
(1.浙江大学动物科学学院,浙江杭州 310058;2.宁波市振宁牧业有限公司,浙江宁波 315600)
全基因组关联分析(Genome-Wide Association Study,GWAS)是指在全基因组范围内找出存在的序列变异,即单核苷酸多态性(Single Nucleotide Polymorphisms,SNPs),并从中筛选出与目的性状相关的SNPs。GWAS 研究最先被应用于医学,Klein 等[1]在《Science》杂志上首次发表了利用GWAS 研究人类视网膜黄斑病的文章,自此之后,GWAS 被广泛应用于人类疾病致病基因的筛选,并成功鉴定了糖尿病、抑郁症等疾病的主要致病基因[2-3]。近年来,GWAS 开始逐渐应用于畜禽数量性状的研究,鉴定出大量与猪繁殖性状、鸡生长及蛋品质等性状相关的候选基因[4-6]。随着高通量测序和高分辨代谢检测技术不断发展,以及多种生物信息学技术和统计学方法发展,使复杂性状基因变异的定位更加准确,为畜禽重要经济性状主效基因和疾病性状致病基因挖掘提供了更加有效的方法[7]。
宁海黄鸡和广西黄鸡是我国南方地区的2 个优质地方鸡种,相比而言,宁海黄鸡产蛋率高,广西黄鸡生长速度快。由于表型差异通常由基因差异引起,因此探究影响2 个品种性状特征的基因将有利于地方品种鸡的遗传改良和配套利用。前人研究表明,鸡的大多数性状受多个微效基因控制[8]。近年来,GWAS 被大量应用于中低密度的单核苷酸多态性与鸡生长、繁殖性状相关性的研究[9-10]。高密度(600K)的基因芯片可以使目标基因的筛选更加准确有效[11],但其目前主要应用于鸡体重、疾病和脂肪沉积等方面的研究[6,12-13]。为此,本研究采用鸡600K SNP 芯片技术对宁海黄鸡和广西黄鸡进行基因分型和GWAS 分析,筛选鉴定与繁殖性状相关的SNP 位点和候选基因。
1 材料与方法
1.1 实验动物 宁海黄鸡和广西黄鸡种母鸡各59 只由浙江省宁波市振宁牧业有限公司提供,个体笼养,营养和环境条件一致,其中产蛋期光照时间每天14 h。
1.2 性状测定 记录每只母鸡在300 日龄内的产蛋数(Egg Number at the Age of 300 d,EN300)。28~29周龄为选留下一代种蛋期,依据种蛋大小、形状和蛋壳厚度等选出合格种蛋,然后转移到机器孵化器进行系谱孵化。记录每只母鸡28~29 周龄合格种蛋的总数,记为入孵蛋数(Setting Eggs,SE)。在孵化第5 天,照蛋除去未受精的蛋,记录每只母鸡剩余受精蛋的数量,并将其与对应母鸡产蛋数的比率计为受精率(Fertility Rate,FR)。孵化至第18 天时,把孵化器里的受精蛋移到出雏器里继续孵化,统计移盘数并记为移盘数(Transferring Eggs,TE)。在出壳当天,将每只母鸡的雏鸡数计为出雏数(Hatching Number,HN),将未孵化出雏鸡的受精蛋的数量计为死胚蛋数(Addle Egg Number,AE)。将出雏数与入孵蛋数及受精蛋数的比例分别计为入孵蛋孵化率(Hatchability Of Setting Eggs,HSE)和受精蛋孵化率(Hatchability Of Fertile Eggs,HFE)。
1.3 基因分型和数据质控 鸡颈静脉采血并储存在-20℃。依据制造商说明,使用Genome DNA Extraction Kit(天根生化科技有限公司,中国北京)提取基因组DNA,并稀释至50~100 ng/μL。SNP 芯片试验的基因组DNA 应达到以下条件:采用NanoDrop2000 紫外分光光度计测量OD 值,A260/A280在1.8~2.1;用1%琼脂糖凝胶进行电泳,条带清晰,且长度大于10 kb。将合格的DNA 送纽勤生物科技(上海)有限公司进行鸡600K SNP 芯片检测和基因分型。采用PLINK(v1.09)进行基因型数据的质控[14]。
1.4 统计分析 利用PLINK(v1.09)软件对所有样本的600K 芯片扫描数据进行质量控制,剔除SNPs 检出率小于90% 且哈迪-温伯格平衡检验P<1×10-6(卡方检验)和最小等位基因频率(MAF)≤0.05 的SNPs,剔除分型成功率小于90% 的样品。使用SPSS 20.0 对表型数据进行统计分析,使用PLINK v1.09 中的主成分分析法(Principal Component Analysis,PCA)对两个群体进行群体分层评估,使用PLINK(v1.09)的线性模型进行全基因组关联分析,所用模型:

其中,Y 是性状表型值向量;β是表型均值、SNPs、群体结构(校正群体结构时,使用前5 个主成分作为协变量)3 个固定效应向量;X 是β 的关联矩阵;e 是残差效应向量,e~ N(0,Iσ2e),其中I 是单位阵,Iσ2e是随机残差方差,最终,每个SNP 位点都能得到一个关联值。为减少多重检验带来的假阳性率,以Bonferroni 校正法[15]对全基因组关联分析结果的P值进行校正。此处独立检验数计算使用PLINK(v1.09)中的“indeppairwise 25 5 0.2”命令(即以25 个SNPs 为一个窗口,5 个SNP 为步移,r2阈值为0.2),估算出连锁不平衡(LD)块和单个独立SNP 数目为149 653,因此Bonferroni校正的达5% 基因组水平显著的P 值阈值为3.341E-07(0.05/149653),即P值低于此阈值的SNPs 则被认为与繁殖性状显著关联;达到基因组水平潜在关联的P值阈值为6.682E-06(1/149653),即P值低于此阈值的SNPs 则被认为与繁殖性状潜在关联。使用R 软件作Quantile-Quantile(QQ)图和曼哈顿图。
2 结果
2.1 表型值的描述性统计 如表1 所示,宁海黄鸡和广西黄鸡的EN300、SE、TE 和HN 存在极显著差异。
2.2 基因分型和数据质控 剔除SNP 检出率小于90%的位点5 604 个、哈迪-温伯格平衡检验P<1×10-6(卡方检验)的位点2 734 个,剔除MAF ≤ 0.05 的SNP 位点78 879 个,剔除分型成功率小于90%的样品0 个,剩余495 704 个SNPs 118 个样本可用于后续与繁殖性状的全基因组关联分析。独立SNPs 计算后,共得到独立SNPs 标记和Block 模块149 653 个。通过统计基因组每100 K 范围内的SNP 数,得到SNPs 在基因各染色体上的分布情况(图1),结果显示基于高密度芯片技术检测到的SNPs 在染色体上分布均匀,SNPs 数据可靠。

表1 宁海黄鸡和广西黄鸡繁殖性状的描述性统计

图1 质控后的SNPs 标记在各染色体上的分布情况
2.3 群体层化分析 在GWAS 分析中,群体分层会导致分析的结果出现假阳性,但主成分分析结果显示,在2个鸡群体中未显示明显的亚群结构(图2),并且繁殖性状的Quantile-Quantile 图结果显示,观测值(纵坐标)和期望值(横坐标)基本吻合(图3),说明关联分析不会因为群体分层而产生假阳性,基于线性模型的关联分析结果可靠。
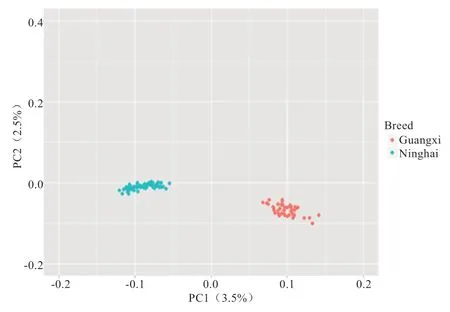
图2 群体结构主成分分析图
2.4 全基因组关联分析 繁殖性状的曼哈顿图见图4,共有4 个SNPs 与繁殖性状的相关性达到Bonferroni 校正的5%基因组显著性水平,其中rs313221983 和rs314844182影响AE,rs14758703 影响FR,rs312721292 影响HSE,rs314844182 和rs312721292 影响HFE。在每个显著的SNP 位点上、下游5 kb 内寻找可能的候选基因,结果共找到3 个可能的候选基因,分别为唾液酸转移酶1(ST8 alpha-N-acetyl-neuraminide alpha-2,8-sialyltransferase 1,ST8SIA1)基因,神经外胚层皮质1(Ectodermal-neural cortex 1,ENC1)基因和LOC101750905基因(表2)。
3 讨 论
从本研究结果可知,宁海黄鸡和广西黄鸡的EN300、SE、TE、HN 存在极显著差异。此外,品种内各个性状都拥有较大的极值差距,显示品种的遗传多样性得到了保持,这种多样性将使GWAS 分析结果更加有效。本研究结果显示,总共4 个SNPs 与繁殖性状的关联性达到了全基因组显著性水平,其中rs313221983和rs314844182 位于1号染色体,rs14758703 位 于Z染色体,rs312721292 位于4 号染色体,值得注意的是,rs314844182 同时与AE、HFE 2 个性状相关联,rs312721292 同时与HSE、HFE 2 个性状相关联。之前的研究通过GWAS 已经鉴定出了许多与鸡繁殖性状相关的QTL、候选区域和SNPs。对白色来航鸡繁殖性状进行GWAS 分析发现,7 号和13 号染色体上各有1 个SNP 分别与21~56 周的产蛋数和初产日龄显著相关[16]。另外,有研究发现5 号染色体上29.5~46.9 Mb 区域与300 日龄产蛋数和孵化率有关[10],并确定了5 号染色体上与21~26 周产蛋数显著相关的6 个基因座[17]。本研究结果所得到的与AE、FR、HSE 和HFE 显著相关的SNPs 位于3 个候选基因的序列附近或内部,分别是ST8SIA1、ENC1和LOC101750905。对ST8SIA1基因的报道主要在于其对乳腺癌细胞的作用,Bobowski 等[18]研究表明,在人乳腺癌细胞中,ST8SIA1的表达量受雌二醇抑制,其核心启动子包含2 个推测的雌激素反应元件,结合本研究结果表明,ST8SIA1基因是影响鸡繁殖性状的候选基因之一。ENC1主要在神经系统中表达,编码一种肌动蛋白结合蛋白,在神经细胞分化和凸起生长中发挥重要作用[19]。Kim 等[20]研究表明,在排卵前颗粒细胞中,ENC1 被促黄体生成素/人绒毛膜促性腺激素(LH/ hCG)诱导与F-肌动蛋白物理结合,可能通过调节细胞骨架组织促进颗粒细胞的分化,这与本研究结果不谋而合,ENC1基因可以作为影响鸡繁殖性状的候选基因之一。有关LOC101750905基因的报道目前较少,GWAS 研究表明,此基因下游的rs14490981位点对活体重、脚重、全净膛重和半净膛重4 个性状显著相关[21]。本研究表明,LOC101750905基因与鸡繁殖性状相关。今后尚需要进一步研究ST8SIA1、ENC1和LOC101750905基因对鸡AE、FR、HSE 和HFE 的调节机制。
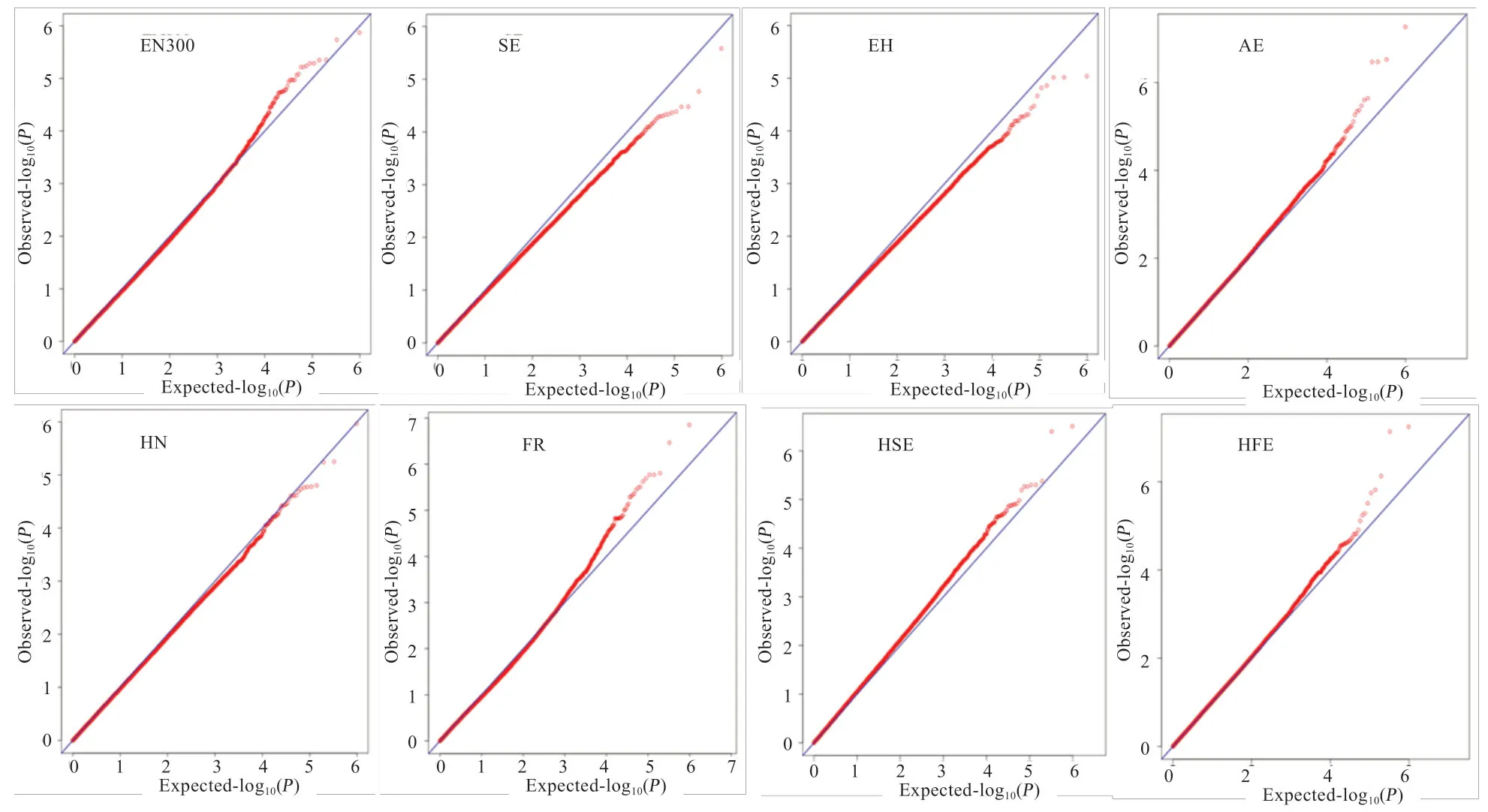
图3 繁殖性状的Quantile-Quantile 图

表2 繁殖性状达到5%基因组显著水平的全基因组关联分析结果
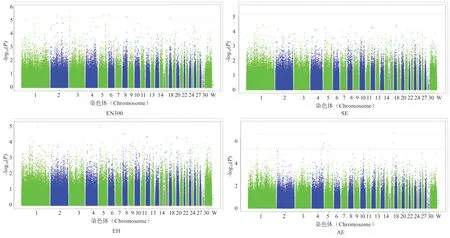
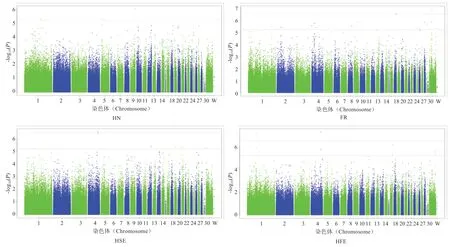
图4 繁殖性状的曼哈顿图
4 结 论
本研究采用600K SNP 芯片检测及GWAS,发现了4 个SNPs 对宁海黄鸡和广西黄鸡繁殖性状关联性达到Bonferroni 校正5%的全基因组显著性水平,并检测到了可能与AE、FR、HSE 和HFE 相关的3 个近端基因(ST8SIA1、ENC1和LOC101750905),为进一步了解地方鸡种繁殖性状的遗传基础和基因组选择提供了理论依据。

