普通小麦‘Holdfast’条锈病成株抗性QTL定位
杨芳萍 刘金栋 郭 莹 贾奥琳 闻伟鄂 巢凯翔 伍 玲 岳维云 董亚超 夏先春,*
普通小麦‘Holdfast’条锈病成株抗性QTL定位
杨芳萍1,2,**刘金栋2,3,**郭 莹1贾奥琳2闻伟鄂2巢凯翔4伍 玲5岳维云6董亚超2夏先春2,*
1甘肃省农业科学院小麦研究所, 甘肃兰州 730070;2中国农业科学院作物科学研究所 / 国家小麦改良中心, 北京 100081;3中国农业科学院深圳农业基因组研究所, 广东深圳 518120;4西北农林大学植保学院, 陕西杨凌 712100;5四川省农业科学院作物研究所, 四川成都 610066;6天水农业科学研究所, 甘肃天水 741200
Holdfast是来自英国的小麦品种, 多年来一直保持良好的条锈病持久抗性。本研究目的是发掘Holdfast的条锈病成株抗性基因及其紧密连锁的分子标记, 为小麦持久抗性品种选育提供材料和方法。利用铭贤169和Holdfast杂交后代重组自交系(recombinant inbred lines, RIL)群体, 于2014—2015和2015—2016年度在甘肃甘谷、甘肃中梁和四川成都进行条锈病成株抗性鉴定, 并统计最大严重度(maximum disease severity, MDS)。基于小麦660K SNP芯片和BSA (bulked segregant analysis)技术初步确定抗病基因所在的染色体后, 将目标区域的SNP标记转化为KASP (Kompetitive allele specific PCR)标记, 检测整个RIL群体, 进行基因型分析。最后进行RIL群体条锈病成株抗性的QTL分析, 在5AL和7AL染色体上发现了2个成株抗性QTL。5A染色体长臂上1个条锈病成株抗性QTL, 在所有环境下均存在, 可解释6.5%~9.3%的表型变异;位于标记和之间, 连锁距离分别为0.5 cM和1.1 cM。在7A染色体长臂上定位到1个条锈病成株抗性QTL, 在2015年和2016年甘谷环境中均稳定存在, 分别解释6.2%和7.3%的表型变异;位于标记和之间, 连锁距离分别为0.5 cM和0.7 cM。携带和抗病等位基因家系的MDS显著低于感病等位基因家系的MDS, 表明和可有效降低条锈病严重度, 可应用于小麦抗条锈育种。
普通小麦; 条锈病; 成株抗性; SNP标记; 连锁分析
由小麦条锈菌(f. sp.)引起的小麦条锈病是世界范围内的重要病害, 可导致减产5%~25%[1], 严重威胁小麦生产。虽然条锈病可通过化学药剂等措施防治, 但利用寄主抗性, 培育和推广抗病品种是防治小麦条锈病最为经济、安全和有效的途径。
小麦对条锈病的抗性可分为苗期抗性和成株抗性。苗期抗性一般由单个或少数主效基因控制, 又称主效基因抗性、全生育期抗性或垂直抗性, 对条锈菌生理小种的抗性有高度特异性, 往往呈高抗或免疫。苗期抗性对病原菌有较强的选择压力, 过度应用会引起生理小种变异, 使品种丧失抗性。成株抗性由多个微效基因控制, 又称部分抗性、水平抗性或慢病性。成株抗性品种往往苗期表现感病, 而成株期表现中抗或高抗, 对病原菌无小种专化性或专化性弱。成株抗性对病原菌选择压力较小, 大面积种植可有效减缓生理小种变异, 使抗性持久稳定。长期以来, 大多数小麦育种家利用苗期抗性培育高抗至免疫的品种, 导致品种抗性频繁丧失, 使抗条锈病育种工作长期处于被动状态。前人研究表明, 成株抗性基因聚合是选育兼抗多种病害品种的重要方式, 聚合4~5个成株抗性基因(如、等)可将条锈抗性提升至高抗至近免疫水平, 且抗性持久稳定[2]。
发掘成株抗性基因及其紧密连锁的分子标记是有效利用成株抗性、培育持久抗性品种的重要手段[2-7]。近年来, 随着分子生物学技术的快速发展, 小麦条锈病抗性基因发掘研究取得较大进展。迄今为止, 国际上正式命名的小麦条锈病抗性基因已有80个(~)[8-9]。在已正式命名的基因中, 55个为苗期抗病基因(、等), 其余25个为成株抗性基因(如等),其中、、、和为高温成株抗性基因。除此之外, 还有40多个暂时命名的条锈病抗性基因。目前,、、、、和等[10-13]已被克隆。
近年来, 由于条锈菌生理小种的快速变异, 已定位的苗期抗性基因大多已丧失抗性, 给小麦生产带来严重威胁。因此, 发掘成株抗性基因, 培育持久抗性小麦品种, 对于保证我国粮食安全具有重要意义。基于双亲群体的连锁分析是发掘数量性状位点的有效方法[14]。SNP标记在基因组中广泛存在, 具有标记密度高、覆盖范围广且可高通量检测的优点。随着分子生物学技术的快速发展, SNP标记已逐渐应用于高密度遗传图谱构建和数量性状基因定位研究, 有效提升QTL的检测效率和精度。迄今为止, 国内外已定位了150余个条锈病成株抗性QTL, 在小麦21条染色体上均有分布。
Holdfast是来自英国的小麦品种, 农艺性状优良, 引进国内后一直保持良好的条锈病成株抗性, 迄今仍是我国甘肃重要的抗病亲本。前人研究表明, Holdfast不仅携带苗期抗病基因, 同时还携带成株抗性基因[15-16]。先前针对Holdfast的研究主要集中于不同生理小种的表型鉴定和初步遗传学分析, 其携带的成株抗性基因仍不明确[15-17]。因此, 本研究基于SNP标记, 对Holdfast进行成株抗性QTL定位, 以明确其抗性遗传机制, 为小麦条锈病持久抗性育种提供材料和分子标记。
1 材料与方法
1.1 试验材料
以感病品种铭贤169为母本, 抗锈品种Holdfast为父本, 采用单粒传法构建含有288个株系的F6代重组自交系(recombinant inbred lines, RIL)群体。2014—2015和2015—2016年度于甘肃中梁、甘肃甘谷和四川成都种植该群体及其亲本, 以鉴定条锈病成株抗性。田间试验采用单行区、随机区组排列, 行长1.5 m, 行距25 cm, 3次重复。在试验区垂直方向种植条锈病诱发行。甘肃甘谷和中梁诱发行品种为铭贤169和辉县红, 四川成都点诱发行品种为川育12和SY95-71。
1.2 条锈菌接种
为促进条锈发病, 在甘肃甘谷和中梁, 于小麦三至四叶期利用包含CYR32、CYR33、CYR34、水4、水7、Hybrid46-8和G22-14的混合菌种对诱发行人工喷雾接种(孢子悬浮液, 0.03%吐温20), 接种后用塑料薄膜覆盖保湿24 h。甘肃甘谷和中梁点条锈病混合菌种由甘肃省农业科学院植物保护研究所提供。在四川成都, 利用CYR32、CYR33、Hybrid46-8、G22-12等混合小种对铭贤169幼苗人工接种。随后于植株三至四叶期, 在诱发行中移栽接菌发病的铭贤169幼苗。四川成都点菌种由四川省农业科学院植物保护研究所提供。
1.3 条锈病抗性鉴定
参照改良的Cobb方法[18]鉴定条锈病抗性。目测植株旗叶和倒二叶条锈菌孢子堆面积占叶片面积的百分比, 即病害严重度(disease severity)。感病亲本铭贤169在成株期的条锈病严重度达80%~100%时, 分2次记载严重度, 并用2次调查的最大严重度(maximum disease severity, MDS)作为最终表型数据进行后续分析。根据发病时间的不同, 分别于4月上中旬(四川成都)、5月中下旬(甘肃甘谷)、5月底(甘肃中梁)进行条锈病MDS记载。
1.4 表型数据和等位基因效应分析
利用Microsoft Excel 2016对铭贤169/Holdfast RIL群体不同试验点小麦条锈病MDS进行频率分析和每个QTL等位基因效应的测验; 利用SAS 9.3 (http://www.sas.com/) PROC GLM程序进行方差分析(analysis of variance, ANOVA)。
1.5 基因型分析
采用集群分离分析法(bulked segregant analysis, BSA)分别选择20株高抗家系(MDS<25%)和20株高感家系(MDS>80%)构建两组抗感池。利用小麦660K iSelect SNP芯片(633,562个SNP位点)[20]对构建的2组抗感池和亲本进行扫描(北京博奥生物技术有限公司, http://www.capitalbiotech.com/)。使用Affymetrix公司的GTC软件(http://www.affymetrix. com/estore/)进行SNP分型和聚类分析。为保证结果准确性, 剔除无多态性及低质量的SNP标记(缺失率>20%)。
基于SNP分型数据, 确定铭贤169/Holdfast RIL群体抗感池间差异SNP主要集中区域。根据差异SNP及其所在的染色体物理位置, 利用在线平台Poly Marker网站(http://polymarker.tgac.ac.uk/)设计KASP特异引物。随后, 在2条正向KASP引物5¢端添加FAM或者HEX荧光接头序列, FAM接头序列为5¢-GAAGGTGACCAAGTTCATGCT-3¢, HEX接头序列为5¢-GAAGGTCGGAGTCAACGGATT-3¢。所有引物均由华大生物技术有限公司合成(北京, http://www.genomics.cn/)。将合成后的引物配置成引物Mix (2条正向引物为12 mmol L–1, 反向引物为30 mmol L–1), 并用亲本和抗感池验证标记的多态性,然后进行遗传群体的基因型分析。
KASP检测基于LGC Genomics公司(https://www. lgcgroup.com/cn)提供的方法。反应体系为2.5 μL 2×KASP V4.0 Mastermix, 0.056 μL引物mix, 20~50 ng μL–1的DNA模板1 μL, ddH2O补足至5 μL。PCR程序为95 ℃预变性5 min; 94℃变性20 s, 65℃退火20 s, 每循环一次降1℃, 9个循环; 95℃变性10 s, 59℃退火60 s, 35个循环, 于4~10℃环境下保存产物。PCR完成后放在酶标仪中读取终端荧光数据, 随后导入Kluster Caller软件进行基因分型。
1.6 遗传连锁图谱及QTL分析
采用JoinMap v4.0, 基于Kosambi函数(Kosambi, 1943)计算遗传距离, 构建连锁图。基于遗传图谱, 利用QTL Cartographer v2.5 (http:// statgen.ncsu.edu/qtlcart/WQTLCart.ht)的复合区间作图法(composite interval method, CIM)对不同环境下条锈病抗性及其均值进行QTL分析。若单个QTL可在2个及以上的环境下重复检测到, 则认为该QTL稳定存在[14]。
2 结果与分析
2.1 铭贤169/Holdfast RIL群体条锈病MDS表型分析
成株抗性品种Holdfast在成都2015、甘谷2015、中梁2015、成都2016和甘谷2016五个环境下平均MDS分别为2.0%、20.1%、19.8%、20.0%和17.0%, 铭贤169在5个环境下平均MDS分别为50.8%、73.3%、69.0%、78.3%和65.3%。铭贤169/Holdfast RIL群体在5个环境下平均MDS值分别为46.7%、70.1%、73.7%、72.9%和67.6%, 变异范围分别为0~90.0%、20.0%~100.0%、10.5%~100.0%、20.0%~ 100.0%和15.0%~90.0%。RIL家系MDS值在多数环境间相关系数达显著或极显著水平(表1), 表型数据相对稳定。
铭贤169/Holdfast RIL群体的MDS值在5个环境下均呈现连续性变异, 表明该群体条锈病成株抗性为典型的数量性状遗传(图1)。此外, 方差分析表明, 该群体后代MDS在不同环境、不同株系、环境内不同重复以及基因型与环境互作间差异均达到极显著水平(< 0.001)(表2), 其MDS的广义遗传力为0.65。以上结果表明抗病基因和环境共同影响条锈病成株抗性。
**显著性水平(< 0.01);*显著性水平(< 0.05)。**Significance at< 0.01;*Significance at< 0.05.
a: 中梁2015; b: 成都2015; c: 甘谷2015; d: 成都2016; e: 甘谷2016; f: 总平均。
a: Zhongliang 2015; b: Chengdu 2015; c: Gangu 2015; d: Chengdu 2016; e: Gangu 2016; f: mean MDS across five environments.

表2 铭贤169/Holdfast RIL群体小麦条锈病MDS方差分析
**代表0.01显著水平。**Significance at< 0.01.
2.2 铭贤169/Holdfast RIL群体抗感池及其亲本SNP芯片分析
利用小麦660K SNP芯片对基于条锈病抗性构建的2组抗感池和双亲进行扫描。参照660K芯片SNP公共遗传图谱[19], 共计1748个具有染色体位置信息的多态性SNP位点在抗感池和亲本间均存在差异。在小麦21条染色体中, 7A染色体上的多态性位点最多且分布集中(421.1~439.8 Mb区段), 其次为5B和5A染色体(图2)。
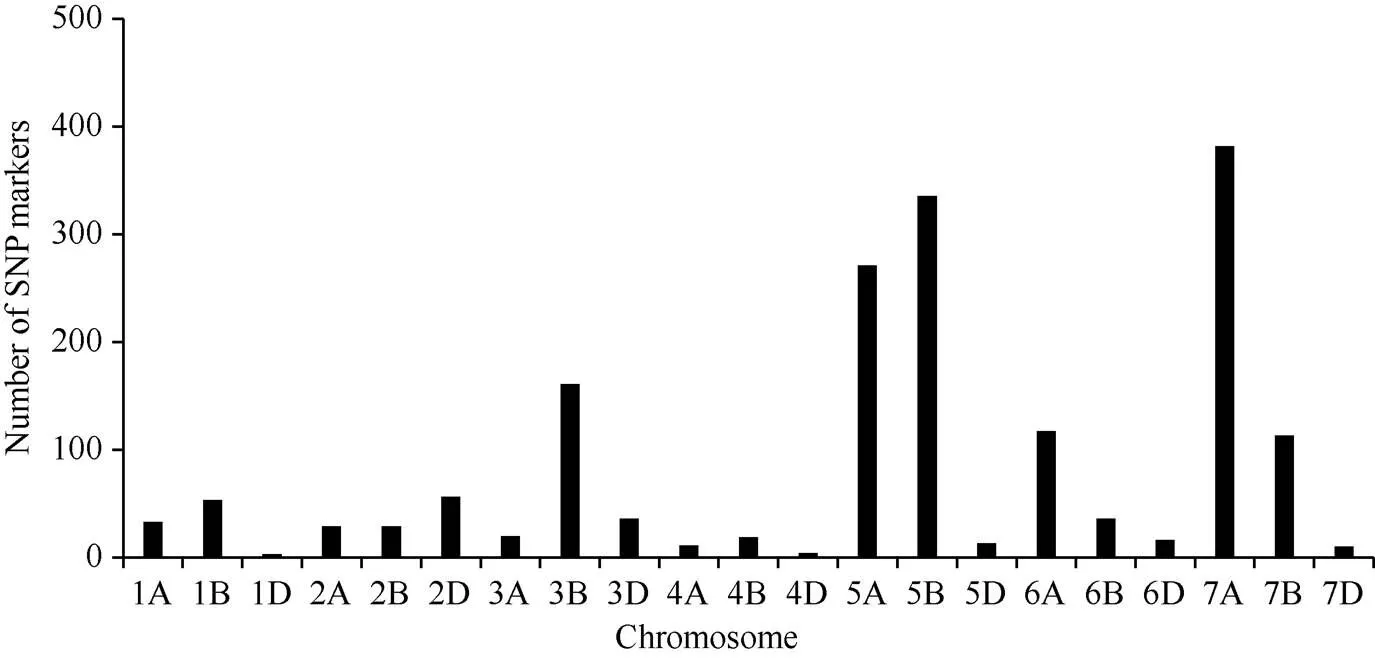
图2 铭贤169/Holdfast RIL群体中多态性SNP在基因组中的分布
2.3 基因型信息分析
参照小麦IWGSC v1.0参考基因组(https://urgi. versailles.inra.fr/jbrowseiwgsc/gmod_jbrowse/?data=my Data% 2FIWGSC_RefSeq_v1.0), 利用本地Blast获取差异SNP侧翼序列。基于差异SNP侧翼序列, 在Poly Marker网站设计特异性引物。选择位于4B、5B、5A、6A和7B染色体上的96对KASP引物, 利用抗、感池和亲本对96对引物进行筛选。用筛选后的多态性引物对铭贤169/Holdfast RIL群体目标区段进行扫描, 最终在5A染色体上有3个可用标记(、和), 7A染色体上有15个可用标记(、、、、、、、、、、、、、和)。在5B染色体上开发了21个KASP标记, 但通过酶标仪对抗感株系检测时, 多态性标记分散, 无法构建完整的连锁图, 在5B染色体上未检测到QTL。
2.4 遗传连锁图谱和成株抗性QTL分析
结合基因型数据和条锈病MDS表型数据, 对铭贤169/Holdfast RIL群体进行条锈病成株抗性基因定位。结果在5A染色体长臂上定位到1个条锈病成株抗性位点, 与KASP标记和紧密连锁, 距离分别为0.5 cM和1.1 cM, 命名为, 在中梁2015、成都2015、成都2016、甘谷2015年和甘谷2016以及均值环境下均存在, 可解释6.5%~9.3%的表型变异。在7A染色体长臂上定位到1个稳定的条锈病成株抗性QTL, 命名为, 在甘谷2015和甘谷2016两个环境中存在, 分别解释6.2%和7.3%的表型变异, 两侧标记分别为(0.5 cM)和(0.7 cM)。

图3 铭贤169/Holdfast RIL 群体条锈病成株抗性QTL (5AL和7AL)的LOD值曲线图
对含有和抗病等位基因和感病等位基因的家系分析发现, 含有抗病等位基因家系的MDS要显著低于含有感病等位基因家系的MDS值(图4, 附表1)。

图4 铭贤169/HoldfastRIL群体中Qyr.gaas-5AL和Qyr.gaas-7AL对条锈病抗性的等位基因效应
*显著性水平(<0.05)。*Significant at<0.05.
3 讨论
近年来, 我国小麦条锈菌生理小种变异加速, 多个具有较大推广面积、携带苗期抗性基因的品种丧失抗性, 严重威胁我国小麦生产安全。寻找新的抗病资源, 发掘抗性基因及其紧密连锁标记, 对于解析条锈病抗性遗传机制, 加快抗病育种进程具有重要作用。甘肃陇南地区是小麦条锈菌最大的越夏区和新小种发源地, 为小麦主产区条锈病的发生提供大量的菌源[20-21]。因此, 甘肃陇南地区是我国小麦条锈病防治的关键地带。发掘新的抗病基因, 培育适宜于甘肃陇南地区种植的抗病品种是防治我国小麦条锈病的重要措施[22]。成株抗性小麦品种成株期病害严重度低, 田间病情动态消长慢, 病斑面积小, 潜育期长, 对产量的影响较小, 且抗性可维持时间较长。当前, 国际上多数国家和地区已将成株抗性的利用作为小麦抗病育种的主攻方向[23-26]。20世纪60年代至今, 国际玉米小麦改良中心(International Maize and Wheat Improvement Center, CIMMYT)选育出一大批成株抗性品种, 如Parula、Trap、Jupateco 73R、Pavon 76、Attila等, 均携带或及其他2~3个微效基因[27-28]。美国、澳大利亚和欧洲也对条锈病成株抗性进行了深入研究, 并成功用于生产实践。近年来, 我国在成株抗性研究和应用方面也取得了快速进展, 对部分优良成株抗性品种(如咸农4号、平原50、Libellula、鲁麦21、百农64等)进行遗传分析, 并利用分子标记辅助选择(Molecular marker assisted selection, MAS)培育出一批优良的成株抗性品系[29-30]。Holdfast在甘肃陇南地区长期种植, 携带多个抗性基因, 在小麦成株抗性育种中被广泛应用[15-17]。中梁30是由天水市农业科学研究所以Holdfast为母本选育而成的冬小麦新品种, 农艺性状优良, 对小麦条锈病具有持久抗性[31]。发掘Holdfast中的条锈病成株抗性基因, 对于培育适宜于甘肃陇南地区种植的持久抗性品种具有重要意义。
前人研究报道, Holdfast不仅携带苗期抗性基因, 还同时携带成株抗性基因[15-16]; 但关于Holdfast的抗病基因研究主要集中于表型鉴定和初步遗传学分析, 其携带的成株抗病基因未见定位报道[15-17]。本研究基于BSA方法, 利用660KSNP芯片, 在铭贤169/Holdfast RIL群体中快速定位到2个条锈病成株抗性QTL:和, 在多个环境下稳定存在, 分别解释6.5%~9.3%和6.2%~7.3%的表型变异。
3.1 QYr.gaas-5AL
小麦5AL染色体上存在多个成株抗性基因, 如[32]、[33]、[34]、[26]和[3]。其中,的供体亲本是Wawht2046, 连锁标记为。来自于PI610750, 与春化基因共分离, 定位于5A长臂的端粒附近, 两侧标记为和遗传距离分别为4.4 cM和0.3 cM。对平原50/铭贤169 DH群体进行条锈病成株抗性定位, 在5AL染色体上检测到1个抗性位点, 标记区间为–, 可解释5.0%~19.9%的表型变异;的供体亲本为Opata 85, 与和紧密连锁, 距离远端26 cM处, 可解释15.0%的表型变异[27,34];供体亲本为Pau 14087, 在远端68.7 cM处, 标记区间为-[3,34]。基于IWGSC物理图谱及Maccaferri等[36]构建的整合遗传图谱,与以上QTL均不同, 该位点有可能是一个新的条锈病抗性QTL。
3.2 QYr.gaas-7AL
小麦7AL染色体上也存在多个条锈病抗性位点。来自于法国品种Recital, 位于7AL染色体上, 连锁标记区间为b–[37]; Zwart等[38]在小麦品种CPI133872检测到一个条锈病抗性QTL, 位于–区间;是来自Avocet感病植株的一个效应微小的QTL, 两侧标记为和1[39];与位于同一遗传区域(726.5 Mb处), 与紧密连锁, 而的遗传标记为[40]。基于IWGSC小麦物理图谱及Maccaferri等[36]构建的整合遗传图谱,与以上位点位置不同, 可能是一个新的条锈病抗性QTL。
前人研究表明, 聚合4~5个条锈病成株抗性QTL的小麦品种在田间表现高水平抗性[2,29,41], 是实现品种持久抗性的重要方法。在本研究中,和均表现出稳定的条锈病成株抗性。和及其紧密连锁的KASP标记, 可以用于分子标记辅助选择, 进行小麦条锈病成株抗性QTL聚合育种。
3.3 KASP分型的优势
近年来, KASP技术发展迅速, 广泛应用于QTL精细定位、种子质量检测、分子标记辅助育种和种质资源鉴定工作, 可快速准确确定品种的基因型。KASP平台是基于PCR的高通量SNP分型检测技术, 通过引物末端碱基特异性匹配来实现对SNP位点的检测。在实际应用中, 样品经PCR扩增后可立即放入酶标仪中进行荧光信号检测, 大大缩短了操作时间。与SSR标记相比, 每人每天至少可完成384×20个样品的KASP标记检测, 是SSR标记分析的20倍, 大大提高了工作效率[42]。本研究结合基因芯片和KASP平台, 利用BSA方法, 在短时间内完成了铭贤169/Holdfast RIL群体条锈病成株抗性QTL定位工作。KASP平台可有效提升数量性状基因发掘效率, 大幅促进小麦分子标记辅助选择育种进程。
4 结论
利用铭贤169/Holdfast RIL群体, 在多个环境下对条锈病MDS进行鉴定, 结合660K SNP芯片和BSA快速基因定位技术, 在5A和7A染色体上检测到2个条锈病成株抗性位点和, 分别解释6.5%~9.3%和6.2%~7.3%的表型变异。RIL群体中携带和优异抗病等位基因家系的MDS显著低于感病等位基因家系。
附表 请见网络版: 1) 本刊网站http://zwxb.chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodical-zuowxb. aspx。
[1] Wellings C R. Global status of stripe rust: a review of historical and current threats., 2011, 179: 129–141.
[2] Singh R P, Huerta-Espino J, William H M. Genetics and breeding for durable resistance to leaf and stripe rusts in wheat., 2005, 29: 121–127.
[3] Chhuneja P, Kaur S, Garg T, Ghai M, Kaur S, Prashar M, Bains N S, Goel R K, Keller B, Dhaliwal H S, Singh K. Mapping of adult plant stripe rust resistance genes in diploid A genome wheat species and their transfer to bread wheat., 2008, 116: 313–324.
[4] Lu Y M, Lan C X, Liang S S, Zhou X C, Liu D, Zhou G, Lu L Q, Jing J X, Wang M N, Xia X C, He Z H. QTL mapping for adult-plant resistance to stripe rust in Italian common wheat cultivars Libellula and Strampelli., 2009, 119: 1349–1359.
[5] Bennett F. Resistance to powdery mildew in wheat: a review of its use in agriculture and breeding programmes., 1984, 33: 297–300.
[6] Liu W, Frick M, Huel R, Nykiforuk C L, Wang X M, Gaudet D A, Eudes F, Conner R L, Kuzyk A, Chen Q, Kang Z S, Laroche A. The stripe rust resistance geneencodes an evolutionary-conserved and unique CC-NBS-LRR sequence in wheat., 2014, 7: 1740–1755.
[7] Krattinger S G, Lagudah E S, Spielmeyer W, Singh R P, Huerta-Espino J, McFadden H, Bossolini E, Selter L L, Keller B. A putative ABC transporter confers durable resistance to multiple fungal pathogens in wheat., 2009, 323: 1360–1363.
[8] McIntosh R A, Dubcovsky J, Rogers W J, Morris C, Appels R, Xia X C. Catalogue of gene symbols for wheat: 2015–2016 (Supplement). (https://shigen.nig.ac.jp/wheat/komugi/genes/macgene/supplement 2015.pdf).
[9] Nsabiyera V, Bariana H S, Qureshi N, Wong D, Hayden M J, Bansal U K. Characterization and mapping of adult plant stripe rust resistance in wheat accession Aus27284., 2018, 131: 1459–1467.
[10] Marchal C, Zhang J P, Zhang P, Fenwick P, Steuernagel B, Adamski N M, Boyd L, McIntosh R, Wulff B B H, Berry S, Lagudah E , Uauy C. ED-domain-containing immune receptors confer diverse resistance spectra to yellow rust., 2018, 9: 662–668
[11] Klymiuk V, Yaniv E, Huang L, Raats D, Fatiukha A, Chen S S, Feng L H, Frenkel Z, Krugman T, Lidzbarsky G, Chang W, Jääskeläinen M J, Schudoma C, Lars P, Laine P, Bariana H, Sela H, Saleem K, Sørensen C K, Hovmøller M S, Distelfeld A, Chalhoub B, Dubcovsky J, Korol A B, Schulman A H, Fahima T. Cloning of the wheatresistance gene sheds light on the plant tandem kinase-pseudokinase family., 2018, 9: 3735.
[12] Lagudah E S, Krattinger S G, Herrera-Foessel S, Singh R P, Huerta-Espino J, Spielmeyer W, Brown-Guedira G, Selter L L, Keller B. Gene-specific markers for the wheat genewhich confers resistance to multiple fungal pathogens., 2009, 119: 889–898.
[13] Fu D L, Uauy C, Distelfeld A, Blechl A, Epstein L, Chen X M, Sela H N, Fah T. A kinase-START gene confers temperature-dependent resistance to wheat stripe rust., 2009, 323: 1357–1360.
[14] Veldboom L R, Lee M. Genetic mapping of quantitative trait loci in maize in stress and non-stress environments: I. Grain yield and yield components., 1996, 36: 1310–1319.
[15] 仝淑玫, 蔺瑞明, 何月秋, 徐世昌. 小麦抗源Holdfast和Flinor抗条锈病主效、微效基因的遗传分析. 中国农业科学, 2006, 39: 2243–2249. Tong S M, Lin R M, He Y Q, Xu S C. Genetic analysis of major and minor gene(s) resistant to stripe rust in resource wheat cultivars Holdfast and Flinor., 2006, 39: 2243–2249 (in Chinese with English abstract).
[16] 张振羽, 冯晶, 白玉路, 蔺瑞明, 彭云良, 徐世昌. 小麦重要抗源Holdfast抗条锈性遗传分析. 中国农业大学学报, 2011, 16(5): 1–5. Zhang Z Y, Feng J, Bai Y L, Lin R M, Peng Y L, Xu S C. Genetic analysis of resistance to stripe rust in wheat cultivar Holdfast., 2011, 16(5): 1–5 (in Chinese with English abstract).
[17] 孙建鲁, 王吐虹, 冯晶, 蔺瑞明, 王凤涛, 姚强, 郭青云, 徐世昌. 100个小麦品种资源抗条锈性鉴定及重要抗条锈病基因的SSR检测. 植物保护, 2017, 43(2): 64–72. Sun J L, Wang T H, Feng J, Lin R M, Wang F T, Yao Q, Guo Q Y, Xu S C. Identification of resistance to wheat stripe rust and detection of known resistance genes in 100 wheat cultivars with SSR markers., 2017, 43(2): 64–72 (in Chinese with English abstract)
[18] Peterson R F, Campbell A B, Hannah A E. A diagrammatic scale of estimating rust intensity on leaves and stems of cereals., 1948, 26: 496–500.
[19] Cui F, Zhang N, Fan X L, Zhang W, Zhao C H, Yang L J, Pan R Q, Chen M, Han J, Zhao X Q, Ji J, Tong Y P, Zhang H X, Jia J Z, Zhao G Y, Li J M. Utilization of a Wheat 660K SNP array-derived high-density genetic map for high-resolution mapping of a major QTL for kernel number., 2017, 3788: 1–12.
[20] 陆宁海, 郑文明, 王建峰, 詹刚明, 黄丽丽, 康振生. 陇南地区小麦条锈菌群体遗传多样性SSR分析. 中国农业科学, 2009, 42: 2763–2770. Lu N H, Zheng W M, Wang J F, Zhan G M, Huang L L, Kang Z S. SSR Analysis of population genetic diversity off. sp.in Longnan region of Gansu, China., 2009, 42: 2763–2770 (in Chinese with English abstract).
[21] 周祥椿, 吴立人, 宋建荣, 金社林. 陇南小麦条锈病的品种遗传多样性控制. 植物保护学报, 2008, 35(2): 97–101. Zhou X C, Wu L R, Song J R, Jin S L. Control of wheat stripe rust based on genetic diversity of cultivars in Longnan., 2008, 35(2): 97–101 (in Chinese with English abstract).
[22] Line R F, Chen X M. Success in breeding for and managing durable resistance to wheat rusts., 1995, 79: 1254–1255.
[23] Chen X M, Line R F. Gene action in wheat cultivars for durable high-temperature adult-plant resistance and interactions with race-specific, seedling resistance to stripe rust caused by., 1995, 85: 567–572.
[24] Bariana H S, Kailasapillai S, Brown G N, Sharp P J. Marker assisted identification ofin the National Cereal Rust Control Program in Australia. In: Slinkard A E. Proceedings of 9th International Wheat and Genetic Symposium. University of Saskatchewan, Saskatoon: University of Extension Press, 1998, 3: 83–91.
[25] Keller M, Keller B, Schachermayr G, Winzeler M, Schmid J E, Stamp P, Messmer M M. Quantitative trait loci for resistance against powdery mildew in a segregating wheat×spelt population., 1999, 98: 903–912.
[26] Boukhatem N, Baret P V, Mingeot D, Jacquemin J M. Quantitative trait loci for resistance against yellow rust in two wheat-derived recombinant inbred line populations., 2002, 104: 111–118.
[27] Singh R P, Rajaram S. Genetics of adult-plant resistance to leaf rust in ‘Frontana’ and three CIMMYT wheats., 1992, 35: 24–31.
[28] William H M, Singh R P, Huerta-Espino J, Ortiz-Islas S, Hoisington D. Molecular marker mapping of leaf rust resistance geneand its association with stripe rust resistance genein wheat., 2003, 93: 153–159.
[29] 何中虎, 兰彩霞, 陈新民, 邹裕春, 庄巧生, 夏先春. 小麦条锈病和白粉病成株抗性研究进展与展望. 中国农业科学, 2011, 44: 2193–2215. He Z H, Lan C X, Chen X M, Zou Y C, Zhuang Q S, Xia X C. Progress and perspective in research of adult-plant resistance to stripe rust and powdery mildew in wheat., 2011, 44: 2193–2215 (in Chinese with English abstract).
[30] 刘金栋, 杨恩年, 肖永贵, 陈新民, 伍玲, 白斌, 李在峰, Garry M, Rosewarne G M, 夏先春, 何中虎. 兼抗型成株抗性小麦品系的培育、鉴定与分子检测. 作物学报, 2015, 41: 1472 –1480. Liu J D, Yang E N, Xiao Y G, Chen X M, Wu L, Bai B, Li Z F, Rosewarne G M, Xia X C, He Z H. Development, field and molecular characterization of advanced lines with pleiotropic adult-plant resistance in common wheat., 2015, 41: 1472–1480 (in Chinese with English abstract).
[31] 宋建荣, 岳维云, 吕莉莉, 张耀辉, 刘鸿燕, 王娜, 南海, 周喜旺. 抗条锈丰产冬小麦新品种——中梁30号. 麦类作物学报, 2011, 31: 186. Song J R, Yue W Y, Lyu L L, Zhang Y H, Liu H Y, Wang N N, Nan H, Zhou X W. Zhongliang 30: a new cultivar of winter wheat with resistance to stripe rust and high yield., 2011, 31: 186 (in Chinese with English abstract).
[32] Bariana H S, Parry N, Barclay I R, Loughman R, McLean R J, Shankar M, Wilson R E, Willey N J, Francki M. Identification and characterization of stripe rust resistance genein common wheat.2006, 112: 1143–1148.
[33] Lowe I, Jankuloski L, Chao S, Chen X M, See D, Dubcovsky J. Mapping and validation of QTL which confer partial resistance to broadly virulant post-2000 North American races of stripe rust in hexapliod wheat., 2011, 123: 143 –157
[34] Lan C X, Liang S S, Zhou X C, Liu D, Zhou G, Lu Q L, Xia X C, He Z H. Quantitative trait loci mapping for adult-plant resistance against stripe rust in Chinese wheat cultivar Pingyuan 50., 2010, 100: 313–318.
[35] Qureshi N, Bariana H S, Zhang P, McIntosh R, Bansal U K. Genetic relationship of stripe rust resistance genesandin wheat and identification of linked KASP markers., 2018, 102: 413-420.
[36] Maccaferri M, Ricci A, Salvi S, Milner S G, Noli E, Martelli P L, Casadio R, Akhunov E, Scalabrin S, Vendramin V, Ammar K, Blanco A, Desiderio F, Distelfeld A, Dubcovsky J, Fahima T, Faris J, Korol A, Massi A, Mastrangelo A M, Morgante M, Pozniak C, N’Diaye A, Xu S, Tuberosa R. A high-density, SNP-based consensus map of tetraploid wheat as a bridge to integrate durum and bread wheat genomics and breeding., 2015, 13: 648–663.
[37] Dedryver F, Paillard S, Mallard S, Robert O, Trottet M, Nègre S, Verplancke G, Jahier J. Characterization of genetic components involved in durable resistance to stripe rust in the bread wheat ‘Renan’., 2009, 99: 969–973.
[38] Zwart R S, Thompson J P, Milgate A W, Bansal U K, Williamson P M, Raman H, Bariana H S. QTL mapping of multiple foliar disease and root-lesion nematode resistances in wheat., 2010, 26: 107–124.
[39] Rosewarne G, Singh R, Huerta-Espino J, Herrera-Foessel S, Forrest K, Hayden M, Rebetzke G. Analysis of leaf and stripe rust severities reveals pathotype changes and multiple minor QTLs associated with resistance in an Avocet × Pastor wheat population., 2012, 124: 1283–1294.
[40] Liu W Z, Maccaferri M, Chen X M, Laghetti G, Pignone D, Pumphrey M, Tuberosa R. Genome‑wide association mapping reveals a rich genetic architecture of stripe rust resistance loci in emmer wheat (ssp.)., 2017, 130: 2249–2270.
[41] 白斌. 普通小麦条锈病成株抗性QTL定位与白粉病成株抗性QTL聚合. 西北农林科技大学博士论文, 陕西杨凌, 2014. Bai B. Mapping of QTLs for Adult-plant Resistance to Stripe Rust and Pyramiding of QTLs for Adult-plant Resistance to Powdery Mildew in Common Wheat. PhD Dissertation of Northwest A&F University, Yangling, Shaanxi, China, 2014 (in Chinese with English abstract).
[42] 吴建辉. 基于BSR-Seq和芯片技术的抗条锈基因候选基因分析及普通小麦成株期抗条锈QTL定位. 西北农林科技大学博士学位论文, 2017, 陕西杨凌. Wu J H. QTL Mapping for Adult-plant Resistance to Stripe Rust in Common Wheat and Candidate Gene Analysis ofBased on BSR-Seq and SNP Array. PhD Dissertation of Northwest A&F University, Yangling, Shaanxi, China, 2017 (in Chinese with English abstract).
QTL mapping of adult-plant resistance to stripe rust in wheat variety holdfast
YANG Fang-Ping1,2,**, LIU Jin-Dong2,3,**, GUO Ying1, JIA Ao-Lin2, WEN Wei-E2, CHAO Kai-Xiang4, WU Ling5, YUE Wei-Yun6, DONG Ya-Chao2, and XIA Xian-Chun2,*
1Wheat Research Institute, Gansu Academy of Agricultural Sciences, Lanzhou 730070, Gansu, China;2Institute of Crop Sciences, Chinese Academy of Agricultural Sciences / National Wheat Improvement Center, Beijing 100081, China;3Institute of Shenzhen Agricultural Genome, Chinese Academy of Agricultural Sciences, Shenzhen 518120, Guangdong, China;4College of Plant Protection, Northwest A&F University, Yangling 712100, Shaanxi, China;5Crop Research Institute, Sichuan Academy of Agricultural Sciences, Chengdu 610066, Sichuan, China;6Tianshui Institute of Agricultural Sciences, Tianshui 741200, Gansu, China
Holdfast is an elite wheat cultivar from the United Kingdom with well durable resistance to stripe rust for many years. The aim of the present study was to identify adult-plant resistance (APR) genes to stripe rust in Holdfast and closely linked molecular markers to provide materials and methods for breeding wheat cultivars with durable resistance. A recombinant inbred lines (RIL) population, generated from the cross between Mingxian 169 (highly susceptible) and Holdfast, was planted in Zhongliang and Gangu of Gansu province, and Chengdu of Sichuan province during 2014−2015 and 2015−2016 cropping seasons for evaluating maximum disease severities (MDS) of stripe rust. The chromosomal locations of QTL were firstly determined based on the wheat 660K SNP array and bulked segregant analysis. Then kompetitive allele specific PCR (KASP) markers were developed to genotype the entire RIL population. Finally, the quantitative trait loci (QTLs) mapping of adult-plant resistance to stripe rust in the RIL population were performed, and two QTLs for APR to stripe rust were mapped on chromosomes 5AL and 7AL, respectively. The QTL on chromosome 5AL, designated, was identified in all environments, including Zhongliang 2015, Chengdu 2015, Chengdu 2016, Gangu 2015, Gangu 2016, explaining phenotypic variances ranging from 6.5% to 9.3%.was flanked by markersandwith genetic distances of 0.5 cM and 1.1 cM, respectively. The 7AL QTL, designated, was detected in two environments Gangu 2015 and Gangu 2016, explaining 6.2% and 7.3% of the phenotypic variances, respectively.was mapped between markersandwith genetic distances of 0.5 cM and 0.7 cM, respectively. The RILs containing the resistance allele atandloci were more resistant to stripe rust and had significantly lower MDS than those without the resistance alleles ofand, indicating thatandcan effectively reduce stripe rust severities, thus may be used in wheat breeding for improvement of stripe rust resistance.
common wheat; stripe rust; adult-plant resistance; SNP markers; linkage analysis
本研究由国家自然科学基金项目(31360336), 兰州市科技计划项目(2019-1-81)和甘肃省农业科学院青年基金项目(2019GAAS41)资助。
This study was supported by the National Natural Science Foundation of China (31360336), the Program of Science and Technology of Lanzhou (2019-1-81), and the Youth Fund of Gansu Academy of Agricultural Sciences (2019GAAS41).
夏先春, E-mail: xiaxianchun@caas.cn
**同等贡献(Contributed equally to this work)
杨芳萍, E-mail: yfp1023@163.com; 刘金栋, Liujindong_1990@163.com
2019-04-02;
2019-06-24;
2019-07-19.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20190719.1457.006.html
10.3724/SP.J.1006.2019.91026

