杭州地区相同分子型别的单核细胞增生李斯特菌临床和食品分离株基因组分析
俞 骅,董华丽,汪皓秋,楼秀芹,刘 涛,张 蔚,王旭初,潘劲草
单核细胞增生李斯特菌(Listeriamonocytogenes)是一种常见的食源性致病菌,在肉、蔬菜、水果、冷冻饮品等食品中均能检出,可引起人畜疾病。免疫力低下者摄入该菌会引起李斯特菌病,症状主要为脑膜炎、败血症和流产等,严重的可以造成死亡。李斯特菌病平均潜伏期为2~3周,最长可超过70 d[1-2]。一旦发病,特别是散发病例,进行食物来源溯源非常困难。近年来在欧美发达国家造成多起由其引起的食物中毒严重暴发事件,死亡率甚至可达20%~30%[3]。而在国内散发病例(如造成孕产妇流产)较为常见[4]。
引起李斯特菌病的血清型主要是1/2a、1/2b和4b,分子分型和溯源一般采用脉冲场凝胶电泳(Pulse-field gel electrophoresis,PFGE)。但传统的分子分型方法受限于分析位点限制,分辨力有限。随着高通量测序和生物信息学技术的快速发展,基于细菌核心基因组或全基因组SNPs位点进行溯源,因其分辨率高、重复性好,被越来越广泛使用[5]。本研究对杭州地区一起食物暴露不明确的李斯特菌病散发事件的临床分离株,和流行病学无关联但具有相同PFGE型别的食品分离株进行了基因组测序,分析基因组序列差异。并与NCBI Assembly公共数据库中相同多位点序列分型(Multi-locus Sequence Typing, MLST)型别的菌株进行基因组SNPs位点进化分析,研究可能的污染食品溯源与传播途径。
1 材料与方法
1.1实验与分析菌株 选择PFGEApaI和AscI酶切图谱完全一致(见图1)的杭州地区2007-2011年单核细胞增生李斯特菌食品分离株6株和2012年临床分离株1株(王某,女,22y,中孕,胎死宫内,高热入院,B超宫内死胎,分离自血培养需氧厌氧双瓶培养瓶)。多位点序列分型(Multi-locus sequence typing, MLST)结果均为ST8型,菌株背景信息参见表1。对NCBI Assembly公共数据库中613株单核细胞增生李斯特菌基因组进行MLST分析,获得ST8型的基因组。
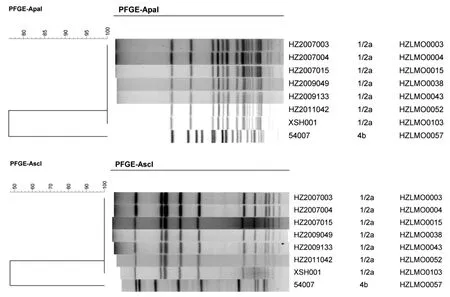
图1 7株杭州本地食品与临床来源ST8型单核细胞增生李斯特菌PFGE分型图谱,CMCC 54007为参考菌株Fig.1 Seven local food and clinical isolates of ST8 type of Listeria monocytogenes PFGE cluster, CMCC 54007 was the reference strain
1.2DNA提取、基因组测序、质控和组装 采用QIAamp DNA mini Kit试剂盒(德国Qiagen公司,货号:51304)提取基因组DNA;采用 Nextera XT DNA文库构建试剂盒(美国illumina公司,型号:FC-131-1024)进行文库构建;基因组测序应用illumina Miseq平台,使用Miseq Reagent Kit v3试剂盒(美国illumina公司,型号:MS-102-3003)。以上操作均按试剂盒说明书进行。对测序reads使用FastQC[6]验证测序质量,用Trimmomatic[7]去除接头序列。使用shovill[8]和Spades[9]进行基因组拼接。基因组拼接序列使用Quast[10]验证质量。
1.3毒力基因、ncRNA鉴定和质粒扫描 基因组测序短序列使用srst2[11],拼接序列使用MLST[12]工具扫描获得ST型别;用Prokka[13]注释拼接基因组,使用Infernal[14]以Rfam[15]为数据库鉴定ncRNAs。VFDB(http://www.mgc.ac.cn/VFs)数据库中66个单核细胞增生李斯特菌毒力基因做为鉴定数据库,用blastn扫描基因组毒力基因。对本地菌株的基因组拼接序列提交到NCBI blast进行比对,鉴定是否携带已知质粒。
1.4基因组共线性和SNPs分析 用Sibelia[16]将杭州本地临床分离株12-103基因组分别与杭州地区ST8型食品分离株,以及公共数据库中2株临床来源的基因组完成图的菌株Lm-N1546,IZSAM_Lm_15_17439_A144和1株临床来源的基因组草图的菌株LM05-00401进行共线性比对。以LM05-00401为参考基因组,用Harvest[17]比对获得ST8型全基因组SNPs序列。使用Gubbins[18]去除重组,RAxML[19]构建基于SNPs的最大似然法进化树(模型采用GTR+G,bootstrap=1 000),使用ggtree[20]绘制进化树。
2 结 果
2.1基因组测序与公共数据库菌株 7株杭州地区ST8型单核细胞增生李斯特菌基因组测序数据及拼接结果参见表1。采用mlst扫描后,NCBI Assembly公共数据库中613个单核细胞增生李斯特菌基因组中可获得已知ST型别的有600个,其中有41株ST8型单核细胞增生李斯特菌株,提取这41株ST8型菌株在NCBI Biosample数据库中的背景信息,具体参见表2。
2.2毒力基因、ncRNAs序列和质粒分布 毒力基因进行扫描结果显示除了Lm-N1546和IZSAM-Lm-15-17439-A144缺失virR基因外,其他ST8型菌株均携带毒力基因(表3)。杭州本地菌株中12-103的lmo1136多一个9 bp的重复序列,12-004和12-015的lmo2396少204 bp的片段。杭州地区菌株的ncRNAs分子只有rli48、rli62和rliG在分布上有差异。公共数据库中的菌株只有LM66和LM71缺少rli31。12-003和12-004未发现携带质粒,其余5株携带pLmA144(KU513859)质粒。菌株的ncRNAs和质粒差异见表3。
2.3基因组共线性和SNPs分析 杭州地区临床分离株12-103与6株食品分离株和3株国外临床分离株大于15 kbp片段的共线性均一致,没有大片段重组现象(共线性结果参见图2)。基因组SNPs进化树结果杭州地区分离株与BCW_3902形成一个的分支。总体上12-103与杭州食品分离株高度近源(进化树参见图3),12-004与临床株12-103的进化上最接近,只有16个SNPs差异,相差最多的是24个SNPs位点的12-015。
表1 杭州地区单核细胞增生李斯特菌分离株及基因组测序结果
Tab.1L.monocytogenesisolates and genome sequencing results in Hangzhou
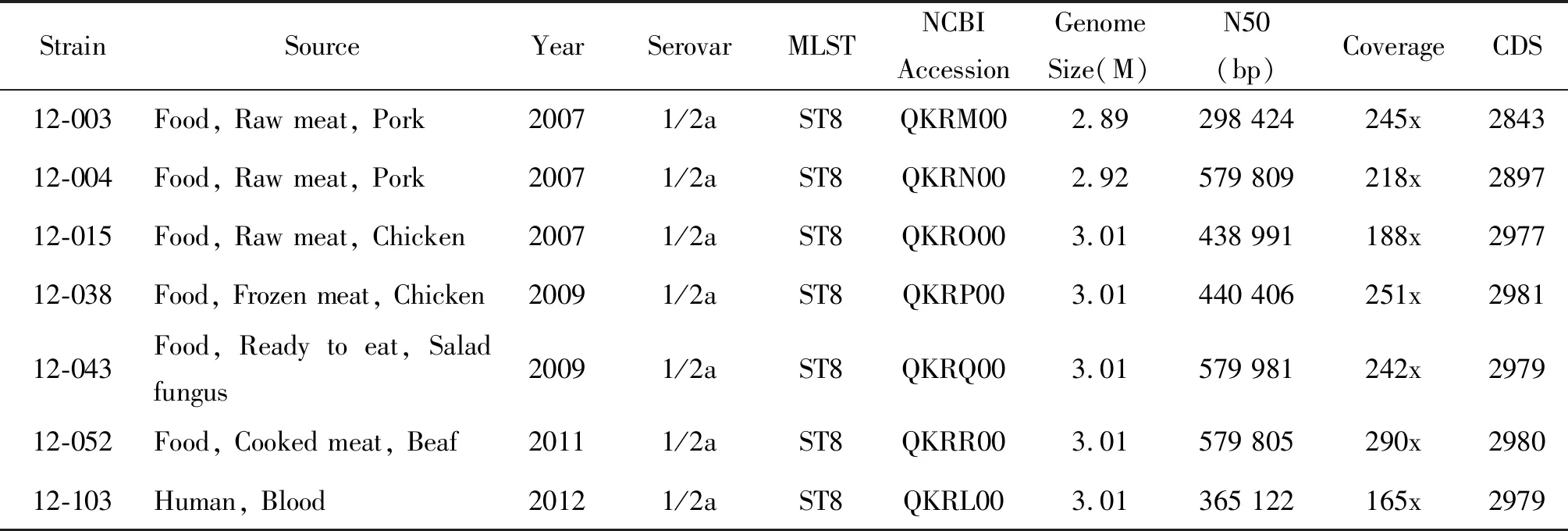
StrainSourceYearSerovarMLSTNCBI AccessionGenomeSize(M)N50(bp)CoverageCDS12-003Food, Raw meat, Pork20071/2aST8QKRM002.89298 424245x284312-004Food, Raw meat, Pork20071/2aST8QKRN002.92579 809218x289712-015Food, Raw meat, Chicken20071/2aST8QKRO003.01438 991188x297712-038Food, Frozen meat, Chicken20091/2aST8QKRP003.01440 406251x298112-043Food, Ready to eat, Salad fungus20091/2aST8QKRQ003.01579 981242x297912-052Food, Cooked meat, Beaf20111/2aST8QKRR003.01579 805290x298012-103Human, Blood20121/2aST8QKRL003.01365 122165x2979
表2 NCBI基因组拼接数据库中ST8的菌株基因组
Tab.2 Genomes information of ST8 in NCBI assembly database
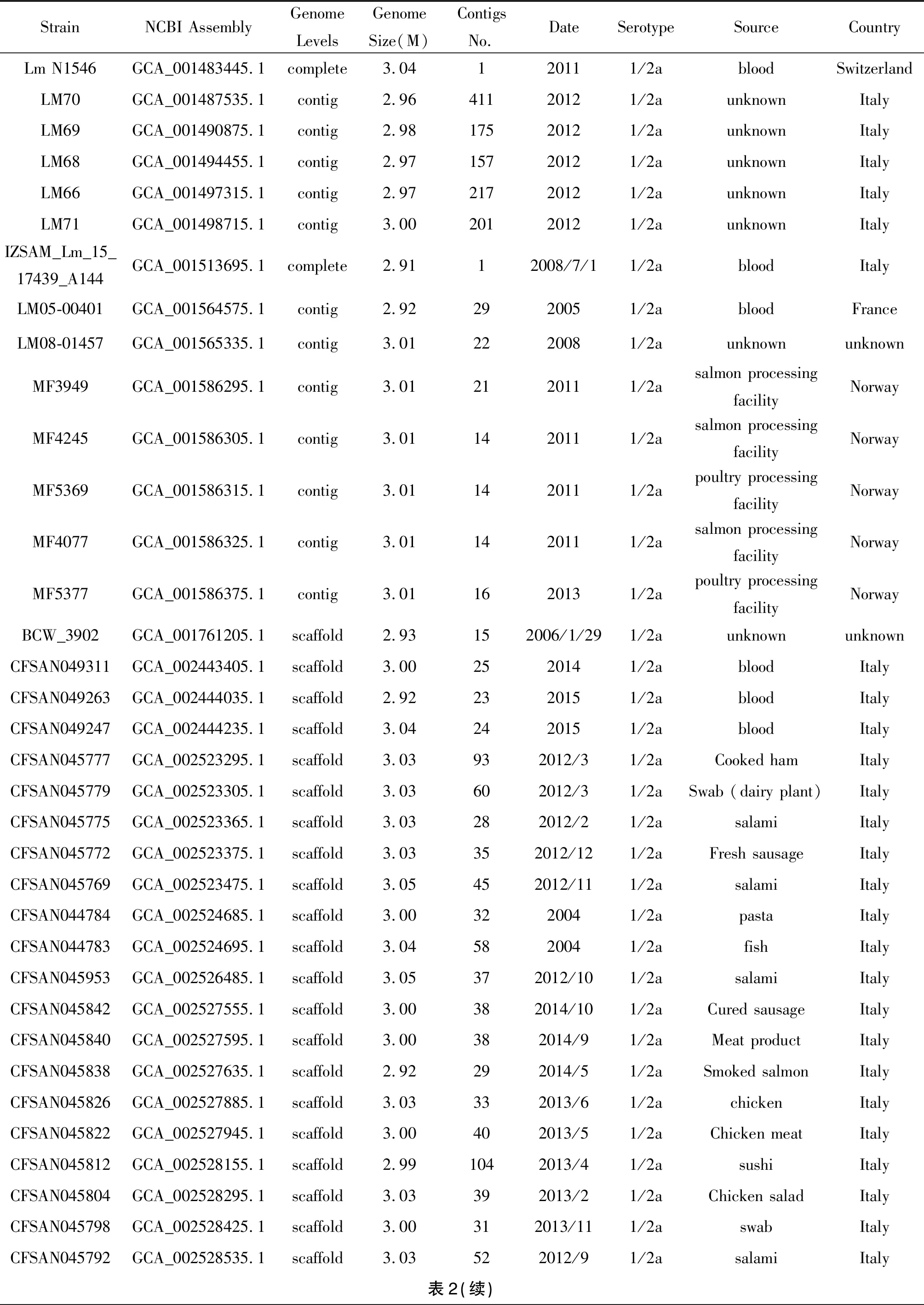
StrainNCBI AssemblyGenome LevelsGenome Size(M)Contigs No.DateSerotypeSourceCountryLm N1546GCA_001483445.1complete3.04120111/2abloodSwitzerlandLM70GCA_001487535.1contig2.9641120121/2aunknownItalyLM69GCA_001490875.1contig2.9817520121/2aunknownItalyLM68GCA_001494455.1contig2.9715720121/2aunknownItalyLM66GCA_001497315.1contig2.9721720121/2aunknownItalyLM71GCA_001498715.1contig3.0020120121/2aunknownItalyIZSAM_Lm_15_17439_A144GCA_001513695.1complete2.9112008/7/11/2abloodItalyLM05-00401GCA_001564575.1contig2.922920051/2abloodFranceLM08-01457GCA_001565335.1contig3.012220081/2aunknownunknownMF3949GCA_001586295.1contig3.012120111/2asalmon processing facilityNorwayMF4245GCA_001586305.1contig3.011420111/2asalmon processing facilityNorwayMF5369GCA_001586315.1contig3.011420111/2apoultry processing facilityNorwayMF4077GCA_001586325.1contig3.011420111/2asalmon processing facilityNorwayMF5377GCA_001586375.1contig3.011620131/2apoultry processing facilityNorwayBCW_3902GCA_001761205.1scaffold2.93152006/1/291/2aunknownunknownCFSAN049311GCA_002443405.1scaffold3.002520141/2abloodItalyCFSAN049263GCA_002444035.1scaffold2.922320151/2abloodItalyCFSAN049247GCA_002444235.1scaffold3.042420151/2abloodItalyCFSAN045777GCA_002523295.1scaffold3.03932012/31/2aCooked hamItalyCFSAN045779GCA_002523305.1scaffold3.03602012/31/2aSwab (dairy plant)ItalyCFSAN045775GCA_002523365.1scaffold3.03282012/21/2asalamiItalyCFSAN045772GCA_002523375.1scaffold3.03352012/121/2aFresh sausageItalyCFSAN045769GCA_002523475.1scaffold3.05452012/111/2asalamiItalyCFSAN044784GCA_002524685.1scaffold3.003220041/2apastaItalyCFSAN044783GCA_002524695.1scaffold3.045820041/2afishItalyCFSAN045953GCA_002526485.1scaffold3.05372012/101/2asalamiItalyCFSAN045842GCA_002527555.1scaffold3.00382014/101/2aCured sausageItalyCFSAN045840GCA_002527595.1scaffold3.00382014/91/2aMeat productItalyCFSAN045838GCA_002527635.1scaffold2.92292014/51/2aSmoked salmonItalyCFSAN045826GCA_002527885.1scaffold3.03332013/61/2achickenItalyCFSAN045822GCA_002527945.1scaffold3.00402013/51/2aChicken meatItalyCFSAN045812GCA_002528155.1scaffold2.991042013/41/2asushiItalyCFSAN045804GCA_002528295.1scaffold3.03392013/21/2aChicken saladItalyCFSAN045798GCA_002528425.1scaffold3.00312013/111/2aswabItalyCFSAN045792GCA_002528535.1scaffold3.03522012/91/2asalamiItaly表2(续)
表3 杭州本地分离株主要毒力基因、non-coding RNA、质粒分布和SNPs位点差异
Tab.3 Distribution of virulence genes, non-coding RNA, plasmid and SNPs differences in local isolates in Hangzhou

isolatelmo1136bplmo2396bpril48ril62133 bp copy130 bp copyrliGplasmidSNPs Numbers12-0031 6292 415-++missND①2312-0041 6292 211③-+-seedND①1612-0151 6292 211③-++fullpLmA144(KU513859)2412-0381 6292 415+--seedpLmA144(KU513859)2012-0431 6292 415-+-seedpLmA144(KU513859)1812-0521 6292 415---seedpLmA144(KU513859)2312-1031 638②2 415+--seedpLmA144(KU513859)/
注:① ND表示未检测到(Not detected)。
② 多一段9bp重复序列5′-gtagatccg-3′。
③ 缺失一段204bp的序列。
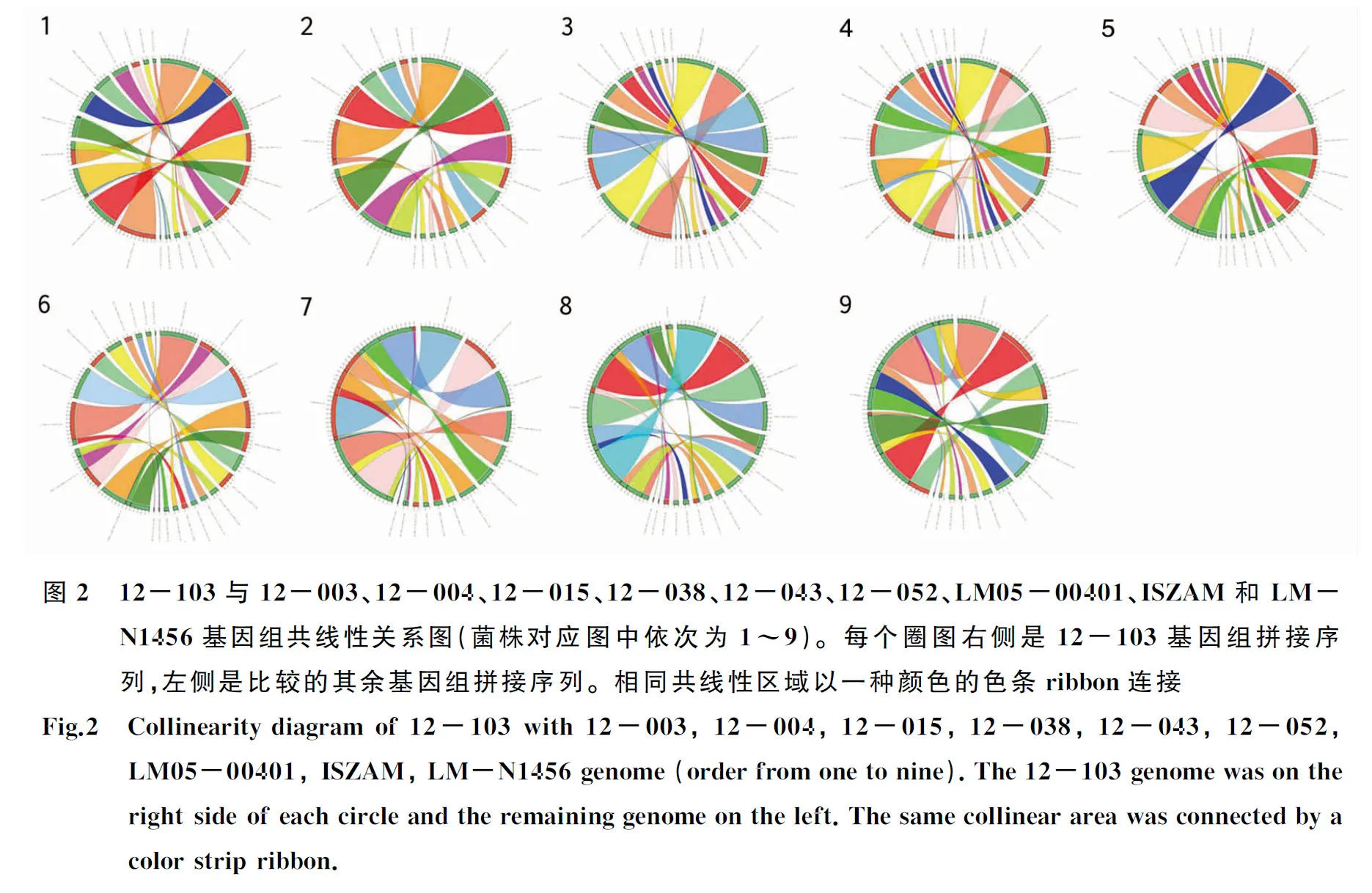
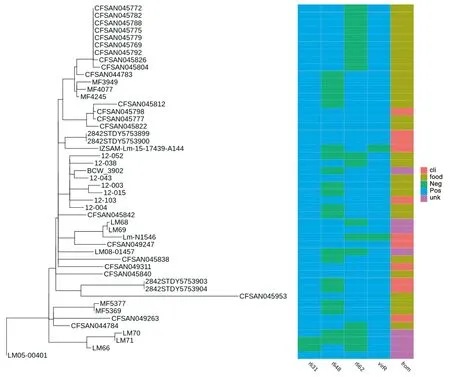
图3 ST8型单核细胞增生李斯特菌基因组SNPs位点进化树图Fig.3 Isolates of L. monocytogenes ST8 genomic SNPs phylogenic tree
3 讨 论
单核细胞增生李斯特菌由于其潜伏期长的特点,散发病例难以从流行病学调查摄入食品找到污染病原。缺乏流行病学证据,可以通过病原细菌溯源“金标准” —— PFGE分子分型的方法,以实验室证据给现场流行病学提供判定依据。但对于一些细菌的特定亚型,PFGE、MLVA等传统的分子分型技术存在分辨效果不够的问题。对于杭州地区具有相同PFGE型别的临床分离株与食品分离株要进行溯源分析,则应采用如高通量测序技术这种精度更高的方法,从基因组层面获得更为全面准确的信息。
研究表明ST8型菌株主要在肉制品或中间加工过程中分离率较高,具有较强的致病潜力,是一类高度克隆化的型别[21]。PFGE带型相同的食品分离株均来自不同年份不同地区的生禽、生畜肉,可能这类菌株对肉制品加工流程的环境适应性比较强。对于共线性结果表明12-103与其他杭州食品分离株和国外临床分离株在基因组层面没有出现大片段的重组或丢失等现象,这也与杭州地区的菌株PFGE结果带型一致相符。对于基因组contigs序列排列是否与国外临床株一致,还需要通过进一步实验验证。单核细胞增生李斯特菌毒力基因携带率很高[22],毒力调控因子virR基因起重要的调控作用,但2株临床分离株(Lm-N1546和IZSAM-Lm-15-17439-A144)却缺失该调控因子。有实验表明缺失virR基因会明显降低菌株的耐药性[23]。本研究中不同来源的ST8型菌株毒力基因分布相同,但部分基因序列结构存在差别。与致死pH值环境中的菌株酸耐受相关[24-25]的lmo0036基因序列中临床分离株12-103比其他食品分离株序列多了9 bp的富亮氨酸重复(Leucine-rich repeat)序列(gtagatccg)。lmo2396基因是internalin内化素基因,与单核细胞增生李斯特菌靶细胞识别有关[26]。食品分离株12-004和12-015此基因缺失了一个204 bp的区域。ncRNA作为一种在转录阶段调控整个生物体表达的RNA小分子,其快速调控的特点与细菌环境适应,致病性以及针对宿主免疫应答逃逸功能相关[27]。通过对本地ST8型菌株ncRNA分布扫描,结果表明ncRNAs分布高度一致,只在调控功能与细菌基因水平转移相关的rli48, rli62和rliG这3个A118 prophage类区域序列[28]位点上有差异。这些序列的差异在临床株与食品株,以及食品株之间无明显内在规律。
比较单核细胞增生李斯特菌PFGE和基因组分析的结果,基于参考基因组的ST8型菌株进化关系表明,杭州地区2007-2011年食品分离株与2012年临床分离株基因组SNPs差异在16~24个之间,基因组核心序列在进化上高度同源。结合国外分离株的基因组,显示ST8型核心基因组序列高度保守,因此提示PFGE方法可能不适合应用于该型别的分型和溯源。虽然ST8型菌株也存在一些ncRNAs分子和质粒分布上的分子差异,这些差异与致病性相关需要后续进一步进行研究。
本文针对一例临床散发单核细胞增生李斯特菌分离株与PFGE带型相同的食品分离株进行基因组序列分析,判断溯源关系。结果表明杭州市场近年肉类食品中存在遗传进化高度相似的一群ST8型单核细胞增生李斯特菌,杭州孕妇的感染非常可能来自这群菌株,因此肉制品中ST8型单核细胞增生李斯特菌引起李斯特菌病的安全风险应引起关注。
利益冲突:无

