桑树NBS.LRR类基因家族的全基因组鉴定及其调控microRNAs分析
刘潮 褚洪龙 韩利红 杨云锦 高永 唐利洲


摘要:利用生物信息学方法,全面分析了桑树NBS-LRR类基因家族组成、结构、进化、组织表达,并对该家族基因的调控microRNA( miRNA)进行了预测。共筛选到112个桑树NBS-LRR类基因,根据功能域主要分为NBS、CC-NBS、CC-NBS-LRR、NBS-LRR 4种类型。内含子数和相位分析结果显示,基因结构类型多样。聚类分析结果显示不同聚類组间存在较多的类型交叉现象。该家族基因存在组织表达特异性。大部分桑树NBS-LRR类基因均具有被miRNAs调控的可能性,miR472b和miR482b在调控该家族基因表达中可能发挥了主要作用。桑树NBS-LRR家族基因结构和进化的复杂性决定了其功能的多样性,miRNA在控制该家族基因表达的适应性成本中发挥作用。
关键词: 桑树;NBS-LRR;生物信息学;miRNA
中图分类号: S792.99
文献标识码:A
文章编号: 1000-4440( 2019) 03-0544-10
植物在与病原的长期协同进化过程中,发展出了一整套高度复杂的免疫系统,以识别并对抗病原的侵袭。植物先天免疫系统包括2层防御系统:第1层是植物通过细胞表面的模式识别受体( Patternrecognition receptors,PRRs)对保守的病原/微生物相关分子模式( Pathogen/microbe associated molecularpattern,P/MAMP)的识别,称为病原相关分子模式引发的免疫(PAMP-triggered immunity,PTI)[1-3];第2层的免疫一般发生在细胞内部,主要依靠抗性基因( Re-sistance gene)编码的多态性抗病蛋白识别无毒因子而实现,这些蛋白质多数是胞内富含亮氨酸重复序列和核苷酸结合位点蛋白( Nucleotide binding site andleucine-rich repeat proteins, NBS-LRR proteins),能直接或间接识别病原特异性效应蛋白,而激发相似的防御反应,称为效应子引发的免疫(Effector-triggeredimmunity,ETI)[1]。与PRRs相比,R蛋白在进化上出现相对较晚,在与病原互作中持续出现了大量新成员。典型的NBS-LRR类基因编码的蛋白质具有一些共同的结构域:位于中心区的NBS结构域、C端的LRR结构域、N端与Toll/白细胞介素受体(Toll-inter-leukin-l receptor,TIR)同源的结构域或卷曲螺旋( Coiled coil,CC)结构域。其中中心区的NBS作为一个信号转换器,通过与ADP或ATP的结合而改变蛋白质的钝化或激活状态,使信号关闭或开放;C端的LRR结构域是植物R蛋白中最具多态性的部分,与其识别效应子的特异性相关[4];N端的CC或TIR结构域则在下游信号的激活中发挥关键作用[5-6]。NB-ARC结构域是在细菌和真核生物中发现的信号基序,存在于植物抗病基因和动物细胞凋亡调节基因中[7],这一结构域无论是在植物还是动物的NBS-LRR蛋白中都十分保守[4],并成为克隆各种作物抗病基因类似物( Resistance gene analogs,RGAs)的NBS区域重要的序列依据。
有研究结果表明,小RNA在植物的先天免疫中发挥作用[8]。植物抗病基因存在适应性成本,NBS-LRR的高表达对植物的生长是不利的[9]。MicroRNA是一类21 nt的小分子RNA.miRNA通过转录或转录后水平调控NBS-LRR类基因的表达[1O-11]。在进化上,NBS-LRR类基因与其调控的miRNA在基因组水平上保持动态平衡,从而控制了NBS-LRR的潜在适应性成本[12]。同一家族miRNA往往有靶向保守区域,因此一个miRNA可靶向多个NBS-LRR类基因[13-14]。
川桑(Morus notabilis)是桑科桑属落叶乔木,分布于中国中部和北部各省。桑叶养蚕在中国有着悠久的历史,是中国丝绸文化的基础。桑树木材可用于制作器具,树皮可作为造纸原料,桑椹可供食用、酿酒。田间栽培中,经常出现桑萎缩病、疫病、褐斑病、赤锈病和青枯病等多种病害,往往造成植株发育不良、桑叶产量降低和品质变劣等问题,严重影响蚕桑产业的健康发展。目前,随着川桑基因组数据的公布[15],大量桑树抗病基因的研究结果先后发表[16-19],然而关于桑树NBS-LRR家族基因的研究鲜见报道。本研究利用生物信息学方法对桑树基因组中NBS-LRR类基因进行鉴定,并分析该家族基因结构、系统发育、组织表达及其调控miRNA特征,为NBS-LRR类基因功能的揭示和开发利用提供借鉴。
1 材料与方法
1.1 桑树NBS-LRR类基因的获取
以拟南芥NBS-LRR类基因序列为查询序列,搜索桑树基因组数据库( https://morus. swu. edu. cn/morusdb/),下载桑树NBS-LRR类基因序列。
1.2 桑树NBS-LRR类基因的鉴定与分类
通过GenBank数据库在线工具CDD( https://www. ncbi. nlm. nih. gov/Structure/cdd/wrpsb. cgi)鉴定蛋白质结构域。使用Pfam( http://pfam. xfam. org/)和COILS Server(http://www. ch. embnet. org/software/COILS_form. html)对候选NBS-LRR类基因编码蛋白质结构域进行确认,包括NBS、TIR、LRR、CC等,排除不含NBS结构域的基因。根据蛋白质结构域类型,将NBS-LRR类基因进行分类。
1.3 桑树NBS-LRR类基因和编码蛋白质结构分析
所有桑树NBS-LRR类基因碱基序列和编码的氨基酸序列均从桑树基因组数据库下载。使用GSDS在线软件( http://gsds. cbi. pku. edu. cn/index. php)绘制基因结构示意图。通过MEME SUITE在线工具(ht-tp://meme-suite. org/tools/meme)预测桑树NBS-LRR蛋白质氨基酸序列的保守Motif,参数为默認设置。
1.4 桑树NBS-LRR蛋白氨基酸序列比对与系统发育分析
对所有蛋白质使用ClustalX进行氨基酸序列比对。应用MEGA 5.0软件,采用邻接法(Neighbor-Joining,NJ)构建系统进化树。
1.5 桑树NBS-LRR类基因表达分析
通过桑树基因组数据库下载获得候选NBS-LRR类基因转录组数据,分别对其在根、树皮、冬芽、雄花和叶中表达进行分析。
1.6 miRNA靶标NBS-LRR类基因预测
根据序列互补原则对NBS-LRR类基因的调控miRNA进行预测。使用psRNATarget在线软件(ht-tp://plantgrn. noble. org/psRNATarget/)对文献中111个桑树miRNA[ 20-21]的靶标NBS-LRR类基因进行预测。
2 结果与分析
2.1 桑树NBS-LRR类基因的鉴定与分类
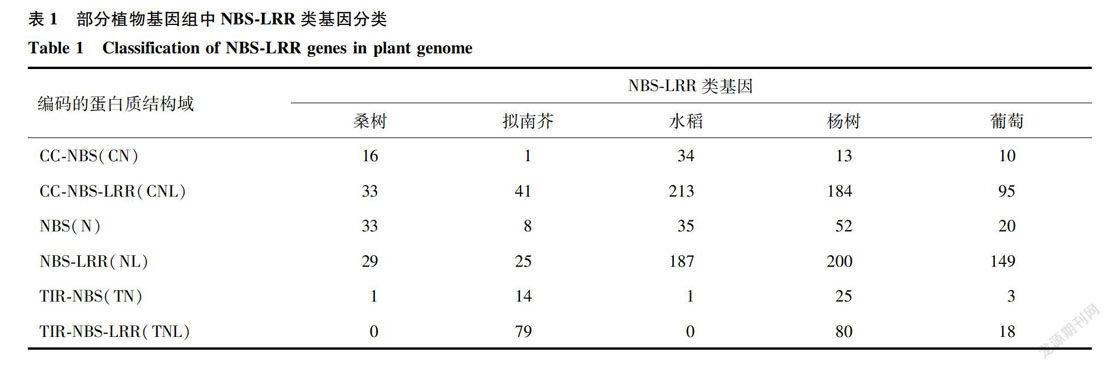
通过搜索桑树基因组数据库,共获得含有NBS核心结构的桑树NBS-LRR类候选基因112个,占桑树基因组总基因数(29 261个)的0.4%,其中编码的蛋白质具有NBS、LRR和CC完整结构域的基因共33个,占NBS-LRR类基因的29.5%(表1)。编码的蛋白质含有LRR结构域的有63个,不含LRR结构域的基因有49个。桑树NBS-LRR类基因比例与拟南芥比较接近,而明显少于其他一些物种[12];物种间NBS-IRR类基因数量存在较大差异,这可能与植物在进化过程中NBS-IRR类基因经受的选择压力有关。
研究发现,桑树NBS-LRR类基因主要分为NBS、CC-NBS、CC-NBS-LRR、NBS-LRR4种结构类型。除了具有NBS、TIR、LRR、CC结构域之外,还包含DUF、P-loop、zf-RVT和RPW8等结构域类型。发现L848_014445、L848_013225、L848_013226含有RPW8结构域,RPW8为拟南芥白粉病广谱抗性蛋白家族功能域,这些基因可能与植物的白粉病抗性有关。功能注释分析结果显示,编码抗病蛋白RPM1基因34个,编码抗病蛋白RGA基因24个,编码抗病蛋白RPP13基因12个,此外还有非活性疾病易感蛋白LOVI、晚疫抗性蛋白、TMV抗性蛋白N等。
2.2 桑树NBS-LRR类基因结构与系统发育分析
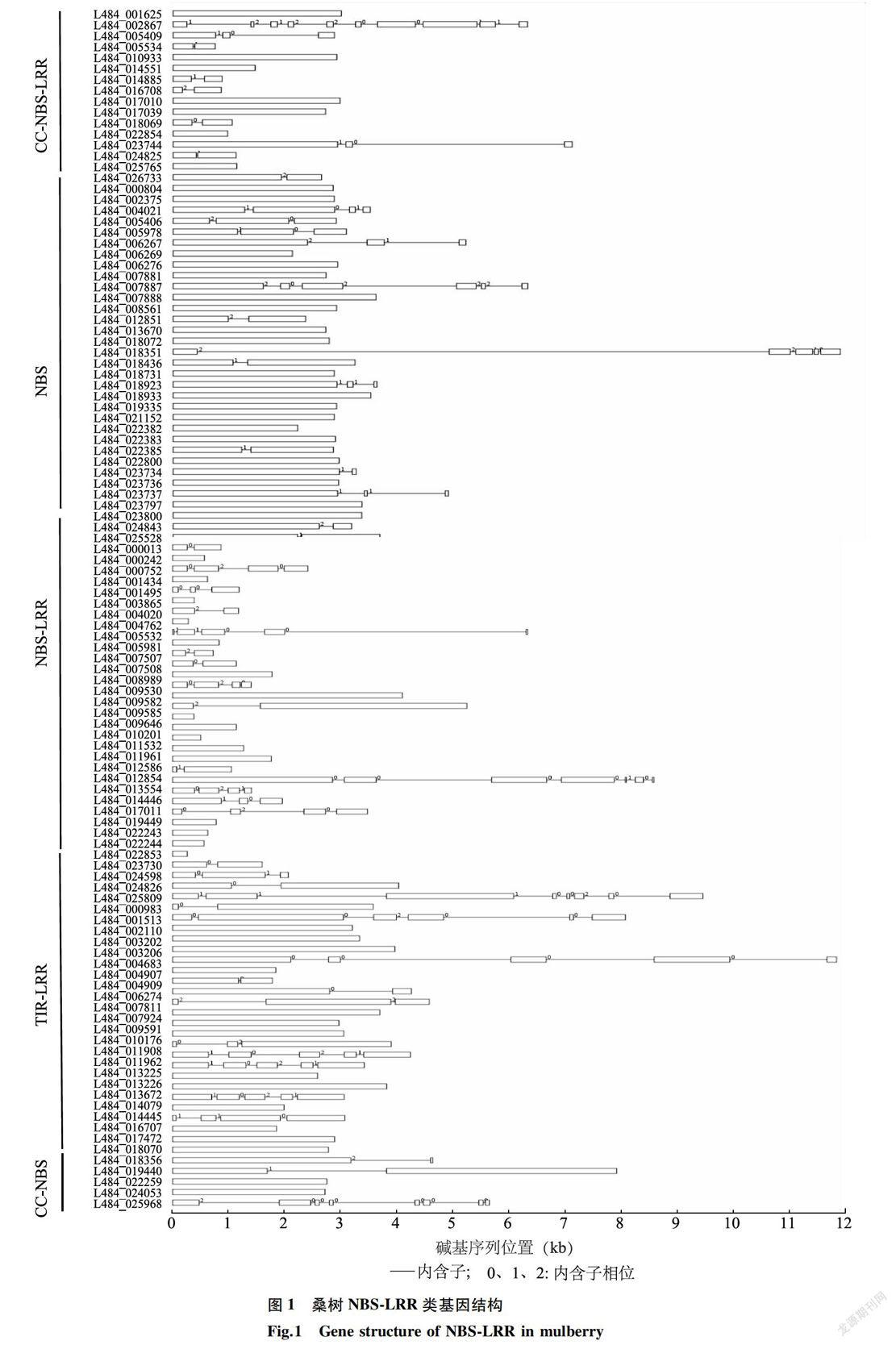
利用基因结构显示系统对桑树NBS-LRR类基因结构进行分析,发现该家族基因结构类型多样。大部分CC-NBS-LRR类型基因只含有1个外显子,编码序列长度一致性较高,而NBS类型基因内含子数和内含子相位类型较复杂,编码序列长度差异较大(图1)。
使用MEME在线软件对桑树NBS-LRR蛋白质基序进行分析(表2)。Motif 1位于NBS功能域N末端,也是AAA功能域(ATPases associated witha variety of cellular activities)的一部分,参与膜融合、蛋白质水解和DNA复制等多种细胞过程;Motif 2- Motif 5和Motif 10位于NBS功能域中间部分,也是AAA功能域的组成部分;Motif 7位于NBS功能域的C端,大部分桑树NBS-LRR蛋白均含有这些Motif,也有少部分桑树缺少该类Moti;Motif 6位于LRR功能域内,也是SCOP dlfqva2功能域的一部分,属于RNI-like超家族,由相似重复构成的规则结构,形成右手卢-a超螺旋的LRR功能域,NBS-LRR蛋白质中含有O至多个Motif 6,最多的IA84_025528蛋白质含有8个;Motif 8和Motif 9分别位于CC结构的N端和中部,在下游信号的激活中发挥作用,CC-NBS和CC-NBS-LRR类蛋白质含有至少1个该类Motif。
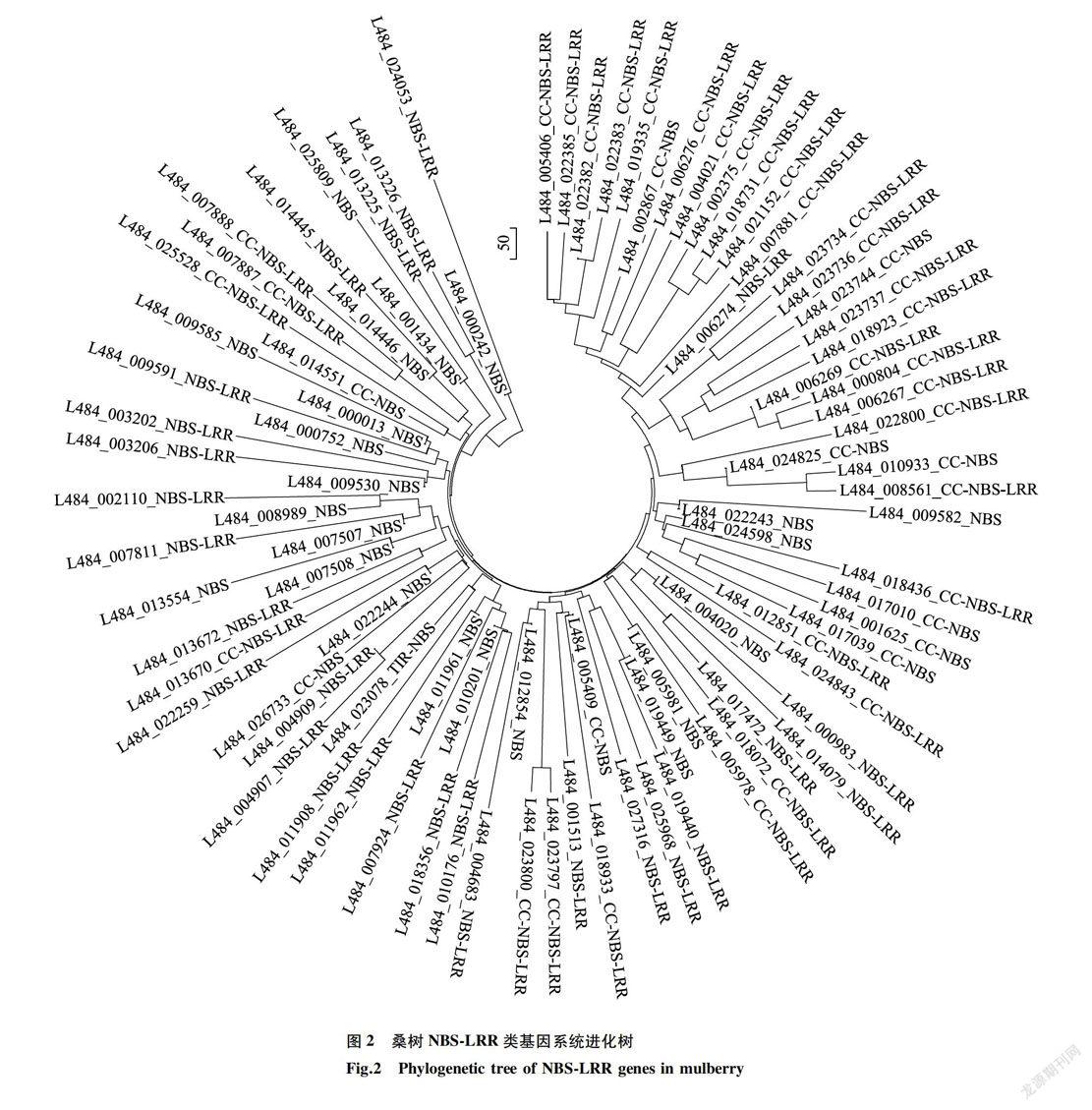
去掉部分相似度较低的序列,选择92个桑树NBS-LRR类基因构建NJ系统进化树,对基因进化关系进行分析。结果(图2)显示,桑树NBS-LRR类基因主要分为4个大的聚类组,4种主要NBS-LRR类基因聚类存在交叉现象,部分CC-NBS和NBS-LRR类基因聚类到CC-NBS-LRR组中,部分CC-NBS-LRR和NBS类基因聚到CC-NBS组中,部分NBS、CC-NBS-LRR和CC-NBS类基因聚到NBS-LRR组中,NBS组中含有多个其他3种基因类型成员。说明进化过程中不同类型的NBS-LRR类基因间可能存在遗传信息的交换或共进化现象。每个小聚类组中,NBS类型基因的进化枝长要短于同组其他类型基因,表明该组基因可能来源于该NBS类型基因。
2.3 桑树NBS-LRR类基因的组织表达
为了解桑树NBS-LRR类基因的组织表达特征,下载并分析了桑树基因组数据库中转录组数据。共检测到93个基因在不同组织中有表达(图3)。其中基因L484_013225和L484_ 014445在所有检测组织中均有较高表达,这2个基因均含有RPW8结构域,属于RPW8-NBS-LRR结构域类型,可能在桑树基础抗病中发挥主要作用。L484一022383、L484一013226、L484_017039、LA84_ 025528、LA84一018436、LA84_017010、LA84一010201等基因在树枝树皮、雄花和叶中表达量较高,而在根(除L484一022383和LA84_ 013226外)和冬芽中表达量稍低。基因L484一013672仅在根和树枝树皮中检测到较高表达。部分基因仅在特定组织中检测到较低表达。总体而言,较多的NBS-LRR类基因在树枝树皮、雄花和叶等组织中表达值较高,其次是根,而冬芽中多数NBS-LRR类基因的表达较低或未检测到表达。说明不同的NBS-LRR类基因在桑树的不同组织中发挥作用,部分基因存在组织表达特异性。
2.4 miRNA对NBS-LRR类基因的调控分析
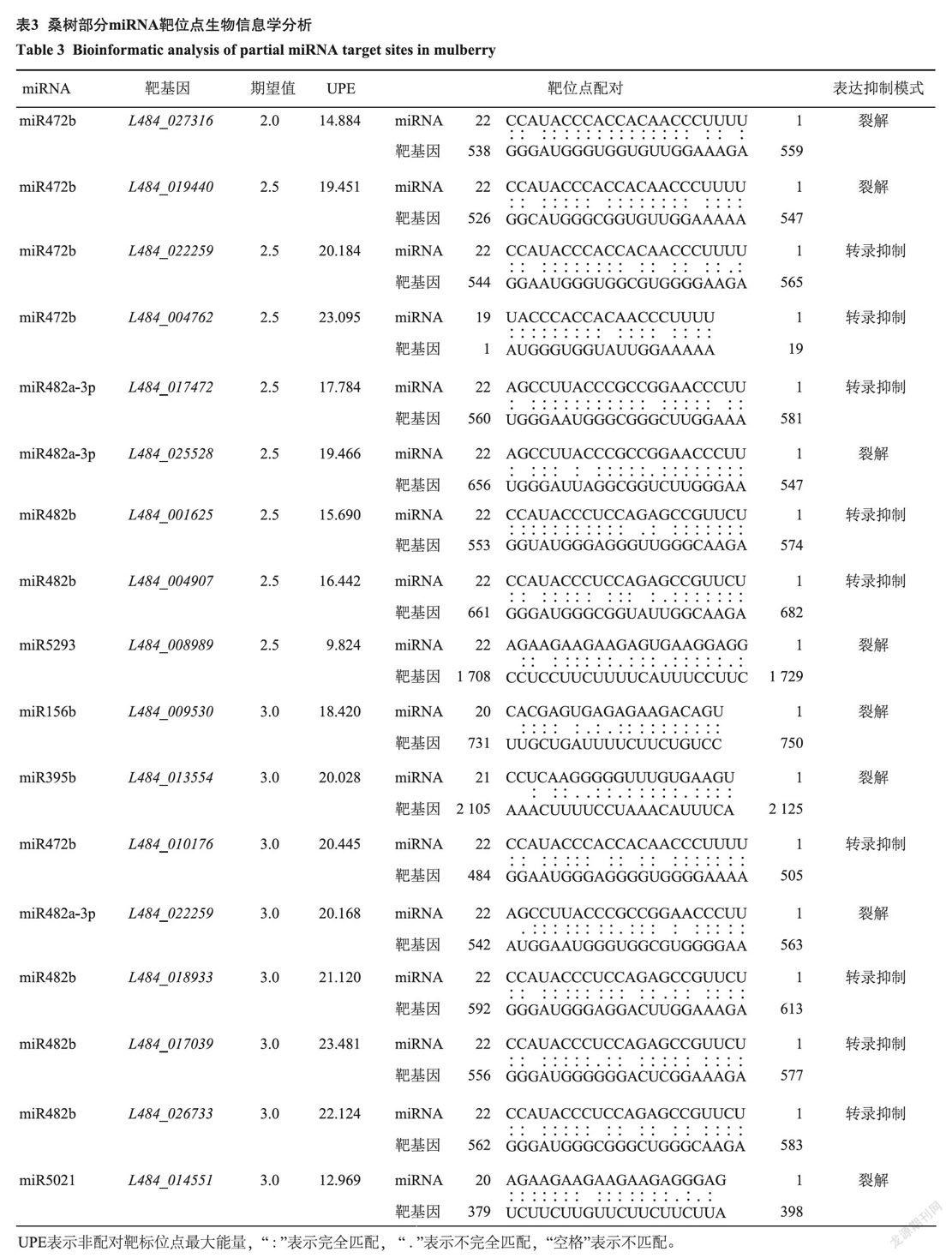
根据序列互补原则,利用psRNATarget在线软件对桑树miRNA与NBS-LRR类基因的调控关系进行序列比对分析,筛选到614个miRNA与NBS-LRR类基因存在调控关系,发现101个桑树miRNA可调控106个NBS-LRR类基因。较低的期望值表示miRNA与靶基因序列匹配较好。非配对靶标位点最大能量( UPE)为解开靶基因mRNA靶位点二级结构所需的能量,较低的UPE值表示miRNA结合或裂解靶基因的可能性较高。本研究选择期望值小于3的部分比对结果进行展示(表3)。miR472b和miR482b可对多个NBS-LRR类基因靶标位点进行结合,LA84一022259同时被miR472b和miR482a-3p
2个miRNA识别。miRNA对靶标NBS-LRR类基因的调控有转录抑制和序列裂解2种方式,这2种抑制基因表达的方式比例相当,但不同类型miRNA的调控方式不同,miR472b可通过转录抑制和裂解2种方式调控不同靶基因的表达,miR482主要通过转录抑制方式调控靶基因表达(表3)。
3 讨论
植物NBS-LRRS存在群体水平上的多态性,其在直系同源基因的序列组成和旁系同源基因的总体数量上都有差异,宿主中NBS-LRRS基因的数量与物种病原效应子的多态性有关[22]。自1994年第一个NBS-LRR类基因被成功克隆后[23],大量NBS-LRR类基因陆续被研究和报道,研究集中在NBS-LRR类基因的多样性及进化上[24]。本研究通过搜索桑树基因组数据库,共获得112个具有NBS核心结构域的桑树NBS-LRR类候选基因,其中63个同时具有NBS和LRR功能域。NBS结构域参与信号转导,具有高度保守和序列一致的基序,而LRR结构域参与病原体配体的识别,通常是高度可变的[25]。进化分析结果显示,桑树NBS-LRR类基因聚类存在类型间的交叉,成员较少的聚类组中基因类型一致性较高,而成员较多的聚类组中往往有多个类型的基因。聚类组中类型和结构一致的基因可能是由共同的NBS-LRR类祖先基因复制而来,而同一聚类组中不同类型的基因可能是由异位重组而来,异位重组导致该聚类组基因的扩张[24]。有些抗病基因需要2个NBS-LRR类基因共同介导,例如水稻中Pikml TS和Pikm2 TS共同介导抗病基因Pikm[26] ,RRS1和RPS4在十字花科植物多种细菌性病害抗性中共同发挥作用[27]。这可能与NBS-LRR类基因的适应性成本有关,也是该家族基因进化复杂性的体现。
多个NBS-LRR类基因被鉴定为广谱抗性基因,过表达NBS-LRR类基因,显著增强了植物的抗病性[28]。本研究中,NBS-LRR類基因的组织表达存在差异,基因L484一013225和IA84一014445在多个组织中均有较高表达,这2个基因编码的蛋白质均含有白粉病抗性蛋白功能域RPW8,可能在桑树的基础抗病中起作用,而L484一022383等基因在部分组织中表达较高,可能在桑树的特定组织器官或特定时期生长发育或抗病中发挥作用,也有些基因组织表达水平较低或未检测到表达,这些基因可能参与植物特定时期的抗病过程或者处于非激活状态。
miRNA作为调节子在NBS-LRR类基因的表达调控中发挥作用[24]。植物NBS-LRR类基因的高表达导致能量损耗而影响植物生长,植物进化出miR-NA-NBS-LRR系统来调控NBS-LRR的表达[12]。Lu等[29]首次报道了miRNA对NBS-LRR类基因的调控,随后大量miNRA与其靶NBS-LRR类基因的作用关系被明确[8]。番茄miR482通过靶NBS-IRR受体的P-loop,与168个抗性基因中的58个发生作用30]。在番茄、马铃薯和烟草中至少搜索到10个miRNA家族及其靶抗性基因之间的作用关系,说明miRNA调节抗性基因表达在植物基因组中普遍存在[31]。通过对桑树miRNA与NBS-LRR类基因调控关系的分析,发现大部分桑树NBS-LRR类基因均具有被miRNA调控的潜在可能,有17个miRNA与靶标序列匹配期望值小于3,其中miR472b和miR482b均有5个靶NBS-LRR预测结合位点,这些miRNA在桑树生长发育和抗病中的调控作用值得进一步深入研究。
参考文献
[1]JONES J D G, DANGL J L. The plant immune system[ J] . Nature,2006. 444 ( 7117 ) : 323-329.
[2]BOLLER T,HE S Y. Innate immunity in plants: an arms race be-tween pattern recognition receptors in plants and effectors in micro-bial pathogens [ J] . Science , 2009 , 324( 5928) : 742.
[3]SANABRIA N M, HUANG J C, DUBERY I A. Sejf/non-self per-ception in plants in innate immunity and defense [J]. Self/nonself, 2010, 1( 1) : 40-54.
[4]QI D , INNES R W. Recent advances in plant NLR structure , func-tion , localization , and signaling [J] . NLR-protein functions in im-munity, 2015 ,4 :348.
[5] BERNOUX M . VE T, WILLIAMS S, et al. Structural and functionalanalysis of a plant resistance protein TIR domain reveals interfacesfor self-association, signaling, and autoregulation [J] . Cell HostMicrobe. 2011, 9 : 200-211.
[6]MAEKAWA T, CHENC W. SPIRIDON L N, et al. Coiled-coil do-main-dependent homodimerization of intracellular barley immunereceptors defines a minimal functional module for triggering cejldeath[J] . Cejl Host Microbe , 2011, 9 : 187-199.
[7]VAN DER BIEZEN E A,JONES J D C. The NB-ARC domain: anovel signalling motif shared by plant resistance gene products andregulators of cell death in animals [J] . Current Biology, 1998, 8(7) : 226-228.
[8]FEI Q,ZHANG Y, XIA R, et al. Small RNAs add zing to the Zig-Zag-Zig model of plant defenses [J] . Molecular Plant-microbe In-teractions, 2016, 29(3) : 165-169.
[9]STOKES T L, KUNKEL B N, RICHARDS E J. Epigenetic variationin Arabidopsis disease resistance [J]. Genes & Development,2002, 16(2) : 171.
[10]HA M , KIM V N. Regulation of microRNA biogenesis [J] . NatureReviews Molecular Cejl Biology, 2014, 15( 8) : 509-524.
[11] MA W, CHEN C. LIU Y, et al. Coupling of micro RNA-directedphased small interfering RNA generation from long noncodinggenes with alternative splicing and alternative polyadenylation insmall RNA - mediated gene silencing [J]. New Phytologist,2018 , 217 (4) : 1535-1550.
[12] ZHANG Y.XIA R, KUANG H, et al. The diversification of plantNBS-LRR defense genes directs the evolution of microRNAs thattarget them[ J] . Molecular Biology and Evolution . 2016. 33 ( 10) :2692-2705.
[13] ZHAI J,JEONC D H.DE P E.et al. MicroRNAs as master regula-tors of the plant NB-LRR defense gene family via the production of phased, trans-acting siRNAs [J] . Cenes & Development, 2011,25( 23) : 2540-2553.
[14]KHALFALLAH Y. BOUKTILA D, HABACHI-HOUIMLI Y . et al.Regulation of NBS-LRR genes by microRNAs in wheat: Computa-tional identification of candidate MIR-2118 genes and evidence offlexibility [J] . Cereal Research Communications , 2017 , 45 (1) :1-10.
[15]HE N,ZHANC C , QI X, et al. Draft genome sequence of the mul-berry tree Morus notabilis [J] . Nature Communications, 2013,4: 2445.
[16] HUANC Y, ZOU Q, WANC Z B. Computational identification ofmiRNA genes and their targets in mulberry[ J] . Russian Journal ofPlant Physiology , 2014 . 61(4) : 537-542.
[17]BARANWAL V K.NEGI N .KHURANA P. Genome-wide identifi-cation and structural. functional and evolutionary analysis ofWRKY components of mulberry [J] . Scientific Reports, 2016,6: 30794.
[18]劉潮 .韩利红 ,宋培兵 ,等.桑树 WRKY转录因子的全基因组鉴定及生物信息学分析 [J] .南方农业学报 . 2017 . 48 (9) :1691-1699.
[19]GAI Y P, ZHAO H N, ZHAO Y N, et al. MiRNA-seq-basedprofiles of miRNAs in mulberry phloem sap provide insight into thepathogenic mechanisms of mulberry yellow dwarf disease [J] . Sci-entific Reports, 2018, 8( 1) : 812.
[20]WU P , HAN S.ZHAO W . et al. Genome-wide identification of abi-otic stress-regulated and novel microRNAs in mulberry leaf [J] .Plant Physiology & Biochemistry. 2015 , 95 : 75-82.
[21] JIA L.ZHANC D,QI X,et al. Identification of the conserved andnovel miRNAs in Mulberry by high-throughput sequencing [J] .PLoS ONE. 2014. 9( 8) : e104409.
[22] KUANC H, WOO S S, MEYERS B C. et al. Multiple geneticprocesses result in heterogeneous rates of evolution within the majorcluster disease resistance genes in lettuce [J] . Plant Cell , 2004 ,16(11) : 2870-2894.
[23]WHITHAM S.DINESH-KUMAR S P , CHOI D.et al. The productof the tobacco mosaic virus resistance gene N : similarity to toLl andthe interleukin-l receptor[ J] . Cell, 1994 , 78( 6) : 1101-1115.
[24]YANC X . WANC J. Cenome-wide analysis of NBS-LRR genes insorghum genome revealed several events contributing to NBS-LRRgene evolution in grass species [J] . Evolutionary Bioinformatics,2016, 12 : 36433.
[25]MACE E,TAI S.INNES D. et al. The plasticity of NBS resistancegenes in sorghum is driven by multiple evolutionary processes [J] .BMC Plant Biology, 2014, 14(1) : 253.
[26]ASHIKAWA I.HAYASHI N . YAMANE H , et al. Two adjacent nu-cleotide-binding site-leucine-rich repeat class genes are required toconfer Pikm-specific rice blast resistance [J] . Cenetics, 2008,180(4) : 2267-2276.
[27]NARUSAKA M.SHIRASU K.NOUTOSHI Y.et al. RRSI and RPS4provide a dual resistance-gene system against fungal and bacterialpathogens[ J] . The Plant Journal, 2009 , 60( 2) : 218-226.
[28] ZHANC C. CHEN H.CAI T, et al. Overexpression of a novelpeanut NBS-LRR gene AhRRS5 enhances disease resistance toRalstonia soLanacearum in tobacco [J]. Plant BiotechnologyJournal, 2017, 15(1) : 39-55.
[29]LU C, KULKARNI K. SOURET F F. et al. MicroRNAs and othersmall RNAs enriched in the Arabidopsis RNA-dependent RNA pol-ymerase-2 mutant [J]. Cenome Research. 2006. 16 (10) :1276-1288.
[30]SHIVAPRASAD P V.CHEN H M.PATEL K. et al. A microRNAsuperfamily regulates nucleotide binding site-leucine-rich repeatsand other mRNAs [J] . The Plant Cell. 2012 . 24( 3) : 859-874.
[31] 11 F, PIGNA'ITA D. BENDIX C. et al. MicroRNA regulation ofplant innate immune receptors [ J] . Proceedings of the National A-cademy of Sciences. 2012. 109(5) : 1790-1795.

