基于k-spaced氨基酸对构成的附子靶点预测
文/丛春雨
1 引言
近年来,随着越来越多的天然产物成功地通过认证而上市,中药 (traditional Chinese medicines,TCMs) 作为天然产物的重要组成部分,在现代药物研发中受到了越来越多的关注和重视。但中药药效物质基础不明确、作用机制不清楚,很难从整体到组织器官、细胞和分子水平进行全面系统地研究。因此,中药研究急需新的研究方法的出现。网络药理学的兴起对中医药是一个很好的契机,其所强调的整体性与系统性和中医药理论不谋而合。因此,在分子水平上,中药方剂用于治疗疾病的机制与网络药理学(network pharmacology)或多向药理学是相通的。由于中药品种繁多,成分复杂,若通过生物学实验手段确定各个化学成分的作用靶点,不仅耗时长,而且任务重,难以实现。相比之下,建立药物—靶标预测模型是一种快速高效的方法,能够减少成本,快速预测成分靶标,为中药网络药理学的应用奠定基础。
2 方法Method
2.1 数据集Dataset
从DrugBank数据库下载2388个美国FDA已经批准的小分子化学药的分子结构及其相应靶蛋白受体,根据对应关系,获得9275条药物—靶点配对,作为本研究的阳性样本集。将阳性样本集中的药物-靶点配对拆开,重新组合配对,剔除阳性样本,从中随机挑选出 2倍(18550条)于阳性样本集的配对结果,即为阴性样本集。从数据集中随机选取阳性样本1275条,阴性样本2550条,组成独立测试集,剩余的阳性样本和阴性样本组成训练数据集,训练数据24000条,测试数据3825条。
2.2 分子描述符和特征选择
本模型应用PowerMV(www.niss.org/PowerMV)计算药物分子,得到6122个描述符,用k-spaced氨基酸对构成(CKSAAP)编码每一个靶蛋白。在本模型中,我们采用k=0,1,2,3,4对应的CKSAAP编码,那么5-spaced特征向量的维数就是2205。基于以上两种编码方法组合的配对数据合计8327维。模型采用主成分分析法(principle component analysis,PCA)对上述数据进行降维处理,按保留原始信息97% 的信息量进行降维,获得数据矩阵分别为24000*995,3825*995。
2.3 实验结果及分析
在我们的方法中,使用支持向量机算法建立预测模型,预测模型中最优参数(c,g)分别是(362.039,0.00552427)。模型在训练数据集上的平均准确率可达79.74%,在独立测试集上准确率达82.41%。
2.4 附子作用靶点预测
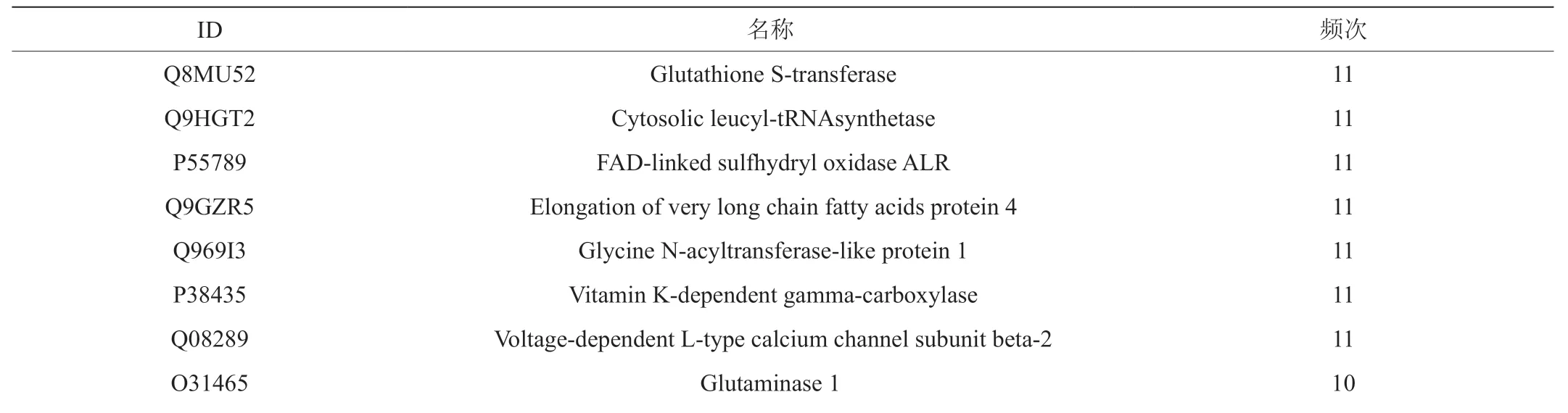
表1:与附子化学成分作用频次较高同时决策值也较高的55个潜在作用靶点

Q9GZZ6 Neuronal acetylcholine receptor subunit alpha-10 10 P49888 Estrogen sulfotransferase 10 P01579 Interferon gamma 10 P07237 Protein disulfide-isomerase 9 Q14643 Inositol 1,4,5-trisphosphate receptor type 1 9 P49411 Elongation factor Tu,mitochondrial 9 Q53707 MecA PBP2'' 9 Q05655 Protein kinase C delta type 9 Q14571 Inositol 1,4,5-trisphosphate receptor type 2 9 O00264 Membrane-associated progesterone receptor component 1 9 P12259 Coagulation factor V 9 O60909 Beta-1,4-galactosyltransferase 2 9 Q9BXA5 Succinate receptor 1 8 O00341 Excitatory amino acid transporter 5 8 O75879 Glutamyl-tRNA 8 Q9UBX3 Mitochondrial dicarboxylate carrier 8 P37059 Estradiol 17-beta-dehydrogenase 2 7 Q27218 Acetylcholine receptor subunit beta-type lev-1 7 P23743 Diacylglycerol kinase alpha 7 Q16760 Diacylglycerol kinase delta 7 Q8TC12 Retinol dehydrogenase 11 7 Q9HBH5 Retinol dehydrogenase 14 7 Q9NYR8 Retinol dehydrogenase 8 7 Q6NUM9 All-trans-retinol 13,14-reductase 7 O75911 Short-chain dehydrogenase/reductase 3 7 Q5JTZ9 Alanine--tRNA ligase,mitochondrial 7 Q6IB77 Glycine N-acyltransferase 7 P02461 Collagen alpha-1 7 Q99418 Cytohesin-2 6 P29475 Nitric oxide synthase,brain 6 Q81F54 Glyoxalase family protein 6 P08149 Penicillin-binding protein 2 6 Q9NR33 DNA polymerase epsilon subunit 4 6 Q86VB7 Scavenger receptor cysteine-rich type 1 protein M130 6 O43766 Lipoyl synthase,mitochondrial 6 Q9Y2J8 Protein-arginine deiminase type-2 6 Q9UQD0 Sodium channel protein type 8 subunit alpha 6 P08123 Collagen alpha-2 6 P98164 Low-density lipoprotein receptor-related protein 2 6 P20333 Tumor necrosis factor receptor superfamily member 1B 6 P50406 5-hydroxytryptamine receptor 6 6 P02775 Platelet basic protein 6 Q06830 Peroxiredoxin-1 5 Q12882 Dihydropyrimidine dehydrogenase [NADP 5 Q9NY56 Odorant-binding protein 2a 5 Q01118 Sodium channel protein type 7 subunit alpha 5 Q9UGM1 Neuronal acetylcholine receptor subunit alpha-9 5
从TCMSP数据库和附子相关文献中收集和整理已报道的附子所含化学成分,共计64个。模型基于python语言和rdkit包,通过计算分子的Morgan指纹,进行附子成分和drugbank数据库中美国FDA已经批准的小分子化学药的相似性比对,从而在64个成分中挑选出了24个与训练集中小分子药物化学空间(chemical space)相近的化合物,将其与drugbank中已知靶点进行组合,构建预测集。
预测集按训练集相应的方法计算分子描述符、归一化和降维等处理,然后带入模型进行靶点预测。本研究选取了与附子化学成分作用频次较高同时决策值(dec_value,decision value是该样本属于该类的隶属度,分值越大,代表属于该类的置信度越大)也较高的前205个潜在作用靶点,总频次1522次,但由于篇幅原因,表1只列出了其中部分潜在靶点。
3 结论
预测出的潜在靶点需要通过实验进一步验证,这是本文研究的意义所在。本项目组建立的预测模型有较高的准确率,该方法可望用于虚拟筛选中药有效成分及其作用靶点,为从分子水平上研究中药的作用机制提供一定的方法学参考。

