以细胞系为中心的基因-突变-疾病语义网络构建研究*
吴 萌 李 姣 侯 丽
(中国医学科学院医学信息研究所 北京 100020)
1 引言
语义网广义上来说是对未来网络的一个设想,狭义上来说是一种智能网络,不但能够理解词语和概念,而且还能够理解它们之间的逻辑关系[1]。随着语义网概念的提出,互联网逐步从仅包含网页之间超链接的文档万维网转变为描述各种实体之间关系的数据万维网。基于此,知识图谱这一概念于2012年5月由谷歌率先提出,其目标在于描述真实世界中存在的各种实体和概念,及实体与概念之间的关联关系,从而改善搜索结果。同时, 资源描述框架(Resource Description Framework, RDF)及其模式(Resource Description Framework Schema, RDFS)在语义Web中处于核心地位, 是实现Web信息共享和数据交换的基础。
伴随生物医学领域测序技术的飞速发展和精准医学概念的提出,越来越多的科学研究开始关注于疾病发生的内在复杂机制,以及各个生物医学实体之间的网络调控通路和关联关系,以提供个性化的治疗方案。知识图谱等语义网络技术为多源异构的生物医学数据的整合和复杂关系网络的建模提供新的解决方案,通过利用统一的数据表示标准,为生物医学数据的检索、分析、挖掘提供基础。在癌症生物学的研究过程中,人类癌症细胞系作为一种易于获取、方便使用的生物模型,广泛应用于探索癌症的分子特征以及相应的治疗反应。由于临床试验复杂且昂贵,而借助细胞系进行临床前实验有助于极大地提高临床实验的成功率。目前,许多项目都致力于为细胞系及其遗传学和基因组学数据提供系统的整合方案,例如癌症体细胞突变目录(Catalogue of Somatic Mutation In Cancer,COSMIC),使用户在进行生物实验和药物测试时可以选择更合适的癌症细胞系,也为生物研究提供临床依据[2]。本研究从NCBI gene、ClinVar、COSMIC、Cellosaurus、OMIM与NCIt 6个数据库中分别获取基因、突变、细胞系与疾病及其间的语义关系数据,拟以细胞系数据为核心,构建一个包含基因、突变与疾病数据及其语义关系的RDF语义网络。旨在对疾病基因组学等相关领域中的生物医学数据进行建模与整合,以期为进一步发现新的医学实体语义关系,理解与分析疾病的致病机制提供数据支撑。
2 研究现状
2.1 语义模型在医学数据领域的应用
2.1.1 生物医学语义模型 随着2015年美国总统奥巴马提出精准医学计划,世界多个国家陆续开始部署精准医学项目,更多的科学研究开始关注于疾病发生过程中内在的分子机制,而所催生的大量多来源异构的生物医学数据,迫切需要统一的数据整合方案。语义模型技术为多来源异构的生物医学数据整合提供方案,并致力于提供一套统一的生物医学实体表示标准,使机器和人都可以理解,其灵活性、可扩展性以及可对语义关系进行模型等特点非常适用于表示复杂的生物医学网路数据。如上海曙光医院构建的中医药知识图谱、医学系统命名法-临床术语(Systematized Nomenclature of Medicine-Clinical Terms,SNOMED-CT)和IBM Watson Health等系统[3]。基于链接数据,也可以识别出新的语义关系。如Dalleau等对药物、疾病和基因相关的6个数据库进行整合与链接,构建药物基因组学相关的RDF格式语义网络,共包含2 640 793个3元组。基于构建的链接数据,分别用两种基于图的机器学习的方法——随机森林和图核,对药物与基因是否相关进行预测,从而发现新的药物-疾病关系[4]。
2.1.2 细胞系相关语义模型 细胞系目前已在许多生物医学实验和研究中被广泛使用。复杂疾病,如癌症的发生通常开始于一系列体细胞DNA变化所导致的失控的细胞增殖,这些大部分变化指的是突变等特定的DNA序列变化。研究认为细胞从正常状态转变到完全的恶性形态的过程,必须积累5~10个体细胞突变,每一种突变都会引发不一样的细胞功能改变[5]。对癌症细胞系进行基因组测序,可以发现引发细胞机制发生变化的重要突变,整合并分析肿瘤发生过程中细胞系发生的突变信息,有利于更好地理解肿瘤发生的内在分子机制,从而发现新的治疗方案。而现有医学数据的语义模型中,整合细胞系、突变和疾病等相关实体及其语义关系的语义模型还非常少见。大多数模型只联系突变和疾病,或细胞系和疾病等实体。如细胞系本体(The Cell Line Ontology,CLO),主要描述细胞系、癌症、细胞和有机体之间的关联[6]。COSMIC数据库整合细胞系、突变和疾病等实体之间的关联,但是对于疾病的描述没有采用通用的表示方法,不利于与其他疾病数据库进行映射,其表示方式没有采用语义模型。
2.2 关系数据向语义数据的转化
随着下一代测序技术的发展,越来越多丰富的组学数据被生产、注释出来。其大多数存储形式为关系型数据库或类似的表格文件形式。而如何将关系型数据的数据资源和语义关系信息转化为RDF语义网络格式,成为一种普遍需求。许多项目如Bio2RDF[7],the EBI platform[8],PDBj[9]以及Linked Open Drug Data (LODD)[10]等都致力于推动健康科学数据转化为统一的链接数据形式。其中,Bio2RDF是一个开源的项目,采用语义网技术构建并提供生命科学数据的链接数据网络。Bio2RDF定义一套简单的规则,为多来源异构的数据集合创建RDF(S)兼容的链接数据形式。目前已为clinicaltrials.gov,dbSNP,GenAge等35个数据库提供RDF链接数据结构[7]。R2RML[11]是W3C RDB2RDF工作组于2012年9月发布的一种映射语言,可以定义关系型数据库与RDF格式数据之间的映射规则,从而将关系型数据转化为RDF 3元组形式。基于R2RML的常用工具如D2RQ、db2triples、OpenLink Virtuoso等都可以实现关系型数据库向RDF的转化。
由此可见,在生物医学领域,构建不同实体间的语义模型对于数据的整合、复杂网络的表示及发现新的语义关系等都具有突出作用。而目前的研究多集中在药物、疾病、症状等传统医学的关系层面,围绕基因、突变、细胞系等精准医学领域实体的语义网络尚不多见。上述关系数据转化技术中,对比发现D2RQ有明显的优势, 它支持任何关系数据库的数据转换、通用性强, 支持灵活的映射配置文件, 提供一种标准的转换方式生成一个虚拟的RDF (S), 确保数据库的内容更新便捷[12]。鉴于此,本文采用D2RQ工具将数据资源转化为RDF格式。
3 研究思路与框架
3.1 语义模型
基因、突变、细胞系与疾病之间存在多种语义关系。对语义关系进行有效以及规范的定义,是语义模型构建的基础,也为后续语义网络在文本挖掘等领域中的应用提供潜能。Verspoor等人提出一种人类变异组信息注释模式,对11种实体类型和关系进行规范。将这种模式应用于一个小型的以肠癌主题的全文预料库中,通过使用这个模式进行全文注释,注释结果的一致性得到显著提高[13]。本研究参考这种模式以及多种数据库对语义关系的命名方式,最终确定4种语义类型Gene、Mutation、Cell-line、Disease之间的6种语义关系的定义模式。根据收集的数据库中的数据形式与数据内容,选择部分基本信息进行提取,例如基因的类型、突变的位点和疾病的别名等。这样,每种语义类型都有多种相关属性进行更为全面的描述。具体语义关系模型设计,见图1。
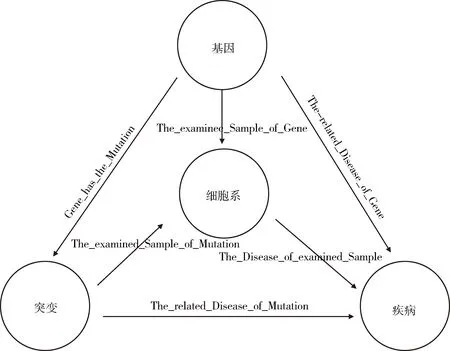
图1 语义关系模型及数据来源
3.2 实验流程
根据所设计的语义模型,选取NCBI gene、ClinVar、COSMIC、Cellosaurus、OMIM与NCIt等6个数据库作为基因、突变、细胞系与疾病等实体数据的来源。通过数据筛选、格式转化及融合等数据预处理流程,形成待处理数据集,将数据集存入MySQL数据库中,依据数据特性共存储为4个实体表,以及6个关系表。其后,利用D2RQ映射工具,根据本研究设计的语义模型,定义关系型数据与RDF数据的映射规则。最后,将关系型数据库转化为RDF语义格式并利用D2RQ工具部署本地Web应用,实现对语义网络的生成、检索与分析。实验流程,见图2。
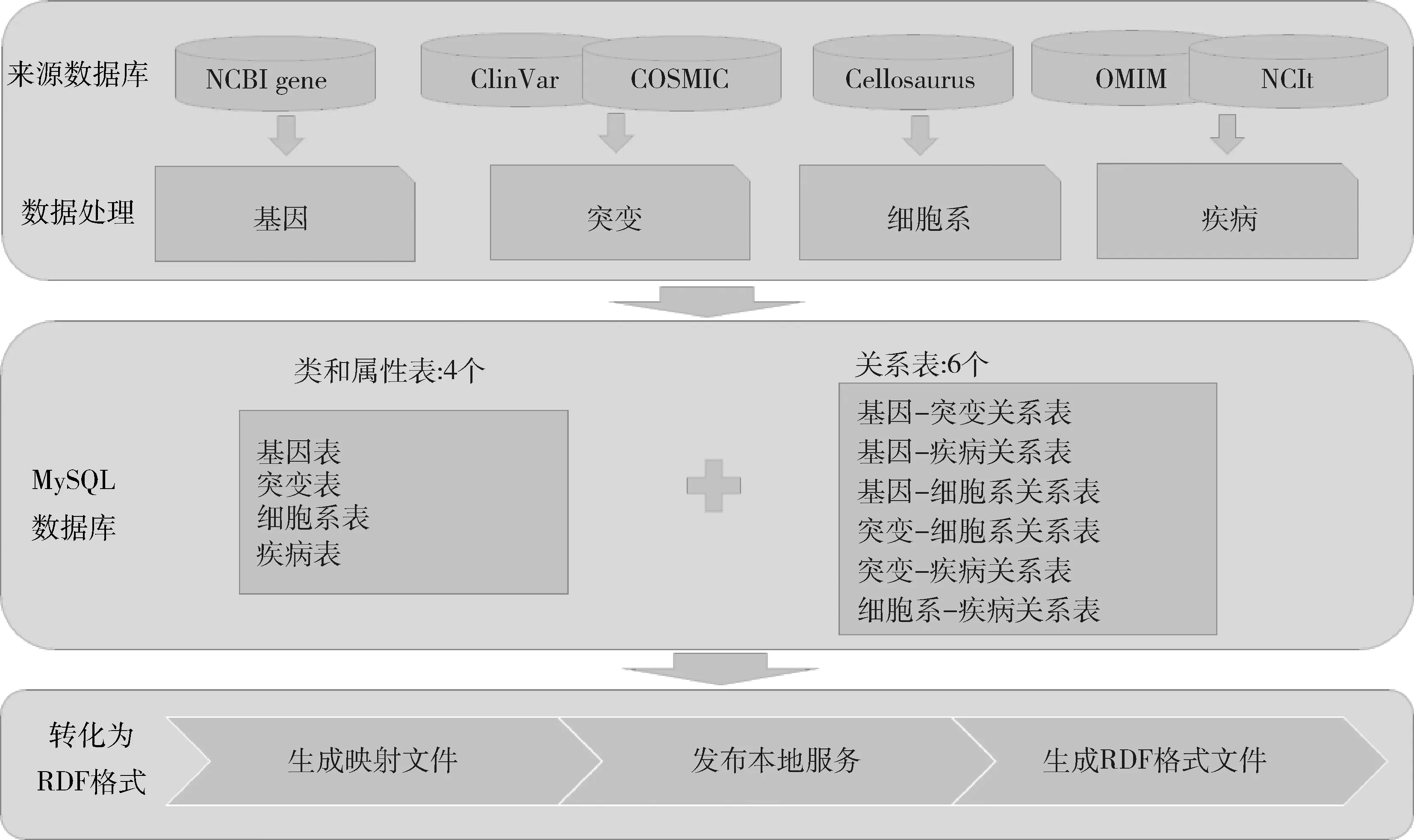
图2 语义关系模型的构建
4 实验过程与结果
4.1 数据处理
4.1.1 数据提取 (1)基因数据选自NCBI gene数据库。NCBI gene数据库提供了多物种的基因序列信息,包括序列、表达、结构、功能以及引用等信息,基因的唯一标识——Entrez_ID,在美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)研发的所有数据库中都是通用的[14]。本研究选择NCBI gene人类物种的基因数据作为实验数据的基因部分,共60 195条,包括基因ID、基因名称、在染色体上的位置和基因类型等信息。以NCBI gene数据库的Entrez_ID为基因数据的标准标识。(2)突变数据与突变-基因、突变-疾病之间的关系数据选自ClinVar数据库。ClinVar 是一个公开的数据库,其中收集与疾病相关的人类遗传变异[15]。本研究选择突变概要文件variant_summary.txt中基因组参考序列版本为GRCh38的突变数据,共254 030条,筛选所在基因、突变名称、突变类型、所在染色体、相关疾病等信息。ClinVar中突变所在的基因使用NCBI gene的Entrez_ID进行标识,可与NCBI gene进行链接。突变相关的疾病整合了OMIM 的疾病ID标识,所以也可与OMIM中的疾病链接。ClinVar的突变标识以RCV000000000.0.形式表示,在突变对应多个疾病的情况下,一个突变会对应多个突变标识所以本研究自定义突变的唯一标识,并保留RCVaccession的信息。(3)疾病数据与疾病-基因之间的关系数据选自OMIM数据库。在线人类孟德尔遗传数据库(0nline Mendelian Inheritance in Man,OMIM)是一个关于人类基因和表型的权威数据库,目前每日更新并支持免费获取[16]。本研究选择morbidmap.txt文件中7 326条疾病数据,保留疾病名称、基因名称、基因的OMIM ID以及基因的位置信息。利用mim2gene.txt中OMIM基因与NCBI基因的映射,获取疾病-基因之间的关系。由于有些疾病的OMIM ID缺失,所以自定义疾病的唯一标识。(4)细胞系的数据选自Cellosaurus数据库。Cellosaurus数据库由瑞士生物信息研究所的团队在Biocuration 2016大会上提出[17]。是目前整合细胞系信息较为全面的数据库。下载并处理68 406条细胞系数据为标准格式,筛选细胞系名称、编码、别名、相关疾病等信息,自定义细胞系的唯一标识。
4.1.2 数据关联 基于以上数据处理的步骤,可以得到大部分实验数据。但疾病-细胞系、突变-细胞系、基因-细胞系的关系是无法直接从已整合的数据库获取的,需要引入中间数据库,才能将这些实体进行关联。Cellosaurus中存在疾病-细胞系的关系,但是Cellosaurus中的疾病数据使用NCIt[18]的疾病术语进行表示,提供其在NCIt中ID编码。所以利用NCIt将Cellosaurus细胞系与OMIM疾病进行关联。参考一体化医学语言系统(Unified Medical Language System,UMLS)中已对NCIt的疾病术语和OMIM的疾病术语进行整合的信息。利用两个来源的术语在UMLS数据中是否在同一个概念下进行同义判断,将NCIt与OMIM的疾病术语进行映射。对于突变-细胞系与基因-细胞系的关系,利用COSMIC数据库作为中间数据库来获取这些信息。COSMIC是世界上最大最全的研究人类体细胞突变对癌症影响的数据资源[2],其中包含细胞系和原代细胞的基因测序信息和识别的突变信息。本研究重点关注对细胞系与突变和基因的关联信息的获取。获取突变数据与细胞系的关系,先将COSMIC的细胞系与Cellosaurus的细胞系的名称以及别名进行匹配,再根据突变在染色体中的位置,以及突变的类型,将COSMIC中的突变信息与ClinVar中的突变信息进行映射,以获得ClinVar突变与Cellosaurus细胞系之间的关联。而COSMIC中存在NCBI基因与细胞系关系,利用之前COSMIC的细胞系与Cellosaurus的细胞系的映射,可获得NCBI基因与Cellosaurus细胞系之间的关联。
4.2 数据映射
4.2.1 数据存储 本研究采用D2RQ工具将融合的数据资源转化为RDF格式。D2RQ是一个开源的平台,提供以虚拟只读的RDF数据形式访问关系数据库的功能。通过D2RQ,可以使用SPARQL语言对关系型数据进行检索,通过其自带Web应用浏览数据,也可以使用工具获取生成RDF格式的文件。根据D2RQ工具所需要的输入文件格式,将基因、突变、细胞系、疾病4种实体类型以及相关属性存为MySQL数据库中相应的4张表,6种实体间的关系存为数据库中相应的6张表。
4.2.2 映射规则 D2RQ提供映射语言来描述关系型数据库模式向RDFS转换的映射规则。一个D2RQ映射文件本身就是用Turtle语法编写的RDF文档。D2RQ提供一些便利的工具,例如generate-mapping工具可以自动生成一个映射文件mapping.ttl。但是自动生成的映射文件只包含一些基础的规则设置,更多复杂的映射规则可以参考D2RQ的映射语言[19]。
本研究首先通过声明一个数据库为d2rq:Database类来设置数据库的相关属性,包括JDBC数据库的URL,JDBC驱动程序类名,数据库用户名密码等。声明一个相应的实体为d2rq:ClassMap类来设置类的属性,包括URI的模式,例如设置gene的URI模式为“gene/@@gene.Gene_ID|urlify@@”,其中gene.Gene_ID为数据库中gene表的Gene_ID列。利用d2rq:PropertyBridge来设置属性,例如属性名称、属性值。实体之间的语义关系,利用其中的d2rq:refersToClassMap来定义,例如基因-突变之间的语义关系“Gene_has_the_Mutation”,将两个实体进行链接。
4.3 实验结果
4.3.1 实验结果浏览与获取 本研究根据基因、突变、细胞系、疾病4种实体类型以及之间的6种语义关系对应的表格,编写D2RQ映射语言文件mapping.ttl。利用生成的文件,运行d2r-server工具,启动D2RQ部署的本地服务http://localhost:2020/。通过这个Web应用,可以对数据进行浏览,也可以利用SPARQL执行搜索并设定展示的条目数量。以AKT3基因为例,数据浏览结果,见图3。基因AKT3的信息页面包含多种信息。其中,该基因相关的突变,用字段“Gene_has_the_mutation”表示;检测该基因的细胞系样本,用字段“The_examined_Sample_of_Gene”表示,其值对应的链接,可以链接到相应的突变和细胞系的信息页面。其他属性,例如,基因所在的染色体、基因全名和基因ID等信息也在该页面中详细列出。

图3 AKT3基因信息浏览页面
此外,D2RQ也提供通过命令行进行SPARQL语言搜索的功能。通过dump-rdf命令可以将生成的RDF文件导出生成“TURTLE”、“RDF/XML”、“RDF/XML-ABBREV”、“N3”以及 “N-TRIPLE”等多种RDF语法格式。实验最终构建的语义网络,共包含基因60 195个,突变254 030个,细胞系68 406个,疾病7 326个,共构建3元组726 236个。其中,基因与突变之间的3元组254 030个,基因与疾病之间的3元组15 477个,基因与细胞系之间的3元组287 342个,细胞系与突变之间的3元组195个,细胞系与疾病之间的3元组36 377个,突变与疾病之间的3元组132 815个。
4.3.2 实验结果对比分析 对NCBI Gene,ClinVAR,Cellosaurus,COSMIC等几个数据库进行分析,对数据类型和数据格式进行调研,与本研究模型进行对比,发现本研究构建的语义关系模型,其细胞系相关的数据类型覆盖程度更为全面,方便用户对细胞系及相关信息进行浏览和查询,而不用跨越多个数据库,缘于已有效地将细胞系相关的数据进行整合。每种数据类型都采用业内通用的数据库的名称和标识,提供良好的互操作性,为细胞系的研究提供帮助。相比于NCBI Gene,本研究构建的语义网络还整合了突变和细胞系的信息,较ClinVar多细胞系信息,比 Cellosaurus数据库的信息主要多基因和突变的数据,而在数据类型覆盖层面,COSMIC数据库同样覆盖了基因、突变、细胞系和疾病的信息,但同时本语义网络在疾病数据的整合方面提供疾病术语命名和编码的标准,见表1。当然该语义网络中的许多数据是从其中几个数据库中提取并整合的,因此整合更多细胞系相关的新数据、发现更多的语义关系也是本语义网络应该继续努力的方向。

表1 本研究模型数据类型覆盖范围与其他几个相关数据库对比
注:*即COSMIC没有提供表型标准名称以及与其他数据库的映射
本研究通过D2RQ部署的语义网络的Web应用,目前仅限于内部实验使用,后期将持续完善细胞系语义网络浏览平台,发布为外网可访问的形式并提供多种语义网络获取方式。
5 结语
本研究利用基于RDB到RDF映射语言(R2RML)的D2RQ映射工具,对以细胞系数据为中心的包含基因、突变与疾病数据以及语义关系的数据进行建模与整合,分析不同来源生物医学数据的特性,发现数据融合的有效方法,所构建的语义网络数据类型覆盖更为全面,可为用户提供更加便捷的服务。但是仍有许多不足之处有待改进,今后的工作将从以下内容展开:进一步优化多来源异构数据的融合方法,多方面考虑数据的不同特性,利用实体相似计算方法提高数据的映射成功率。研究突变对于癌症等复杂疾病带来的内部细胞机制的变化,丰富实体之间的语义关系。最终将数据来源扩展至文献、电子病历等形式,从中挖掘更多的生物医学实体之间的语义关系,从而对语义网络进行扩展,提高语义模型的实用性。

