中文疾病知识图谱构建初探*
吴思竹 修晓蕾 王安然 钱 庆
(中国医学科学院医学信息研究所 北京100020)
1 引言
知识图谱是结构化的语义知识库,用于以符号形式描述物理世界中的概念及其相互关系。其基本组成单位是“实体-关系-实体”三元组,以及实体和其相关属性-值对实体间通过关系相互关联构成网状的知识结构[1]。知识图谱是一种新型知识管理和服务模式,其活跃得益于Google的Knowledge Graph项目。后来,Google将Freebase并入 WikiData,转而基于 WikiData的API构建知识图谱,将细粒度语义信息资源关联起来,用于优化已有的搜索引擎和知识发现。随着Linking Open Data 等项目的全面展开,语义Web数据源的数量激增,大量关联数据被发布,大量开放关联数据如DrugBank、LinkedCT、DailyMed、DBpedia、Diseasome、RDF-TCM、RxNorm等被应用于知识图谱的构建。百度、搜狗、微软等搜索引擎公司也建立知心、知立方等知识图谱用以提升搜索质量。
虽然已有不少知识图谱被构建,但是在行业或专业领域知识图谱投入的研究力度并不足够,准确性和可用性也有待提高。在国外的医学领域,荷兰自由大学的黄智生教授构建乳腺癌知识图谱[2],MS Desarkar等[3]构建可对医疗应用程序进行知识图谱框架构建的Med-Tree,Travis Goodwin等人[4]提出一种基于电子病历的信念状态自动构建医学知识图谱的方法。在国内,开放医疗与健康联盟建立开放知识图谱平台[5],收集包括中国中医科学院根据UMLS构建的中药知识图谱、中医医案知识图谱、中医养生知识图谱、中医经方知识图谱,中国科学院自动化研究所构建的脑科学关联知识图谱等知识图谱资源。国内的知识图谱探索多是基于以前的知识库技术进行静态可视化实现,并没有充分利用现有知识组织体系进行实体数据组织、没有结合语义数据转换与开放关联数据资源或外部资源相互关联。因此,为更好地推动基于语义网技术的生物医学领域知识图谱研究,本研究探索以疾病为中心,包含疾病、药物、药企、医生、检查检验、手术操作和医院等7大类实体和实体之间关联的中文疾病知识图谱构建。进行可视化检索和浏览,帮助用户更方便地进行疾病识别、治疗药物和救治医院等多种关联的查找以及新的实体关联发现。
2 总体框架和思路
中文疾病知识图谱构建思路,见图1,首先从医学文献、医学健康网站、医学资源数据库和医学知识组织体系等多来源渠道应用多种方式采集抽取7大类数据,主要包括疾病、药物、检查检验、手术操作、医生、医院和药企,部分数据采集后需进行数据清洗、加工和审核,将数据存储在关系数据库中。构建7类数据之间的语义关联需进行数据对齐和整合,最终通过自建的IMI R2RML映射转换工具进行数据转换,基于R2RML标准将关系数据库中存储的数据转换为RDF数据,通过IMI R2RML映射转换工具进行基本数据检索。而后将RDF数据存储到Openlink Virtuoso数据库中,通过Relfinder实现图谱数据可视化和知识发现。整个架构相对简化,目标是初步探索实现中文疾病知识图谱的构建流程并发现构建过程中的主要问题,在取得一定成果和经验的基础上,进行更深一步的探索和实践。
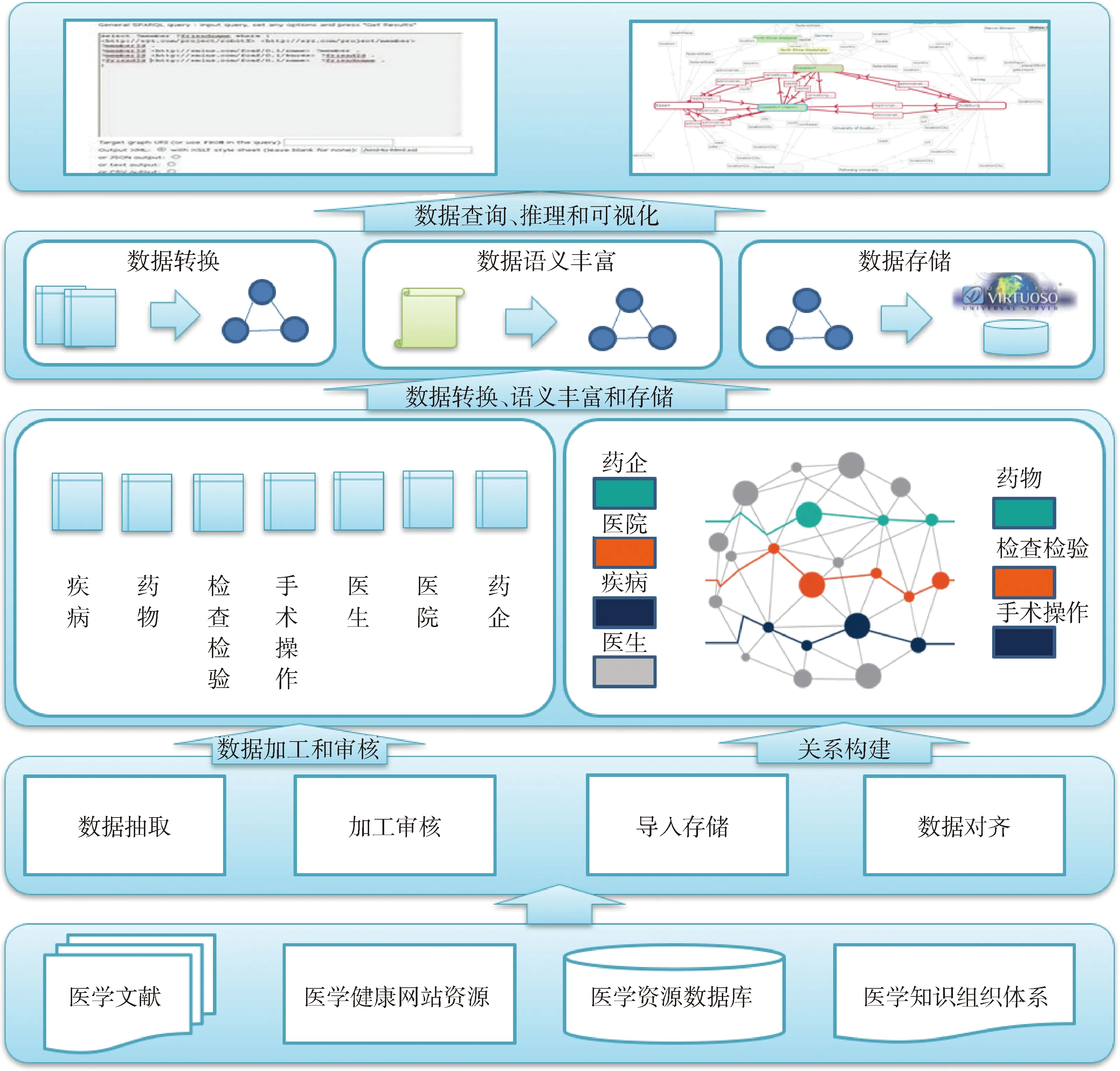
图1 中文疾病知识图谱构建框架
3 数据抽取和建模
3.1 数据抽取及处理
3.1.1 实体定义 本研究的主要目标是确定包括疾病在内的7大类实体,除疾病外,还包括药物、检查检验、手术操作、医生、医院和药企。其中,在药物实体下设立药物产品作为二级实体,由此构建以疾病为主的疾病知识图谱。7大类实体被定义为:(1)疾病。指机体在一定的条件下,受病因损害作用后,因自稳调节紊乱而发生的异常生命活动过程,并引发一系列代谢、功能、结构、空间、大小的变化,表现为症状、体征和行为的异常[6]。(2)药物。指用于预防、治疗、诊断人的疾病,有目的地调节人的生理机能并规定有适应症或者功能主治、用法和用量的物质等[7]。(3)检查检验。指为客观掌握人体的健康状态及发病原因、病情发展程度所使用的各种医疗检验手段。(4)手术操作。指医生用医疗器械对病人身体进行切除、缝合等治疗,以刀、剪、针等器械在人体局部进行操作,是外科的主要治疗方法。(5)医生。指掌握和运用医学领域专业知识、技能或技术手段治疗疾病的人。(6)医院。指以向人提供医疗护理服务为主要目的医疗机构。(7)药企。指生产药物和经营药品的企业,本文数据主要是指获得国家药监局批号的药物产品生产企业。
3.1.2 数据处理 在中文疾病知识图谱中,考虑到症状的情况比较复杂,本研究中未将症状单独列出作为一种实体,而是将其作为疾病的属性,在后续研究中将对其进行重点研究。相较于Google、Baidu等基于Wikipedia、百度百科等半结构化站点和英文数据构建知识图谱,本研究主要基于中文数据,分别从网络资源(健康网站、国家食品药品监督管理总局、临床医学知识库、医学百科等)、医学资源数据库、医学文献和书籍、医学知识组织体系等采集和抽取数据,采集整理后进行数据清洗、数据加工和质量控制。处理后的数据被导入并存储在MySQL数据库的不同实体数据表中,每类实体均包括不同的属性信息。(1)疾病属性。包括疾病名称、分类编码、概述、症状、病因、诊断、临床表现和治疗科室。(2)药物属性。药物名称、适应症、不良反应、禁忌症、注意事项、性状和用法与用量。(3)药物产品属性。商品名称和产品分类。(4)检查检验属性。检查名称、分类、概述、适应症和临床意义。(5)手术操作属性。手术操作名称、分类编码、基本术式和部位。(6)医生属性。医生名称、医生简介、所在科室和主治疾病。(7)医院属性。医院名称、医院等级、特色科室、电话、地址和邮编。(8)药企属性。药企名称、电话、网址、地址、地区和邮编。
3.2 数据建模
中文疾病知识图谱的数据建模采用自底向上的方式,根据图谱构建的目的和采集的数据确定图谱的模型,包括7大类实体,并确定实体对应的9类不同关系类型,见表1,每类实体包括3.1.2节中列出的对应属性。以疾病为主的实体之间的关系和相关属性建模,见图2。通过本体编辑器Protege创建该模型,后续图谱构建中依据此模型进行数据组织和关联构建。

表1 实体类型之间的关联关系类型
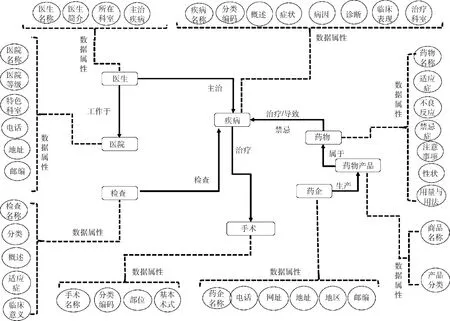
图2 以疾病为主的实体之间的关系和相关属性
3.3 关联构建
由于数据采集的来源不同,通过仔细分析每个实体数据和其属性,建立不同实体之间的关联。表1列出不同实体间存在的关联关系类型。不同实体之间的关联构建主要采用多种匹配算法并结合人工审核的方式。匹配算法主要采用3种方式:(1)精确匹配。通过不同实体表中共同的字段名称进行完整匹配。此外,还可以通过完整包含进行匹配,如疾病和药物间的关联,通过寻找精确匹配的疾病名称是否出现在药物属性的适应症的文字描述中匹配。(2)模糊匹配。由于中文名称拼写差异较大,如“西安交通大学医学院第一附属医院”和“西交大医学院第一附属医院”指向相同实体名称,但仅通过精确匹配并不能从数据中挖掘和建立两个实体间的关联。因而需要采用语义相似度算法(如余弦相似度)并通过人工审核和筛选确定实体关联。(3)基于别名扩展词表的匹配。使用疾病、药物规范名称和别名词表进行扩展构建药物、疾病和其他实体之间的关联,匹配后由人工审核并修正匹配结果。在本研究中,疾病和药物的名称和分类编码分别使用ICD-10和ICD-9-CM-3标准术语,一方面可用于规范实体名称和编码,另一方面可用于建立和外部资源的关联。由此建立表1中的多个实体间的关联关系,共构建9张实体对关系表。明确实体和关联关系后,根据构建的数据模型进行数据组织和映射。通过实例显示构建知识图谱模式层和实例数据之间是如何进行映射对应的,见图3。
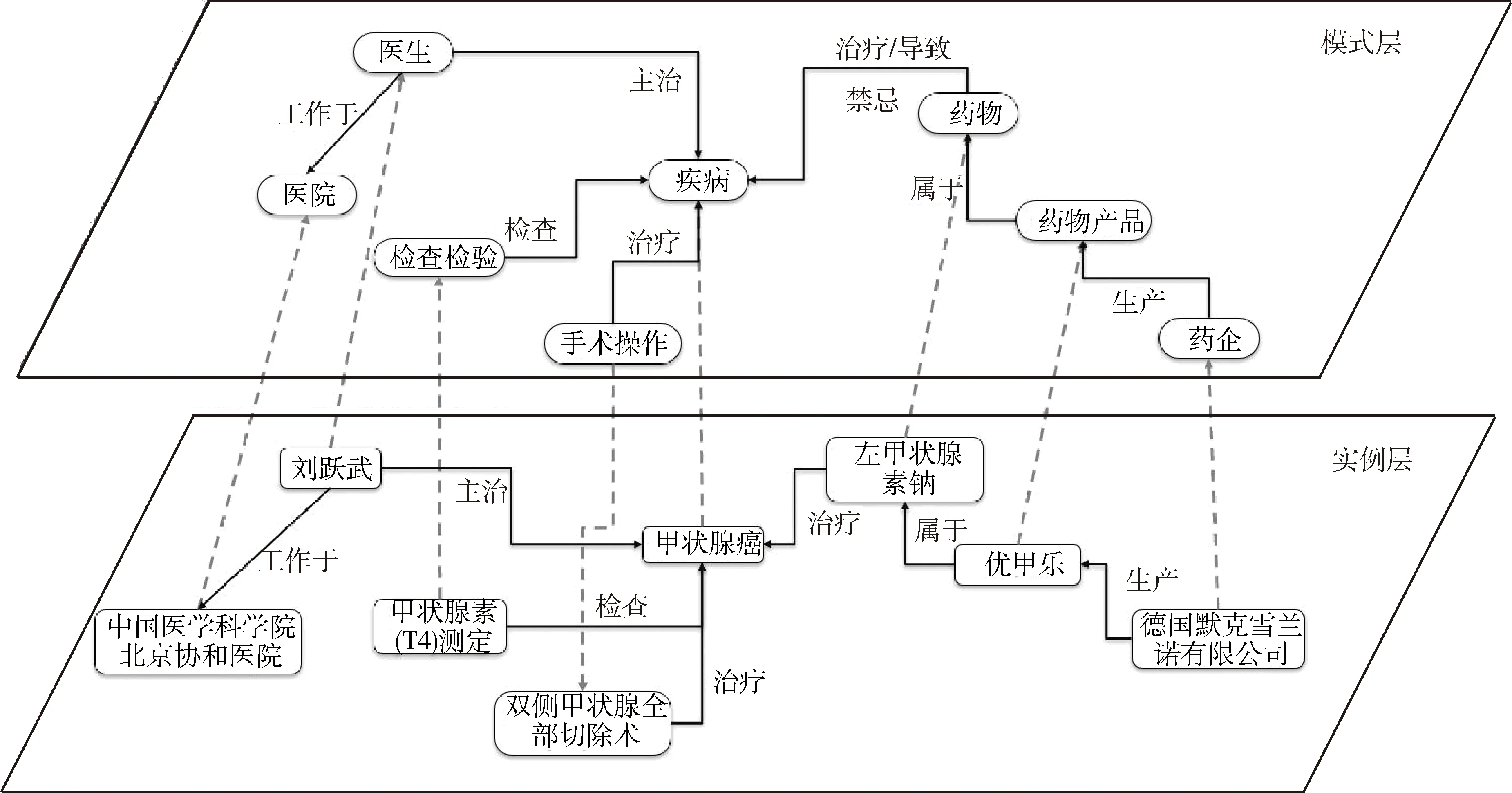
图3 模式层和实例层的映射
4 知识图谱生成与可视化
4.1 RDF数据转换
4.1.1 URI模式定义 本研究使用自建的IMI R2RML映射转换工具,基于W3C推荐的 R2RML映射标准实现关系数据到RDF数据的映射转换。首先需要为映射数据创建基础URI,URI是根据特定语法规范形成的字符串,为数据资源提供唯一标识。URI模式定义,见表2,每类实体都被分配一个URI,以疾病为例:“http://data.imicams.ac.cn/diseaseKG/disease/{疾病名称}”, 其中疾病名称为数据表中的列名,最终3元组被表示为“http://data.imicams.ac.cn/diseaseKG/disease /肺炎”。

表2 URI模式定义
4.1.2 RDF词汇 在本中文疾病知识图谱构建中,尽量使用已有RDF词汇描述实体类型和谓语关系,其提供的术语定义给数据类型和关系描述提供清晰的语义。表3列出本图谱构建中使用的一些主要RDF词汇。但是,由于一些谓语并没有找到适合的RDF描述词汇,因此采用自定义词汇进行描述。自定义RDF词汇的URI统一表示为“http://data.imicams.ac.cn/diseaseKG/vocab/{自定义描述词汇}”,描述词汇采用表1中列出的关系描述词。在数据映射规则撰写中,可以为“http://data.imicams.ac.cn/diseaseKG/vocab/”定义一个前缀dkgvocab用以简化书写,表示“治疗”的谓语就可以表达为“dkgvocab:treats”。
4.1.3 表间关系自定义映射 IMI R2RML映射转换工具提供3种映射模式,分别为直接映射、自定义映射和领域语义映射。本研究选择自定义映射模式进行实现,先进行实体表、关系表之间的数据映射定义,见图4。

表3 使用的已有RDF词汇

图4 映射工具中的表间关系自定义映射
建立各表的映射关系后,可在工具的映射核查视图中进行谓语和数据类型的RDF词汇定义。如为表示“属于的关系类型”自定义谓语“belongs_to”,治疗科室的谓词定义引入外部RDF词汇表VIVO的http://vivoweb.org/ontology/core#Department。完成映射核查和谓语定义后,IMI R2RML映射工具根据设定项自动生成R2RML映射规则文档。对于实体映射,如疾病实体映射,从疾病表中选择数据,创建一个TriplesMap,包括一个subjectMap和多个predicateObjectMap。subjectMap以疾病名称作为主语,定义类型为领域模型中的dkg:disease,将特定疾病归并到疾病类型下。predicateObjectMap通过rr:predicateMap定义谓语为http://ww.w3.org/2000/01/rdf-schema#label,以属性列值作为宾语,具体规则:
rr:logicalTable [rr:sqlQuery """select 疾病名称,治疗科室,分类编码,概述,临床表现,疾病治疗,病因,症状,诊断 from 疾病"""];
rr:subjectMap [rr:class dkg:disease;rr:template "http://data.imicams.ac.cn/diseaseKG/disease/{疾病名称}";];
rr:predicateObjectMap [rr:predicateMap [rr:constant
rr:objectMap [rr:column "疾病名称"; rr:datatype xsd:string];];
实体间的关系映射包括通过表的主、外键关系实现的映射,如药物实体和疾病实体的关联,可以通过药物_疾病_导致表的疾病名称和疾病表的疾病名称通过R2RML的rr:joinCondition语句关联,指定rr:child和rr:parent。映射规则:
rr:logicalTable [rr:sqlQuery """select 药物名称,疾病名称from 药物_疾病_导致"""];
rr:subjectMap [rr:template "http://data.imicams.ac.cn/diseaseKG/vocab/drug/{药物名称}";];
rr:predicateObjectMap [rr:predicateMap [rr:constant dkgvocab:causes;]];
rr:objectMap [rr:parentTriplesMap
rr:joinCondition [rr:child "疾病名称" ;rr:parent "疾病名称" ; ]; ];].
生成的R2RML可编辑修改,在最终确定映射规则后,在IMI R2RML映射转换工具执行数据转换按钮,共生成161 337个RDF 3元组,见图5,其中药物和手术操作创建和外部资源ICD-10和ICD-9-CM-3本体的关联,如“B超引导下盆腔穿刺术”,其分类编码可以直接链接到ICD-9-CM-3本体。

图5 RDF数据片段
4.2 数据存储
在IMI R2RML 映射转换工具中,转换后的数据直接存储为RDF文件,工具支持SPARQL语义检索和可视化功查询。但为了更好地进行知识图谱数据的数据查询和知识关联的发现,本研究进一步使用Openlink Virtuoso 进行RDF三元组数据的存储[8]。而后使用Relfinder进行可视化和知识发现[9-11]。
图6主要以高血压疾病为例展示构建的疾病知识图谱中的部分内容,其中显示高血压的相关检查检验方式、治疗的药物、生产该药物的药企、医院主治该疾病的医生等。高血压疾病可以采用甘油三酯(TG)测定、总胆固醇(TC)测定、高密度脂蛋白胆固醇(HDL-C)测定等方法进行检验。尼群地平、硝苯地平、替米沙坦等药物可以用于治疗高血压,这些药物均有相关产品。在高血压防治中,患有高血压疾病的患者需要谨慎使用一些药物,如甘草酸二铵、地诺前列素、醋酸泼尼松龙等。从图谱中还可以知道徐州医学院附属医院的陈浩,中日友好医院的许杼、王倩、高焱莎,中国医学科学院阜外心血管病医院的马文君、张慧敏等医生擅长治疗高血压疾病。Relfinder的可视化界面能够动态显示实体和关系,可以通过设置参数来展示两个实体间的不同路径长度的关系和发现新的关系。但Relfinder在可视化的过程中也存在一些不足,如将连接两实体之间的边表示为双向箭头,而实际为单向,在后续研究过程中将对该问题进行优化。
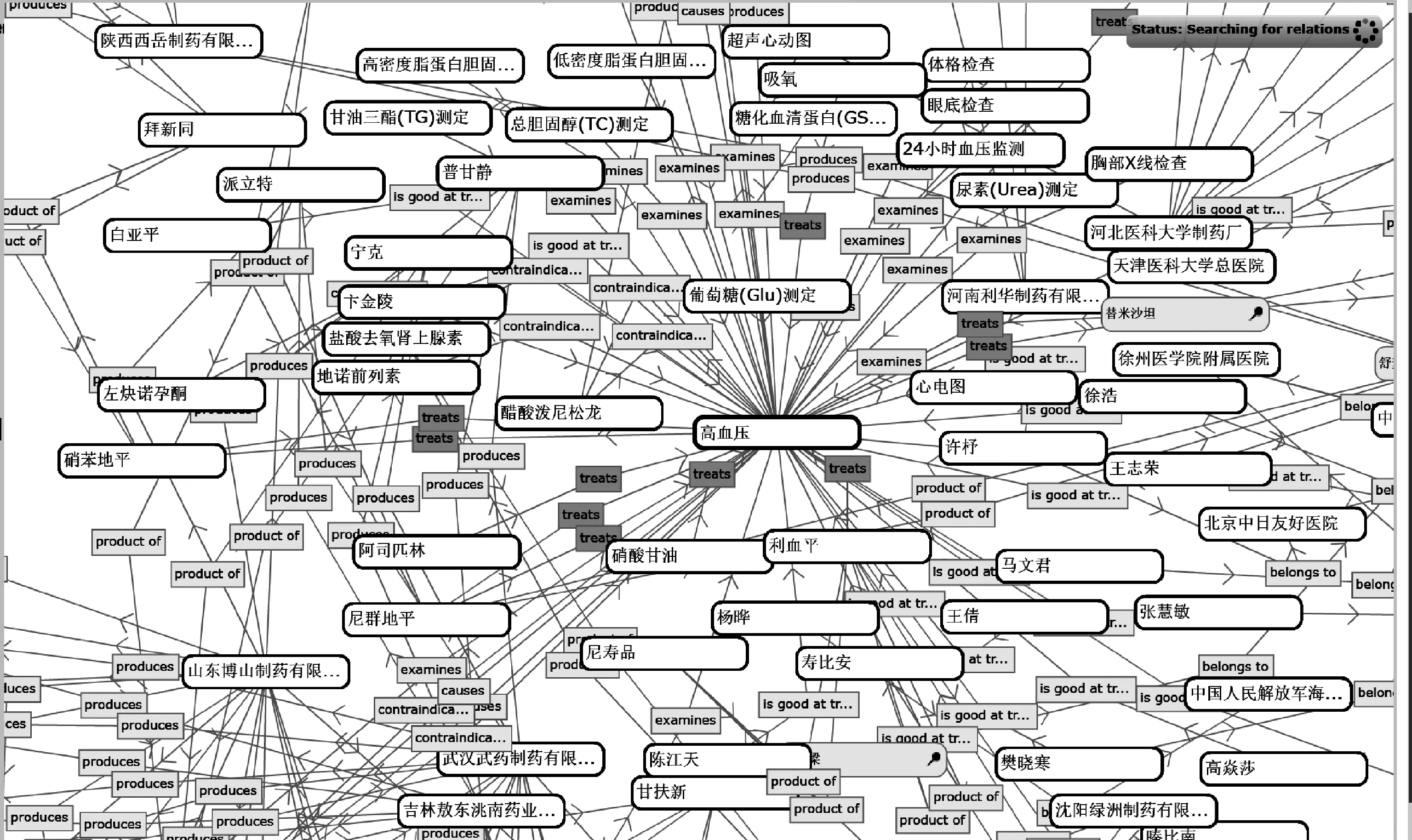
图6 中文疾病图谱的可视化浏览和关系发现
5 重点问题探讨
5.1 数据关联建立存在的问题
虽然本研究采用多种匹配方式进行实体数据关联构建,包括精确匹配、相似度计算和别名扩展词典,但由于疾病、药物等实体数据存在不同的命名方式,难以穷尽,导致生成的关联数量有限。各实体相关的中文同义词表也相对缺乏,影响中文实体名称规范和实体对齐的效果。因此,在数据对齐、数据关联构建方面还需进一步研究,积累多种实体名称和构建中文实体同义词表,加强数据名称规范,提升数据对齐的数量和质量。
5.2 RDF词汇复用与制动以词汇
中文疾病知识图谱构建中,生成的谓语可以使用已有本体或RDF词汇表中的词汇进行规范和丰富,本研究中使用部分RDF词汇予以描述,为便于理解使用自定义的谓语描述。但不足之处在于构建的是中文疾病知识图谱,而中文RDF词汇表相对缺乏,为保证一定的一致性,本研究采用英文RDF词汇作为谓语的表达。通用领域RDF词汇较容易获取,在本研究中使用的已有RDF词汇多来自FOAF、DC等。在面向领域能够复用和表达必要含义的英文RDF词汇中也很难找到应用较广泛且适合的词汇,因此,本研究采用自定义词汇(dkgvocab前缀的词汇)。虽然没有采用中文谓语描述,但是为更好地理解,在生成的图谱中将自定义RDF词汇也转换为中文描述并转换为三元组,用以必要时的机器语义理解和复用,如
6 结语
本研究基于R2RML标准针对中文疾病知识图谱建设的主要流程进行初步探索,取得预期效果。但是医学是一门复杂学科,在中文疾病知识图谱构建过程中,发现当前研究中的不足,包括各类数据实体的质量控制问题(实体消歧和对齐)、领域建模的完善和优化问题(领域模型结构相对简单,还可以扩展症状和科室实体,丰富每类实体类的结构层次)、实体关系和外部关联的尚待丰富等问题。下一步我们将进一步优化领域模型,更好地融合不同类型的实体数据和表示其之间的关联,进行更广泛的数据采集、优化数据质量、丰富实体和不同资源之间关联关系。后续将探索整体知识图谱构建自动化流程和服务平台的建设,建立数据在线浏览、SPARQL查询等一体化功能。对内容展示和可视化效果进行优化,改进IMI R2RML 映射转换工具对大数据查询和可视化的效果,优化Relfinder工具在显示关系上的指向缺陷。进一步探索中文疾病知识图谱数据扩展、更新、维护等问题,以便更好地实现语义数据管理和知识发现。

