双向聚类方法的文献计量分析*
杨 虎,易丹辉,张艳宏,吕晓颖,白文静**
(1.中央财经大学信息学院 北京 100081;2.中国人民大学统计学院 北京 100872;3.中国中医科学院中医临床基础医学研究所 北京 100700)
1 引言
近年来,随着信息技术在社会、经济、生活等各个领域不断渗透和推陈出新,拓展了人类创造和利用信息的范围。在新计算机技术的支撑下,人们获取信息、存储信息、处理信息等能力不断提高、成本大大降低,使得包括中医在内的医学领域积累了大量有待分析的临床数据、生化检测数据和互联网咨询健康数据[1-5]等。由于传统聚类方法多用于挖掘数据的全局结构信息,较少用于识别数据的局部结构信息方面,因而双向聚类方法被提出并用于挖掘数据的局部结构信息。
双向聚类(Biclustering)一词最早由Mirkin于1996年提出[6],而双向聚类方法的相关研究则最早可追溯到1972年[7]。双向聚类是一种能够同时对数据矩阵的行和列进行聚类的数据挖掘算法,也被称为块聚类(Block Clustering)[8]、协同聚类(Co-clustering)[9]、或二模态聚类(Two Model Clustering)[10]。根据双向聚类方法的不同原理,可以分为四类:基于传统聚类的双向聚类方法[11-13]、基于贪心迭代搜索算法的双向聚类方法[14,15]、基于穷举策略的双向聚类方法[16-18]和基于数学模型的双向聚类方法[19,20]。经过几十年的长足发展,双向聚类方法在健康领域数据分析中已经得到广泛应用。特别是在基因数据分析方面,双向聚类被用于实现重大疾病分类,同时识别疾病分类相关的基因标志物。虽然双向聚类方法的应用方兴未艾、不断演进,但目前在中医药领域数据分析方面的应用仍不多见。双向聚类方法作为一种无监督学习算法,能够在未事先确定样本分类的情况下挖掘数据的全局结构信息和局部结构信息。在中医药领中,双向聚类方法可以用来挖掘中医证候分类与核心处方、中医证候分类与症状体征表现等之间的全局或局部结构关系[21]。此外,随着基因检测技术的普及,双向聚类方法还可以用于挖掘中医证候分类、核心处方、症状体征与基因标志物之间的结构关系。
为拓展双向聚类方法在中医药领域中的应用,本文拟采用文献计量学分析来展示该方法的研究进展和前沿动态。文献计量学是通过互联网采集已发表文献的样本数据,并采用统计分析工具和方法评价样本数据的现状并预测其演化趋势的图书情报学的分支学科,已被广泛应用于信息科学[22]、再生医学[23]、神经科学与心理学[24]、战略管理[25]、循环经济[26]、世界经济[27]和高等教育[28]等领域。由于文献计量方法具有提取文献特征,进行深层次分析的优点,因此本文将借助该方法研究主题词“双向聚类”的文献,旨在掌握双向聚类方法相关研究的发展趋势、研究热点领域和未来的发展方向。最后,结合中医药领域数据的特点,探讨了双向聚类方法在中医药领域数据分析中的潜在应用价值。
2 双向聚类文献统计分析
本文采用CiteSpace文献计量分析软件作为双向聚类方法文献计量的可视化分析工具。CiteSpace软件是由美国德雷塞克大学信息科学与技术学院(College of Computing and Informatics,Drexel University)基于JAVA平台研发的文献计量可视化软件,它采用共引分析方法(Co-Citation)[29,30]和关键路径算法(path finder)[31,32]等,对特定领域文献进行计量分析,进而绘制出科学知识图谱,直观地展现科学知识领域的信息全景,识别某学科领域中的关键文献、热点研究和前沿的演进历程等[33]。
本文以Web of Science核心数据库(包括SCI和SSCI等)为数据源进行主题检索,检索主题为biclustering、block clustering、two-mode clustering或co-clustering等与双向聚类有关的文献,检索时间跨度在2008年至2017年之间,学科范围设置为所有学科,文献类型限定为期刊论文,语言类型设定为英语,检索时间为:2017年6月。
2.1 增长趋势分析
论文的发表量通常被认为是衡量学科发展水平和科技产出的一项重要指标,也是对科研成就与贡献的一种度量,发表量的变化趋势反映了学科知识量的变化趋势,从而判断研究领域的成熟度[34,35]。本研究通过近10年来双向聚类的文献发表量变化,反映双向聚类方法研究领域的发展现状(图1)。
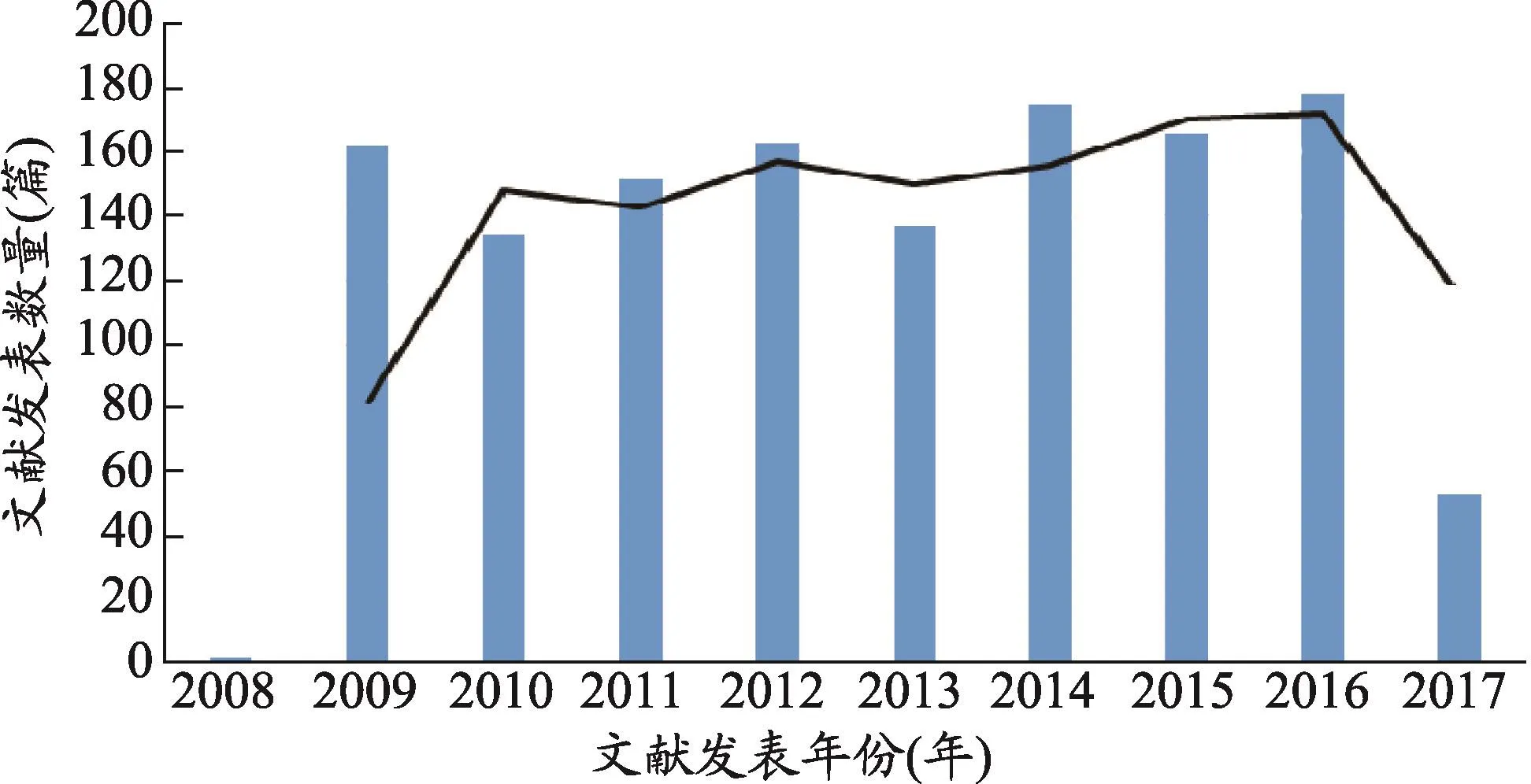
图1 双向聚类每年论文发表量
分析2008年至2017年间年论文发表量可以看出,双向聚类的相关论文在2009年后开始迅速增长,且近年来的文献发表量呈现小幅增长态势,每年的论文发表在140篇上下。双向聚类研究的论文发表数量相对稳定,波动不大,这说明双向聚类方法的科学知识已经积累到一个较为稳定的状态,该方法的研究已经趋于成熟(图1)。
2.2 主要国家与研究机构分析
在CiteSpace中,以“Country”和“Institution”为网络节点(Node Types),阙值选择(Selection Criteria)中选Top50(每个时间切片中出现或被引频次的前50项),网络裁剪(Pruning)中选择“Pathfinder”和“Pruning the merged network”,其他选项保持默认。运行Citespace软件,得到国家与研究机构合作的混合网络,如图2所示,其中有节点238个,连线722条,网络密度为0.010 9。
由图2可见,美国(USA)和中国(CHINA)作为最大的节点在国家/机构混合网络排名靠前。结合表1,从各个国家的论文发文数量上看,USA是文献贡献率最大的国家,其次是CHINA,分别发表论文316篇和288篇。其后依次是法国(110篇)、印度(87篇)、和日本(78篇)。研究机构中论文发表量最高的机构是Osaka prefecture university(大阪府立大学)和Chinese Acad Univ(中国科学院),论文发表量分别是28篇和24篇。
结合图2和表1可知,我国在双向聚类方面的研究水平处于国际领先的地位。本文采用中心度指标来评价我国学术论文的影响力,我国发表的双向聚类方面的论文中心度较低,仅为0.19,小于发文量相当的美国的中心度(0.28)。这说明我国在双向聚类方法的研究成果虽然不少,但多是是国内学者之间的合作,尚未与国外其他科研机构广泛开展合作。可见,在双向聚类方法的研究方面,我国还需扩大与国外科研机构的交流合作,进一步提升我国在双向聚类方法研究方面的学术影响力。
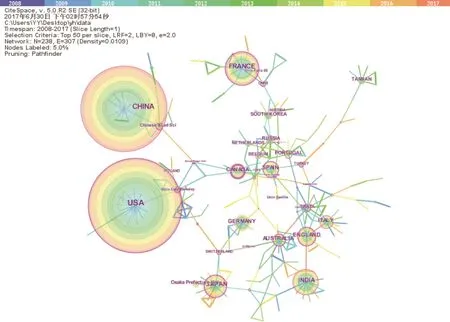
图2 国家/研究机构的混合网络图谱
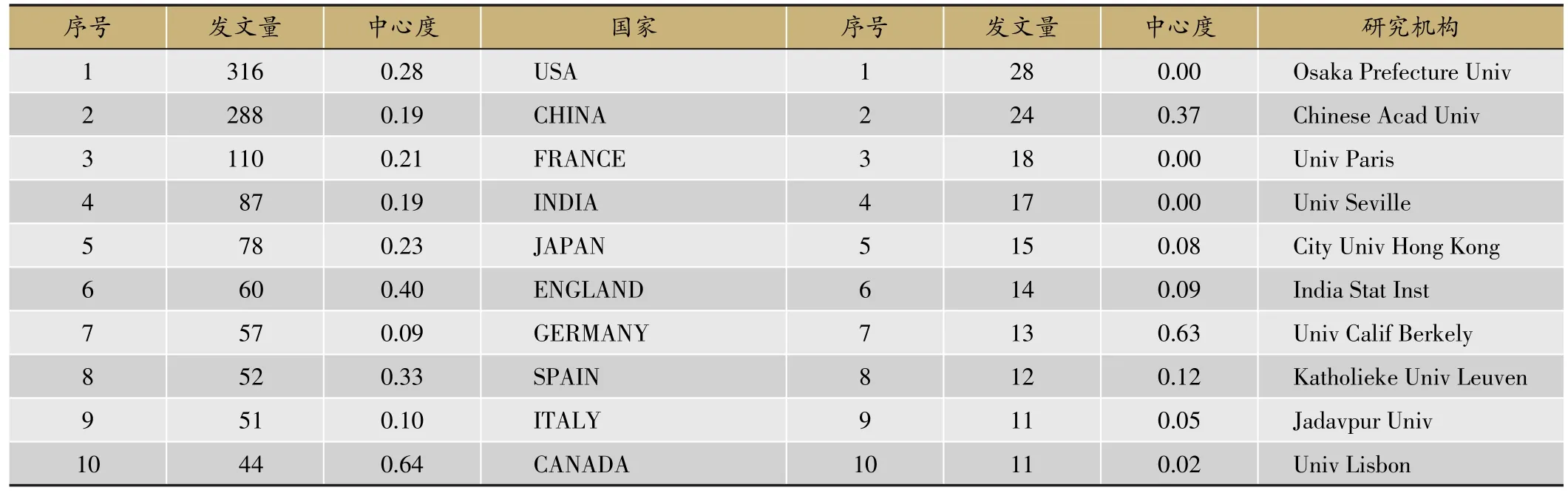
表1 国家/研究机构发表量Top10
3 双向聚类文献的共引分析
通过对双向聚类文献的统计分析可以发现双向聚类的发展趋势,从而判断该领域是否处于成熟阶段,不同国家/机构在双向聚类研究上的成熟程度;而共引分析则着重考察文献的贡献率。本研究采用citespace构造双向聚类相关研究的引文网络,进行引文聚类、识别网络中的重要节点及动态演化分析,目的是识别双向聚类的动态发展变化和热点问题。
3.1 关键节点文献分析
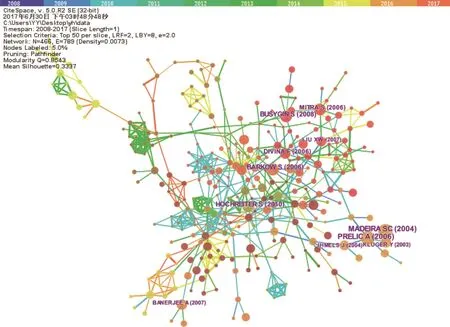
图3 共被引网络图谱
在文献共/被引网络中,不同类别之间通过某些具有“桥梁”作用的节点文献相连接。陈超美等将关键节点定义为图谱中连接两个及以上不同聚类且中心度和被引频次相对较高的节点。这些节点是共引网络中由一个文献发表时段向另一个文献发表时段过渡的关键点[29,30]。因此,我们对“双向聚类”领域研究进行关键节点文献分析,有利于识别该领域的核心研究学者和经典文献。本文将主题词来源设定为文献标题(Title)、摘要(Abstract)、作者关键词(Descriptors)、增补关键词(Keywords Plus),阙值选择(Selection Criteria)中选Top50,网络裁剪(Pruning)中选择“Pathfinder”和“Pruning the merged network”,其他选项保持默认设置,得到共被引网络图谱(图3)。图3中有466个节点,789条连线,网络密度为0.007 3。对该网络图谱中进行汇总,得到10个具有主导型地位和学术影响力的关键节点(文献的平均被引频次≥35)(表2)。
从共引网络中可以发现,有10篇影响力高的双向聚类的研究文献,研究内容的简要介绍如下:
葡萄牙贝拉英特拉大学Madeira教授2004年在IEEE/ACM Transactions on Computational Biology&Bioinformatics发表的论文《Biclustering algorithms for biological data analysis:a survey》是双向聚类方法的重要研究,该论文被引用157次[36]。文章从双向聚类在生物信息分析中应用的角度,讨论了双向聚类的发展演化过程、分类、评估方法及潜在应用场景。研究指出,双向聚类在信息提取和数据挖掘中已得到广泛应用,该方法的优势在于发现数据中的微观结构,能够更为准确的解释样本和特征之间的局部结构信息。
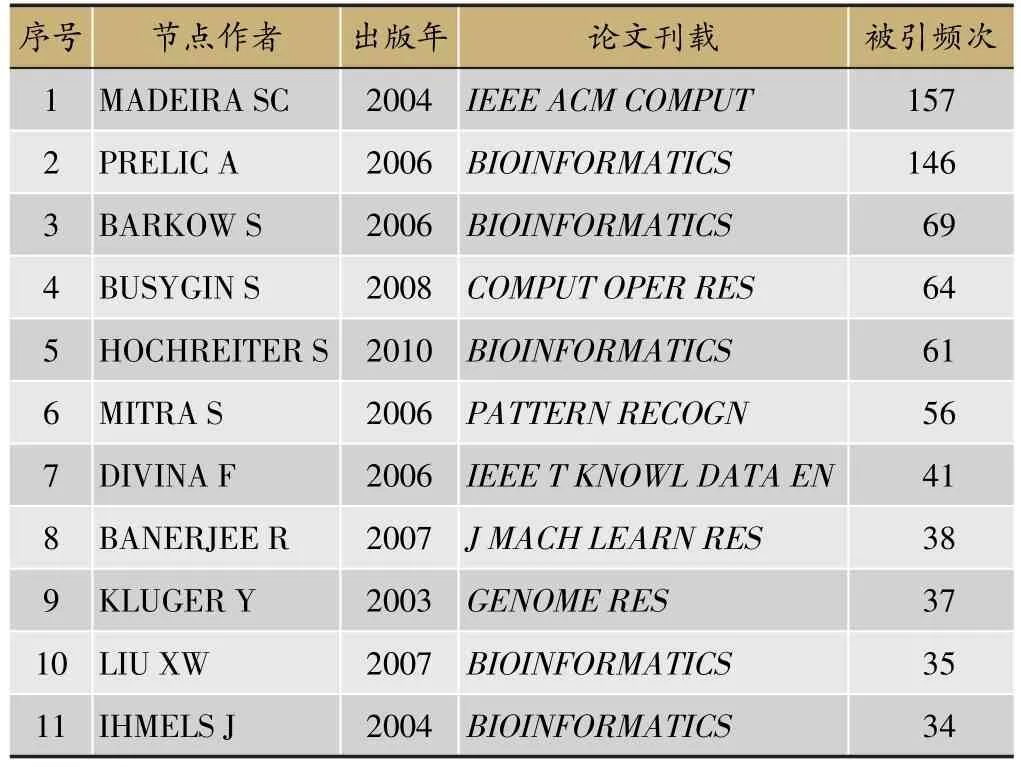
表2 共被引网络图谱关键节点信息
苏黎世联邦理工大学Prelic等2006年在Bioinformatics发表的论文《A systematic comparison and evaluation of biclustering methods for gene expression data》是第一篇对双向聚类有效性进行评价的文章[37]。该文章提出了一种对比和检验双向聚类算法有效性的方法,该方法的优点在于能够简单准确的判别最优的分组方法,对五种聚类方法的性能进行了比较。Barkow等2006年在Bioinformatics发表的论文《BicAT:a biclustering analysis toolbox》介绍了进行双向聚类的软件平台[38]。该平台是一个开源工具箱,集成了多种双向聚类方法和技术,并且提供了统一的图形化界面。此外,该平台还提供一些数据实例、检查和预处理的工具,能够用于实现基因数据的双向聚类分析。佛罗里达大学Busygin等2008年在Computers&Operations Research发表的论文《Biclustering in data mining》系统地介绍了双向聚类的算法[39]。该文献从双向聚类的定义、数学形式、可视化及实现双向聚类的算法的角度对双向聚类算法进行了综述研究,并指出双向聚类算法在生物医学、文本挖掘、协同过滤推荐算法和市场行为分析等方面具有较高的应用价值。
上述三篇论文是双向聚类的重要综述类文献,因此受到引用和关注的程度相对较高。其他重要的文献则集中在算法改进方面,不断提升算法的准确性。如:Hochreiter等2010年在Bioinformatics发表的论文《FABIA:factor analysis for bicluster acquisition》提出了一种新的生成模型,用于实现双向聚类[40]。该模型基于乘法模型,来计算基因表达与实验条件之间的线性依赖关系,并且捕获了真实世界转录组学数据所观察到的厚尾分布。Mitra等2006年在Pattern Recognition发表的论文《Multi-objective evolutionary biclustering of gene expression data》提出了一种多目标的双向聚类算法框架,优化了双向聚类的局部信息搜索过程[41]。Divina等2006年在IEEE Transactions on Knowledge&Data Engineering发表的论文《Biclustering of expression data with evolutionary computation》提出了基于进化计算来实现双向聚类[42]。Banerjee等2007年在Journal of machine Learning Research发表的论文《A generalized maximum entropy approach to bregman co-clustering and matrix approximation》提出了一种广义最大化信息熵的双向聚类计算框架,兼具一般性和灵活性[43]。2003年Kluger在Genome Research发表的论《Spectral biclustering of microarray data:coclustering genes and conditions》通过谱双向聚类发现基因表达数据矩阵中的独特“棋盘”模式[44]。Liu等2007年在Bioinformatics发表的论文《Computing the maximum similarity bi-clusters of gene expression data》优化了双向聚类算法的搜索过程,大大减少了双向聚类算法的运算时间[45]。Ihmels等2004年在Bioinformatics发表的论文《Defining transcription modules using large-scale gene expression data》引入签名算法,克服了聚类参数未知的问题[46]。
综上所述,双向聚类方法的应用场景主要是在基因数据分析方面。基因数据具有维度高、样本小的特点。利用传统聚类方法对基因数据进行聚类,仅能够从样本或者基因二选一地分析数据的全局结构信息,难以同时识别样本和基因的聚类、及数据的局部结构信息。利用双向聚类方法分析基因数据,则可以分析样本和特征之间的局部结构关系,挖掘具有相似基因表达的相似样本,实现精准的亚组分析。
3.2 研究热点的共现分析
研究热点是指在某一时间段内,有内在联系的、数量相对较多的一组论文所研究的科学问题或专题。我们对双向聚类领域研究热点的演变进行了分析,更好地把握该领域发展过程中的焦点内容[47]。在CiteSpace节点类型(Node Types)中选择“Keyword”,每两年为一个分割时间片段,在阙值设置(Selection Criteria)中选择“Top N per slice”,且设定为Top50,其他选项保持默认。运行CiteSpaceⅤ,绘制关键词共现网络,为了更好地展现双向聚类方法研究中的时间分布及相互关系,我们选择“Time Zone”时区网络视图,得到基于词频统计的各年份关键词分布,如图4所示,其中网络节点137个,连线173条,网络密度为0.018 6。在此基础上对全部关键词进行EM聚类,得到关键词的年度变化频次≥15的关键词(表3)。
从关键词的共现网络时区图和关键词频数表可以看出,与双向聚类方法有关的词语主要是数据挖掘领域的热点词汇,如pattern、model、classification、clustering等,其次是生物信息学数据分析有关的热点词汇,如gene expression data、microarray、gene expression、network。从关键词可以看出,双向聚类方法主要应用于分析基因表达数据、基因微阵列数据和基因调控网络数据等。这充分说明,双向聚类方法在基因数据分析中已经等到广泛的应用,这也符合基因数据高维的复杂结构特点。此外,通过关键词的词频统计分析发现,当前的研究前沿术语主要有:“data-analysis”、“fuzzy-clustering”、“room-temperature”、“data-set”、“real-world”、“pattern-mining”、“expression-patterns”。随着大数据技术,基因测序为代表的生物信息学技术的快速发展,机器学习、数据挖掘和大数据等方法也将用于生物信息学研究。
4 双向聚类在中医数据分析中的潜在应用价值及讨论

图4 关键词共现网络时区视图
通过对双向聚类方法的描述统计和文献共引分析,我们发现:双向聚类方法的研究已趋于稳定,发展相对较为成熟;该方法已被广泛应用于基因数据分析,在生物领域数据分析、信息检索和数据挖掘等方面得到应用。与生物信息学相比,中医学是以中医药理论与实践经验为主体,研究人类生命活动中健康与疾病间的转化规律及预防、诊断、治疗、康复和保健的综合性科学[48]。中医更关注疾病的证、治、效之间的关联关系[49]。由于中医辨证论治过程中,存在医师差异、患者个体差异性、疾病症状及临床表现不一、复杂干预手段等影响因素,要客观科学地刻画症、治、效间关系必然更需要采用双向聚类的分析方法。
结合中医药领域的研究的特点,双向聚类方法在中医数据分析中具有潜在应用价值。
识别具有类症状群的患者的人群特征、探索症状分类与患者特征之间的关系。中医证候反映疾病发生和演变过程中某阶段以及患者个体所处特定内、外环境本质,它以相应的症、舌、脉、形、色、神表现出来,能够不同程度地揭示病因、病位、病性、邪正盛衰、病势等病机内容。辨证论治地对患者进行分类治疗是中医的特色[50]。在实际数据采集中,由于患者的四诊信息、临床症状、体征等信息较多,收集的数据具有高维性,因此通过双向聚类方法分析患者症状体征数据,可以识别具有某些特征的患者子人群并判断其典型症状表现,即提取特属某类子人群的局部证候特征(图5),有助于根据疾病证候实现疾病分类。
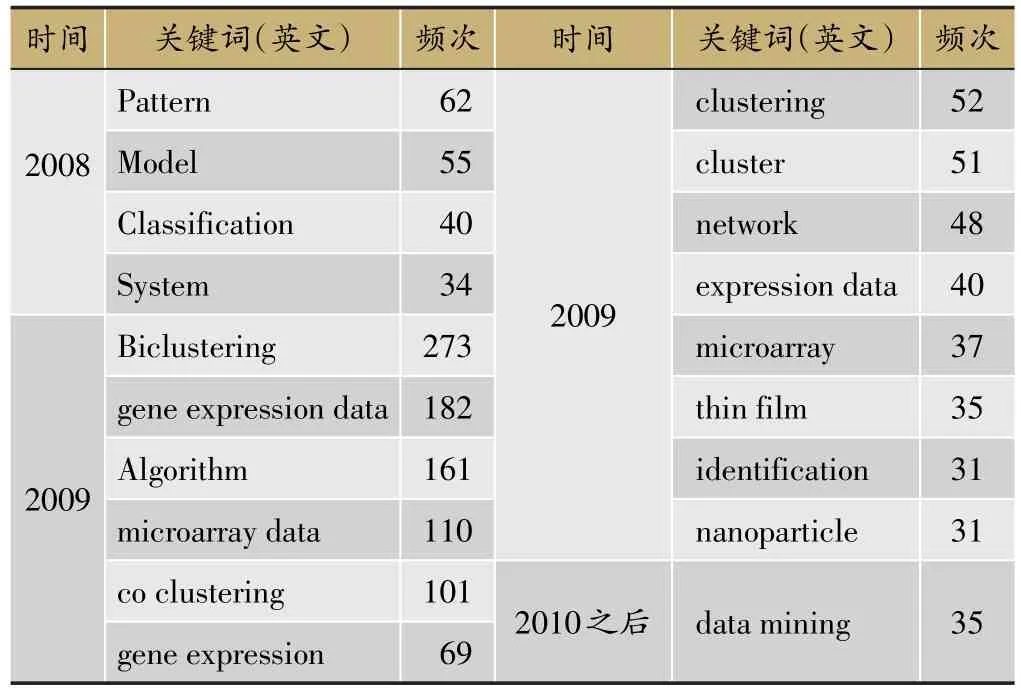
表3 关键词(频次≥30)列表
探索药物配伍特点,实现精细化治疗。根据患者的证候诊断与临床表现,医生会为不同的患者提供不同的中药处方。双向聚类方法能够同时对患者的证候和中药处方进行聚类,探索不同证候患者子人群的核心处方及用药差异性。这有助于探索药物配伍特点,实现对症下药,发现中医证候与核心处方之间的关系,可以探索名老中医的用药特点。对于针刺治疗亦是如此,可通过双向聚类探索穴位组合与治疗效果之间的联系。同理,也可以通过双向聚类来挖掘中医其他治疗手段与症状、患者子人群的联系。

图5 双向聚类的局部结构特征
探索量效关系,实现动态、科学合理用药。例如,在患者的某个或某些症状得到改善的情况下,通过双向聚类方法探索不同患者子人群的症状与中药组方中药物剂量关系或穴位手法组合方面的规律,科学合理的推荐用药方法和治疗指南;若患者的症状没有改善,也可以通过双向聚类方法分析该人群的治疗错位未起效的原因,进行治疗方案的实时、动态调整。此外,若可获取实验室检查等客观指标,则可以通过双向聚类算法探索患者子人群症状改善、药物配伍与其他客观指标变化之间的联系。
最后,随着基因测序等生物信息检测技术的普及,探索基因组学、蛋白组学等分子与中医症候、治疗方案等之间的关系将是双向聚类方法的重要潜在应用场景。对组学数据的深入分析能够有助于实现在分子层面重新定义健康状况、重塑临床实践[51],这对中医发展来说是一个潜在的发展机遇。

