早产相关基因的挖掘与特征分析
刘玄石,李巍
早产相关基因的挖掘与特征分析
刘玄石,李巍
国家儿童医学中心,首都医科大学附属北京儿童医院,遗传与出生缺陷防治中心;北京市儿科研究所,出生缺陷遗传学研究北京市重点实验室;儿科重大疾病研究教育部重点实验室,北京 100045
早产(preterm birth, PTB)指胎儿在完成37周妊娠前出生,是新生儿死亡的主要原因,与多种新生儿疾病和成年发生的慢性病相关。据双生子和家系研究报道,遗传因素约占早产风险的15%~35%,然而早产的分子流行病学机制目前尚不明确。本研究通过挖掘文献数据库和疾病数据库中与早产相关的文献,并结合两重过滤的方法,筛选出355个与早产相关基因。富集分析发现早产相关基因主要分子功能包括:受体配体活性、细胞因子受体结合、细胞因子活性和生长因子活性等;主要通路包括KEGG中富集的糖尿病并发症中的AGE-RAGE信号通路、Chagas病和IL-17信号通路和TNF信号通路等,以及Reactome中富集的多个与免疫相关的通路。早产相关基因与基因组其他基因相比较,转录本数量有差异(α = 0.1,= 0.06),但在GC含量和基因长度上没有明显差异。本研究结果提示早产基因大多集中在免疫相关通路,具备与免疫过程密切相关的分子功能,为早产的遗传机制研究提供了重要资源。
早产;数据挖掘;富集分析;基因特征;转录本数量
早产是指胎儿在完成37周妊娠前出生。2010年,世界卫生组织等国际组织对全世界184个国家的调查发现,新生儿的早产率大致是5%~ 18%[1],中国的早产率大约是7%,每年约有120万早产婴儿,全球排名第二,仅低于印度[2]。除死亡风险外,早产还可能伴有脑瘫、肺部疾病、听觉和视觉缺陷等风险[1,2],甚至有研究发现早产与成年后发生的一些慢性疾病相关,如心血管疾病和糖尿病等[3]。目前,早产的发生机制尚不明确。根据双生子及家系研究的估算,遗传因素对早产风险的影响大约占15%~35%[4~6]。早期对早产遗传机制的研究,通常根据早产病理学特点,选择可能相关的基因展开研究。例如,与新生儿出生体重和月经期有关的[7],参与炎症反应的、[8]和[9],与血管生成有关的[10,11]等。近年来,采用高通量测序技术对早产遗传因素的研究,发现了大量相关的位点和基因,包括采用全基因组关联分析找到的与自发早产相关的3个位点(rs17053026、rs17527054和rs3777722)[12],以及位于、和基因上的与早产相关的位点[13];利用全外显子测序发现与早产最显著相关的位点落在基因外显子上[14];全基因组、转录组和甲基化数据的结果提示和基因与早产相关[15]等。虽然针对早产遗传因素的研究已经积累了大量数据,然而由于早产的遗传机制相当复杂,现有研究结果也缺乏较好的归纳和整合,如Database for Preterm Birth (dbPTB)最后一次更新是2014年,这使得后续采用生物信息学手段对早产遗传信息的挖掘和早产遗传模型的构建变得困难[16]。因此,本研究利用生物信息学方法,通过挖掘文献数据库以及疾病基因数据库中报道的早产相关基因信息,整合并分析早产相关基因的特征,为早产的遗传研究提供重要资源。
1 材料与方法
1.1 数据库和软件
(1)文献数据库:美国国家医学图书馆(PubMed, https://www.ncbi.nlm.nih.gov/pubmed/);(2)疾病数据库:人类孟德尔遗传数据库(OMIM, https://www. omim.org/,下载时间:2019年1月18日)、人类基因组变异数据库(ClinVar, https://www.ncbi.nlm.nih. gov/clinvar/,下载时间:2019年2月11日)以及毒物基因组学数据库(CTD, http://ctdbase.org/,下载时间2019年2月6日);(3)基因特征数据通过Ensembl 数据库收集(http://grch37.ensembl.org/biomart/martview/ b3df3ce0609b9d96d3347ff1d09e4348,数据下载时间:2019年3月10日)。基因数据均统一使用人类参考基因组GRCh37/hg19;(4)统计应用软件R,版本号3.5.1。R包ClusterProfiler (版本3.10.1)用于富集分析[17];(5)网页版文本挖掘工具SciMiner (http:// hurlab.med.und.edu/SciMiner/,使用时间:2019年3月10日)[18]。
1.2 文献数据库的信息挖掘
2019年3月8日,通过计算机检索PubMed数据库,采用关键词检索式“preterm birth”AND“gene”,检索年限为建库至2019年3月。整理出所有文献的PMID,输入文本挖掘工具SciMiner。SciMiner软件通过关键字“preterm birth”,以及软件内置的正则表达规则和基因字典,挖掘文献中与早产相关基因。为避免过度匹配,对SciMiner挖掘结果设置阈值和人工审核的两层过滤方式。首先根据设置的阙值,删除了仅在2篇及以下文献中出现的基因。其次通过人工核查摘要,删除摘要中没有直接提及早产的基因。最后筛选出用于后续分析的基因列表。
1.3 疾病数据库的信息挖掘
通过Shell脚本程序,搜索疾病数据库OMIM,ClinVar和CTD,查找与“preterm birth”或其同义词匹配的记录,提取记录下的基因信息,并合并进文献数据库筛选出的基因列表。
1.4 基因富集分析
采用R软件包ClusterProfiler对筛选出的基因,进行了基因功能(Gene Ontology, GO)和KEGG通路(京都基因与基因组大百科全书数据库,Kyoto Encyclopedia of Genes and Genomes)以及Reactome通路[19]的富集分析,对结果进行多重检验后,获得显著的功能和通路,以FDR<0.05 (false discovery rate)作为显著性的阈值。
1.5 基因特征的收集
采用Ensembl的BioMart,收集了20320个基因的长度,转录本数量,GC含量特征(人基因组版本GRCh37.p13/hg19)。根据筛选出的基因列表,采用Shell脚本程序,从BioMart数据中提取了所需基因的特征信息。
2 结果与分析
2.1 早产相关基因数据库挖掘结果
通过计算机检索PubMed数据库获得来源于800种杂志的2264篇相关文献的摘要,并通过PMID和SciMiner软件挖掘出了文献中与早产可能相关的2149个基因。其中,文献数量居前5%的杂志多数是临床专业期刊(附表1)。经过阈值和人工审核的两层过滤,筛选出在1274篇文献里出现的355个基因(附表2),表1列出了在文献数量中排名前5%的基因。
通过对疾病数据库OMIM、ClinVar和CTD的挖掘,找到1个早产相关基因()。由于该基因已存在于上述355个基因中,因此最终用于分析的基因数目不变。
GO富集分析发现174种显著的生物学功能(FDR<0.05)。根据显著性由高到低排列,前10种生物学功能包括:受体配体活性(receptor ligand activity)、细胞因子受体结合(cytokine receptor binding)、细胞因子活性(cytokine activity)、生长因子活性(growth factor activity)、生长因子结合(growth factor binding)、蛋白酶结合(protease binding)、血红素结合(heme binding)、生长因子受体结合(growth factor receptor binding)、四吡咯结合(tetrapyrrole binding)和脂多糖结合(lipopolysaccharide binding) (图1,附表3)。其中具有受体配体活性功能的基因数量最多,共有61个。
KEGG富集分析发现的显著信号通路达到158个(FDR<0.05)。前10条通路根据显著性由高到低排列分别是:糖尿病并发症中的AGE-RAGE信号通路(AGE-RAGE signaling pathway in diabetic complications),Chagas病(美洲锥虫病),IL-17信号通路(IL-17 signaling pathway),TNF信号通路(TNF signaling pathway),PI3K-Akt信号通路(PI3K-Akt signaling pathway),Toll样受体信号通路(Toll-like receptor signaling pathway),结核(tuberculosis),炎症性肠病(inflammatory bowel disease (IBD)),乙型肝炎(hepatitis B)和流体剪切力和动脉粥样硬化(fluid shear stress and atherosclerosis) (图2,附表4)。
Reactome通路富集分析中前10个显著通路分别是:白细胞介素信号(Signaling by Interleukins),白细胞介素4和白细胞介素-13信号传导(Interleukin-4 and Interleukin-13 signaling),白细胞介素10信号传导(Interleukin-10 signaling),Toll样受体级联(Toll-like Receptor Cascades),Toll样受体4 (TLR4)级联(Toll Like Receptor 4 (TLR4) Cascade),Toll样受体TLR1:TLR2级联(Toll Like Receptor TLR1: TLR2 Cascade),Toll样受体2 (TLR2)级联(Toll Like Receptor 2 (TLR2) Cascade),免疫系统疾病(Diseases of Immune System),与TLR信号级联相关疾病(Diseases associated with the TLR signaling cascade),质膜上启动的MyD88:MAL (TIRAP)级联(MyD88:MAL (TIRAP) cascade initiated on plasma membrane) (图3,附表5)。

表1 筛选出的基因列表中排前5%的早产相关基因
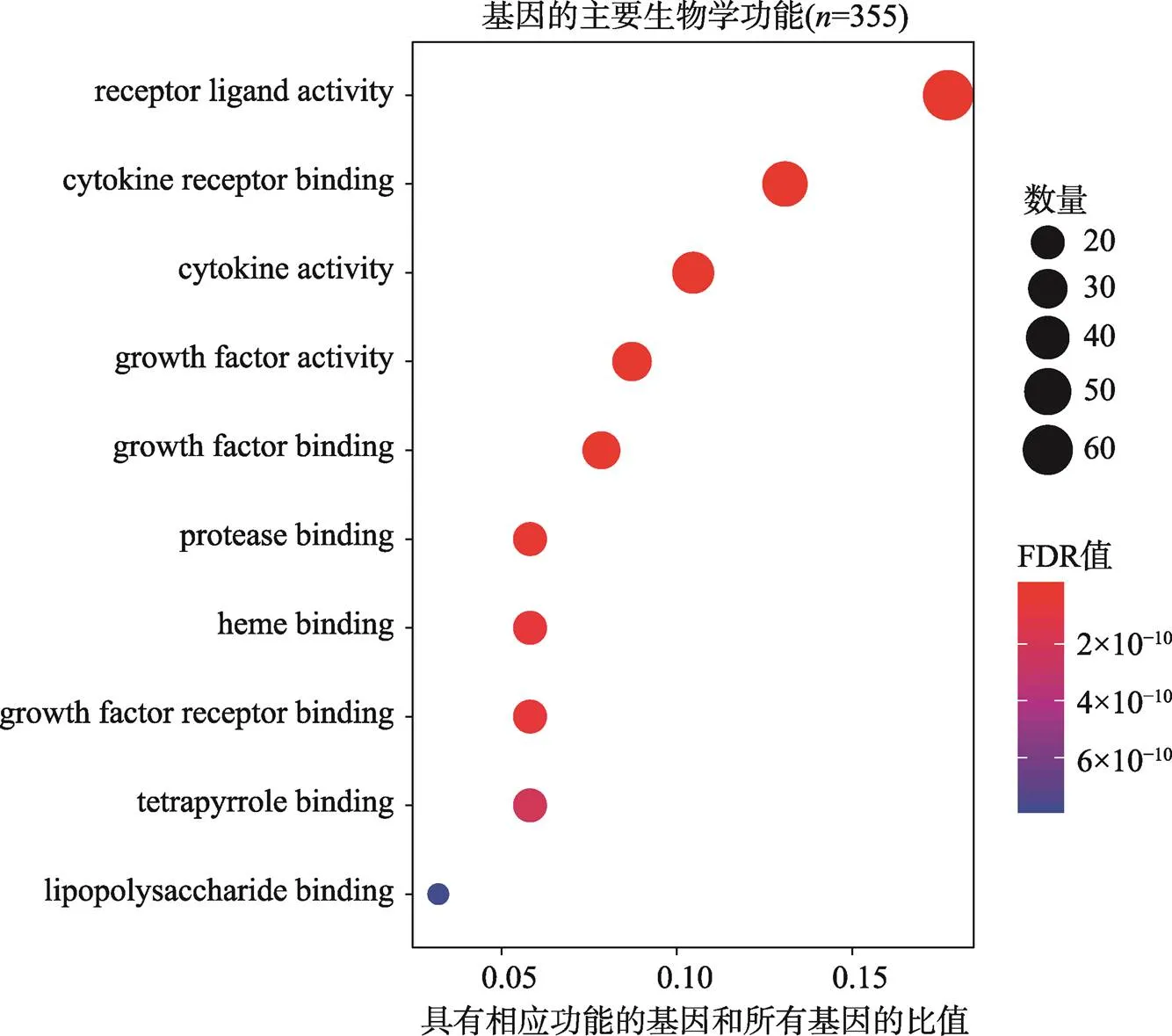
图1 基因分子功能的GO富集
颜色代表FDR值的大小,由蓝色到红色FDR值逐渐变小,圆点的面积代表基因的数量。

图2 基因KEGG通路的富集结果
颜色代表FDR值的大小,由蓝色到红色FDR值逐渐变小,圆点的面积代表基因的数量。
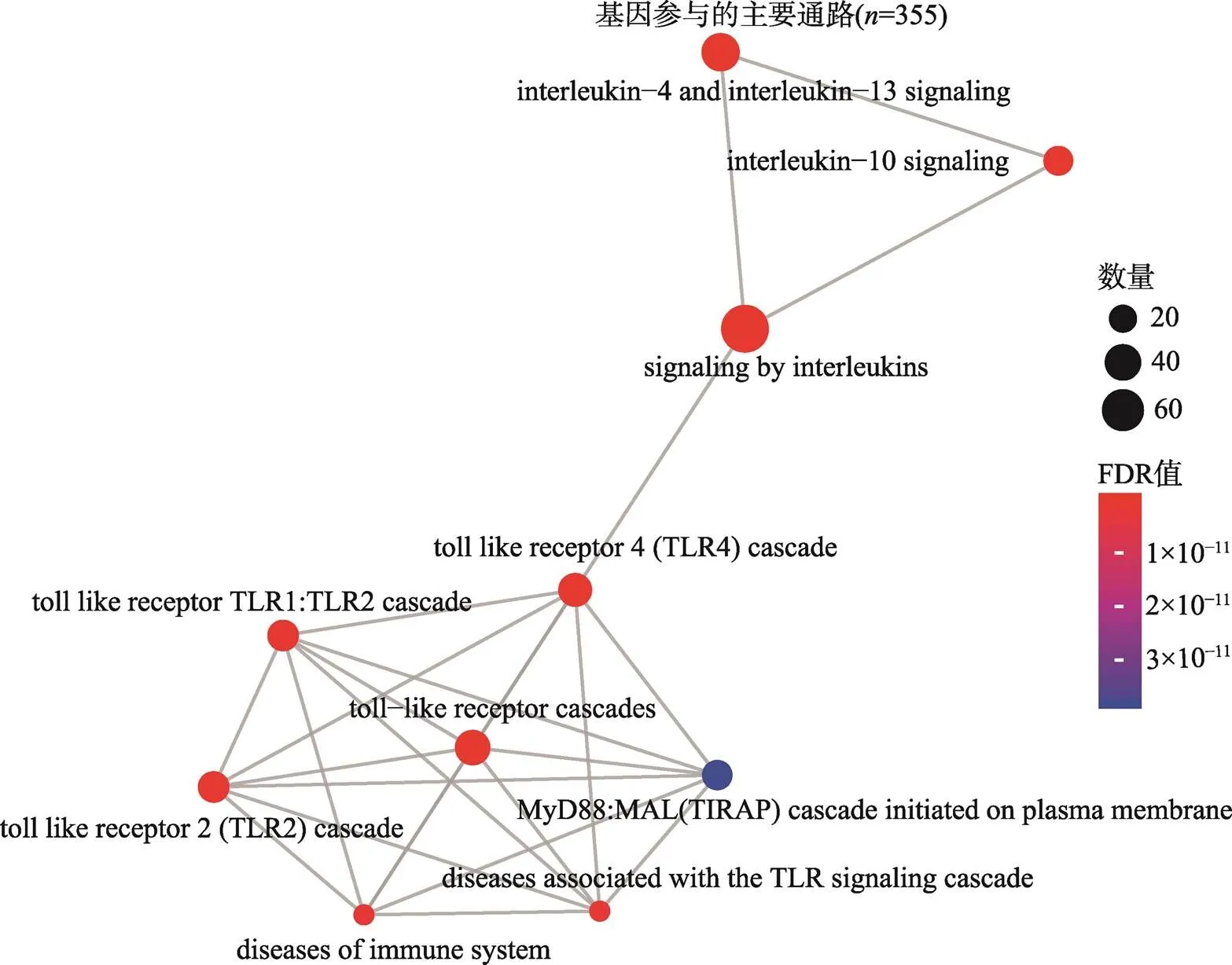
图3 基因Reactome通路的富集
颜色代表FDR值的大小,由蓝色到红色FDR值逐渐变小,圆点的面积代表基因的数量。
2.2 基因特征的收集与分析结果
对比早产基因的每个基因转录本数量和全基因组每个基因的转录本数量,早产基因的转录本数量平均值(8.2)要高于全基因组基因的转录本数量平均值(7.5) (图4A)。在显著性水平α=0.1的情况下,差异显著(检验:=0.06)。针对GC含量的比较,早产基因和全基因组基因之间没有明显差异(检验:=0.70,α=0.1) (图4B)。
在早产基因长度和全基因组编码蛋白的基因长度的比较中发现,早产基因的平均长度为63 100 bp,而全基因组基因的长度平均为61 191 bp (图5)。在显著性水平α=0.1的情况下,差异不显著(检验:=0.73)。
3 讨论
早产是新生儿健康研究领域的一个极其重要的研究方向。虽然关于早产发生发展的分子作用机制尚不明确,但是已有大量研究表明早产的发生与遗传有关,并已产生了大量的数据。本研究通过文本挖掘工具挖掘PubMed中所检索的2264篇早产相关文献中的基因,结合阈值和人工审核的两层过滤以及疾病数据库记录,最终锁定355个早产相关基因。这是目前为止从文献中挖掘的最新的早产相关基因数据集。富集分析表明早产相关基因大多集中在免疫相关通路,基因特征分析发现早产相关基因和全基因组基因对比,GC含量和基因长度没有差异,而转录本数量有差异。
以往的研究发现,免疫和炎症反应对维持妊娠和决定分娩时间起重要作用[8,20,21]。其中,由于父源和母源抗原的同时存在,母胎免疫耐受的维持在妊娠期间起重要作用,而这种稳态的破坏,可能会导致早产的发生[20]。先天免疫细胞通过释放炎性因子来影响妊娠过程和分娩时间,例如巨噬细胞释放的炎性因子可能促进催产素的产生,从而使子宫发生收缩,为分娩做准备[22]。同时,先天免疫和获得性免疫之间的失衡,也可能导致早产发生[23]。本研究采用挖掘得到的早产相关基因进行KEGG和Reactome富集分析,结果发现早产基因大多集中在免疫和炎症反应相关通路,这一点与以往的研究发现相吻合。先天免疫系统反映了对感染的应答作用,包括但不限于巨噬细胞、toll-like受体、噬中性粒细胞和细胞因子等;获得性免疫系统主要是T淋巴细胞和B淋巴细胞[24]。GO富集分析的结果也体现了早产相关基因具备与免疫过程密切相关的分子功能,包括受体配体活性、细胞因子受体活性等。本研究找到的前20个早产相关基因中,大多与免疫直接或间接相关。其中研究基因的文献数目最多,研究包括胎儿肠膜发育和早产介导炎症[25]、环境内分泌物与孕期炎症生物标志物[26]。
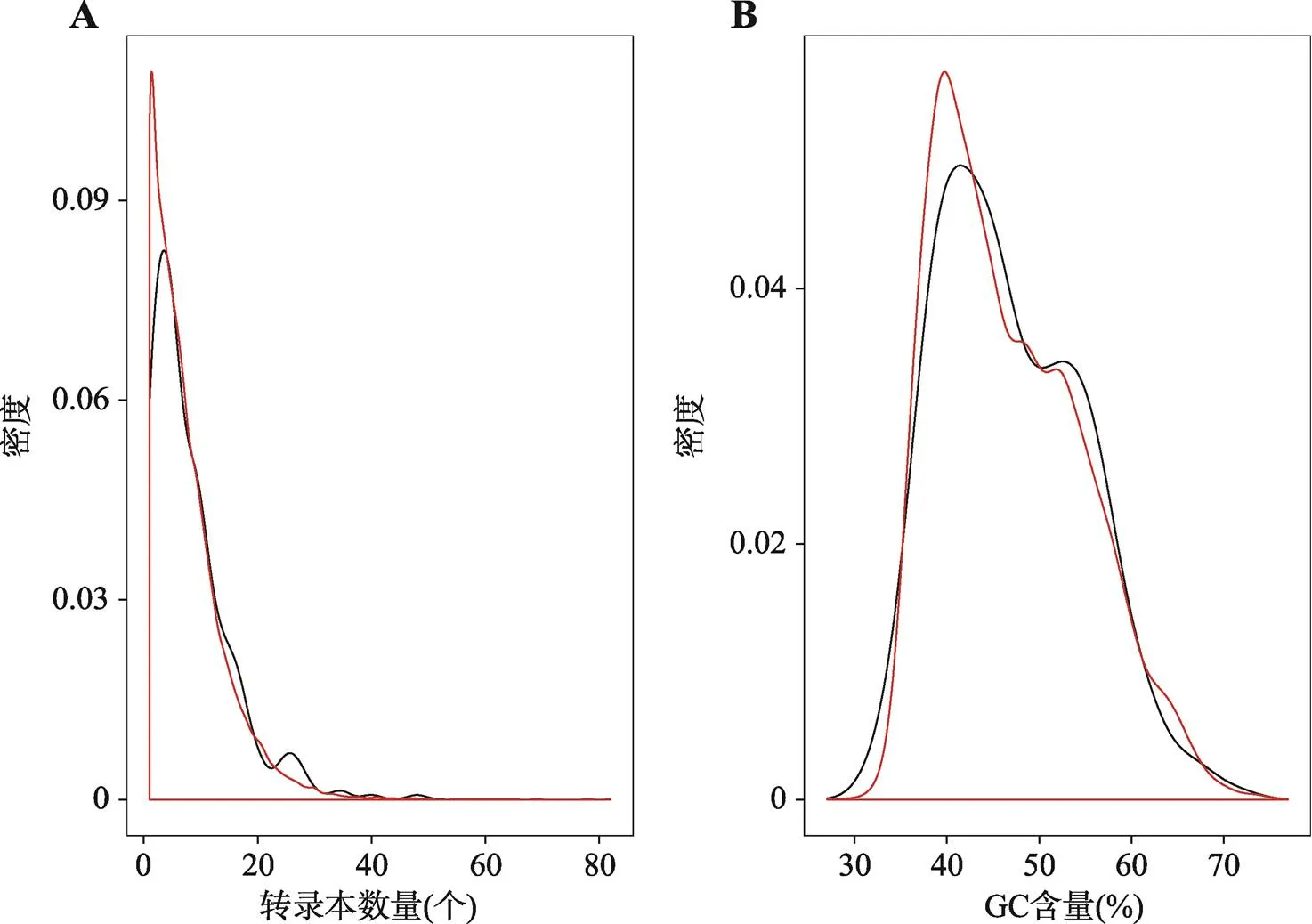
图4 对比早产基因和全基因组基因的转录本数量以及GC含量
A:转录本数量分布(个);B:GC含量分布(%)。红色的曲线代表全基因组,黑色的曲线代表早产基因。

图5 对比早产基因和全基因组编码蛋白基因的长度
红色的曲线代表全基因组,黑色的曲线代表早产基因。
据文献报道,人类基因组可能在疾病中具备一定特征[27,28],如慢性阻塞性肺疾病相关的基因转录本复杂度与对照组显著不同[29],内源性疾病的基因编码区具有高GC含量[30],在神经发育和神经退行性疾病中发现基因的长度扮演重要角色[31],其中在自闭症可能的候选基因中有许多长基因[32]。为进一步探索早产相关基因的基因组特征,本研究对比了早产相关基因与全基因组基因在转录本数量、GC含量和基因长度上的差异。其中,转录本数量存在差异。有研究发现,具有较多转录本数量的基因多为管家基因或必需基因,在生物学上起重要作用[33],然而针对转录本数量较多的早产相关基因,目前尚无文献报道。这些基因在早产所起的作用,仍需要进一步研究。GC含量在本研究中反映的是鸟嘌呤和胞嘧啶在每个基因中所占的比例。本研究并未发现早产相关基因与全基因组基因GC含量上存在显著差异。同时,早产基因在基因长度上与全基因组的所有基因相比,也无明显差异。
然而,本研究也有一定的局限性。首先,在数据库的甄选上,挖掘文献中早产相关基因时,也可以考虑包括中文数据库,例如CNKI,可以挖掘更多与中国人早产相关的研究和相关基因。其次,对基因的特征分析可以引入更多的变量,如种族信息等。对不同种族的研究,或许可以找到疾病相关且种族特异的遗传背景[34]。
综上所述,本研究结合文本挖掘和两层过滤方法以及疾病数据库记录,最终锁定355个早产相关基因,是截止到投稿时,最新的早产相关基因的整合记录。富集分析表明早产相关基因大多集中在免疫相关信号通路,基因特征分析提示了早产相关基因的转录本数量对比全基因组基因有一定差异。本研究对早产基因的挖掘和整合,可以为早产的遗传研究提供重要资源和提示相关研究方向。
附录
附表1~5见文章电子版www.chinagene.cn。
[1] Liu L, Oza S, Hogan D, Chu Y, Perin J, Zhu J, Lawn JE, Cousens S, Mathers C, Black RE. Global, regional, and national causes of under-5 mortality in 2000-15: an updated systematic analysis with implications for the sustainable development goals, 2016, 388(10063): 3027– 3035.
[2] Blencowe H, Cousens S, Oestergaard MZ, Chou D, Moller AB, Narwal R, Adler A, Vera Garcia C, Rohde S, Say L, Lawn JE. National, regional, and worldwide estimates of preterm birth rates in the year 2010 with time trends since 1990 for selected countries: a systematic analysis and implications, 2012, 379(9832): 2162–2172.
[3] Sipola-Leppänen M, Vääräsmäki M, Tikanmäki M, Matinolli HM, Miettola S, Hovi P, Wehkalampi K, Ruokonen A, Sundvall J, Pouta A, Eriksson JG, Järvelin MR, Kajantie E, Cardiometabolic risk factors in young adults who were born preterm, 2015, 181(11): 861–873.
[4] Wu W, Witherspoon DJ, Fraser A, Clark EA, Rogers A, Stoddard GJ, Manuck TA, Chen K, Esplin MS, Smith KR, Varner MW, Jorde LB. The heritability of gestational age in a two-million member cohort: implications for spontaneous preterm birth, 2015, 134(7): 803–808.
[5] Kistka ZA, DeFranco EA, Ligthart L, Willemsen G, Plunkett J, Muglia LJ, Boomsma DI. Heritability of parturition timing: an extended twin design analysis, 2008, 199(1): 43.e1–5.
[6] York TP, Eaves LJ, Lichtenstein P, Neale MC, Svensson A, Latendresse S, Långström N, Strauss JF 3rd. Fetal and maternal genes' influence on gestational age in a quantitative genetic analysis of 244,000 Swedish births, 2013, 178(4): 543–550.
[7] Liang HY, Wu BY, Chen DF, Yang F, Hu HY, Chen L, Xu XP. Association of PON2 Gene Polymorphisms in Neonates with Preterm., 2002, 24(5): 515–518.梁红业, 吴白燕, 陈大方, 杨帆, 胡海燕, 陈栎, 徐希平, 新生儿PON2基因多态性与早产的关系遗传, 2002, 24(5): 515–518.
[8] Annells MF, Hart PH, Mullighan CG, Heatley SL, Robinson JS, Bardy P, McDonald HM. Interleukins-1, -4, -6, -10, tumor necrosis factor, transforming growth factor-beta, FAS, and mannose-binding protein C gene polymorphisms in australian women: risk of preterm birth, 2004, 191(6): 2056–2067.
[9] Krediet TG, Wiertsema SP, Vossers MJ, Hoeks SB, Fleer A, Ruven HJ, Rijkers GT. Toll-like receptor 2 polymorphism is associated with preterm birth, 2007, 62(4): 474–476.
[10] Papazoglou D, Galazios G, Koukourakis MI, Kontomanolis EN, Maltezos E. Association of -634G/C and 936C/T polymorphisms of the vascular endothelial growth factor with spontaneous preterm delivery, 2004, 83(5): 461–465.
[11] Chen BH, Carmichael SL, Shaw GM, Iovannisci DM, Lammer EJ. Association between 49 infant gene polymorphisms and preterm delivery, 2007, 143A(17): 1990–1906.
[12] Zhang H, Baldwin DA, Bukowski RK, Parry S, Xu Y, Song C, Andrews WW, Saade GR, Esplin MS, Sadovsky Y, Reddy UM, Ilekis J, Varner M, Biggio JR Jr. A genome-wide association study of early spontaneous preterm delivery, 2015, 39(3): 217–226.
[13] Zhang GB, Feenstra B, Bacelis J, Liu X, Muglia LM, Juodakis J, Miller DE, Litterman N, Jiang PP, Russell L, Hinds DA, Hu Y, Weirauch MT, Chen X, Chavan AR, Wagner GP, Pavličev M, Nnamani MC, Maziarz J, Karjalainen MK, Rämet M, Sengpiel V, Geller F, Boyd HA, Palotie A, Momany A, Bedell B, Ryckman KK, Huusko JM, Forney CR, Kottyan LC, Hallman M, Teramo K, Nohr EA, Davey Smith G, Melbye M, Jacobsson B, Muglia LJ. Genetic associations with gestational duration and spontaneous preterm birth, 2017, 377(12): 1156–1167.
[14] McElroy JJ, Gutman CE, Shaffer CM, Busch TD, Puttonen H, Teramo K, Murray JC, Hallman M, Muglia LJ. Maternal coding variants in complement receptor 1 and spontaneous idiopathic preterm birth, 2013, 132(8): 935–942.
[15] Knijnenburg TA, Vockley JG, Chambwe N, Gibbs DL, Humphries C, Huddleston KC, Klein E, Kothiyal P, Tasseff R, Dhankani V, Bodian DL, Wong WSW, Glusman G, Mauldin DE, Miller M, Slagel J, Elasady S, Roach JC, Kramer R, Leinonen K, Linthorst J, Baveja R, Baker R, Solomon BD, Eley G, Iyer RK, Maxwell GL, Bernard B, Shmulevich I, Hood L, Niederhuber JE. Genomic and molecular characterization of preterm birth, 2019, 116(12): 5819–5827.
[16] Uzun A, Laliberte A, Parker J, Andrew C, Winterrowd E, Sharma S, Istrail S, Padbury JF. DbPTB: a database for preterm birth, 2012, 2012: bar069.
[17] Yu G, Wang LG, Han Y, He QY. ClusterProfiler: an R package for comparing biological themes among gene clusters, 2012, 16(5): 284–287.
[18] Hur J, Schuyler AD, States DJ, Feldman EL. SciMiner: web-based literature mining tool for target identification and functional enrichment analysis, 2009, 25(6): 838–840.
[19] Fabregat A, Jupe S, Matthews L, Sidiropoulos K, Gillespie M, Garapati P, Haw R, Jassal B, Korninger F, May B, Milacic M, Roca CD, Rothfels K, Sevilla C, Shamovsky V, Shorser S, Varusai T, Viteri G, Weiser J, Wu G, Stein L, Hermjakob H, D'Eustachio P. The reactome pathway knowledgebase, 2018, 46(D1): D649– D655.
[20] Romero R, Dey SK, Fisher SJ. Preterm labor: one syndrome, many causes,, 2014, 345(6198): 760–765.
[21] Macones GA, Parry S, Elkousy M, Clothier B, Ural SH, Strauss JF 3rd. A polymorphism in the promoter region of TNF and bacterial vaginosis: preliminary evidence of gene-environment interaction in the etiology of spontaneous preterm birth, 2004, 190(6): 1509–1519.
[22] Fang X, Wong S, Mitchell BF. Effects of LPS and IL-6 on oxytocin receptor in non-pregnant and pregnant rat uterus, 2000, 44(2): 65–72.
[23] Gomez-Lopez N, StLouis D, Lehr MA, Sanchez- Rodriguez EN, Arenas-Hernandez M. Immune cells in term and preterm labor, 2014, 11(6): 571–581.
[24] Melville JM, Moss TJ. The immune consequences of preterm birth, 2013, 7: 79.
[25] Schreurs R, Baumdick ME, Sagebiel AF, Kaufmann M, Mokry M, Klarenbeek PL, Schaltenberg N, Steinert FL, van Rijn JM, Drewniak A, The SML, Bakx R, Derikx JPM, de Vries N, Corpeleijn WE, Pals ST, Gagliani N, Friese MA, Middendorp S, Nieuwenhuis EES, Reinshagen K, Geijtenbeek TBH, van Goudoever JB, Bunders MJ. Human fetal TNF-α-Cytokine-Producing CD4+effector memory T cells promote intestinal development and mediate inflammation early in life, 2019, 50(2): 462–476.e8.
[26] Ferguson KK, Cantonwine DE, Rivera-González LO, Loch-Caruso R, Mukherjee B, Anzalota Del Toro LV, Jiménez-Vélez B, Calafat AM, Ye X, Alshawabkeh AN, Cordero JF, Meeker JD. Urinary phthalate metabolite associations with biomarkers of inflammation and oxidative stress across pregnancy in Puerto Rico, 2014, 48(12): 7018–7025.
[27] Collins A. The genomic and functional characteristics of disease genes, 2014 16(1): 16–23.
[28] Pengelly RJ, Vergara-Lope A, Alyousfi D, Jabalameli MR, Collins A. Understanding the disease genome: gene essentiality and the interplay of selection, recombination and mutation, 2019, 20(1): 267–273.
[29] Lackey L, McArthur E, Laederach A. Increased transcript complexity in genes associated with chronic obstructive pulmonary disease, 2015, 10(10): e0140885.
[30] Peng Z, Uversky VN, Kurgan L. Genes encoding intrinsic disorder in Eukaryota have high GC content, 2016, 4(1): e1262225.
[31] Zylka MJ, Simon JM, Philpot BD. Gene length matters in neurons, 2015, 86(2): 353–355.
[32] King IF, Yandava CN, Mabb AM, Hsiao JS, Huang HS, Pearson BL, Calabrese JM, Starmer J, Parker JS, Magnuson T, Chamberlain SJ, Philpot BD, Zylka MJ. Topoisomerases facilitate transcription of long genes linked to autism, 2013, 501(7465): 58–62.
[33] Ryu JY, Kim HU, Lee SY. Human genes with a greater number of transcript variants tend to show biological features of housekeeping and essential genes, 2015, 11(10): 2798–2807.
[34] Rappoport N, Toung J, Hadley D, Wong RJ, Fujioka K, Reuter J, Abbott CW, Oh S, Hu D, Eng C, Huntsman S, Bodian DL, Niederhuber JE, Hong X, Zhang G, Sikora-Wohfeld W, Gignoux CR, Wang H, Oehlert J, Jelliffe-Pawlowski LL, Gould JB, Darmstadt GL, Wang X, Bustamante CD, Snyder MP, Ziv E, Patsopoulos NA, Muglia LJ, Burchard E, Shaw GM, O'Brodovich HM, Stevenson DK, Butte AJ, Sirota M. A genome-wide association study identifies only two ancestry specific variants associated with spontaneous preterm birth, 2018, 8(1): 226.
Mining and characterization of preterm birth related genes
Xuanshi Liu, Wei Li
Preterm birth (PTB) refers to birth before 37 completed gestational weeks. PTB is the leading cause of neonatal deaths and is associated with various neonatal complications and adult-onset chronic diseases. According to twin and family studies, genetic variants account for about 15% to 35% of the incidence of PTB. However, the molecular epidemiology of PTB is still unclear. By mining the PTB-related researches in the literature database and the disease databases, and combining two filtering methods, 355 PTB-related genes were selected. The enrichment analyses of molecular function revealed that the main functions of PTB-related genes include: receptor ligand activity, cytokine receptor binding, cytokine activity, growth factor activity, etc.; the main pathways from KEGG enrichment were the AGE-RAGE signaling pathway in diabetic complications, Chagas disease, and the IL-17 signaling pathway, the TNF signaling pathway, etc, as well as several immune-related pathways from Reactome enrichment. There were differences in the number of transcripts between PTB-related genes and other genes in the genome (α = 0.1,= 0.06), but there was no significant difference in GC content and gene lengths. The results suggest that PTB-related genes are mostly in immune-related pathways, and have molecular functions closely related to immunity. Our work provides an important resource for the study of the genetical mechanisms of PTB.
preterm birth; data mining; enrichment analysis; gene features; transcript number
2019-03-21;
2019-05-08
刘玄石,博士研究生,助理研究员,专业方向:生物信息学。E-mail: liuxs2017bioinf@163.com
李巍,博士,教授,博士生导师,研究方向:医学生物化学,医学遗传,细胞生物学,产前诊断以及遗传咨询。E-mail: liwei@bch.com.cn
10.16288/j.yczz.19-078
2019/5/10 15:23:07
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190510.1522.002.html
(责任编委: 方向东)

