基于线粒体COI基因序列的蒙古鲌群体遗传结构分析
谢佳燕,颜 渊,杨钰慧,林少卿
(1.武汉轻工大学生物与制药工程学院,武汉 430023;2.西藏自治区畜牧总站水产科,拉萨 850007)
蒙古鲌(Chanodichthysmongolicus)隶属于鲤形目(Cypriniformes)鲤科(Cyprinidae),为凶猛肉食性鱼类。广泛分布于我国的江河、湖泊、水库等各种水体,是长江流域中重要的经济鱼类[1-2]。蒙古鲌曾是长江中下游湖泊鱼类群落的优势物种和渔业生产的主要对象[1]。然而, 由于过度捕捞、生境破坏和水污染等原因, 天然水域中蒙古鲌资源严重衰退,分布区域缩小[3]。为弥补天然资源的不足和满足市场需求,近年来长江中下游一些湖泊已开始实施蒙古鲌人工增殖放流和人工繁殖的研究[4-5]。对放流群体进行监测后发现蒙古鲌生长速度较快, 放流次年可长至500 g以上的商品鱼, 是湖泊增殖放流的优良品种[5]。然而,在人工繁殖的实践中,规模化人工养殖的首要问题是解决苗种来源。因此,对物种天然种群进行种质资源鉴定和遗传组成的分析,对于合理利用物种种质资源及水产养殖业结构调整具有重要的实践意义。
线粒体COI(cytochrome oxidase subunit I)基因为位于线粒体DNA的蛋白质编码基因,已广泛应用于物种鉴定和群体遗传学等方面的研究[6-8]。彭居俐等[6]用COI基因序列探讨了鲌属鱼类物种间的系统发育关系。王丹等[7]揭示了三峡库区鲌属鱼类具有较高的线粒体COI基因序列遗传多样性。目前, 国内外学者对蒙古鲌的生物学特性、遗传组成、人工繁殖技术和营养成分等方面已进行了相应的研究[2,4,6-9],但对蒙古鲌自然种群间遗传多样性和遗传结构分析方面的研究较少。因此,本研究对我国长江中下游流域湖北段和江苏段不同河段或湖泊中的蒙古鲌种群进行COI基因序列测序,分析不同水域蒙古鲌种群的遗传组成和遗传关系,以期为长江流域蒙古鲌自然种质资源现状的评价、保护和合理开发利用提供理论依据。
1 材料与方法
1.1 样本采集
蒙古鲌样本分别采自于湖北省丹江口市丹江口水库(DJ) 、湖北省荆门市漳河镇漳河(JM) 、武汉市磨山东湖(DH) 、武汉市安山镇梁子湖(LZH) 和江苏省丹阳市(JS)。取新鲜肌肉样于95%乙醇中固定,-20 ℃保存。
1.2 基因组DNA提取、PCR扩增与序列测定
采用标准酚/氯仿法提取蒙古鲌基因组DNA[10],利用Ward等[11]的引物FishF1/FishR2扩增COI基因序列。PCR扩增体系为30L,包括1.5 U的Taq DNA聚合酶和1L 的DNA模板。扩增反应条件为94 ℃预变性3 min,随后进行35个循环。每个循环由94 ℃ 50 s,52 ℃ 1 min,72 ℃ 1.5 min组成。之后72 ℃延伸10 min,4 ℃结束。产物送测序公司完成测序工作。
1.3 数据分析
采用ClustalX 1.83[12]和Seaview[13]软件进行COI基因片段核苷酸组成和序列分析。用DnaSP v5[14]软件计算群体单倍型,Arlequin 3.5[15]进行遗传多样性、群体的分子方差分析(Analysis of molecular variance, AMOVA)、遗传距离和基因流Nm值的计算。使用Network 软件基于Median Joining方法构建单倍型之间的系统关系图[16]。从NCBI下载鲤属鲤(Cyprinuscarpio,序列号KJ511883)和海南鲌(C.recurviceps,序列号KJ609181)的线粒体DNA全长序列,与蒙古鲌个体的DNA序列进行比对,识别COI基因序列并进行比较分析。在MEGA 6.0软件[17]中以Kimura双参数法(Kimura-2-parameter)为替代模型,采用NJ法(Neighbour-joining)构建蒙古鲌不同种群的分子聚类树,分支置信度采用自引导法(Bootstrap analysis) l 000次重复检测。
2 结果
2.1 蒙古鲌COI基因片段核苷酸组成特点
对蒙古鲌样本的线粒体COI基因序列进行比对,获得COI基因其5′端长度为652 bp的同源序列。其中,多态位点9个,无碱基插入或缺失,7个位点为碱基转换,2个位点发生碱基颠换,但翻译后所有氨基酸组成均相同,表明COI基因的编码在蒙古鲌鱼种中的保守性。序列中(A+T)碱基的含量为0.524高于(G+C)的含量0.475,但碱基组成在蒙古鲌不同种群间无显著差异。
2.2 蒙古鲌COI基因序列的单倍型分析
蒙古鲌不同地理种群个体的COI基因序列共定义了10种单倍型,各单倍型在各地理种群间的分布见表1。6种单倍型为单个蒙古鲌种群所特有, 占个体总数的13.2%,另外的4种单倍型在不同种群间共享。其中,单倍型h9分布最广,在蒙古鲌所有群体中均有分布,其个体数占总数的54.7%。种群间单倍型数最多是东湖种群(DH)为6种,荆门种群(JM)最少仅有1种。对蒙古鲌种群的单倍型进行网络图分析(图1)。结果表明,所有单倍型以种群单倍型2为中心和起点,其他单倍型则经过1~2步突变与其相连,其他单倍型呈星状分布态势或进化形成新的分支。各单倍型之间的突变步数为1~2步,各单倍型并未依据采样地而形成各自的分支。
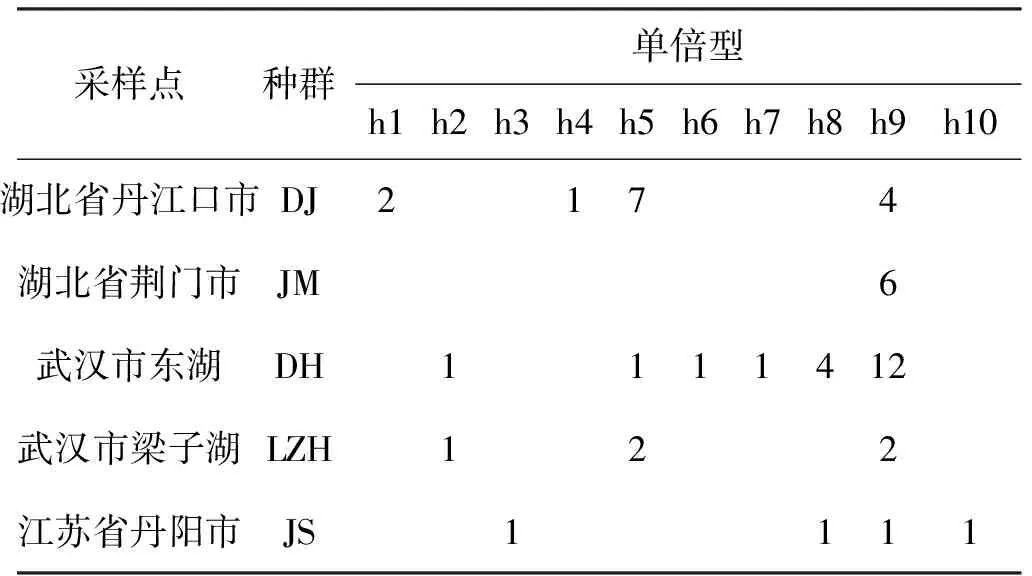
表1 蒙古鲌不同种群COI基因序列的单倍型分布
2.3 不同蒙古鲌种群的遗传多样性
对蒙古鲌不同地理种群的遗传多样性进行检测(表2)。结果表明,蒙古鲌总群体的基因多态性(H)为0.664,核苷酸多态性()为0.002。不同种群间遗传多样性存在一定差异,其中,江苏(JS)和梁子湖(LZH)种群间的基因多态性和核苷酸多态性较高,而荆门(JM)种群仅有一种单倍型,遗传多样性指数值为零。
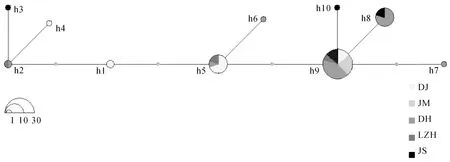
图1 蒙古鲌不同种群线粒体COI基因的单倍型网络图
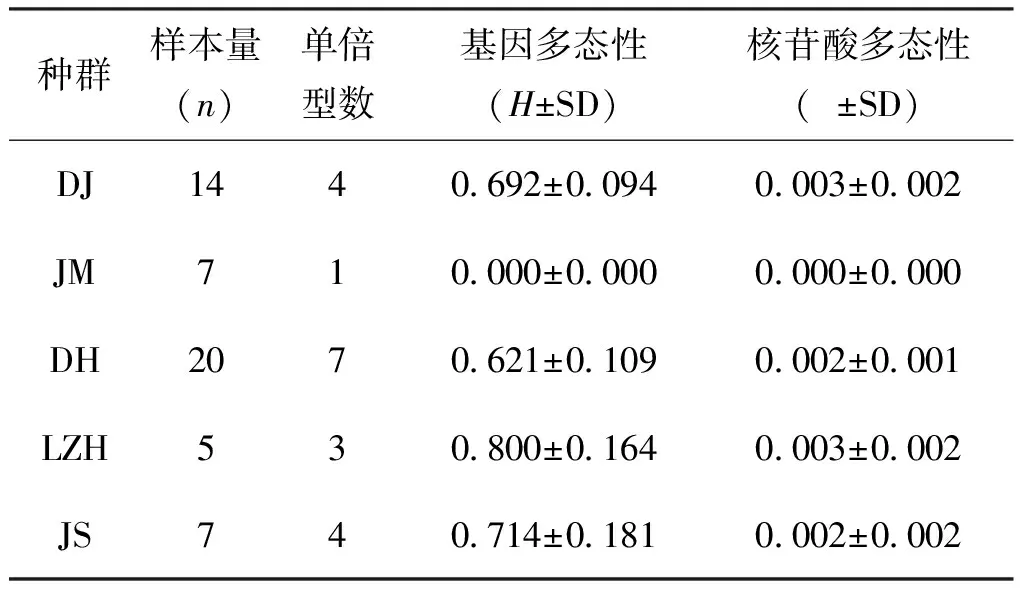
表2 蒙古鲌种群的基因多态性和核苷酸多态性
2.4 蒙古鲌的种群结构与遗传差异
对蒙古鲌不同种群之间的遗传距离进行检测(表3)。结果表明,种群间的遗传距离在-0.076~0.383之间,除DJ群体与其他群体间的遗传距离存在显著差异(P<0.05)外,其余的群体间无明显差异,种群间存在一定的基因流,整个野生种群的基因流值为7.841。对蒙古鲌不同种群进行分子变异分析(AMOVA)表明,遗传变异主要发生在种群内,占整个种群的79.7%。根据群体间遗传距离的结果,将所有种群分为两组(DJ群体组和其他个体组)进行分子变异分析。结果表明,不同组间遗传变异为30.1%,组间无明显差异(P>0.05)。
基于Kimura双参数法,计算不同蒙古鲌种群间的遗传距离,以鲤属鲤(Cyprinuscarpio,序列号KP993137)的线粒体DNA序列作为外群,构建NJ聚类树(图2)。结果表明,蒙古鲌种群个体呈现一种混杂的分布格局, 未检测到与采样地点严格对应的分支, 不能依据地理区系进行分群。
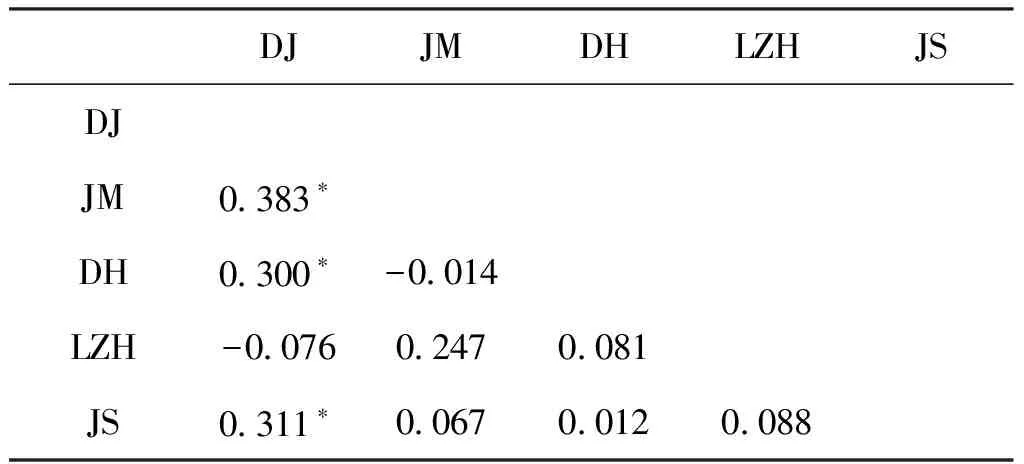
表3 不同蒙古鲌种群间的遗传距离
注:*为群体间的遗传距离存在显著差异,P<0.05
3 讨论
本研究中长江中下游流域湖北段和江苏段不同河段或湖泊中的蒙古鲌自然种群的遗传多样性水平中等,不同种群间遗传多样性存在一定差异。单个种群特有的单倍型较少,较多个体属于共享单倍型。这可能与近年来长江中下游流域生境破坏、过度捕捞和水污染等致使天然水域中蒙古鲌资源严重衰退有关[3]。
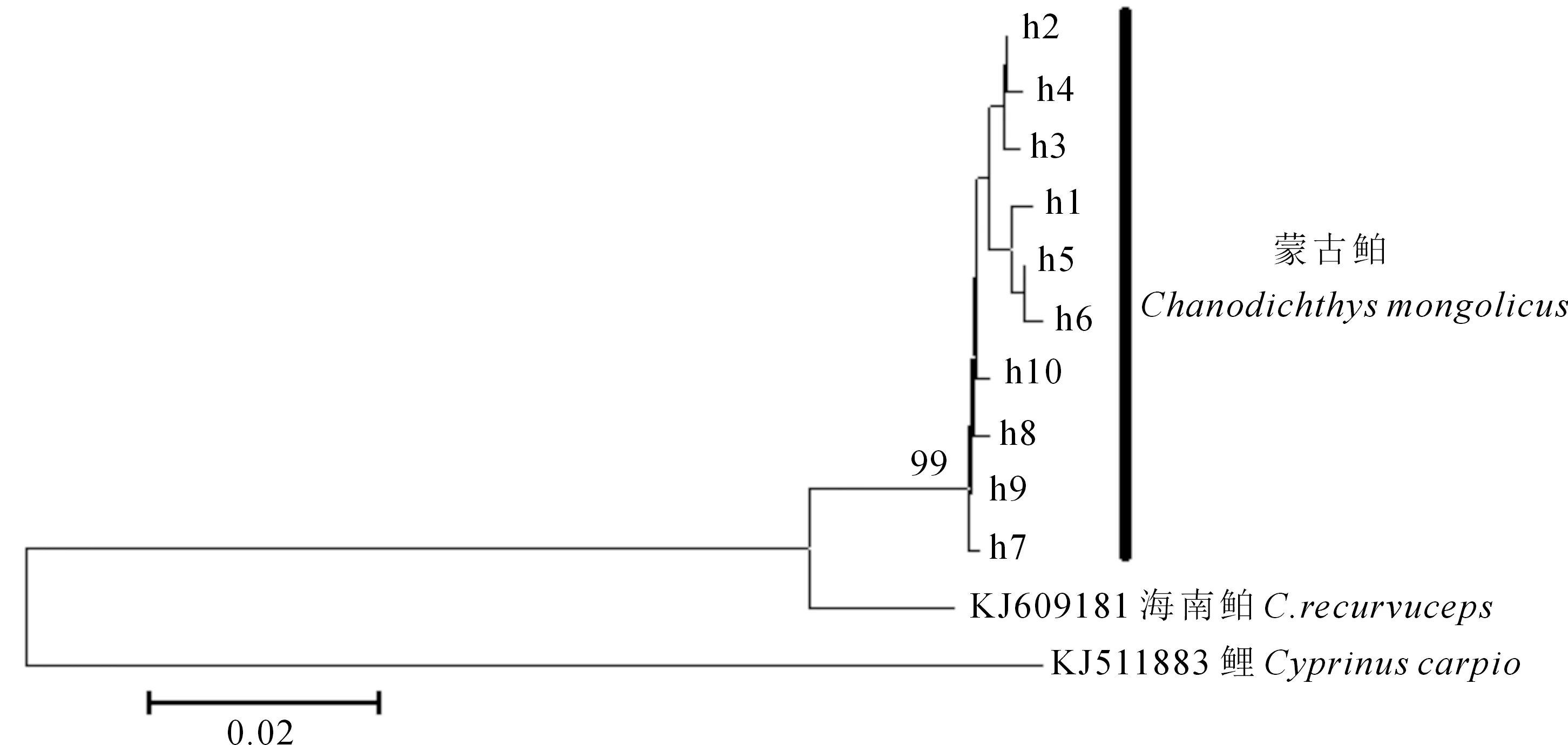
图2 蒙古鲌线粒体COI基因序列单倍型的NJ聚类树
种群分子方差分析表明了蒙古鲌不同野生种群间显示出一定程度的遗传不同。地理隔离、栖息地差异、迁移、人类活动等因素均可能导致群体间遗传差异的出现[18-20]。一些湖泊群体历史上与长江干流连通,近年来与干流处于相对可控连通的状态[21-22],可能促进了栖息地间的不连续性和地理隔离。同时,蒙古鲌繁殖时所产卵具一定黏性,而栖息于江河流水或水库上游环境中的个体可产漂流性卵[23],但水体间长距离迁移的削弱,可能加速了由于地理距离导致的遗传变异的产生。此外,栖息地水体环境污染日益严重,梯级水利设施的大规模构建和过度捕捞等,也可能促进了种群间的遗传差异[24-25]。汉江为长江最大支流,于长江中游武汉段汇入长江。丹江口群体位于汉江上游,地理位置上的不同可削弱支流上游群体与干流上中游群体间的迁移和基因交流;此外,20世纪70年代建成了丹江口大坝。大坝隔离和构建后水力学条件的显著变化也可削弱支流上游群体向干流群体的迁移,从而可促进其遗传差异的产生和固定。一些研究也发现丹江口大坝建成后汉江中下游经济鱼类的早期资源发生明显的变化,经济鱼类卵苗径流量明显下降,产卵场数量减少[24-25]。
虽然不同蒙古鲌种群间显示了一定程度的遗传差异,但聚类树也揭示了蒙古鲌不同种群个体仍然呈现一种混杂的分布格局,无法依据地理区系进行分群,未形成明显的系统地理结构, 表明蒙古鲌种群可能正处于谱系重排时期。分子方差分析也表明虽然丹江口群体与其他群体间存在明显差异,但分组分析并未表明丹江口群体可完全解释组间变异的产生。两个因素可能导致物种种群处于谱系重排状态,即种群间的隔离分化较短及种群间基因交流[26]。鲌属鱼类是东亚特有鲤科鱼类中较为重要的一个类群[1],研究认为鲌属鱼类经历了相同的短时间物种暴发,因此作为一群快速分化的物种,其基因序列的变异度相对较小[27]。此外,遗传距离分析也表明大部分的群体间无显著的遗传差异,不同种群间仍然存在一定的基因交流;栖息地水系间相互连通,也促进了种群间相互交流,共享单倍型,削弱因地理距离产生的种群间差异,致使不同种群间缺乏较为明显的按地理单元划分的地理分布格局。

