牧草细胞壁降解菌的群落结构多样性研究
侯玉洁, 徐 俊,王淑清, 刘昌林, 郭小泽,杨宗英, 曾柳根, 雷小青, 熊春贤, 邓敏超, 许亮清*
(1.南昌市农业科学院,江西南昌 330008;2.江西省农业科学院农产品质量安全与标准研究所,江西南昌 330200;3.南昌市动物疫病预防控制中心,江西南昌 330008;4.江西省农业科学院畜牧兽医研究所,江西南昌 330200;5.新建区农业农村局,江西南昌 330100)
纤维在瘤胃微生物作用下降解为挥发性脂肪酸为机体供能,而饲粮是影响瘤胃微生物组成和多样性的关键因素 (Shawrang等,2013;Wilson等,1997),牧草化学组成和解剖学结构的差异造成了瘤胃降解率的不同,而吸附在细胞壁上的细菌是影响降解的关键因素(Saro等,2012)。徐俊等(2015)研究指出,不同牧草组织结构存在显著差异,微生物对细胞壁的吸附和降解与牧草组织结构和类型密切相关,黄色瘤胃球菌(R.flavefaciens)、白色瘤胃球菌(R.albus)和产琥珀酸丝状杆菌 (F.succinogenes)是三种主要纤维分解菌(Koike 等 ,2006;Krause 等 ,2006;Tajima 等 ,2001),他们对植物细胞壁的降解作用已通过电子显微镜观察得到了充分证实 (Jung等,2001;Akin等,1975)。研究发现羊茅草和鸭茅草中纤维分解菌有70%属于F.succinogenes和R.flavefaciens(Cheng 等,1984;Akin 等,1980),分别占细菌总量的0.1% ~6.6%和1.3% ~2.9%(Krause等,1999;Lin 等,1994)。Thoetkiattikul等(2013)利用高通量测序技术研究不同纤维和淀粉比例饲粮对奶牛瘤胃微生物特性的影响发现,瘤胃中主要的细菌包括拟杆菌门、硬壁菌门和变形菌门,且细菌丰富度与饲粮纤维含量密切相关。
目前,研究多集中在对几种主要纤维分解菌数量变化的分析上(Ghasemi等,2012),通过高通量测序手段对嵌在牧草细胞壁内部微生物多样性的研究鲜有报道。 徐俊等(2016、2014、2013)报道的牧草茎秆各时间点降解率变化及扫描电镜图片结果显示,牧草在降解24 h后的组织结构变化最大,因此,本试验通过高通量测序手段进一步从微生物角度探究吸附在牧草细胞壁的微生物多样性,以期更加真实、全面地反映降解牧草细菌的群落结构。
1 材料与方法
1.1 主要试剂和药品 粪便总基因组提取试剂盒 QIAamp DNAstool mini ki(QIAGEN,51504);Pyrobest DNA Polymerase(TAKARA,Japan);琼脂糖(Invitrogen,75510-019);DNA 胶回收纯化试剂盒 Axy Prep DNA Gel Extration kit (Axygen,APGX-500);文库检测及定量试剂盒 Quant-iT PicoGreen dsDNA Assay Kit(Invitrogen,P7589);测序试剂盒 Miseq keagent kit v2 (Illumina,MS-102-2003);构建测序文库试剂盒Agencourt AMPure XP Beads (Illumina,A63881) 和 TruSeqTMDNA Sample Prep kit-SetA (Illumina,MS-102-2003);蛋白酶 K(TaKaRa,DRR006B);无水乙醇(天津科密欧);异丙醇(色谱级);双蒸水。
1.2 主要仪器和设备 -80℃超低温冰箱(BCD-208K ACJN),青岛海尔股份有限公司;制冰机(FMB-70),无锡沃信仪器有限公司;冷冻离心机(5810R),德国 Eppendorf公司;高压灭菌锅(LDZX-50KBS),上海申安医疗器械厂;凝胶成像系统(BS60chamGel 1600),西化仪科技有限公司;DYY-6C电泳仪,北京六一仪器厂;PCR仪,德国Eppendorf公司;酶标仪 (FLX800),美国伯腾(BioTek)仪器有限公司;安捷伦2100分析仪,美国安捷伦(Agilent)科技公司;Illumina-Misep DNA测序平台,美国Illumina公司。
1.3 试验动物和饲养管理 选取3头安装有永久性瘤胃瘘管的荷斯坦奶牛[(600±15)kg],饲喂主要由玉米青贮和秸秆组成的全混合日粮,栓系式饲养,日喂两次,自由饮水。
1.4 试验材料 本试验选用长势和株高相近的初花期紫花苜蓿、抽穗初期燕麦草和羊草以及收割后的稻草秸秆,从根部切割后去掉叶子和叶鞘,剥离出茎秆作为试验材料。
1.5 试验步骤 分别称取3.0 g粉碎好的茎秆样品(2 mm 筛)放入尼龙袋(8 cm×12 cm,网孔孔径300目)中,于晨饲前分别投放到奶牛瘤胃中,将尼龙绳固定在瘘管盖上。瘤胃中降解24 h后取出所有尼龙袋,放入冰水中停止发酵,用冷水冲洗直到流出的水澄清为止,将样品放入-80℃低温冰箱中保存。
根据细菌16SrDNA基因V3区的保守序列,设 计 通 用 引 物 338F:5' -ACTCCTACGGGAGGCAGCAG -3',533R:5' -TTACCGCGGCTGCTGGCAC-3'。试验中12个样品的Barcode序列如表1所示,PCR扩增采用25μL反应体系,体系组成如表2所示。
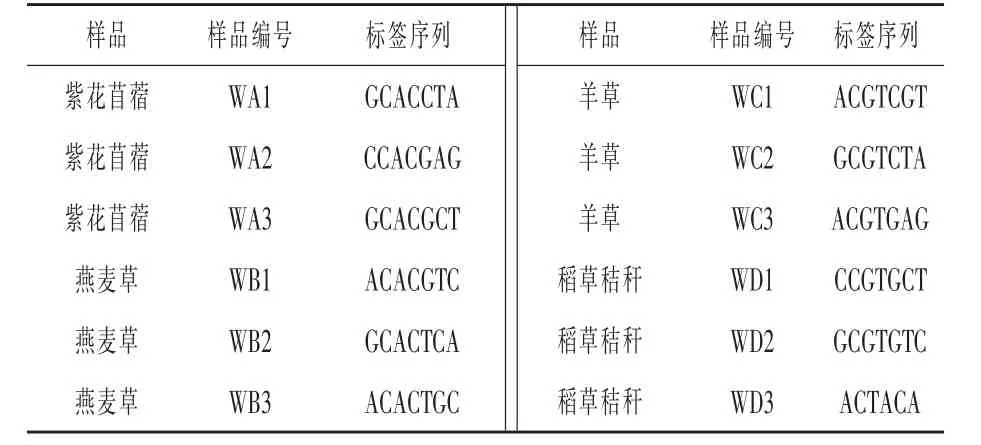
表1 样品Barcode序列
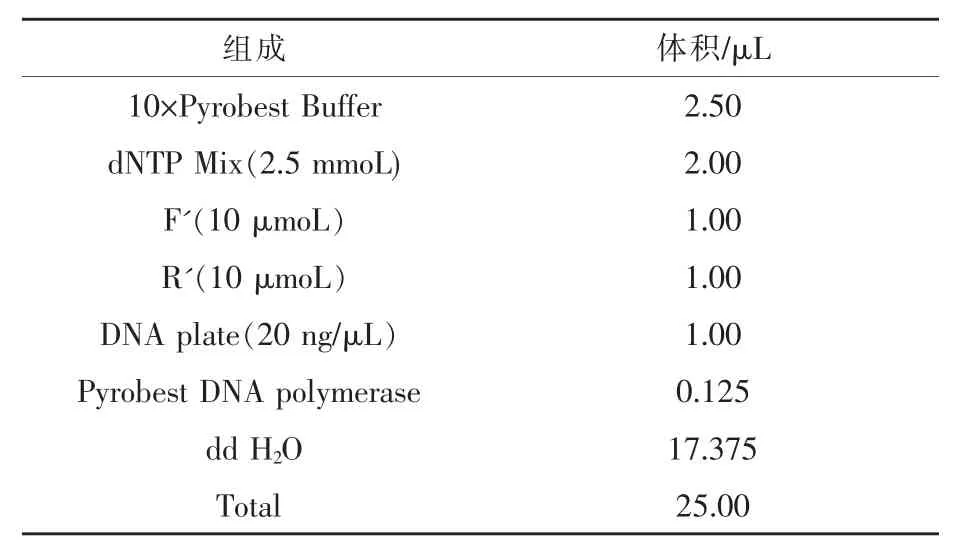
表2 PCR扩增体系
PCR扩增采用25μL反应体系,反应程序为:94℃预变性5 min;94℃变性30 s,58℃退火30 s,72℃延伸30 s,进行21个循环;最后72℃延伸5 min;结束时4℃保存。PCR扩增产物用1.5%琼脂糖凝胶电泳进行检测,然后使用Axygen DNA胶回收纯化试剂盒 (Axy Prep DNA Gel Extration kit,AP-GX-500)对 V3区扩增产物进行切胶回收纯化,经Biotek酶标仪对纯化好的PCR产物进行定量,将样品等量混匀后形成均一混合物。PCR混合物经质量控制后,采用标准的Illumina TruSeq DNA文库制备流程构建Illumina测序文库,最后按照Illumina Miseq平台上机进行Barcoded Illumina Miseq测序。
1.6 数据处理与分析 测序结束后,对原始序列数据进行质量控制得到有效序列,再对有效序列进行去杂处理,丢弃长度短于120 bp、含有模糊碱基,引物碱基含2个以上的错配信息、单个碱基重复数超过6个的序列,最终获得用于后续分析的优质序列。通过Qiime分析平台对优质序列进行生物信息学分析,根据序列相似度为97%的原则,将序列归为多个操作分类单元(OTU)。然后对序列进行聚类分析,选取每个类最长序列为代表序列,采用RDP-classifier,以RDP数据库的序列为训练集,对OTU代表序列进行注释,得到每个OTU的分类学信息。通过软件Mothur(http://www.mothur.org)对生成的OTU信息进行细菌群落多样性和丰富度分析。
数据采用Excel 2007进行整理,结果采用SAS统计软件的PDIFF模块进行方差分析和显著性检验,以P<0.05作为差异显著性判断标准。
2 结果与分析
2.1 细菌丰富度和多样性 通过Illumina Miseq高通量测序后,本研究12个样品共产生有效序列591378条,经质量控制后得到562727条高质量序列,每个样品平均产生46894条序列,平均序列长度为161 bp。
为了研究不同牧草经瘤胃细菌降解后的菌群结构多样性,采用Chao和Ace指数计算群落物种丰富度,用Shannon和Simpson指数计算群落结构多样性。样品在97%相似性水平下的测序覆盖深度用Good’coverage计算。12个样品在97%相似性水平下的序列数和多样性指数如表3所示。
Rarefaction curve和Shannon多样性曲线可以反映该测序量和97%相似度OTU水平下文库的生物多样性。从图1可知,OTU数目随测序量的提高不断提高,当测序量到达一定程度时,OTU增加趋势变得缓慢,但仍未达到饱和,说明随着测序量的深入,可检测到瘤胃中大部分菌种,但仍有少部分新的物种有待进一步发现。上述结果表明,增加测序量仍然可能发现新的菌种,但本试验测序量基本可以反映瘤胃内细菌的多样性情况。
在Rank abundance曲线中,对各样品的OTU进行丰度排序,对丰度值取log2的对数值做出Rank abundance曲线图,如图2可知,12个样品中均发现存在一条很长的尾巴,这说明降解不同牧草的细菌群落中存在大量低丰度的菌种,这也再次说明,使用高通量测序手段可以更加真实、全面地反映降解牧草细菌的群落结构。

表3 12个牧草样品在97%相似性水平下丰富度和多样性指数

图1 97%相似性水平下12个牧草样品的稀释曲线
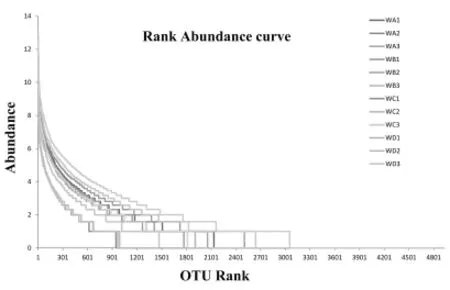
图2 12个牧草样品丰度分布曲线图
2.2 细菌组成和群落结构 本试验采用RDP和BLAST同源性序列比对所有样品序列鉴定得到16个门,27个纲,37个目,63个科和91个属。由图3可知,在门水平上,各牧草组均以硬壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、螺旋体门(Spirochaetes)、纤维杆菌门(Fibrobacteres)和变形菌门(Proteobacteria)为优势菌门,他们的比例均占序列总数的1%以上,其中,硬壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)平均占序列总数的42.22%(39.02% ~46.49%)和19.86%(17.65% ~21.42%)。无壁菌门 (Tenericutes)、蓝细菌门(Cyanobacteria)、酸杆菌门(Actinobacteria)、TM7、疣微菌门(Verrucomicrobia)和 SR1、梭杆菌门(Fusobacteria)、黏胶球形菌门(Lentisphaerae)、浮霉菌门(Planctomycetes)、互养菌门(Synergistetes)和广古菌门(Euryarchaeota)序列含量都低于1%。
由表4可知,门水平下,相对含量大于0.1%的门共有11个,其余5个门含量均低于0.1%,丰度较低。此外,还有平均3.23%(1.22%~6.68%)的序列无法进行归类。统计结果表明,各牧草组均以硬壁菌门 (Firmicutes)和拟杆菌门(Bacteroidetes)比例最高,其中硬壁菌门(Firmicutes)在苜蓿、燕麦草、羊草和稻草中的比例分别占序列总数的46.49%、40.34%、39.02%和43.03%,拟杆菌门 (Bacteroidetes)在上述四组牧草中分别占20.69%、17.65%、21.42%和19.68%,不同牧草中硬壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)比例存在极显著差异 (P<0.01)。剩余的9个门中,除了 TM7、SR1 和疣微菌门(Verrucomicrobia)各牧草组间无显著差异外(P>0.05),其余均存在极显著差异(P<0.01)。由此可见,瘤胃中降解不同牧草的细菌群落结构存在明显差异。
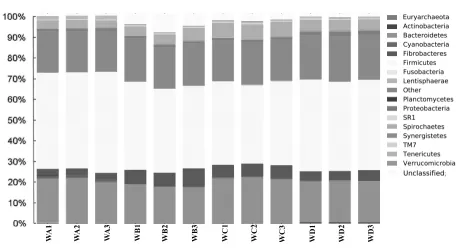
图3 基于门水平12个牧草样品中细菌和古细菌相对含量(序列所占比例)

表4 基于门水平相对含量大于0.1%(序列所占比例)的不同牧草比较分析
3 讨论
瘤胃中细菌和真菌可以分泌多种活性很高的纤维降解酶,其占纤维酶活力的80%以上(Dijkstra等,1995)。尽管真菌的穿透力和降解能力更强,且可以溶解部分木质素,但真菌在瘤胃微生物中的含量较低(占8%),因而对纤维降解的贡献低于细菌,细菌由于数量上的绝对优势,使其在纤维降解过程中起到关键作用。瘤胃中细菌可分为瘤胃液中的游离细菌,与饲料颗粒紧密吸附的细菌和附着于瘤胃壁上的细菌(McAllister等,1994;Czerkawski等,1988)。瘤胃中88% ~91%的葡糖糖内切酶和木聚糖酶,70%的淀粉酶和75%的蛋白酶活通过饲料颗粒中紧密吸附的微生物分泌(Minato 等,1993),因此,与牧草茎秆紧密吸附的微生物才是降解纤维的关键菌群 (Miron等,2001),他们占瘤胃中纤维分解菌数量的70%~80%(Craig 等,1987)。
通过对测序数据的分析发现,在当前测序量前提下,不同牧草样品的稀释曲线仍不能完全进入平台期(图1),上述结果表明,增加测序量仍然可能发现新的种系型,但本试验测序量基本可以反映紧密吸附在牧草纤维上细菌的群落结构多样性。四组牧草中共产生8997个OTU,燕麦草获得的OTU数目最高,羊草最低,在97%相似性水平下丰富度指数Chao和Ace也是燕麦草最高,羊草最低,这可能是因为燕麦草优质序列数更高,降解率远远高于同为禾本科的羊草,且附着在燕麦草和羊草中的细菌数量不同。
序列比对后发现,牧草吸附的细菌中硬壁菌门和拟杆菌门平均占序列总数的42.22%和19.86%,这与奶牛瘤胃液中两菌门也是优势菌群相一致(Jami等,2012)。牧草茎秆中主要成分是纤维素,而硬壁菌门含有大量分解纤维的菌属,如丁酸弧菌属和瘤胃球菌属,这充分说明了硬壁菌门为何在牧草紧密吸附的菌群中占据绝对优势。
4 结论
本试验通过高通量测序法研究了吸附在牧草细胞壁降解菌的群落结构,结果表明:(1)吸附于不同牧草细胞壁中的细菌群落结构丰富度:燕麦草>苜蓿>稻草>羊草;(2)四组牧草样品共鉴定得到16个门,27个纲,37个目,63个科和91个属。门水平上,硬壁菌门、拟杆菌门、螺旋体门、纤维杆菌门和变形菌门为优势菌门。综上:不同牧草吸附的细菌种类和数量存在显著差异。

