角木叶鲽的群体遗传多样性研究和形态学分析❋
宋垚萱, 高天翔, 杨天燕, 韩志强, 宋 娜❋ ❋
(1. 中国海洋大学水产学院,山东 青岛 266003; 2. 浙江海洋大学水产学院,浙江 舟山 316022)
角木叶鲽的群体遗传多样性研究和形态学分析❋
宋垚萱1, 高天翔2, 杨天燕2, 韩志强2, 宋 娜1❋ ❋
(1. 中国海洋大学水产学院,山东 青岛 266003; 2. 浙江海洋大学水产学院,浙江 舟山 316022)
角木叶鲽(Pleuronichthyscornutus)是中国近海重要的经济鱼类,本研究基于线粒体DNA控制区第一高变区部分序列,对采自青岛和舟山近海的角木叶鲽群体进行遗传多样性研究。线粒体DNA控制区序列分析显示:舟山和青岛两群体均呈现较高的单倍型多样度和较低的核苷酸多样度;单倍型邻接关系树与最小跨度树均显示青岛、舟山两个群体个体相互混杂,无显著的分支;群体间遗传分化指数(FST=0.027 6;P=0.030 3)揭示了两群体之间存在微弱的遗传分化;TaJima’sD中性检验和贝叶斯天际线分析的结果显示角木叶鲽群体可能经历过近期扩张事件。基础形态学特征测量结果表明,舟山群体的可量性状比值,如体长/尾柄高、体长/头长、体长/有(无)眼侧胸鳍长等均略高于青岛群体,其中体长/有(无)眼侧胸鳍长差异显著。
角木叶鲽;线粒体DNA控制区;遗传多样性;形态学;青岛群体;舟山群体
角木叶鲽(Pleuronichthyscornutus)隶属鲽形目(Pleuronectiformes)鲽科(Pleuronectidea)木叶鲽属(Pleuronichthys),是暖温性近岸底层鱼类。在我国,角木叶鲽主要分布在渤海、黄海和东海海域,在日本和朝鲜的沿海也较为常见。角木叶鲽营养价值较高,是食用性的海洋经济鱼类[1]。历史上,角木叶鲽是我国沿海渔民的重要渔获对象,但近些年因受海水污染和富营养化、过度捕捞(过密渔具使用)及产卵场遭到破坏等影响[2],野生角木叶鲽的资源正在迅速的衰退。
鱼类群体遗传多样性的研究是评估种质资源现状、制定野生群体保护策略的重要前提,并作为管理野生渔业和其他海洋生物的辅助工具达50年[3]。为了合理保护与利用角木叶鲽这一重要的渔业资源,开展对角木叶鲽的种质资源及遗传多样性现状的研究尤为重要。分子生物技术的发展为揭示海洋物种的系统进化关系、探讨种下水平的微进化研究提供技术支持[4]。其中,分子标记是检测物种遗传多样性和遗传结构的有效方法[5-6]。线粒体DNA序列作为核外遗传物质,具有结构简单、基因排列紧凑和母系遗传等特点,在过去的十几年中已经被广泛应用于动物分子系统发育和群体遗传学的研究[7-10]。而线粒体DNA控制区具有很高的核苷酸替换速率,其进化速率约是线粒体DNA蛋白质编码基因的2~5倍[11],更适合作为海洋鱼类群体遗传学研究的分子标记[12]。
目前关于鲽类的形态与地理分布等方面的研究较多,如圆斑星鲽(Veraspervariegatus)[13]、钝吻黄盖鲽(Pleuronectesyokohamae)[14]、星斑川鲽(Platichthysstellatus)[15]等,高眼鲽(Cleisthenesherzensteini)[16]、角木叶鲽[17]等遗传多样性方面研究也有的相关报道。本研究基于mtDNA控制区第一高变区序列对角木叶鲽两个群体的遗传多样性、遗传结构和历史动态进行了研究,以丰富这一经济物种的相关遗传学资料,为其资源合理利用和管理提供理论依据。
1 材料与方法
1.1 样品采集、形态测定与样品保存
本研究所用角木叶鲽样品分别采自浙江舟山和山东青岛近海,各30尾。经形态学鉴定以后,对其可数性状直接计数。用直尺(精确到0.1 cm)测量可量性状体宽、体长、全长、头长、尾柄长、尾柄高、有眼侧胸鳍长、无眼侧胸鳍长、尾鳍长;用游标卡尺(精确到0.01cm)测量眼径、眼间距、上颌长。用电子天平(精确到0.1g)测量样品重量。将2个群体的10个形态指标比值变量(A-I)进行T检验分析。测量方法按照Meng等[18]的方法进行,数据分析在Excel中完成。传统形态测量实验完成以后,取背部肌肉于95%酒精中保存备用。
1.2 DNA提取和目的基因扩增
取角木叶鲽背部肌肉组织,采用标准酚—氯仿方法[19]提取基因组DNA,4℃保存备用。角木叶鲽控制区高变区序列扩增引物为CR-s:5’-CCCACCACTAACTCCCAAAGC-3’和CR-r:5’-CTGGAAAGAACGCCCGGCATG-3’。PCR反应在热循环仪(Biometra)上进行,PCR反应总体积为25.0 μL,其中:17.5 μL双蒸水,2.5 μL10×Buffer,2 μL dNTP,0.15 μLTaq酶,正反向引物各1 μL和1 μL模板DNA。PCR反应程序为:94℃预变性5 min,94 ℃变性45 s,49 ℃退火45 s,72 ℃延伸45 s,35个循环,最后72℃延伸10 min,4℃保存。同时设阴性对照以排除DNA模板被污染的情况。取1~2 μL PCR扩增产物进行1%琼脂糖凝胶电泳检测。将检测合格的PCR产物送至英潍捷基有限公司切胶纯化,并在自动测序仪(ABI3730DNA)上进行双向测序。
1.3 目的基因序列分析
利用DNAStar(DNASTAR,Inc)软件对已获取的角木叶鲽两群体的序列进行比对,并根据序列峰图辅以人工校正;2个群体的碱基组成、变异位点、转换/颠换值、单倍型多样度性等相关参数由Arlequin3.0[20]软件分析;利用J Modeltest[21]软件计算得到适合角木叶鲽控制区片段的核苷酸最佳替换模型;以同属的长木叶鲽为外群,采用邻接法(Neighbor-Joining; NJ)[22]重建单倍型序列邻接关系树,其中NJ分析利用MEGA4.0[23]软件采用1 000次自展分析检验各节点的置信度;此外,由Arlequin3.0软件输出单倍型之间的关系,并根据结果构建最小跨度树(Minimum spanning tree; MST)分析种内单倍型之间关系。采用Tajima’sD和Fu’sFs检验分析种群历史动态,检验中性假说是否成立,同时利用BEAST.v2.4.3[24]软件包和Tracer v1.6[25]构建贝叶斯天际线(Bayesian skyline plot; BSP)检测群体历史群体大小变动,在Tracer中查看结果时,若所有值均≥200,则直接构建贝叶斯天际线,若有部分均值<200,再根据均值,输入重新运算MCMC链的次数,剔除2次结果中前25%的数据再整合为一,重新获取BSP。
2 结果与分析
测量60尾标本,体长范围为96~121 mm,体重范围为24.1~60.6 g。角木叶鲽体呈卵圆形,侧扁。眼在身体右侧,高凸,前缘有骨突。两眼间隔窄,前后有棘。有眼侧体褐色,通常具有不规则类似云状分布的黑色斑点;无眼侧体为白色。侧线直线形,颞上枝长通常不具有向前的小分枝。尾鳍截形。口小,上颌达眼前缘。本研究中,鱼体体长为体宽的1.66~2.25倍,角木叶鲽可量与可数性状特征分别见表1和2。
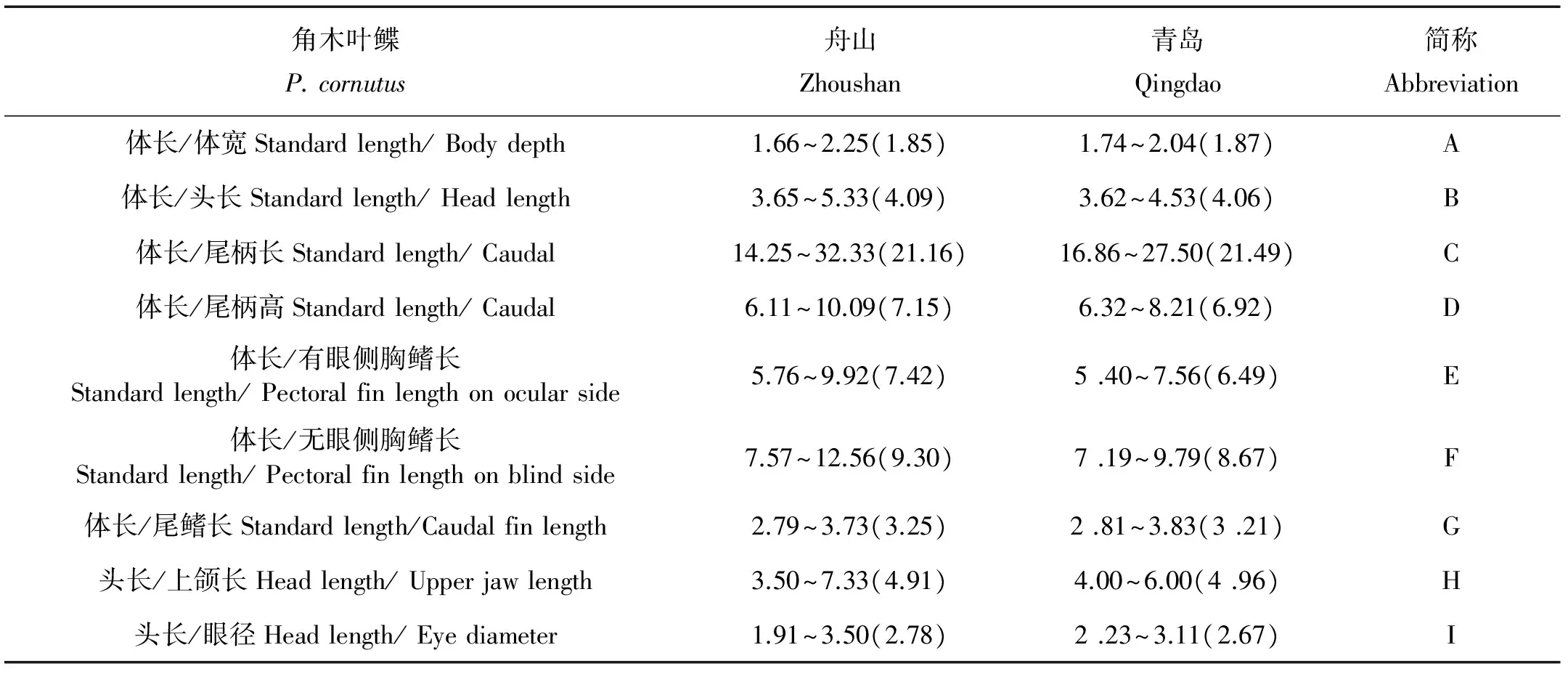
表1 角木叶鲽2个群体的可量性状特征比较Table 1 Proportional measurements of two P. cornustus populations
注:括号内为均值,下同。
Note:Mean values in the brackets, the same below.

表2 角木叶鲽2个群体的可数性状特征比较Table 2 Count measurements of two P.cornutus populations
Note:①P.cornutus;②Zhoushan;③Qingdao;④Dorsal fin rays;⑤Anal fin rays;⑥Caudal-fin rays;⑦Vertebrae;⑧Pectoral-fin rays on ocular side;⑨Pectoral-fin rays on blind side;⑩Pelvic-fin rays on ocular side;Pelvic-fin rays on blind side
对角木叶鲽2个群体9个形态特征比值进行单因素方差分析,对不具有方差齐性的变量采用T检验法进行分析。P=0.05显著性水平下的单因素方差分析结果如表3所示,2个群体在2个变量(E,F)上存在显著性差异。
对舟山和青岛2个群体共60尾样本线粒体控制区进行PCR扩增,得到目的片段长度为504 bp的序列,其中包括18 bp的tRNAPro序列。在tRNAPro序列并未检测到变异位点,去掉tRNAPro序列,60个个体共获得486 bp的控制区的第一高变区部分序列。在线粒体DNA控制区的同源片段上,角木叶鲽舟山群体中共检测到20个变异位点,包括转换16个,颠换4个,共定义20个单倍型;青岛群体中共检测到14个变异位点,包括转换12个,颠换2个,共定义了22个单倍型。青岛和舟山2个群体的单倍型多样度、核苷酸多样度以及两两序列比较的平均碱基数均不存在明显的差异(见表4)。2个群体在碱基含量组成上,均为A(35.39;35.50)>T(30.22;30.23)>C(18.57;18.50)>G(15.82;15.76),呈现出较高的AT含量(65.61;65.75)和明显的反G偏倚现象,这与大多数鱼类一致[26]。通过J Modeltest软件计算得到适合角木叶鲽控制区片段的核苷酸最佳替换模型为Tamura-Nei模型。

表3 角木叶鲽群体T检验分析结果(均值±标准差,P=0.05)Table 3 The results of T-test between P. cornutus populations (Mean±S. E., P=0.05)
注:*:P<0.05.
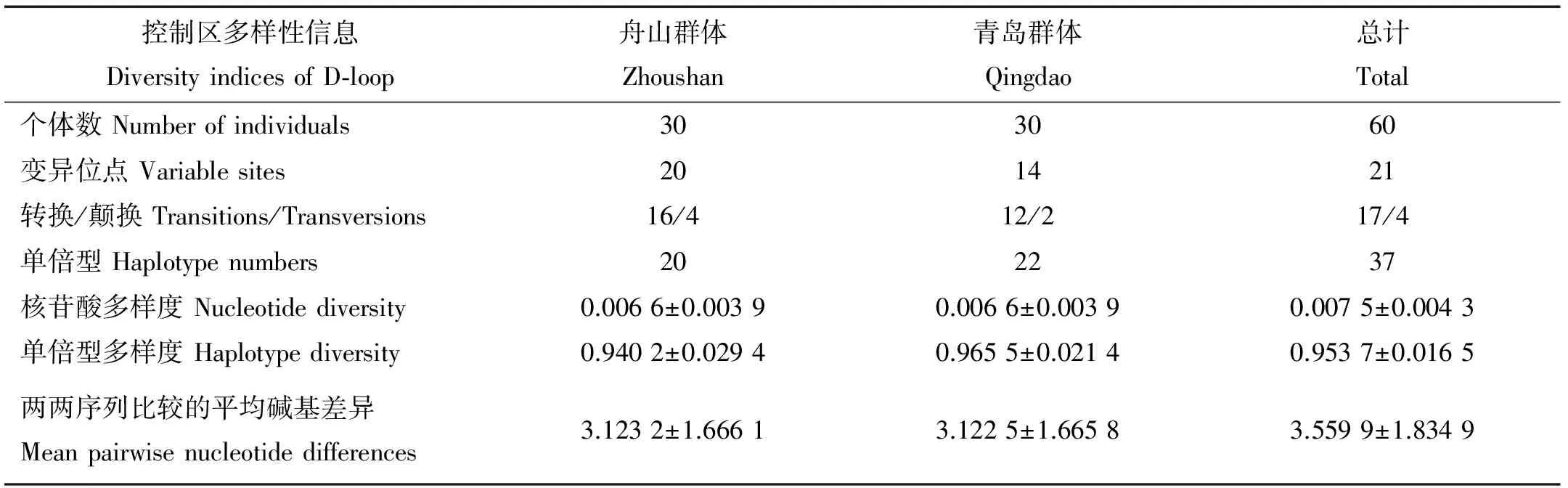
表4 本研究中角木叶鲽群体遗传多样性信息Table 4 Molecular diversity indices for P. cornutus in this study
以长木叶鲽(P.japonicus)(Genbank:KY038655)为外群,构建角木叶鲽舟山和青岛群体控制区单倍型序列的邻接关系树,可以看出舟山与青岛两群体个体相互混杂地分布在单倍型的邻接关系树上,没有检测到与采样地点相对应的分支(见图1)。同样,两群体的单倍型都广泛地分布在单倍型的邻接关系树上且有5个共享单倍型,未检测到明显的谱系结构(见图2)。遗传分化指数FST=0.027 6(P=0.030 3)表明角木叶鲽的舟山群体和青岛群体之间存在微弱而显著的遗传分化。
角木叶鲽的群体历史动态分析显示,TaJima’sD检验结果(D= -0.49,P>0.05)并未显著偏离中性,但是Fu’s检验结果(Fs=-26.11,P<0.01)却是极显著的,这可能是由于Fu’s检验对近期群体扩张更为敏感所致。贝叶斯天际线分析(见图3)表明角木叶鲽群体经历了有效群体大小增加的过程,约在12.5万年前增速较快,约在10万年前趋于平稳。
3 讨论
为了解栖息环境对鱼类生长发育的作用及影响,一种主要的方法是定期观察鱼类的形态并结合统计学进行评估分析。鱼类生长发育状态的监测是保护野生角木叶鲽种质资源的重要方面。形态测量数据结果显示,角木叶鲽2个地理群体之间在体长/有眼侧胸鳍长和体长/无眼侧胸鳍长2个可量性状比值上存在显著差异。胸鳍形态变化是生态学适应的结果,因此推测差异可能是为适应复杂的外界环境形成。

(分支上的数字为1 000次重抽样分析得到的支持率。Bootstrap supports by 1 000 replicates are shown.)
图1 控制区单倍型序列邻接关系树
Fig.1 Neighbor-joining tree for control region haplotypes
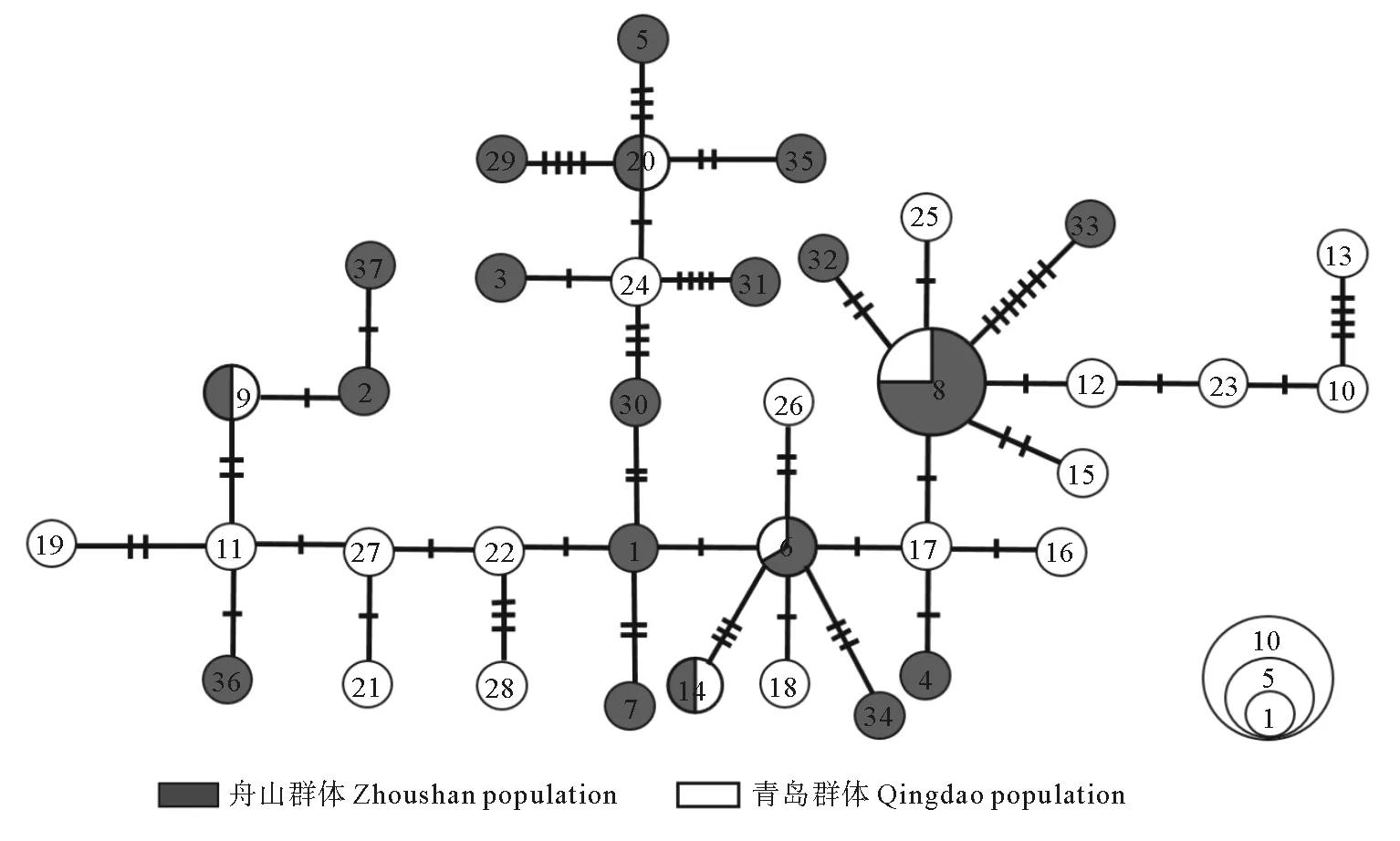
(圆圈面积与单倍型频率成正比,短划线代表单倍型间的核苷酸替换数目,数字表示对应的单倍型。The sizes of circles are proportional to haplotype frequency. Perpendicular tick marks on the lines joining haplotypes represent the number of nucleotide substitutions and the numbers in circles represent haplotypes.)
图2 角木叶鲽控制区单倍型最小跨度树
Fig.2 Unrooted minimum spanning trees for control region haplotypes inP.cornutus

(深色线条代表有效群体大小的中值,浅色线条代表有效群体大小95%置信区间的上限与下限。Dark lines represent median estimates ofNeT; light lines are the upper and lower 95% highest posterior density (HPD) limits ofNeT.)
图3 本研究中角木叶鲽群体的贝叶斯天际线分析
Fig.3 Bayesian skyline analysis ofP.cornutusin this study
物种的遗传多样性是生物适应生存和发展的前提,是在漫长进化过程中不断累积的产物[27]。物种遗传多样性越高,适应生存环境变化的能力就越强[28],反之亦然。群体遗传结构的背景知识对管理经济鱼类渔业具有非常重要的意义。以线粒体DNA为基础,可以分析物种的进化潜力,研究物种的进化史,并提供预测物种发展动向的依据[29]。本研究中角木叶鲽2个群体共检测到37个单倍型,单倍型多样度为0.95,核苷酸多样性为0.007 5,表现出丰富的遗传多样性。这也符合Bowen等人在其研究中提出的鱼类较高的单倍型多样性和较低的核苷酸多样性的类型[30]。推测随着在角木叶鲽群体扩张中数量不断的增加,单倍型多样度也会有所提高,但短期内积累核苷酸产生的变异较少,因此会造成核苷酸多样度较低而单倍型多样度较高的遗传多样性模式,中性检验的结果也为这种推测提供了依据。太平洋油鲽也存在类似的遗传模式[31]。其次,两群体的群体独享单倍型比率较高,影响群体间的遗传差异。
更新世时期气候的剧烈变化使得许多物种的分布范围经历了收缩和扩张,极大地影响着种内遗传多样性的数量和分布[32]。本研究中角木叶鲽群体的贝叶斯天际线结果显示其有效群体大小趋于平缓的时间约在10万年前,处于更新世晚期。这一时期海平面多次大幅度的升降,这些气候和环境的变迁对海水温度、海流模式和海岸栖息地的影响都比较大,末次冰期海平面上升,冰期形成的地理隔离被打破,随着群体之间基因交流的增强导致的物种遗传同质化的影响大于长期冰期隔离效应导致的群体间显著遗传差异的影响,这可能是导致群体未出现明显谱系结构的原因,朱叶等人的研究结果[17]也支持这一假设。
此外,生活史特征也是影响海洋鱼类群体遗传格局不可或缺因素。由于海洋中存在不明显的阻隔扩散障碍,并且海洋鱼类在其生活史的各个阶段都具有较强的扩散潜力,因此海洋鱼类的群体遗传分化通常相对较低[33]。本研究中舟山和青岛2群体之间的遗传分化指数(FST=0.027 6;P=0.030 3±0.006 9)表明群体之间存在微小而显著的遗传分化,这一结果与朱叶等人的研究结果[17]有所不同,这可能因为样本数量造成的差异。角木叶鲽成鱼营底栖沙居生活,近海繁殖,产浮性卵,仔鱼初期营漂浮性生活[34],这与鲽科中其他种类如高眼鲽、长鲽等[16]类似,其幼体可能借助洋流扩散,使不同地理群体间产生基因交流。
4 结语
本研究从形态学和分子标记2种方法对舟山、青岛的角木叶鲽群体遗传多样性进行分析。2个群体在体长/有眼侧胸鳍长和体长/无眼侧胸鳍长2个可量性状有显著的差异;控制区第一高变区序列的分析的结果表明2个群体之间存在微弱而显著的遗传分化。更新世冰期剧烈的气候变化可能对角木叶鲽的群体历史动态产生巨大影响,同时其卵和幼鱼的浮游生活史可能促进了现有不同地理群体间的基因交流,从而形成了角木叶鲽现有的系统地理格局。综上,本文丰富了中国近海角木叶鲽群体遗传多样性研究的结果,并综合传统的形态资料为其种群结构和遗传变异研究提供更充分的基础资料,从而更好地保护与利用角木叶鲽野生资源。
[1] Nelson J S. Fish of the World[M]. (4th ed). New York: John Wiley & Sons. Inc., 2006: 442-452.
[2] Wang Q Y, Qian R, Zhao J P, et al. To promote marine ecology research and civilization[J]. Hebei Fish, 2013, 12: 59-61.
[3] Ovenden J R, Berry O, Welch D J, et al. Ocean’s eleven: a critical evaluation of the role of population, evolutionary and molecular genetics in the management of wild fisheries[J]. Fish & Fisheries, 2013, 16(1): 125-159.
[4] Xiao Y S, Zhang Y, Gao T X. Study of the relationship between GeneraKareiusbicoloratusandPlatichthysstellatusby mtDNA sequence[J]. J Ocean Univ China, 2010, 40(6): 69-76.
[5] Luo K, Lu H X, Wu Z D, et al. Genetic Diversity and population structure analysis of main sweet potato breeding parents in southwest China[J]. Sci Agricul Sin, 2016, 49(3): 593-608.
[6] Ma Z J. Research progress on molecular genetics of the yak chromosome[J]. J China Agricul Univ, 2016, 21(2): 93-99.
[7] Li D H, Shi W, Gong L, et al. The complete mitochondrial genome ofParaplagusiablochii(Pleuronectiformes: Cynoglossidae)[J]. Mitochondr DNA Part A, 2016, 27(1): 92-93.
[8] Tsukagoshi H, Takeda K, Kariya T, et al. Genetic variation and population structure of marbled solePleuronectesyokohamaeand cresthead flounderP.schrenkiin Japan inferred from mitochondrial DNA analysis[J]. Biochem Syst Ecol, 2015, 58: 274-280.
[9] Li Y, Shi Y, Lu J, et al. Sequence and phylogenetic analysis of the complete mitochondrial genome ofLasiopodomysmandarinusmandarinus(Arvicolinae,Rodentia)[J]. Gene, 2016, 593(2): 302-307.
[10] Yoshida R, Nei M. Efficiencies of the NJp, maximum likelihood, and Bayesian methods of phylogenetic construction for compositional and noncompositional Genes[J]. Mol Biol Evol, 2016, 33(6): 1618-1624.
[11] Cann R L, Brown W M, Wilson A C. Polymorphic sites and the mechanism of evolution in human mitochondrial DNA[J]. Genetics, 1984, 106(3): 479-499.
[12] Coscia I, Chopelet J, Waples R S, et al. Sex change and effective population size: implications for population genetic studies in marine fish[J]. Heredity, 2016, 117(4): 251-258.
[13] Chen S Q, Yu D X, Ma A J, et al. Structure and evolution of complete mitochondrial genome of spotted halibutVeraspervariegatus[J]. J Fish China, 2002, 17(10): 25-27.
[14] Zhang Y, Xiao Y S, Gao T X, et al. Comparative analysis of mtDNA gene sequences between two species of Pleuronectes[J]. J Fish China, 2010, 31(5): 15-21.
[15] Wang B, Sun P, Fang H H, et al. Morphological characteristics and parameters measurement of starry flounder (PlatichthysstellatusPallas)[J]. Acta Oceanol Sin, 2010, 32(2): 139-147.
[16] 肖永双. 西北太平洋五种海洋鱼类的分子系统地理学研究[D]. 青岛:中国海洋大学, 2010.
Xiao Y S. Molecular Phylogeography of Six Marine Fishes in the Northwestern Pacific[D]. Qingdao:Ocean University of China, 2010.
[17] Zhu Y, Zhang Q, Li G S, et al. Genetic diversity of fourPleuronichthyscornutuspopulations in coastal waters of China[J]. Mar Sci Bull, 2012, 31(5): 522-556.
[18] 孟庆闻,苏锦祥,缪学祖.鱼类分类学[M].北京:中国农业出版社,1995:962-970.
Meng Q W, Su J X, Miu X Z. Taxonomy of Fishes[M]. Beijing: China Agriculture Press, 1995: 962-970.
[19] Sambrook J, Fritsch E F, Maniatis T. Molecular Cloning[M]. New York: Cold Spring Harbor Laboratory Press, 1989.
[20] Schneider S, Roessli D, Excoffier L. Arlequin: a software for population genetics data analysis[J]. User Manual Ver, 2000, 2: 2496-2497.
[21] Darriba D, Taboada G L, Doallo R, et al. jModelTest 2: more models, new heuristics and parallel computing[J]. Nat Methods, 2012, 9(8): 772.
[22] Nei M, Kumar S. Molecular Evolution and Phylogenetics[M]. Oxford: Univ Press, 2000.
[23] Tamura K, Dudley J, Nei M, et al. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4. 0[J]. Mol Biol Evol, 2007, 24(8): 1596-1599.
[24] Bouckaert R, Heled J, Kühnert D, et al. BEAST 2: a software platform for bayesian evolutionary analysis[J]. Plos Comput Biol, 2014, 10(4): 1003537.
[25] Rambaut A, Drummond A J. Tracer v1. 5. [Z/OL]. [2009-11-30] (2017-02-27). http: //tree. bio. ed. ac. uk/software/tracer/.
[26] Hochachka P W,Mommsen T. Biochemistry and Molecular Biology of Fishes:Environmental and Ecological Biochemistry[M]. [s.1.]: Elsevier Sci, 1995, 2: 1-38.
[27] Soltis P S, Soltis D E. Genetic variation in endemic and widespread plant species: Examples from Saxifragageae and Polystichum (Dryopteridaceae)[J]. Aliso, 1991, 13(1): 215-223.
[28] Huenneke L F. Ecological implications of genetic variation in plant populations[M].//Genetics and Conservation of Rare Plants. New York: Oxford Univ Press, 1991: 31-44.
[29] Chen S H, Qu Y J, Li J. Mitochondrial DNA and its progresses in Fish[J]. Biotechnol Bull, 2011, 3: 13-20.
[30] Grant W A S, Bowen B W. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation[J]. J Hered, 1998, 89(5): 415-426.
[31] Stepien C A. Phylogeographical structure of the Dover soleMicrostomuspacificus: The larval retention hypothesis and genetic divergence along the deep continental slope of the northeastern Pacific Ocean[J]. Mol Ecol, 1999, 8(6): 923-939.
[32] Ren J. Late Pleistocene paleoceanography of the Subarctic Pacific derived from the diatom record[D]. Bremen: University of Bremen, 2015.
[33] Ashe J L, Feldheim K A, Fields A T, et al. Local population structure and context-dependent isolation by distance in a large coastal shark[J]. Mar Ecol-Prog Ser, 2015, 520: 203-216.
[34] 时伟. 应用线粒体全序列研究鲽形目鱼类的分子系统关系及演化[D]. 青岛:中国海洋大学, 2011.
Shi W. Molecular Phylogenetic Analysis of the Order Pleuronectiformes (Teleostei) Based on Complete Mitochondrial DNA Sequences[D]. Qingdao:Ocean University of China, 2011.
ComparativeAnalysisofGeneticDiversityandMorphologyofPleuronichthyscornutusPopulations
SONG Yao-Xuan1, GAO Tian-Xiang2, YANG Tian-Yan2, HAN Zhi-Qiang2, SONG Na1
(1. College of Fisheries, Ocean University of China, Qingdao 266003, China; 2. College of Fishery, Zhejiang Ocean University, Zhoushan 316022, China)
Pleuronichthyscornutusis a commercially important species, and studies on its genetic diversity will exclusively provide the research basis for fishery management board. Mitochondrial DNA control sequence analysis showed that high haplotype diversity and low level of nucleotide diversity existed in Zhoushan and Qingdao populations. From the neighbor-joining tree and the minimum spanning tree constructed based on haplotypes, we observed that the individuals from two populations were mixed with each other and there was no obvious correspondence between the branches. The pairwise fixation index (FST=0.027 6;P=0.030 3) revealed a weak but significant genetic differentiation between two populations. The neutrality tests and Bayesian skyline analysis showed thatP.cornutusmay have experienced a historical sudden and special population expansion. Morphological studies revealed that some proportional measurements of Zhoushan population were slightly higher than those of Qingdao population, such as standard length/caudal, standard length/head length, and standard length/pectoral fin length on blind side. The standard length/pectoral fin length on blind (ocular) side existed conspicuous differentiation between Zhoushan and Qingdao populations. These results provided information for the genetic resource protection and heritable variation study of this species.
Pleuronichthyscornutus; mitochondrial DNA control region; genetic diversity; morphology; Qingdao population; Zhoushan population
S931.3;Q38
A
1672-5174(2018)02-049-07
10.16441/j.cnki.hdxb.20170084
宋垚萱, 高天翔, 杨天燕, 等. 角木叶鲽的群体遗传多样性研究和形态学分析[J]. 中国海洋大学学报(自然科学版), 2018, 48(2): 49-55.
SONG Yao-Xuan, GAO Tian-Xiang, YANG Tian-Yan, et al. Comparative analysis of genetic diversity and morphology ofPleuronichthyscornutuspopulations[J]. Periodical of Ocean University of China, 2018, 48(2): 49-55.
海洋公益性行业科研专项项目(201305043;201405010)资助
Supported by the Public Science and Technology Research Funds Projects of Ocean (201305043;201405010)
2017-02-27;
2017-05-20
宋垚萱(1992-),女,硕士生。E-mail:song871570@163.com
❋ ❋ 通讯作者:E-mail:songna624@163.com
责任编辑 朱宝象

