罗非鱼“粤闽1号”及其繁育群体的遗传多样性和遗传关系分析*
刘志刚 卢迈新 曹建萌 高风英
罗非鱼“粤闽1号”及其繁育群体的遗传多样性和遗传关系分析*
刘志刚 卢迈新①曹建萌 高风英
(中国水产科学研究院珠江水产研究所 农业农村部热带亚热带水产资源利用与养殖重点实验室 广州 510380)
罗非鱼“粤闽1号”是用尼罗罗非鱼()和奥利亚罗非鱼()培育的全雄罗非鱼新品种。采用同工酶电泳技术和线粒体DNA控制区序列分析方法,对罗非鱼“粤闽1号”及其繁育群体的遗传多样性、遗传结构及群体之间的遗传关系进行研究。同工酶电泳结果显示,酯酶(EST)和乳酸脱氢酶(LDH)的酶谱具有组织和群体特异性。肌肉和脾脏中的EST表达量极低,而肝脏中的EST表达量较高,LDH在3种组织中均有较高表达,但酶谱存在差异。在罗非鱼“粤闽1号”及其繁育群体中,共检测到10条EST酶带和5条LDH酶带,多态性位点比例()为12.50%~71.43%,平均观测杂合度(o)为0.0417~0.6143,Hardy-Weinberg遗传偏离指数()为‒0.2347~0.9072。线粒体DNA控制区序列分析结果显示,mtDNA D-loop区的碱基组成无显著差异,均呈现出A+T碱基偏向性(63.39%)。在罗非鱼“粤闽1号”及其繁育群体中,共发现20种单倍型,核苷酸多样性指数(P)为0~0.0519,奥尼罗非鱼雌鱼(WY1)、尼罗罗非鱼雌鱼(XX)、尼罗罗非鱼雄鱼(XY)和罗非鱼“粤闽1号”(XY2)群体的P值(0.0440~0.0519)明显高于超雄尼罗罗非鱼(YY1)、超雄奥尼罗非鱼(YY2)和奥利亚罗非鱼雌鱼(WZ)群体的P值(0~0.0009)。各群体之间的遗传分化指数(ST)为‒0.0115~0.9963,XY2与WY1群体之间、XX与XY群体之间以及WZ和YY2群体之间的遗传分化不显著(ST<0.05,>0.05)。XY2群体与尼罗罗非鱼群体(XX群体和XY群体)和奥利亚罗非鱼群体(WZ群体)之间的平均遗传距离分别为0.0639和0.0695。在NJ系统进化树中,XX、XY、YY1和XY2群体聚为一支,WY1、WZ和YY2群体聚为另一支。本研究揭示了罗非鱼“粤闽1号”的生化和分子遗传特征,为其种质鉴定、繁育群体的构建和优良性状的稳定遗传提供理论基础。
罗非鱼“粤闽1号”;同工酶;线粒体DNA控制区;遗传多样性;遗传关系
罗非鱼“粤闽1号”为本研究团队与福建百汇盛源水产种业有限公司和西双版纳同益水产科技有限公司在漳州市和西双版纳育种基地历经7年(2010~ 2016年)培育出来的全雄罗非鱼新品种,该罗非鱼新品种以尼罗罗非鱼()和奥利亚罗非鱼()为基础群体,通过群体选育、家系选育、杂交育种和遗传全雄罗非鱼(Genetic male tilapia,)技术等复合育种技术培育而成,其培育技术路线见图1。近年来,罗非鱼“粤闽1号”分别在云南、广东、福建、广西和海南等省进行了池塘、网箱和水库中试养殖,结果显示,其具有雄性率高、生长快、抗病力强、出肉率高和饲料转化率高等特点。为了揭示其种质特征,有必要对罗非鱼“粤闽1号”及其繁育群体的遗传多样性和遗传关系进行研究。

图1 罗非鱼“粤闽1号”的培育技术路线
同工酶在生物界广泛存在,变异丰富,呈共显性遗传,由1个或多个基因座位编码,能比较客观地代表基因组的变异,可揭示生物种群的进化和亲缘关系,是一种生化表现型的遗传标志,且有实验条件简单、成本低、结果快速可靠等优点,被广泛应用于水产物种鉴定、种间亲缘关系和遗传进化分析(杨淞等, 2006; 张健东等, 2015; 丁金强等, 2013)。线粒体DNA(Mitochondrial DNA, mtDNA)是细胞核外具有自主复制、转录和翻译能力的环状双螺旋DNA,具有分子量小、结构简单、进化速度快、高度多态、母性遗传等特点,被广泛应用于鱼类种群的进化和遗传研究(Teletchea, 2009; 胡玉婷等, 2015)。mtDNA D-loop区是线粒体基因组中变异最大、进化速度最快的区域,具有较高的突变积累,适用于种内、种群或个体间遗传多样性的研究,也可用于种间遗传差异分析(吴长敬等, 2010; 颉晓勇等, 2014; 刘红等, 2016)。
本研究采用同工酶电泳技术和mtDNA控制区测序分析方法,对罗非鱼“粤闽1号”及其繁育群体进行遗传多样性、遗传结构和遗传关系分析,从而揭示罗非鱼“粤闽1号”的生化和分子遗传特征。
1 材料与方法
1.1 实验材料
罗非鱼“粤闽1号”及其繁育群体共7个罗非鱼群体的样品均采集于本研究团队与福建百汇盛源水产种业有限公司和西双版纳同益水产科技有限公司在漳州市和西双版纳的育种基地。尼罗罗非鱼雌鱼(XX)、尼罗罗非鱼雄鱼(XY)、奥利亚罗非鱼雌鱼(WZ)为以生长速度为群体选育指标的F5代,超雄尼罗罗非鱼(YY1)为F4代尼罗罗非鱼雄鱼(XY)与尼罗罗非鱼伪雌鱼(P-XY)交配后,通过性别标记筛选获得的群体。奥尼罗非鱼雌鱼(WY1)为超雄尼罗罗非鱼(YY1)与F5代奥利亚罗非鱼雌鱼(WZ)杂交后获得的雌鱼群体。超雄奥尼罗非鱼(YY2)为超雄尼罗罗非鱼(YY1)与奥尼罗非鱼雌鱼(WY1)交配后获得的雄鱼群体。罗非鱼“粤闽1号”(XY2)为F5代尼罗罗非鱼雌鱼(XX)与超雄奥尼罗非鱼(YY2)杂交后获得的子代,样品具体信息见表1。无菌解剖,取其肌肉、肝脏和脾脏,置于液氮中冻存,用于研磨后进行同工酶电泳分析;剪取其尾鳍,置于无水乙醇中固定,用于抽提DNA,进行mtDNA D-loop区序列分析。
表1 罗非鱼“粤闽1号”及其繁育群体样品采集信息

Tab.1 Sampling information of tilapia “Yuemin No.1” and its breeding populations
1.2 实验方法
1.2.1 同工酶电泳样品制备 将1.1中冻存的罗非鱼的肌肉、肝脏和脾脏置于0.8%的生理盐水中,洗涤2次,除去血污,用滤纸吸干后分别称取约0.25 g组织,置于无菌离心管中。按1∶6(重量g/体积ml)的比例加入预冷的0.1% mol/L磷酸盐缓冲液(pH= 7.0),采用组织匀浆器粉碎组织,置于低温离心机中,4℃下12000×g离心20 min,其中,肝脏需离心2次。吸取上清液,转移至新的离心管中,分装后置于 ‒80℃冰箱中冻存备用。
1.2.2 酯酶和乳酸脱氢酶电泳 采用Bio-Rad电泳仪和聚丙烯酰胺凝胶垂直电泳槽进行电泳,酯酶和乳酸脱氢酶电泳所用的非变性聚丙烯酰胺凝胶均为3.75%的浓缩胶和7.5%的分离胶,酯酶电泳采用EBT (pH=8.0)电泳缓冲体系,乳酸脱氢酶电泳采用TC (pH=8.0)缓冲体系,聚丙烯酰胺凝胶体系和电泳缓冲体系参照朱蓝菲(1992)和吴燕燕等(2008)的方法操作。样品中加入上样缓冲液,每个孔点15 μl,先在 4℃下100 V预电泳40 min,然后调高电压至200 V,电泳2~4 h。
1.2.3 同工酶染色、固定及酶谱分析 电泳完毕后,取下凝胶,加入染色液,置于37℃染色30 min,直至酶带清晰,酯酶染色液和乳酸脱氢酶染色液参照吴燕燕等(2008)的方法配制。染色后,用蒸馏水漂洗2遍,置于7%的醋酸溶液中固定,扫描,根据酶谱和相对迁移率绘制模式图,并参照李思发(1998)的方法对各酶带进行命名和遗传学分析。
多态位点比例=(/)×100%
平均杂合度的观测值o=1/∑o*
平均杂合度期望值e=1/∑(1‒∑i2)
位点有效等位基因数e=1/∑i
Hardy-Weinberg遗传偏离指数=(o‒e)/e
式中,为多态性位点数,为所测位点总数,o*=单个位点杂合子观测值/该位点观察个体总数,为第位等位基因的频率,i为第个位点的等位基因总数。
1.2.4 基因组DNA提取 采用动物基因组DNA提取试剂盒(Magen)提取罗非鱼尾鳍基因组DNA,操作步骤参照试剂盒说明书。采用1%琼脂糖凝胶电泳检测所提取DNA的完整性,用核酸蛋白定量仪检测其浓度。
1.2.5 mtDNA D-loop区扩增及测序 罗非鱼线粒体DNA D-loop区扩增引物和扩增条件参照杨洁等(2014),PCR产物经1%琼脂糖电泳检测后,由广州艾基生物有限公司进行PCR产物测序。
1.2.6 数据分析 采用Contig-Express软件对所测序列进行拼接,采用Bioedit软件对拼接序列进行校正,Blast分析表明,所获序列为罗非鱼mtDNA D-loop区目的片段。采用ClustalW软件对序列进行多重比对,采用DnaSP 5.0软件计算单倍型多样性(Haplotype diversity,)、核苷酸多样性(Nucleotide diversity,P)、平均核苷酸差异数(Average number of nucleotide differences,)和核苷酸多态位点数(Numbers of polymorphic sites,),采用Arlequin 3.5软件计算遗传分化指数(-statistics,ST)和基因流(m)。采用MEGA 5.0软件中的Kimura 2-Parameter程序计算群体间遗传距离,用邻接法(Neighbor-Joining, NJ)构建群体间的分子系统进化树。
2 结果
2.1 酯酶和乳酸脱氢酶酶谱分析
对罗非鱼“粤闽1号”及其繁育群体中6个罗非鱼群体的肌肉、脾脏和肝脏中的酯酶(EST)进行聚丙烯酰胺凝胶电泳分析。结果显示,肌肉和脾脏中EST的表达量极低,酶谱很浅,无法进行辨别;肝脏中的EST表达量较高,其酶谱和模式图见图2。在6个罗非鱼群体肝脏中,共检测到10条EST酶带,按迁移率的快慢分别命名为EST-1~EST-10。EST-6~EST-9酶带颜色较深,酶活力较强;EST-2、EST-3、EST-7 和EST-9酶带存在于所有罗非鱼群体中。YY2群体的酶带最多(8条),YY1和WZ群体的酶带最少 (5条)。罗非鱼“粤闽1号”的父本(YY2群体)的主带为EST-6、EST-7和EST-9,母本(XX群体)的主带为EST-7、EST-8和EST-9,罗非鱼“粤闽1号”(XY2群体)的主带为EST-6、EST-7、EST-8和EST-9,表现出杂合现象。尼罗罗非鱼群体XX和YY1中有EST-8主带,不含EST-6主带;奥利亚罗非鱼群体WZ与之相反;在奥尼杂交罗非鱼群体中,除了YY2群体,在WY1和XY2群体中,同时出现EST-6和EST-8酶带。
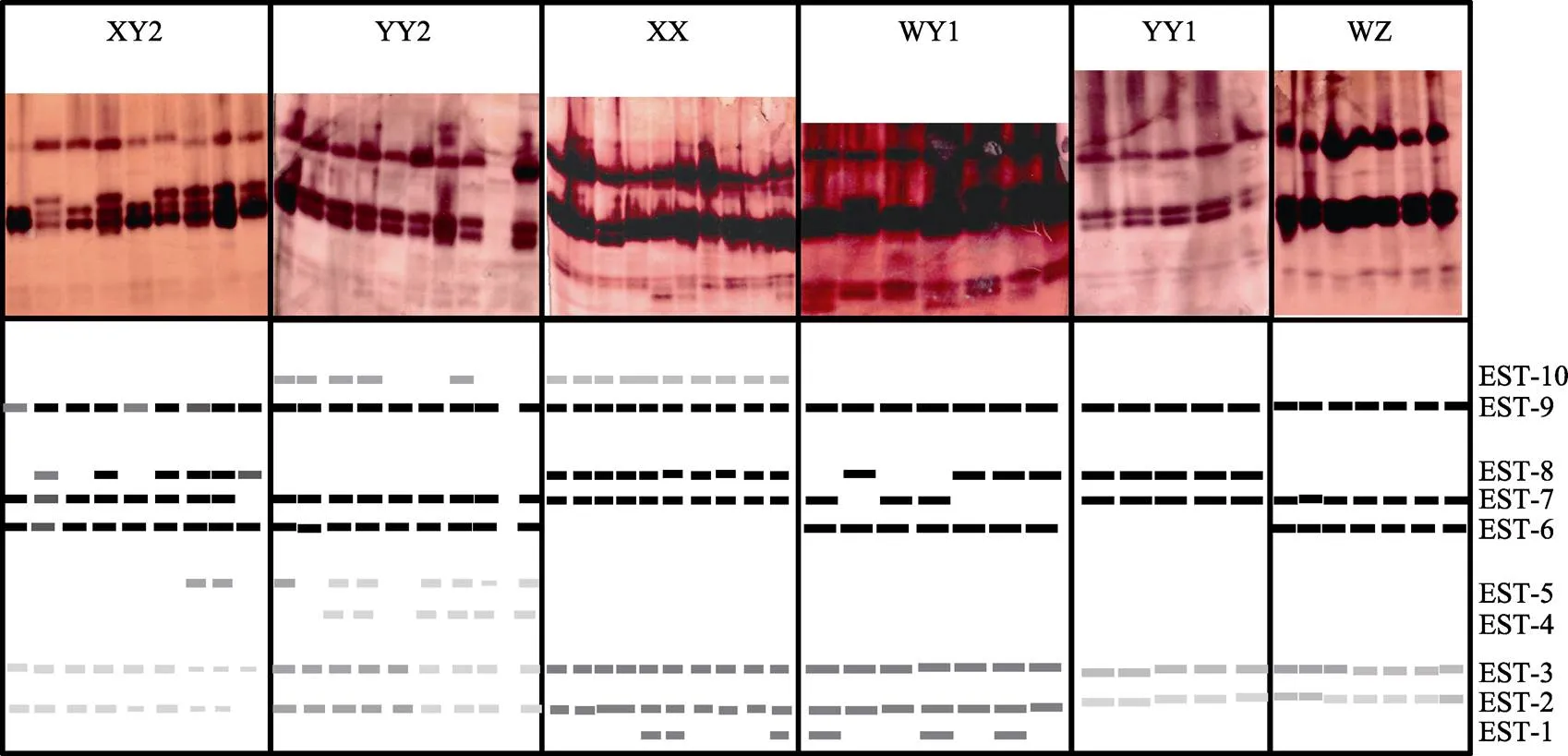
图2 罗非鱼“粤闽1号”及其繁育群体的肝脏中酯酶的电泳图谱和模式图
对6个罗非鱼群体的肌肉、脾脏和肝脏中的乳酸脱氢酶(LDH)进行电泳分析。结果显示,在3种组织中均能检测到清晰酶带,其酶谱和模式图如图3所示,共检测到5条酶带,根据其迁移率快慢分别命名为LDH-1~LDH-5。LDH的表达具有明显的组织特异性,在肌肉中检测到5条酶带(LDH-1~LDH-5),而在脾脏和肝脏中均检测到3条酶带(LDH-2、LDH-3和LDH-5);LDH-2酶带在所有组织中颜色均最深。 6个罗非鱼群体肌肉中均含有LDH-1和LDH-2酶带,WY1群体中含有LDH-3酶带,XX群体中含有LDH-3、LDH-4和LDH-5酶带。XY2、YY2和WZ群体脾脏中的LDH酶谱一致,均含有LDH-2、LDH-3和LDH-5酶带,XX和YY1群体脾脏中的LDH酶谱一致,均含有LDH-2和LDH-3酶带,WY1群体脾脏中只有LDH-2酶带。YY2和YY1群体的肝脏中只有LDH-2酶带,XY2和WZ群体的肝脏中只有LDH-2和LDH-5酶带,XX群体的肝脏中含LDH-2和LDH-3酶带,WY1群体的肝脏中含有LDH-2、LDH-3和LDH-5酶带,且具有个体差异性。
2.2 同工酶遗传结构分析
根据6个罗非鱼群体组织中酯酶和乳酸脱氢酶电泳酶谱,对其群体遗传结构参数进行分析。结果显示,不同群体的基因位点数存在差异(6~9个),XY2和YY2群体中基因位点数最多(9个);6个罗非鱼群体的平均有效等位基因数(A)为1.13~1.71;多态性位点比例()为12.50%~71.43%;平均期望杂合度(e)为0.0347~0.3221;平均观测杂合度(o)为0.0417~ 0.6143;上述4个遗传学参数均在XX群体中的值最大,在WZ群体中的值最小;6个罗非鱼群体中,哈迪-温伯格平衡偏离指数()为‒0.2347~0.9072,其中,WY1群体最小(‒0.2347),XX群体最大(0.9072)(表2)。

图3 罗非鱼“粤闽1号”及其繁育群体的不同组织中乳酸脱氢酶的电泳图谱和模式图
表2 基于酯酶和乳酸脱氢酶的遗传结构分析

Tab.2 Genetic structure analysis based on EST and LDH
2.3 mtDNA D-loop区序列变异和单倍型分析
通过PCR产物双向测序、拼接、比对和人工校正,获得罗非鱼“粤闽1号”及其繁育群体共7个群体的线粒体DNA控制区序列880 bp。7个罗非鱼群体mtDNA D-loop区的碱基组成之间无显著差异,C、T、A和G的平均含量分别为22.28%、31.86%、31.54%和14.33%,A+T含量(63.39%)明显高于G+C含量(36.61%)。在880个位点中包括保守位点737个,增添/缺失位点16个,变异位点127个(3个单碱基变异位点和124个简约信息位点),变异位点在序列中的分布情况见表3。
在7个罗非鱼群体共140条序列中共发现20种单倍型(表3),其在不同群体中的分布情况见表4。 20种单倍型中共享单倍型有8种(40%),12种为群体特异性单倍型(60%)。Hap-2和Hap-19单倍型的共享率最高,为4个罗非鱼群体共享。YY1群体只有 1种单倍型,其他群体的单倍型数为5~6种。WY1、XX、XY2、XY、YY1、WZ和YY2群体的优势单倍型分别是Hap-2/Hap-19、Hap-7、Hap-2、Hap2/Hap7、Hap5、Hap-10和Hap-10。
2.4 群体遗传多样性和遗传分化
通过DnaSP 5.0软件对7个罗非鱼群体的遗传多样性参数进行分析。结果显示,WY1、XX、XY2和XY 4个群体的单倍型多样性(d)、多态性位点数()、平均核苷酸差异数()、核苷酸多样性指数(P)都明显高于YY1、YY2和WZ 3个群体的相应值,其中,YY1群体的遗传多样性最低(表5)。通过用MEGA 5.0软件中的Kimura 2-Parameter模型计算群体内和群体间遗传距离。结果显示,WY1群体内的遗传距离最大(0.0564),YY1群体内的遗传距离最小(0);WZ群体与YY2群体之间的遗传距离最小(0.0008),XX群体与WZ、YY2群体之间的遗传距离最大(0.1043)。XY2群体与奥利亚罗非鱼群体(WZ)之间的遗传距离(0.0695)大于其与尼罗罗非鱼群体(XX、XY)之间的遗传距离(0.0639) (表6)。通过Arlequin 3.5软件计算群体间遗传分化指数(ST)和基因流(m)。结果显示,除了XY2与WY1群体之间、XX与XY群体之间及WZ与YY2群体之间的遗传分化不显著(ST<0.05,>0.05)之外,其他群体之间均存在极显著的遗传分化(ST>0.05,0.01);WY1与XY2群体之间、XY2与XX群体之间、XY与WY1群体之间、XY与XX群体之间以及XY与XY2之间的基因流N>1,而其他群体之间的基因流N<1 (表7)。

表4 罗非鱼“粤闽1号”及其繁育群体的单倍型及其分布
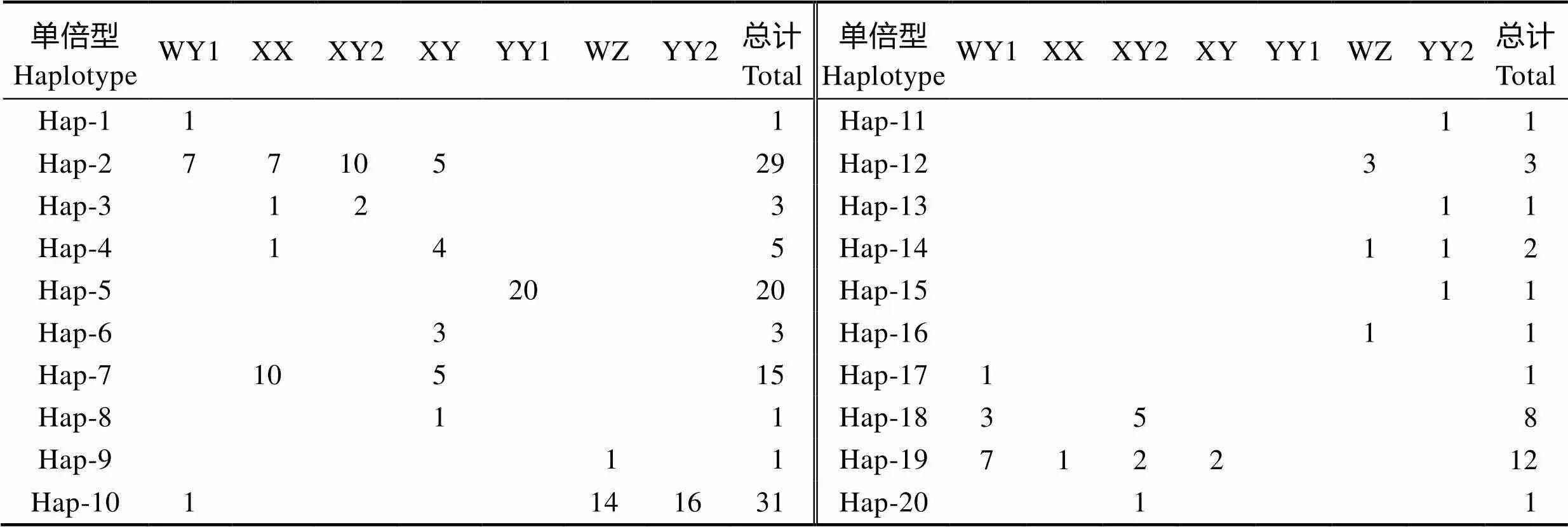
Tab.4 The haplotypes and their distribution in the tilapia “Yuemin No.1” and its breeding populations
2.5 群体遗传进化关系
根据群体之间的遗传距离,采用MEGA 5.0软件中的邻接法构建群体间的分子系统进化树。结果显示,7个罗非鱼群体可以聚为两大支,XX、XY、YY1和XY2群体聚为一支,WY1、WZ和YY2群体聚为另一支,XY2群体与尼罗罗非鱼的亲缘关系比其与奥利亚罗非鱼的亲缘关系近(图4)。
表5 罗非鱼“粤闽1号”及其繁育群体的遗传多样性参数

Tab.5 Genetic diversity paramaters of tilapia “Yuemin No.1” and its breeding populations
表6 罗非鱼“粤闽1号”及其繁育群体间(左下)和群体内遗传距离(对角线)

Tab.6 Genetic distance among (left lower) and within (diagonalline) tilapia “Yuemin No.1” and its breeding populations
表7 罗非鱼“粤闽1号”及其繁育群体遗传分化指数FST(右上)和基因流m(左下)

Tab.7 Gene flow(left lower) and genetic differentiation (upper right) of tilapia “Yuemin No.1” and its breeding populations
** 表示差异极显著,<0.01
** represented highly significant difference,<0.01
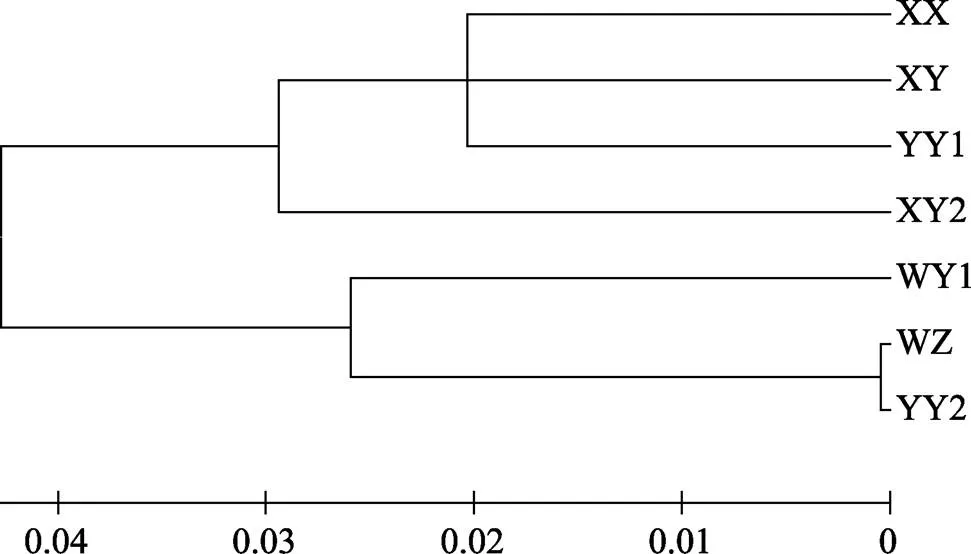
图4 基于mtDNA D-loop区序列构建的罗非鱼“粤闽1号”及其繁育群体的NJ系统进化树
3 讨论
3.1 同工酶酶谱的群体和组织特异性
EST是一种水解酶类,除维持正常的能量代谢外,还可以催化酯类化合物水解为相应的醇和酸 (韩庆等, 2015)。本研究表明,在罗非鱼“粤闽1号”及其繁育群体共6个群体的肌肉和脾脏中EST表达量极低,而肝脏中的EST表达量较高,具有明显的组织特异性,与李思发等(1995)的研究结果类似,这可能与鱼类肝脏中脂类代谢旺盛,具有降解代谢废物和毒素的解毒功能有关(韩庆等, 2015)。大多数鱼类的EST为单体,通常由2个以上的等位基因控制,多态性现象普遍,酶谱复杂(熊全沫, 1992)。杨淞等(2006)在荷那龙罗非鱼()和莫桑比克罗非鱼()中共检测到13条EST酶带。本研究在6个罗非鱼群体肝脏中共检测到10条EST酶带,YY2群体中酶带最多(8条),YY1和WZ群体中酶带最少(5条),且存在个体差异性,这说明不同罗非鱼种类或群体的EST表达具有特异性。罗非鱼“粤闽1号”的EST的酶谱与其父母本群体相比,表现出杂合现象,该现象也同样出现在奥尼杂交罗非鱼群体中,这说明EST-8为尼罗罗非鱼特有谱带,EST-6为奥利亚罗非鱼特有谱带,由不同的等位基因控制,杂交子代中可同时获得这2个谱带。LDH是一种四聚体糖酵解酶,主要参与乳酸的产生和利用,它可在无氧条件下将丙酮酸还原为乳酸,产能以维持机体的能量需求(杨玲等, 2014)。罗非鱼“粤闽1号”及其繁育群体的LDH酶谱具有组织和群体特异性,在肌肉中检测到 5条酶带,而在脾和肝脏中都只检测到3条酶带,这可能与肌肉中无氧代谢较旺盛有关。
3.2 群体遗传多样性
多态性位点比例()和平均杂合度()是评估鱼类群体遗传变异及多态性的重要指标。研究表明,淡水鱼类的多态性位点比例为0.118~0.333,平均杂合度为0.080~0.100,均值为0.043;海水鱼类的平均杂合度为0.029~0.088,均值为0.063 (Gyllensten, 1985; 徐成等, 2001)。本研究对6个罗非鱼群体不同组织中EST和LDH电泳酶谱进行遗传结构分析,结果显示,多态性位点比例()为12.50%~71.43%,平均观测杂合度(o)为0.0417~0.6143,这说明6个罗非鱼群体多样性处于较高水平,遗传多样性较丰富。Hardy- Weinberg遗传偏离指数()用于检验群体的遗传平衡状况,值越趋于0,该群体的基因分布就越接近平衡状态,>0表明杂合子过剩,<0表明杂合子缺失(杨元昊等, 2015)。本研究表明,6个罗非鱼群体的Hardy-Weinberg遗传偏离指数()为‒0.2347~0.9072,其中,WY1群体最小(‒0.2347),XX群体最大(0.9072),说明WY1群体存在杂合子缺失现象,遗传变异较小,而其他群体表现为不同程度的杂合子过剩现象,其中XX群体遗传变异最大,遗传多样性最高。
由于同工酶只能检测基因表达产物,易受外界环境和表达调控的影响,反映的遗传变异和亲缘关系不够全面。因此,本研究进一步利用mtDNA D-loop区部分序列对罗非鱼“粤闽1号”及其繁育群体的遗传多样性进行研究。研究表明,罗非鱼“粤闽1号”及其繁育群体共7个群体的mtDNA D-loop区序列的碱基组成差异较小,均呈现出A+T碱基偏向性(63.39%),这与脊椎动物线粒体DNA序列的特点相一致(Broughton, 2001)。在7个罗非鱼群体中共发现20种单倍型,多数群体的单倍型数为5~6种,YY1群体只有1种单倍型,其单倍型多样性(d)为0,说明YY1群体的遗传多样性较低,这与YY1群体来自尼罗罗非鱼雄鱼与尼罗罗非鱼伪雌鱼交配获得,近亲繁殖严重,子代趋于纯合有关。核苷酸多样性(P)是分子遗传学中评价遗传多样性的重要指标,杨洁等(2014)对8个养殖尼罗罗非鱼群体的研究结果显示,其P值为0.0008~0.0569;本研究中的7个罗非鱼群体的P值为0~0.0519,与其结果相近,WY1、XX、XY2和XY群体的P值(0.0440~0.0519)明显高于YY1、YY2和WZ群体的P值(0~0.0009),说明WY1、XX、XY2和XY群体的遗传多样性较高,而YY1、YY2和WZ群体的遗传多样性较低。虽然YY1、YY2和WZ群体的高度纯合现象有利于其优良性状的稳定遗传,但作为罗非鱼“粤闽1号”的繁育群体,高度纯合不利于这些纯种的保存和复壮。因此,应该扩大选育群体数量,避免过度近亲繁殖和遗传漂变导致群体遗传多样性的下降和优良性状的衰退(吴长敬等, 2010)。
3.3 群体间的遗传差异与亲缘关系
同工酶谱差异是由同工酶基因变异导致同工酶结构差异所引起。一般认为,酶带相似则同工酶的氨基酸组成也相似,因而可能具有较近的亲缘关系;若酶谱差异较大,则表明在生化水平上存在一定分化(Janko, 2007; 魏玉众等, 2017)。XY2、YY2和WZ群体脾脏中的LDH酶谱一致,XX和YY1群体脾脏中的LDH酶谱一致,说明这些酶谱相似的群体之间的亲缘关系较近。
遗传分化指数(ST)是评价群体间遗传分化程度的重要指标,ST<0.05时,表明群体间无遗传分化(Wright, 1978)。本研究表明,7个罗非鱼群体之间的ST为‒0.0115~0.9963,除了XY2与WY1群体之间、XX与XY群体之间以及WZ和YY2群体之间的遗传分化不显著(>0.05)之外,其他群体之间均存在极显著的遗传分化(0.01),这表明罗非鱼“粤闽1号”与尼罗罗非鱼群体和奥利亚罗非鱼群体均存在显著遗传分化,而与奥尼杂交罗非鱼WY1群体遗传分化不明显。XY2群体与尼罗罗非鱼群体(XX和XY群体)之间的基因流m>1,说明它们之间的遗传分化主要由基因流引起,而XY2群体与奥利亚罗非鱼群体(WZ群体)之间的基因流m<1,其遗传分化主要由遗传漂变导致(Slatkin, 1987)。
群体间遗传距离直接反映了各群体之间的亲缘关系,本研究表明,XY2群体与其父母本(YY2群体♂和XX群体♀)的遗传距离分别为0.0696和0.0677,其与母本的亲缘关系较近,这与mtDNA为母系遗传有关。XY2的父本(YY2群体)与奥利亚罗非鱼群体(WZ群体)的亲缘关系明显近于尼罗罗非鱼群体(XX群体和XY群体),这有利于XY2群体最大限度地获得奥利亚罗非鱼的优良性状。本研究还显示,XY2群体与尼罗罗非鱼群体(XX群体和XY群体)的平均遗传距离为0.0639,与奥利亚罗非鱼群体(WZ群体)之间的遗传距离为0.0695,在构建的分子系统进化树中,XY2群体也与尼罗罗非鱼群体(XX和XY群体)聚为一大支,表明XY2群体与尼罗罗非鱼的亲缘关系较近,而与奥利亚罗非鱼的亲缘关系较远。
Broughton RE, Milam JE, Roe BA,. The complete sequence of the zebrafish () mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA. Genome Research, 2001, 11(11): 1958‒1967
Ding JQ, Liu P, Li J,. Genetic variation analysis of four geographic populations ofby isozyme. Progress in Fishery Sciences, 2013, 34(2): 82‒89 [丁金强, 刘萍, 李健, 等. 日本蟳4个地理群体遗传变异的同工酶分析. 渔业科学进展, 2013, 34(2): 82‒89]
Gyllensten U. The genetic structure of fish: Differences in the intraspecific distribution of biochemical genetic variation between marine, anadromous, and freshwater species. Journal of Fish Biology, 1985, 26(6): 691‒699
Han Q, Zhao DH. Comparative study on three isozymes in different tissues of catfish () with different color in Dongting Lake. Guangdong Agricultural Sciences, 2015(2): 104‒107, 113 [韩庆, 赵东海. 洞庭湖不同体色鲇不同组织同工酶的比较研究. 广东农业科学, 2015(2): 104‒107, 113]
Hu YT, Hu W, Jiang H,. Sequence and phylogenetic analysis of the complete mitochondrial genome of Chuzhou Crucian Carp (). Progress in Fishery Sciences, 2015, 36(5): 63‒70 [胡玉婷, 胡王, 江河, 等. 滁州鲫()线粒体全基因组序列分析及系统进化. 渔业科学进展, 2015, 36(5): 63‒70]
Janko K, Flajšhans H, Choleva L,. Diversity of European spined loaches (genusL.): An update of the geographic distribution of thehybrid complex with a description of new molecular tools for species and hybrid determination. Journal of Fish Biology, 2007, 71(Suppl C): 387‒408
Li SF, Cai WQ. Introgression in hatchery stocks ofandin China. Journal of Fisheries of China, 1995, 19(2): 105‒111 [李思发, 蔡完其. 我国尼罗罗非鱼和奥利亚罗非鱼养殖群体的遗传渐渗. 水产学报, 1995, 19(2): 105‒111]
Li SF. Genetical characterization of major freshwater culture fishes in China. Shanghai: Shanghai Scientific and Technical Publishers, 1998, 189‒193 [李思发. 中国淡水主要养殖鱼类种质研究. 上海: 上海科技出版社, 1998, 189‒193]
Liu H, Zhang HQ, Cai SL,. Analysis of the genetic diversity of six populations of culturedbased on the mitochondrial DNA control region. Progress in Fishery Sciences, 2016, 37(1): 63‒73 [刘红, 张海强, 蔡生力, 等. 基于线粒体DNA控制区序列的6个凡纳滨对虾()养殖群体的遗传多样性分析. 渔业科学进展, 2016, 37(1): 63‒73]
Slatkin M. Gene flow and the geographic structure of natural populations. Science, 1987, 236(4803): 787‒792
Teletchea F. Molecular identification methods of fishspecies: Reassessment and possible applications. Reviews in Fish Biology and Fisheries, 2009, 19(3): 265‒293
Wei YZ, Zhang GR, Huo B,. A comparative study on lactate dehydrogenase isozymes in six species of. Freshwater Fisheries, 2017, 47(5): 3‒8 [魏玉众, 张桂蓉, 霍斌, 等. 雅鲁藏布江中游6种裂腹鱼乳酸脱氢酶同工酶的比较研究. 淡水渔业, 2017, 47(5): 3‒8]
Wright S. Evolution and the genetics of populations: Variability within and among natural populations. Chicago: University of Chicago Press, 1978, 580
Wu CJ, Zou ZY, Yang H,. Structure of the mitochondrial DNA D-Loop region and analysis of genetic diversity in different strains of tilapia (). Chinese Journal of Zoology, 2010, 45(5): 121‒128 [吴长敬, 邹芝英, 杨弘, 等. 罗非鱼mtDNA D-loop区部分序列结构和种群遗传多样性分析. 动物学杂志, 2010, 45(5): 121‒128]
Wu YY, Li LH, Han JL. Studies on four isozyme from several tissues of Jifu tilapia. Biotechnology Bulletin, 2008(Suppl), 319‒323 [吴燕燕, 李来好, 韩君莉. 吉富罗非鱼不同组织中4种同工酶研究. 生物技术通报, 2008(增刊): 319‒323]
Xie XY, Li SF. Comparison of base sequence diversity of Cytb and D-loop gene of Nile tilapia. Genomics and Applied Biology, 2014, 33(5): 982‒985 [颉晓勇, 李思发. 罗非鱼选育群体Cytb与D-loop序列变异信息对比分析. 基因组学与应用生物学, 2014, 33(5): 982‒985]
Xiong QM. Isozymesanalysis of fishes (Vol. 1). Hereditas (Beijing), 1992, 14(2): 41‒44, 48 [熊全沫. 鱼类同工酶谱分析(上). 遗传, 1992, 14(2): 41‒44, 48]
Xu C, Wang KL, You F,. Biochemical genetics ofpopulation Ⅰ biochemical genetic analysis of isozymes. Oceanologia et Limnologia Sinica, 2001, 32(1): 42‒49 [徐成, 王可玲, 尤锋, 等. 鲈鱼群体生化遗传学研究Ⅰ同工酶的生化遗传分析. 海洋与湖沼, 2001, 32(1): 42‒49]
Yang J, He AY, He XJ,. Genetic diversity and relationships of mitochondrial DNA control region from the eight domesticated populations of Nile tilapia. Journal of Fishery Sciences of China, 2014, 21(4): 693‒699 [杨洁, 何安元, 何学军, 等. 尼罗罗非鱼8个养殖群体线粒体控制区遗传多样性和遗传关系分析. 中国水产科学, 2014, 21(4): 693‒699]
Yang L, Lu H, Liu YQ,. Study on the tissue specificity of 8 isozymes infrom Dongping Lake. Chinese Agricultural Science Bulletin, 2014, 30(14): 26‒32 [杨玲, 卢红, 刘羽清, 等. 东平湖鲤8种同工酶的组织特性研究. 中国农学通报, 2014, 30(14): 26‒32]
Yang S, Huang ZH, Ye X,. Preliminary study on three isozymes ofand. Journal of Dalian Fisheries University, 2006, 21(2): 122‒126 [杨淞, 黄樟翰, 叶星, 等. 荷那龙罗非鱼和莫桑比克罗非鱼3种同工酶的分析. 大连水产学院学报, 2006, 21(2): 122‒126]
Yang YH, Zhou JS, Li L,. Preliminary analysis on isozymes in different tissues and population genetic structure of. Freshwater Fisheries, 2015, 45(1): 25‒29 [杨元昊, 周继术, 李蕾, 等. 兰州鲇不同组织同工酶及群体遗传结构初步分析. 淡水渔业, 2015, 45(1): 25‒29]

Zhu LF. Gradient gel electrophoresis on polyacrylamide of isozymes and proteins of fishes. Acta Hydrobiologica Sinica, 1992, 16(2): 183‒185 [朱蓝菲. 鱼类同工酶和蛋白质的聚丙烯酰胺梯度凝胶电泳法. 水生生物学报, 1992, 16(2): 183‒185]
Genetic Diversity and Genetic Relationship Analysis of Tilapia “Yuemin No.1” and Its Breeding Populations
LIU Zhigang, LU Maixin①, CAO Jianmeng, GAO Fengying
(Pearl River Fisheries Research Institute, Chinese Academy of Fisheries Science, Key Laboratory of Tropical & Subtropical Fishery Resource Application & Cultivation, Ministry of Agriculture and Rural Affairs, Guangzhou 510380)
“Yuemin No.1” is a novel holandric species of tilapia, which was bred withand. In order to reveal the genetic characteristics of tilapia “Yuemin No.1,” the genetic diversity, genetic structure, and genetic relationships among groups of tilapia “Yuemin No.1” and its breeding populations were studied by isozyme electrophoresis and sequence analysisof the D-loop region of mtDNA. The results of isozyme electrophoresis showed that the zymogram of esterase (EST) and lactate dehydrogenase (LDH) werepopulation- and tissue-specific in tilapia. The expression level of EST in the muscle and spleen was extremely low, whereas that in the liver was high. LDH was highly expressed in all the tissues, as revealed by the different zymograms. Ten bands of EST and five of LDH were detected in tilapia “Yuemin No.1” and its breeding populations. The proportion of polymorphic loci () ranged from 12.50% to 71.43%, the average observed heterozygosity (o) ranged from 0.0417 to 0.6143, and the Hardy-Weinberg genetic deviation index () ranged from ‒0.2347 to 0.9072. The result of sequence analysis of the D-loop region of mtDNA revealed that no significant difference occurred in base composition, and that A+T content (63.39%) bias was widespread in all the populations. There were 20 haplotypes in tilapia “Yuemin No.1” and its breeding populations. The nucleotide diversity index (P) ranged from 0 to 0.0519. ThePvalue of WY1, XX, XY2, and XY populations (0.0440~0.0519) were significantly higher than that of YY1, YY2, and WZ populations (0~0.0009). The genetic differentiation index (ST) among populations ranged from ‒0.0115 to 0.9963. There was no significant genetic differentiation (ST<0.05,>0.01) between the XY2 and WY1, XX and XY, and WZ and YY2 populations. The average genetic distance between XY2 population and(XX and XY populations) was 0.0639, whereas that between the XY2 population and(WZ population) was 0.0695. Cluster analysisrevealed two clusters; one cluster included XY, XX, YY1, and XY2 populations, and the othercluster included WY1, WZ, and YY2 populations. This study revealed the biochemical and molecular genetic features of tilapia “Yuemin No.1,” and provide research data for germplasm identification, establishment of breeding populations, and maintainence of stable inheritance of desirable traits.
Tilapia “Yuemin No.1”; Isozyme; Mitochondrial DNA control region; Genetic diversity; Genetic relationship
LU Maixin, E-mail: mx-lu@163.com
刘志刚, 卢迈新, 曹建萌, 高风英. 罗非鱼“粤闽1号”及其繁育群体的遗传多样性和遗传关系分析. 渔业科学进展, 2018, 39(6): 31–41
Liu ZG, Lu MX, Cao JM, Gao FY. Genetic diversity and genetic relationship analysis of tilapia “Yuemin No.1” and its breeding populations. Progress in Fishery Sciences, 2018, 39(6): 31–41
* 现代农业产业技术体系专项资金(CARS-46)、广东省自然科学基金(2016A030313146)和广州市科技计划项目(201803020034)共同资助[This work was supported by China Agriculture Research System (CARS-46), Natural Science Foundation of Guangdong Province(2016A030313146), and Science and Technology Program of Guangzhou(201803020034)]. 刘志刚,E-mail: wenliugang@163.com
卢迈新,研究员,E-mail: mx-lu@163.com
2018-04-28,
2018-05-21
10.19663/j.issn2095-9869.20180428001
S917.4
A
2095-9869(2018)06-0031-11
(编辑 马璀艳)

