基于Red重组系统的阴沟肠杆菌基因重组技术
,,,,, ,*
(1.上海大学生命科学学院,上海 200444; 2.中国科学院上海高等研究院,生物炼制实验室,上海 201210; 3.广东本科检测有限公司,广东汕头 515071)
Red重组系统是大肠杆菌(Escherichiacoli)中最常用的基因重组系统,该系统从λ噬菌体衍化而来,主要由Exo、Bet、Gam三个基因组成。Exo基因编码一个5′-3′核酸外切酶,该酶可以作用于双链DNA,产生3′端突出。Bet基因编码一种单链DNA结合蛋白,该蛋白具有介导互补单链DNA退火的功能,防止单链DNA被核酸酶降解[1]。Gam基因编码的蛋白能够与宿主菌的RecBCD核酸外切酶结合,抑制其对外源DNA的降解。由于该系统可以利用具有36碱基同源臂的抗性盒进行基因重组,使得同源臂可以直接合成在引物上,而不需要将目的基因克隆出,使得整个操作过程简单、高效[2]。目前,已经有学者将Red重组系统改造修饰后用于多种细菌及真菌的基因替换,例如:痢疾杆菌(Bacillusdysenteriae)[3]、铜绿假单胞菌(Pseudomonasaeruginosa)[4]、天蓝色链霉菌(Streptomycescoelicolor)[5]、霍乱弧菌(Vibriocholerae)[6]、克雷伯氏肺炎菌(Klebsiellapneumoniae)[7]及耶尔森氏菌(Yersiniaenterocolitica)[8]等。
阴沟肠杆菌(Enterobactercloacae)是一种重要的工业微生物,在自然界中广泛存在,属于革兰氏阴性菌,其生长力旺盛,耐受能力强,培养条件简单、底物广泛且底物利用率高。有学者报道利用该菌作为底盘生物发酵葡萄糖可以合成乙偶姻[10]、2,3-丁二醇[11]、双乙酰[12]和乙醇[13]等化学品;在环境保护方面,E.cloacae也可以用于降解有机磷农药[14]。阴沟肠杆菌(E.cloacae)[11]、克雷伯氏肺炎杆菌(Klebsiellapneumoniae)[15]、枯草芽孢杆菌(Bacillussubtilis)[16]等多种微生物可以合成2,3-丁二醇。这些微生物中2,3-丁二醇的合成途径都相同。丙酮酸在乙酰乳酸合成酶的催化下形成乙酰乳酸,乙酰乳酸在脱羧酶的催化下形成乙偶姻,乙偶姻在丁二醇还原酶的催化下形成2,3-丁二醇,这三个酶的编码基因分别为budB(AlsS)、budA和budC[17]。在课题组前期的研究中,K.pneumoniae的乙酰乳酸脱羧酶(budA)失活后菌株合成乙偶姻和2,3-丁二醇的能力丧失,但是该突变株在低pH高溶氧条件下可以合成高水平的2-酮基葡萄糖酸[18],在中性pH、低溶氧条件下可以合成α-酮异戊酸[19]。现有的报道中,E.cloacae中最常用的基因重组方法是自杀型质粒法[11]。该方法周期较长,敲除效率低,且抗性标记不易消除。为了建立一种适用于E.cloacae的基因重组方法,本文将Red重组系统引入E.cloacae中,并对抗性盒同源臂长度进行了优化。进一步的研究中,研究了利用Flp重组酶系统消除突变株的抗性标记基因。
利用Red重组系统对E.coli进行基因缺失时,常用的质粒为pKD46,其上带有Red重组酶基因,而在消除抗性标记时常用的质粒为PCP20,其上带有Flp重组酶基因,两种质粒所带抗性基因均为氨苄青霉素[9]。本文所使用的E.cloacae是从自然界筛选得到,天然带有氨苄青霉素抗性,不能直接将两种质粒用于E.cloacae的基因重组,因此选用pSC-MSC质粒作为Red和Flp重组酶的表达载体,该质粒的启动子为温控型启动子,在基因敲除时可以较方便的诱导重组酶的表达。
1 材料与方法
1.1 材料与仪器
质粒提取试剂盒、细菌基因组提取试剂盒、PCR清洁试剂盒 Axygen公司;DNA Maker(10kb)、T4 DNA连接酶、限制性内切酶(BamH I、Nco I及Dpn I) Thermofisher公司;kod-plus-Neo DNA聚合酶 TOYOBO公司;Taq DNA聚合酶 康为世纪公司;其余试剂 均为国产分析纯,购于国药;发酵种子用LB培养基进行培养,配方为氯化钠10 g/L,蛋白胨10 g/L,酵母提取物5 g/L。发酵用发酵培养基进行发酵,组成成分为:葡萄糖50 g/L,酵母提取物15 g/L,玉米浆粉12 g/L,硫酸铵15 g/L,氯化钾1.2 g/L,醋酸钠9 g/L,七水合硫酸镁0.3 g/L,七水合硫酸亚铁0.06 g/L,一水合硫酸锰0.03 g/L;实验所用菌株及来源见表1,实验所用引物及序列见表2。

表1 菌株与质粒Table 1 Strains and plasmids

表2 PCR所用引物及其序列Table 2 Oligonucleotides used for PCR
ZHJH-C1115C型超净工作台 上海智城分析仪器制造有限公司;S1000 Thermal Cycler PCR仪、MicroPulser电转仪、1 mm电转杯、PowerPac Basic电泳仪、Gel Dox XR+凝胶成像仪 BIO-RAD;WFJ 2100型可见分光光度计 尤尼柯(上海)仪器有限公司;ZWY-2102C型恒温培养振荡器 上海智城分析仪器制造有限公司;RID-10A/SPD-20A高效液相色谱 SHIMADZU;MLS-3780型高压蒸汽灭菌器 SANYO;Biostat A Plus发酵罐 Sartorius。
1.2 实验方法
1.2.1 重组质粒pSC-MSC-red及pSC-MSC-flp的构建 以质粒pSC-MSC-red的构建为例,利用表2中的引物red-s及red-a以pIJ790质粒为模板扩增red基因,通过TA克隆将red基因连接到pMD18-T载体上,获得质粒pMD18-T-red。利用限制性内切酶BamH Ⅰ和Nco I分别同时处理pMD18-T-red质粒和pSC-MSC质粒,割胶回收获得带有粘性末端的red基因片段及线性载体pSC-MSC。在T4 DNA连接酶的作用下,载体与片段发生连接获得重组质粒pSC-MSC-red,化转到DH5α感受态细胞中扩增后提取该质粒,PCR验证并测序。
构建pSC-MSC-flp质粒时,扩增flp基因用表2中的引物flp-a和flp-s,模板为pDK6-flp,其余步骤与pSC-MSC-red质粒的构建相同。
1.2.2 带长同源臂抗性盒的构建 以E.cloacae的基因组为模板,利用表2中的引物budAk-s及budAk-a扩增目的基因budA-Ec,所扩基因包含budA原始基因及其上下游500 bp外围基因。将目的基因budA-Ec连接到pMD18-T载体上获得质粒pMD18-T-budA-Ec。制作E.coliDH5α/PIJ790的感受态细胞,将菌株划线于LB固体平板,待长出单克隆后挑取单克隆接种于LB液体试管,过夜培养后转接至50 mL液体LB培养基中,当细菌生长至OD600=0.5时,冰域放置30 min;4 ℃,5000 r/min离心10 min收集感受态细胞,用预冷的无菌水清洗感受态细胞3次,调整细胞终浓度为OD600=10获得E.coliDH5α/PIJ790的感受态细胞。设置电转仪2.0 kV,200 Ω,25 μF将pMD18-T-budA-Ec质粒电击转化入E.coliDH5α/PIJ790,获得含有双质粒的菌株命名为E.coliDH5α/PIJ790/pMD18-T-budA-Ec。以PIJ773质粒为模板,利用表2中的引物FRT-s1及FRT-a1扩增出带有39 bp同源臂的抗性盒。
制作菌株E.coliDH5α/PIJ790/pMD18-T-budA-Ec的感受态(培养初期时加入10 mmol/L的阿拉伯糖诱导pIJ790质粒上的Red重组酶表达),将含有39 bp同源臂的抗性盒电击转化入感受态细胞中,在Red重组酶的作用下,抗性盒与pMD18-T-budA-Ec质粒上的目的基因发生同源重组,获得重组质粒pMD18-T-budA-Ec-773。
1.2.3 扩增不同长度同源臂的抗性盒 以pMD18-T-budA-Ec-773质粒为模板,用表2中的引物budAk100-a和budAk100-s;budAk200-a和budAk200-s;budAk300-a和budAk300-s;budAk400-a和budAk400-s;budAk500-a和budAk500-s进行PCR,获得中间为安普霉素抗性基因两端带有不同长度同源臂的抗性盒。所得PCR产物用Dpn I限制性内切酶处理消除质粒模板。
1.2.4E.cloacae/pSC-MSC-red的构建 将活化好的E.cloacae接种到50 mL的LB液体培养基中,37 ℃,200 r/min培养,当细胞浓度达到OD600=0.5~0.8时,将培养物置于冰上冰浴30 min。冰浴结束后5000 r/min,4 ℃离心10 min收集细胞,用预冷的无菌水清洗细胞三次,然后将其悬浮于无菌水,调整细胞终浓度为OD600=30,获得感受态细胞。每100 μL感受态细胞中加入1 μg的pSC-MSC-red质粒,冰浴静置10 min后进行电击转化[20]。电转仪设置为2.0 kV,200 Ω及25 μF,将电转杯置于电转仪中进行电击转化[7]。电击转化完成后在感受态细胞中加入适量LB培养基,并将其置于37 ℃,200 r/min摇床复苏1 h。复苏完成后5000 r/min离心3 min收集菌体,将菌体全部涂布于含有卡那霉素抗性(100 μg/mL)的固体LB培养基上,37 ℃过夜静置培养。将平板上生长起来的阳性单克隆命名为E.cloacae/pSC-MSC-red。
1.2.5 缺失E.cloacae的budA基因 制作E.cloacae/pSC-MSC-red的电击转化感受态细胞,当细菌生长至OD600=0.1时,将培养摇床温度改为42 ℃,以诱导pSC-MSC-red质粒上的Red重组酶表达,其余过程与1.2.4所述相同。将带有不同长度同源臂的抗性盒分别电击转化入E.cloacae/pSC-MSC-red感受态细胞中,复苏后涂布于带有安普霉素抗性(50 μg/mL)的固体LB培养基上37 ℃ 过夜静置培养。待单克隆生长起来后用表2中的引物budAy-a及Test773做菌落PCR验证,budAy-a引物以budA-Ec的下游基因为模板设计,Test773引物以安普霉素抗性基因为模板设计。通过测序确认budA基因被成功敲除后将基因缺失菌命名为E.cloacaeΔbudA-773/pSC-MSC-red,该菌株带有卡那霉素抗性和安普霉素抗性。
1.2.6 消除E.cloacaeΔbudA-773/pSC-MSC-red菌株的抗性标记 消除抗性标记需要Flp重组酶和FRT序列的共同作用[2]。由于pSC-MSC-flp质粒和pSC-MSC-red质粒所用载体均为pSC-MSC,在转化pSC-MSC-flp质粒前需消除E.cloacaeΔbudA-773/pSC-MSC-red的pSC-MSC-red质粒。方法为将E.cloacaeΔbudA-773/pSC-MSC-red接种于不含抗性的LB液体培养基中,传代3次后划线分离得到数个单克隆。将每一个单克隆分别接种到两种LB培养基中,一种含有100 μg/mL的卡那霉素,另一种不含抗生素。用菌落PCR验证能在无抗性培养基上长起来且在含卡那霉素抗性培养基上长不起来的单克隆。
将消除质粒的菌株命名为E.cloacaeΔbudA-773,该菌只带安普霉素抗性,制作该菌的感受态细胞,按照1.2.4的方法将pSC-MSC-flp质粒电击转化入该菌中,获得菌株E.cloacaeΔbudA-773/pSC-MSC-flp。将E.cloacaeΔbudA-773/pSC-MSC-flp接种到液体LB培养基中42 ℃传代5次,与消除质粒方法相同筛选出消除了抗性标记的E.cloacaeΔbudA-773/pSC-MSC-flp,将其命名为E. cloacae ΔbudA/pSC-MSC-flp。
按照消除pSC-MSC-red质粒的方法消除掉E.cloacaeΔbudA/pSC-MSC-flp的pSC-MSC-flp质粒,得到无抗性标记的budA基因缺失菌,将其命名为E.cloacaeΔbudA,该菌只带有氨苄霉素抗性。
1.2.7E.cloacaeΔbudA生理特性研究 将E.cloacae及E.cloacaeΔbudA分别接种到50 mL液体LB培养基中,37 ℃,200 r/min培养8 h获得发酵种子,将该种子接种到总体积为5 L的发酵罐(内含3L培养基)中进行批次发酵。发酵条件为通风量2 L/min,转速300 r/min,温度37 ℃,用30%的NaOH溶液控制发酵过程的pH为6.8。发酵过程每3 h取样检测葡萄糖、乙偶姻、琥珀酸、乳酸、乙酸、2,3-丁二醇和乙醇等组分的含量变化。发酵液组分测定采用高效液相色谱法,色谱柱为HPX-87H,流动相为0.005 mol/L的稀硫酸溶液,流速为0.8 mL/min,柱温箱为60 ℃,使用视差检测器进行检测[21]。
2 结果与分析
2.1 pSC-MSC-red及pSC-MSC-flp质粒的构建
pSC-MSC-red和pSC-MSC-flp的构建按照材料与方法描述进行。提取的质粒使用引物pSC-s及pSC-a进行PCR验证。重组质粒pSC-MSC-red的PCR结果见图1a,重组质粒pSC-MSC-flp的PCR结果见图1b。图1a中,泳道2为实验组在2500~3000 bp位置处有一目的条带,red基因原始长度为1885 bp,PCR产物中包含的质粒序列为718bp,其总长与目的条带大小相符,证明pSC-MSC-red质粒构建成功。同理,图1b中,泳道4为实验组在1500~2000 bp位置处有一目的条带,flp基因原始长度为1272 bp,其PCR产物中质粒序列为718 bp,其总长也与图中目的条带相符,证明pSC-MSC-flp质粒也构建成功。图1a及图1b中阴性对照(泳道1和泳道3)是以pSC-a和pSC-s为引物,以pSC-MSC空质粒为模板扩增得到,说明实验组的目的条带为特异性扩增的阳性克隆。两重组质粒上目的基因序列测定结果与参考序列完全一致。

图1 重组质粒pSC-MSC-red和pSC-MSC-flp的PCR验证Fig.1 PCR results of recombinant plasmid pSC-MSC-red and pSC-MSC-flp注:泳道1和泳道3为以空载质粒pSC为模板扩增得到的条带,泳道2为以pSC-MSC-red质粒为模板扩增得到的条带,泳道4为以pSC-MSC-flp质粒为模板扩增得到的条带,Marker为10 kp。
2.2 不同长度同源臂抗性盒的构建
应用Red重组系统进行基因敲除时,不同菌株中所需同源片段长度不同,大肠杆菌为36 bp,而在其他细菌中则稍微长一些。在霍乱弧菌中,50 bp或100 bp的同源臂便足以对目的基因进行敲除,但是其同源臂长度增加到1000 bp时,重组效率显著提高[6]。在K.Pneumoniae中200 bp同源臂可获得重组子,500 bp同源臂重组效率显著提高[7]。为了研究不同长度同源臂对于基因重组效率的影响,本文构建了带有39、100、200、300、400、500 bp同源臂的抗性盒用于E.cloacae的基因敲除。
按照材料与方法描述过程将带39 bp同源臂的抗性盒转入到E.coliDH5α/PIJ790/pMD18-T-budA-Ec中,用安普霉素抗性平板进行筛选。待菌落生长出来后,挑取部分单克隆使用通用引物M13-47和M13-48进行菌落PCR验证,结果见图2。未重组时,以质粒pMD18-T-budA-Ec为模板扩增出来的条带大小应为1710 bp,图中泳道7和9对应的单克隆未发生重组。同源重组发生后,部分基因被抗性编码基因替换,扩增条带大小应为2410 bp。泳道1、2、3、6、8对应的单克隆成功重组,pMD18-T-budA-Ec-773质粒构建成功。
图2中,泳道4、5、10含有双条带,其原因是pMD18-T载体为高拷贝质粒,转化入抗性盒后只有一部分pMD18-T-budA-Ec质粒与抗性盒发生了重组,另外一部分pMD18-T-budA-Ec保持未重组状态。为了解上述问题,在后期的实验中可以尝试通过Overlap PCR直接扩增出带有不同长度源臂的抗性盒。

图2 重组质粒pMD18-T-budA-Ec-773的PCR验证Fig.2 PCR results of recombinant plasmid pMD18-T-budA-Ec-773 注:泳道1~10为带39 bp抗性盒与E.coli DH5α/PIJ790/pMD18-T-budA-Ec重组后各单克隆菌落PCR验证结果,Marker为10 kb。
以pMD18-T-budA-Ec-773质粒为模板,扩增出带有不同长度同源臂的抗性盒,结果见图3。图3中泳道1、2、3、4、5分别对应了带有500、400、300、200、100 bp同源臂的抗性盒;泳道6为以pIJ790质粒为模板使用引物FRT-s1和FRT-a1扩增得到的带有39 bp同源臂的抗性盒。

图3 不同长度同源臂的抗性盒的PCR结果Fig.3 PCR results of different length homologous arms注:泳道1、2、3、4、5、6分别为带有500、400、300、200、100、39 bp同源臂的抗性盒的。
2.3 不同长度同源臂抗性盒的重组效率
为了确定E.cloacae中基因重组所需同源臂的最适长度,分别将带有39、100、200、300、400、500 bp同源臂的抗性盒转化入E.cloacae/pSC-MSC-red。待单菌落生长起来后,计算各长度同源臂抗性盒重组的效率。基因重组的效率与所加入的抗性盒DNA的量有关,可以用每1 μg抗性盒DNA转化入宿主菌后抗性平板上生长起来的单菌落中阳性克隆的个数来表示敲除效率。各长度同源臂抗性盒重组效率见表3。从表中可以看出,同源臂长度为39 bp和100 bp时重组效率为零;同源臂长度增加到200、300及400 bp时,重组效率分别为6.1、14.3和38.8 CFU/μg;同源臂长度增加到500 bp时,重组效率有较大提高,为131.5 CFU/μg。基于此结果,500 bp长度的同源臂抗性盒更适用于E.cloacae的基因重组。
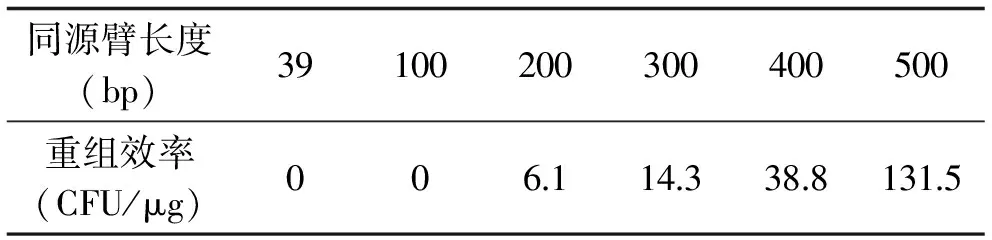
表3 不同长度同源臂抗性盒的重组效率Table 3 Recombination ratios of different length homologous extensions
Red重组酶应用于E.cloacae的基因最高重组效率为131.5 CFU/μg,略低于大肠杆菌(500 CFU/μg)[7],其原因可能是Red重组酶中的Gam蛋白来源于E.coli,其对于E.cloacae的RecBCD核酸外切酶抑制能力较弱。在K.pneumoniae的基因重组中,同样存在重组效率(266.8 CFU/μg)低于E.coli的情况,通过抑制宿主菌的RecBCD核酸外切酶活性,可以提高Red重组系统在K.pneumoniae中的敲除效率[22]。
2.4 利用Flp重组酶消除E. cloacae ΔbudA-773/pSC-MSC-red菌株的抗性标记
按照1.2.6所述方法消除E.cloacaeΔbudA-773/pSC-MSC-red的pSC-MSC-red质粒。用表2中的引物pSC-a和pSC-s PCR验证能在无抗性培养基上长起来且在带卡那霉素抗性培养基上长不起来的单克隆,结果见图4a。图中泳道2为消除质粒前的对照,在2500 bp左右处有一目的条带;消除了pSC-MSC-red质粒后扩增不出条带(泳道1),说明pSC-MSC-red质粒已经成功消除。

图4 消除宿主菌外源质粒及抗性标记的PCR验证Fig.4 PCR results of eliminating the plasmid and resistance marker from E. cloacae ΔbudA-pSC-MSC-flp注:泳道1、3、5为消除菌株质粒或抗性标记基因后的PCR结果,为实验组;泳道2、4、6为消除菌株质粒或抗性标记基因前的PCR结果,为对照组。
按照1.2.6所述方法传代培养E.cloacaeΔbudA-773/pSC-MSC-flp,使用表2中的引物budAy-a及Test773 PCR验证能在无抗性培养基上长起来且在带安普霉素抗性培养基上长不起来的单克隆,结果见图4b。图中泳道4为消除抗性标记前的对照,在1500 bp左右处有一目的条带;消除抗性标记后扩增不出条带(泳道3),说明E.cloacaeΔbudA-773/pSC-MSC-flp菌株的安普霉素抗性标记已成功消除。
按照1.2.6所述方法消除E.cloacaeΔbudA-pSC-MSC-flp的pSC-MSC-flp质粒。用表2中的引物pSC-a和pSC-s PCR验证能在无抗性培养基上长起来且在带卡那霉素抗性培养基上长不起来的单克隆,结果见图4c,其中6号泳道为消除质粒前的对照,在1500~2000 bp处有一目的条带,消除pSC-MSC-flp质粒后,扩增不出条带(泳道5),说明pSC-MSC-flp质粒已经成功消除。
2.5 E. cloacae ΔbudA生理特性研究
为了考察敲除E.cloacae的budA基因后其生理特性的变化,将E.cloacae及E.cloacaeΔbudA分别接种到发酵罐中进行批次发酵,其结果见图5,其中图5(a)和图5(b)为E.cloacae的发酵结果;图5(c)和图5(d)为E.cloacaeΔbudA发酵结果。E.cloacae发酵过程中合成了一定量的乙偶姻且2,3-丁二醇产量逐渐增加,而E.cloacaeΔbudA整个发酵过程都不再合成乙偶姻和2,3-丁二醇。此外,敲除E.cloacae的budA基因后,菌株的生长变缓,野生型菌株在9 h左右底物全部耗尽,菌体生长进入平稳期,OD600为13.79。而突变株发酵9 h还有16.69 g/L的葡萄糖残余,OD600只有12.03。细菌代谢糖类合成乙酸或乳酸等酸类物质时,大量有机酸会对菌株的生长产生抑制作用,而醇类物质对菌体的抑制作用要低很多[23]。在E.cloacaeΔbudA中,敲除了乙偶姻的合成基因,2,3-丁二醇的合成被阻断,细菌合成较多有机酸使得生长变缓。

图5 E. cloacae及E. cloacae ΔbudA批次发酵结果Fig.5 Batch fermentation of E. cloacae and E. cloacae ΔbudA注:(a)和(b)为E. cloacae发酵结果;(c)和(d)为E. cloacae ΔbudA发酵结果。
发酵15 h后发酵液中各组分具体含量见表4。从表中可以看出,野生型菌株合成了(0.97±0.08) g/L的乙偶姻和(12.25±0.71) g/L的2,3-丁二醇以及其他一些有机酸等代谢产物,而E.cloacaeΔbudA的发酵液中检测不到乙偶姻和2,3-丁二醇,主要代谢产物为琥珀酸和乳酸。发酵结果进一步说明E.cloacae的budA基因已被成功缺失。

表4 E. cloacae和E. cloacae ΔbudA发酵结果Table 4 Batch fermentation of E. cloacae and E. cloacae ΔbudA
3 结论
本文以budA基因为例,详细地叙述了Red重组系统在E.cloacae基因重组中的应用。构建了重组质粒pSC-MSC-red质粒和pSC-MSC-flp质粒,在Red重组酶的作用下成功敲除了E.cloacae的budA基因,并通过Flp重组酶成功消除了突变株的抗性标记。运用本文的方法对E.cloacae进行基因缺失的周期一般为3个周。除了budA基因外,运用该系统还成功地缺失了E.cloacae的一些其他基因。总的来说,该系统的建立,有利于快速构建E.cloacae的单基因和多基因重组突变株,将促进E.cloacae的代谢工程改造和其在多方面的应用。

