原位整合型枯草芽孢杆菌芽孢表面展示载体的构建
杨亚威 ,杨明明,龚月生
(西北农林科技大学 动物科技学院,陕西 杨凌 712100)
枯草芽孢杆菌虽为益生菌[1],但其在动物肠道中停留时间短暂且易被机体降解,而芽孢能有效抵抗胃肠道中的酸碱环境,因此延长芽孢在肠道中的停留时间能够有效地发挥枯草芽孢杆菌的益生特性。
在环境恶劣,营养缺乏等极端条件下,枯草芽孢杆菌属的营养细胞会形成一种休眠体,即芽孢。枯草芽孢杆菌的芽孢包括中心内核和几种蛋白层结构,内核主要由高度失水的基因组构成,蛋白层结构由至少70种蛋白质组成,由内到外分为皮质层、基底层、芽孢内壳、芽孢外壳和crust层[2](图1)。
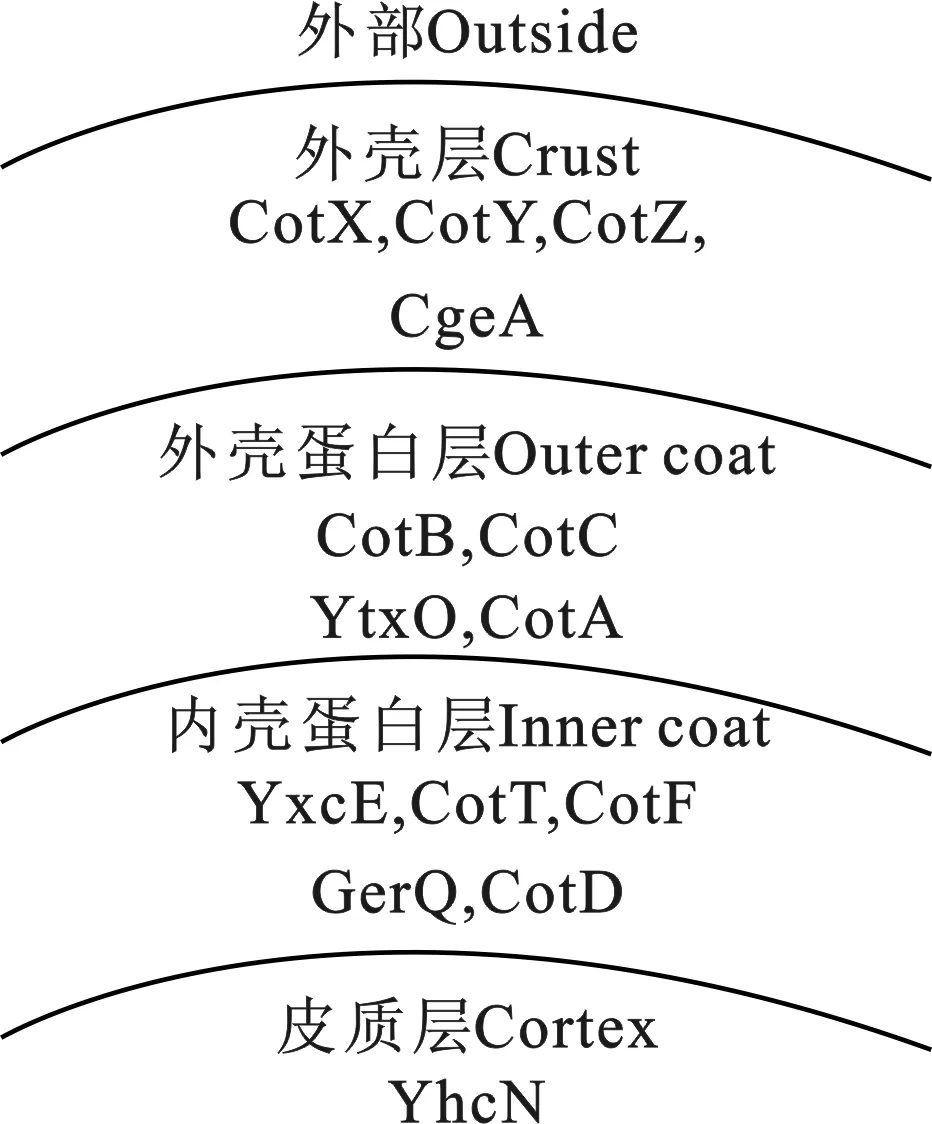
图1 芽孢外壳结构示意图Fig.1 Structure diagram of spore coat
芽孢用钌红染色后在电镜下能观察到crust层[3],外壳蛋白层主要有保护芽孢的作用,使其免受各种蛋白酶、有毒物质和恶劣环境因子等的影响。
在芽孢表面展示高粘附因子能够增强芽孢粘附肠道细胞的能力,但绝大多数已经报道的研究常采用异位整合的方式实现外源蛋白与芽孢衣壳蛋白的融合表达,表达效率低[4]。本研究尝试利用原位整合的方法构建一套芽孢表面展示载体,寻找一种新的芽孢表面展示方法,选用两种芽孢衣壳蛋白CotB和CotZ作锚定蛋白,通过原位整合的方式尝试实现绿色荧光蛋白在枯草芽孢杆菌芽孢表面的展示。
1 材料与方法
1.1 试验材料
1.1.1 引物、菌株、质粒和主要试剂 大肠杆菌(E.coliDH5α)和枯草芽孢杆菌(BacillussubtilisPY79)为本实验室保存,分别作为克隆宿主菌和表达宿主菌。携带有绿色荧光蛋白编码基因的质粒pEYW24为本实验室保存。各种限制性内切酶、T4 DNA连接酶、pGEM T-easy载体购自美国Promega公司;普通Taq DNA聚合酶、基因组提取试剂盒、DNA纯化回收试剂盒、PCR纯化回收试剂盒等购自北京全式金生物技术有限公司,质粒小提试剂盒购自OMEGA,兔源GFP抗体及羊抗兔二抗购自西安嘉城生物科技有限公司。用于扩增外壳蛋白编码基因引物见表1,用于扩增芽孢衣壳蛋白编码基因的下游引物均不含终止密码子。
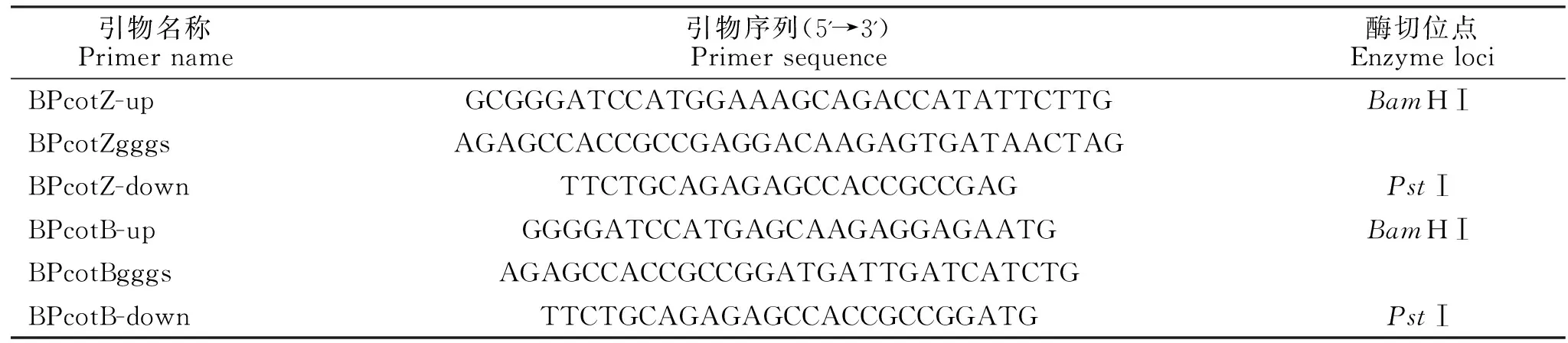
表1 引物名称及序列Table 1 Primers and sequences
1.1.2 培养基 大肠杆菌和枯草芽孢杆菌均用普通LB培养基培养,在需要时分别加入氨苄青霉素(100 μg/mL)和壮观霉素(50 μg/mL)。
1.2 方法
1.2.1 原位整合型重组质粒的构建 以BacillussubtilisPY79基因组为模板,分别以BPcotB-up和BPcotBgggs,BPcotZ-up和BPcotZgggs为引物,扩增芽孢衣壳蛋白基因(在上游加入了BamHⅠ酶切位点,下游加入了GGGS连结肽),体系回收PCR产物,再以PCR产物为模板,BPcotB-up/down,BPcotZ-up/down为引物扩增芽孢衣壳蛋白基因(在下游加入PstⅠ酶切位点),体系回收PCR产物。上述PCR产物纯化后连接于pGEM T easy,酶切鉴定正确后送样测序。以BamHⅠ和PstⅠ酶切与pGEM T easy连接的两种重组质粒,以PstⅠ和SacⅠ酶切质粒pEYW24,回收cotB、cotZ和gfp片段,克隆于枯草芽孢杆菌整合型质粒pLX-2上,得到重组质粒pEYW25和pEYW26,构建流程图见图2。
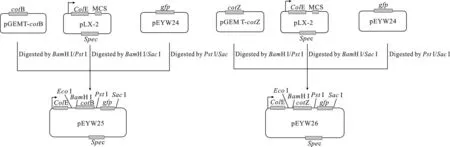
图2 原位整合载体pEYW25和pEYW26的构建流程 cotB,cotZ为芽孢外壳蛋白编码基因;ColE为大肠杆菌复制子;MCS为多克隆酶切位点; Spec为壮观霉素抗性基因;gfp为绿色荧光蛋白编码基因Fig.2 Flow diagram of construction strategy for single crossover integration vectors pEYW25 and pEYW26 cotB,cotZ:the coding genes of spore coat protein;ColE:replicon of E.coli;MCS:abbreviation of multiple cloning site;Spec:Spectinomycin resistance gene;gfp:the coding gene of green fluorescence protein
1.2.2 原位整合型重组菌的构建 将鉴定正确的整合型重组质粒pEYW25和pEYW26转化枯草芽孢杆菌,质粒上带有的基因片段cotB和cotZ与枯草芽孢杆菌基因组上的同源片段发生单交叉同源重组,整个质粒被线性化地整合到染色体上(图3)。
1.2.3 分子生物学操作 DNA的酶切、连接和转化大肠杆菌均按照分子克隆实验指南第三册的方法进行,质粒DNA的提取,酶切产物回收均按照试剂盒说明进行操作。
1.2.4 枯草芽孢杆菌的转化 采用Spizizen法[5]制备BacillussubtilisPY79感受态细胞,-80 ℃保存。转化时将感受态细胞于45 ℃融化,加入适量的DNA,37 ℃,80 r/min培养30 min,将感受态细胞涂布在含壮观霉素的LB平板上,37 ℃过夜培养后进行筛选鉴定。

图3 重组菌BSYW25和BSYW26的构建流程图 cotB,cotZ为芽孢外壳蛋白编码基因;ColE为大肠杆菌复制子;MCS为多克隆酶切位点; Spec为壮观霉素抗性基因;gfp为绿色荧光蛋白编码基因Fig.3 Flow diagram of construction strategy for recombinant strains BSYW25 and BSYW26 cotB,cotZ:the coding genes of spore coat protein;ColE:replicon of E.coli; Spec:Spectinomycin resistance gene;gfp:the coding gene of green fluorescence protein
1.2.5 枯草芽孢杆菌芽孢的制备及芽孢衣壳蛋白的提取 采用营养耗尽法[6]诱导枯草芽孢杆菌生孢,5 000 r/min离心10 min收集芽孢,重悬于1 mL无菌水中,加入终浓度为2 mg/mL的溶菌酶37 ℃处理1 h杀死营养体细胞,离心取上清,1 mL无菌水重悬。
取芽孢悬浮液离心去上清,加入适量的SDS-DTT溶液,37 ℃水浴保温2 h,用pH 8.0的1 mol/L Tris-HCl缓冲液洗涤数次,芽孢用等体积的裂解缓冲液重悬,置于冰上超声破碎,低温下8 500 r/min离心20 min收集芽孢,沉淀用PBS重悬。
1.2.6 枯草芽孢杆菌芽孢的显微拍摄 取10 μL芽孢悬浮液滴于载玻片上,盖上盖玻片,置于NIKON正置荧光显微镜Ni-U载物台上,将激发光调至蓝光档观察绿色荧光,利用NIS-Elements图像处理软件中的Color DS-Ri2调整曝光时间,在100倍油镜下观察荧光。
1.2.7 Western blot 分析 取200 μL的芽孢外壳蛋白提取液,加入等体积的上样缓冲液,95 ℃水浴10 min,10 000 r/min离心10 min取上清进行SDS-PAGE,电泳完毕后,将分离胶浸在TBST转膜缓冲液5~10 min,电转采用恒流电转,电流大小由膜面积而定。电转使蛋白转移到PVDF膜上,然后将膜在封闭液中封闭1 h,加入兔源GFP一抗,室温反应2 h,再加入HRP标记的羊抗兔二抗,用HRP-DAB底物试剂盒进行显色反应。
2 结果与分析
2.1 原位整合型重组质粒的构建
BamHⅠ和SacⅠ酶切鉴定质粒pEYW25和pEYW26,分别出现大小约为1.5 kb,1.3 kb的目的条带和1.8 kb大小的载体片段,与预期结果一致,表明含有融合基因cotB-gfp和cotZ-gfp的重组质粒构建成功,结果如图4。

图4 质粒pEYW25和pEYW26酶切鉴定 1,4:EcoRⅠ和SacⅠ双酶切质粒pEYW25, pEYW26; 2:Trans 1 Kb DNA Ladder;3:Trans 2Kb Plus DNA LadderFig.4 pEYW25 and pEYW26 digested by restriction enzyme 1,4:pEYW25 and pEYW26 digested by EcoRⅠ and SacⅠ; 2:Trans 1Kb DNA Ladder; 3:Trans 2Kb Plus DNA Ladder
2.2 融合表达CotB-GFP、CotZ-GFP的原位整合型重组菌的构建
分别用BPcotB-up/down,PSGfp-up/down,BPcotB-up和PSGfp-down验证cotB-gfp片段是否插入染色体中,用BPcotZ-up/down,PSGfp-up/down,BPcotZ-up和PSGfp-down验证cotZ-gfp片段是否插入染色体中,PCR鉴定结果如图5,分别出现了大小约为800 bp的cotB,750 bp的gfp,1.5 kb的cotB-gfp,600 bp的cotZ,750 bp的gfp,1.3 kb的cotZ-gfp的目的条带,证明融合片段成功地整合到染色体中,每个融合基因的转录都受自身启动子的调控。鉴定正确的菌种分别命名为BSYW25和BSYW26。
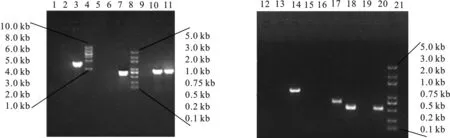
图5 重组菌BSYW25和BSYW26基因组的PCR鉴定
4:Trans 1 Kb DNA Ladder;8,21: Trans 2K Plus DNA Ladder;1,2,3:分别以ddH2O,BacillussubtilisPY79,BSYW25 genome为模板,以BPcotB-up/PSGfp-down为引物对融合基因片段cotB-gfp的扩增产物;5,6,7:分别以ddH2O,BacillussubtilisPY79,BSYW25 genome为模板,以PSGfp-up/down为引物对基因片段gfp的扩增产物;9,10,11:分别以ddH2O,BacillussubtilisPY79,BSYW25 genome为模板,BPcotB-up/down为引物对基因片段cotB的扩增产物;12,13,14:分别以BacillussubtilisPY79, ddH2O, BSYW26 genome为模板,BPcotZ-up/PSGfp-down为引物对融合基因片段cotZ+gfp的扩增产物;15,16,17:分别以BacillussubtilisPY79,ddH2O,BSYW26 genome为模板,PSGfp-up/down为引物对基因片段gfp的扩增产物;18,19,20:分别以BacillussubtilisPY79,ddH2O,BSYW26 genome为模板,BPcotZ-up/down为引物对基因片段cotZ的扩增产物
Fig.5 PCR-amplificated production of recombinant strain BSYW25 and BSYW26
4:Trans 1 Kb DNA Ladder;8,21:Trans 2 K Plus DNA Ladder;1,2,3:Amplification of fusion gene fragmentcotB-gfpusing ddH2O,BacillussubtilisPY79, BSYW25 genome as template and BPcotB-up/PSGfp-down as primer;5,6,7:Amplification of gene fragment gfp using ddH2O,BacillussubtilisPY79, BSYW25 genome as template and PSGfp-up/down as primer;9, 10,11:Amplification of gene fragment cotB using ddH2O,BacillussubtilisPY79,BSYW25 genome as template and BPcotB-up/down as primer;12,13,14:Amplification of fusion gene fragmentcotZ-gfpusingBacillussubtilisPY79,ddH2O,BSYW26 genome as template and BPcotZ-up/PSGfp-down as primer;15,16,17: Amplification of gene fragment gfp usingBacillussubtilisPY79,ddH2O,BSYW26 genome as template and PSGfp-up/down as primer;18,19,20:Amplification of gene fragmentcotZ
usingBacillussubtilisPY79,ddH2O,BSYW26 genome as template and BPcotZ-up/down as primer
2.3 重组菌BSYW25和BSYW26芽孢表面展示GFP的鉴定
将鉴定正确的重组菌BSYW25和BSYW26按1:50~100的比例接种于液体LB培养基中培养,分别在24 h和48 h取菌液,制备芽孢在荧光显微镜下观察,以BacillussubtilisPY79为对照。培养20 h的重组菌和BacillussubtilisPY79均为营养细胞,检测不到荧光信号(结果未显示),培养48 h的重组菌和BacillussubtilisPY79大部分为芽孢,BSYW26在荧光显微镜下能观察到荧光,而BSYW25和BacillussubtilisPY79均不能观察到荧光,结果如图6。
2.4 Western blot检测重组菌芽孢表面展示的GFP
为了进一步验证GFP是否被锚定在重组菌芽孢上,Western blot分析重组菌芽孢外壳蛋白提取液,以BacillussubtilisPY79的芽孢外壳蛋白为对照,结果见图7,重组菌BSYW26的芽孢中出现大小约为55 kDa的目的条带(CotZ-GFP融合蛋白,条带1和2),对照组没有出现对应的目的条带(条带3和4),表明GFP锚定在了重组菌芽孢上。

图6 重组菌芽孢表面GFP的荧光检测(10×100 油镜) a,b:分别为重组菌BSYW26在荧光和白光下;c,d:分别为重组菌Bacillus subtilis PY79在荧光和白光下Fig.6 Detection of GFP on spore surface of recombinant strain(10×100 oil lens) a,b:spores of recombinant strain BSYW26 under fluorescence and visible light;c,d:spores of Bacillus subtilis PY79 under fluorescence and visible light

图7 Western blot 分析重组菌BSYW26芽孢表面GFP 1,2.重组菌BSYW26;3,4.Bacillus subtilis PY79Fig. 7 Analysis of GFP on spore of recombinant strain BSYW26 by Western blot 1,2. the spore of recombinant strain BSYW26; 3,4. the spore of Bacillus subtilis PY79
3 讨 论
影响枯草芽孢杆菌芽孢表面展示效率的因素包括:目的蛋白的表达量、稳定性和属性;锚定蛋白的定植位点和特性;锚定蛋白与目的蛋白的连接方式;表达载体和宿主菌的类型等。锚定蛋白的选择在芽孢表面展示的成功中起着关键作用,因为锚定蛋白的定植位置和特性影响芽孢表面展示的效率,同一种目的蛋白通过不同的锚定蛋白来展示,其最终的展示效率存在很大差异,Hinc等[7]利用芽孢外壳蛋白CotB、CotC和CotG来展示外源蛋白UreA,结果表明外壳蛋白CotC的展示效率最高。Wang等[8]利用Crust层蛋白CotY和CotZ展示外源蛋白β-半乳糖苷酶(β-galactosidase),结果显示CotZ展示β-半乳糖苷酶的效率明显高于CotY。Hinc等[9]在外壳蛋白的编码基因cotB的3'端分别使用两种不同的连结肽(GGGGS和GGGEAAAKGG G)与UreA编码基因的5'端融合,结果显示CotB-GGGGS-UreA融合蛋白无法成功地展示在枯草芽孢杆菌芽孢表面,原因可能为短的连结肽没能形成二级结构,不能有效地稳定融合蛋白的结构,而较长的连结肽GGGEAAAKGGG在融合蛋白翻译后修饰的过程中可以形成稳定的α螺旋结构,该结构能有效地提高表面展示的效率,Negri等[10]在相关的研究中也得出同样的结果。目的蛋白的大小和属性是决定展示成功的决定因素,当展示的目的蛋白较大且比较复杂时芽孢表面展示则可能失败[11]。
枯草芽孢杆菌芽孢外壳蛋白CotB和CotZ已在研究中被证实能够用于展示外源蛋白[7-8,12-14],Imamura等[2]利用抗GFP抗体证明了蛋白CotZ位于Crust层,CotB位于outer coat层并且抗体无法与CotB-GFP蛋白结合,通过cotZ的敲除证明外壳蛋白CotZ在Crust层蛋白沉积在芽孢周围的过程中起关键作用,cotZ基因的缺失会导致Crust层蛋白在芽孢周围分布不均匀。cotB基因的缺失则对outer coat 层的形成没有影响,但是否影响融合蛋白CotB-GFP的沉积过程还需进一步验证。上述大部分研究均采用淀粉酶基因位点异位整合的方式,而有关原位整合方式在枯草芽孢杆菌芽孢表面展示应用的报道较少。原位整合与异位整合的不同在于原位整合可使芽孢衣壳蛋白编码基因的阅读框被破坏,相应地,使得芽孢衣壳蛋白的合成受阻,重组菌内合成的融合蛋白在生孢过程中的定植可能出现紊乱,造成表面展示的失败。
本研究采用原位整合的方式,外壳蛋白的结构被破坏,这能初步证明外壳蛋白的破坏是否影响相应融合蛋白的展示。重组菌BSYW26芽孢表面能检测到荧光表明CotZ蛋白的破坏没有影响融合蛋白CotZ-GFP在芽孢表面的展示。而重组菌BSYW25芽孢表面展示GFP蛋白的失败则可能存在两方面原因:第一,由于各种芽孢外壳蛋白在装备到芽孢表面的过程中存在“优先性”,融合蛋白CotB-GFP在这一过程中则可能被蛋白酶降解,进而导致表面展示的失败;第二,融合蛋白CotB-GFP之间的连结肽较短,不能形成有效的二级结构,融合蛋白结构不稳定。
4 结 论
本研究表明,原位整合的方式能使芽孢的Crust层蛋白CotZ作为锚定蛋白展示外源蛋白,而芽孢的Outer coat 层蛋白CotB在芽孢表面展示方面的应用还需进一步的改进。

