改进的β-NMF在基因聚类中的应用
游春芝,崔 建
(山西医科大学 汾阳学院 基础医学部,山西 吕梁 032200)
基因芯片技术的快速发展使得越来越多的实验人员可以对成千上万的基因表达水平进行研究,因此受到很多生物学家的青睐.这也使得基因表达数据聚类成为生物信息中比较活跃的一个领域,如发掘基因路径、基因聚类和基因功能预测等.目前用于基因表达数据聚类的方法有K-means聚类[1]、主成分(PCA)[2]聚类法、独立成分分析[3](ICA)聚类算法等.其聚类的结果与实验时所用的数据和聚类时所用的相似度函数有很大的相关性,带有一定的主观性.虽然PCA和ICA都能进行数据的特征提取聚类,但是他们只能提取到整体的特征,而且对数据进行描述时允许数据中存在负数.而NMF[4]不但能够提取数据的整体特征而且也可以提取部分特征并对原数据进行纯加性的线性组合描述,所以NMF被认为是一种对数据分析很有用的技术,它被广泛地应用到语音信号处理[5]、数据聚类[6]、图像分析[7]等研究领域.但是这种算法不能够产生基于部分的表示结果,这就使得利用分解所得矩阵在聚类时产生一定的困难,而且标准NMF算法效率较低.文章针对基因表达数据高维、高噪等特点提出了一种改进的基于正交约束的β散度非负矩阵分解算法,用于基因表达数据,并进行聚类实验.
1 非负矩阵分解
对于一个m×n的基因表达数据矩阵V,NMF的目标是把它分解成两个大小分别为m×k和k×n的非负矩阵W和H,满足
V≈WH
(1)
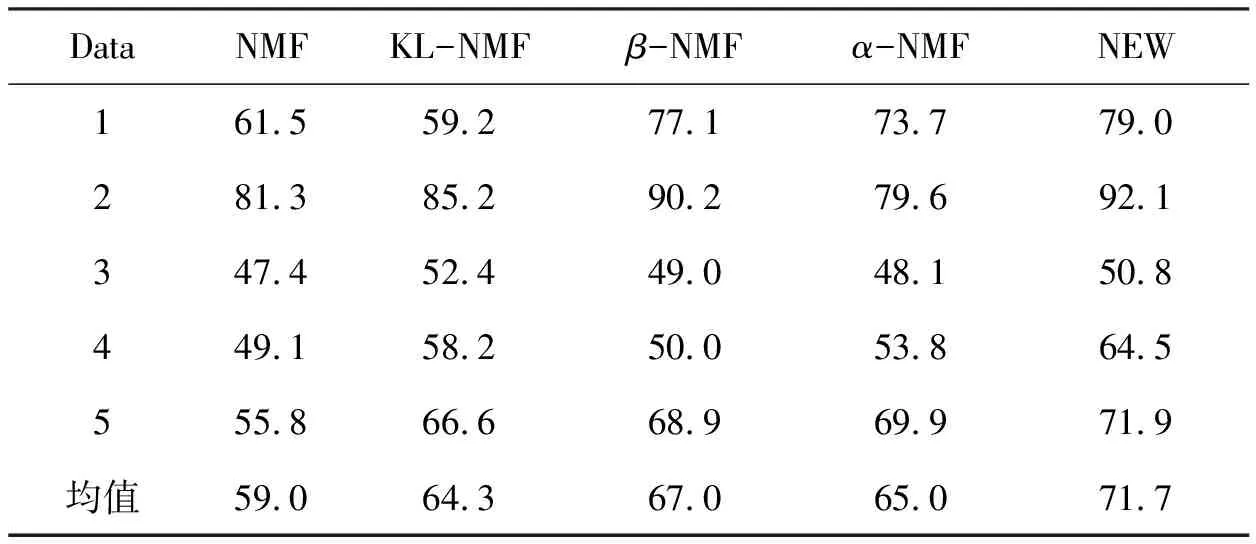
其中分解因子k满足(m+n)k NMF求解实际是一个NP问题,可以转化成优化问题,利用迭代方法求解W和H,为判断迭代收敛性,NMF常用的目标函数有两种,一种是欧几里得距离(ED): (2) 当且仅当V=WH时达到最小值0.另一种是KL散度[8]: (WH)ij) (3) KL散度是V和WH的相对熵,因为它不是对称的,所以不是一个距离,同式(2)一样,当且仅当V=WH时达到最小值0.该优化问题,就是在约束条件W>0,H>0下对D(V‖WH)最小化.对于上述两个目标函数中只能对W或者H凸,而不是同时对二者凸,找到一个全局最优解是不现实的,所以只能得到局部最优解. Lee和Seung[4]提供了一种基于梯度下降的乘法更新算法,对于式(2)的更新规则为 (4) (5) 对于式(3)的更新规则为 (6) (7) 其中i=1,2,…,m;j=1,2,…,n;⊗表示对应的元素乘积.目前提出的非负矩阵分解的目标函数有L2范数平方距离、KL散度、α散度[9]、β散度[10]等. 非负矩阵分解的重要性在于它能产生直观的而且是基于部分的表示,这样能够对原有的基因表达数据作出很好的描述.Cichocki[10]等人提出了β散度的非负矩阵分解,其目标函数为 (8) 在W>0,H>0的限制条件,使得准则函数也就是目标函数最小.β取值为1时,式(8)与式(2)等价,即为KL散度.用标准的梯度下降方法,式(8)有如下迭代规则:β→0,有 (9) (10) 为了进一步提高β散度的非负矩阵分解的局部表示能力,对β散度矩阵分解引入正交约束[11-12],其目标函数如下: (11) 其中I表示为单位矩阵,λ为权重系数.将式(11)目标函数分别对W和H求偏导得到: 2λ(WWTW)-2λW (12) (13) 用标准的梯度下降法,有 (14) (15) 为了让矩阵W和H保持其非负性,分别取步长ηW和ηH为 (16) (17) 分别代入,得到迭代规则如式(18)(19): (18) (19) 基于上述新的乘积更新原则,将该算法归纳为如下: ① 初始化:输入V,k待定,初始矩阵W1和H1为非负随机矩阵,并设定参数β,λ; ② 迭代更新:将初始矩阵W1和H1带入式(18)和式(19),求解W和H; ③ 终止条件:如果目标函数值小于阈值或者是最大的迭代次数,则终止算法;否则返回②. 在验证改进算法的有效性时,选择5种标准的肿瘤基因数据作为实验对象,其数据来自于http://www.gems-system.org/.由于DNA微阵列数据中每个点的信号强度是前景信号减去背景信号,因此会出现负值,而负值没有生物意义,取其绝对值作为基因表达水平.为了能够体现算法的有效性,将每个样本随机排列,其中每一个数据集中第一列为真实的聚类标签,其余列为有效基因表达数据.实验数据如表1所示. 表1 肿瘤基因表达数据Table 1 Tumor gene expression data 目前对于基因表达数据聚类效果评价的方法有聚类准确率、归一化互信息[13-14],也有文章用聚类的稳定性来衡量聚类效果[15].本文取聚类准确率作为实验结果,来衡量新算法的有效性.在计算聚类准确率时,通过对比获得的样本标签与给定数据集的标签一致性来实现.对于给定的包含m个样本的属于c类的微阵列数据集,实验中类别c是给定的,对于给定样本,cn是通过预测得到的样本标签,rn是数据集中附带的肿瘤类标签.聚类的准确率用式(20)计算: (20) 其中,I(X,Y)表示一个符号函数,当X=Y时,I(X,Y)=1,否则I(X,Y)=0,map(cn)是一个从聚类实验所得标签到已知标签之间的一个最大映射. 实验中将改进的算法(NEW)与基于二范数的NMF(ED),KL-NMF,α-NMF,β-NMF 5种方法分别对基因表达数据矩阵Vm×n分解.其中m表示样本数,n表示特征维数即基因个数.将得到的矩阵Wm×k作为特征矩阵用于聚类实验,取分解因子k为10,参数β取值0.2,根据文献[11],λ为5.考虑到初始矩阵选取对聚类结果的影响,取随机矩阵20次分别对其进行分解,最后将分解所得矩阵分别用K-means聚类实验,取其平均的聚类效果作为实验结果,如表2所示. 表2 各算法下的聚类精度Table 2 Clustering accuracy of each algorithm 从表2中可以看出,改进的算法平均聚类精度达到71.7%,比传统的NMF高出12个百分点,比KL-NMF高出7个百分点,比β-NMF提高了4个百分点,比α-NMF高出6个百分点.在数据2上聚精度达到92.1%.总体分析,改进的算法聚类效果要比其他4种算法明显. 在β散度的非负矩阵分解基础上,对该算法进行了改进.通过增加正交性约束,使得低维特征具有很好的区分能力.实验结果表明改进的算法具有很好的聚类效果.虽然该算法在应用中取得一定的优势,但还存在不足,例如初始矩阵的随机选择,导致得到的分解矩阵结果不稳定,如何选择更合适的参数β和更稳定有效地方法还有待研究.

2 改进的β散度的非负矩阵分解
2.1 β散度的非负矩阵分解
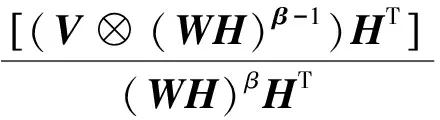
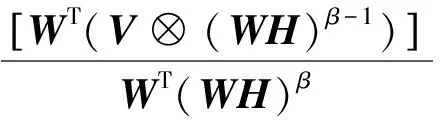
2.2 改进的β散度的非负矩阵分解
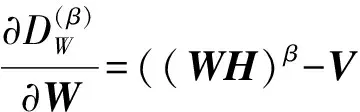
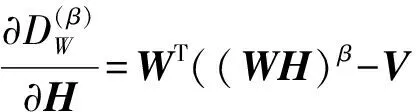
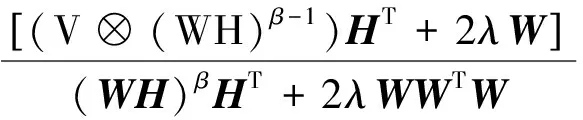
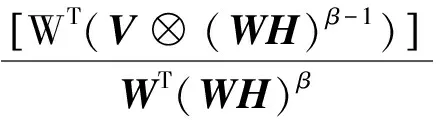
3 实 验
3.1 数据的预处理

3.2 基因表达数据聚类效果评价方法
3.3 实验结果分析
4 结 语

