异源表达Hvsusiba2水稻对稻田甲烷排放及土壤相关菌群的影响*
苏 军, 单 贞, 陈在杰
异源表达2水稻对稻田甲烷排放及土壤相关菌群的影响*
苏 军, 单 贞, 陈在杰
(福建省农业科学院生物技术研究所/福建省农业遗传工程重点实验室 福州 350003)
2是调控大麦淀粉合成和光合产物分配的转录因子。前期研究我们将2导入粳稻L.subsp),2粳稻稻田甲烷排放显著下降, 胚乳淀粉含量显著提高。为进一步明确2对稻田甲烷排放的影响, 本研究我们将2导入籼稻(L.subsp.), 研究异源表达2籼稻全生育期甲烷排放和稻田主要甲烷菌及甲烷氧化菌变化。采用静态箱法测定2水稻稻田甲烷排放通量, 结果显示2稻田全生育期的大部分时段甲烷排放量显著(<0.05)或极显著(<0.01)低于对照株系。2水稻甲烷减排率幅度为54.7%~3.8%, 减排率最高的时期为幼穗分化期。2个2水稻株系生长季累计甲烷排放量分别为5 060.16mg×m-2和5 250.60 mg×m-2, 比对照减排30.30%和27.58%。采用荧光定量PCR法检测水稻关键生长期根土6类产甲烷菌和2类甲烷氧化菌以及土壤总细菌的丰度变化。结果显示: 在整个生长期内2水稻根土6类产甲烷菌菌群丰度的总体趋势是前期高、后期低; 甲烷古菌(Archaea,ARC)、甲烷鬃菌科(Methanosaetaceae,Mst)和甲烷微菌目(Methanomicrobiales,MMb)3类菌群丰度的高峰出现在分蘖盛期, 甲烷八叠球菌科(Methanosarcinaceae,Msc)菌群丰度的高峰出现在幼穗分化穗期, 普通产甲烷菌(Methanogens,MET)和甲烷杆菌目(Methanobacteriales, MBT)分蘖期最高。2水稻产甲烷菌丰度在分蘖期、抽穗期和开花期显著或极显著地低于野生型对照。在大部分测试时间段内2水稻的2类甲烷氧化菌群丰度比对照有显著(<0.05)或极显著(<0.01)下降;2水稻土壤总细菌丰度在水稻的分蘖期、抽穗期和开花期也显著低于野生型水稻。稻田中产甲烷菌的丰度依次是甲烷鬃菌科(Mst)>甲烷古菌(ARC)>普通产甲烷菌(MET)>甲烷微菌目(MMb)≥甲烷八叠球菌科(Msc)>甲烷杆菌目(MBT); 2类甲烷氧化菌中Ⅰ型甲烷氧化菌(MBAC)丰度极显著大于Ⅱ型甲烷氧化菌(TYPEⅡ)。结合之前的研究结果, 我们认为2可能是通过改变水稻光合同化物分配生理, 减少向土壤有机质的输送, 降低土壤相关菌群的丰度达到稻田甲烷减排的。
水稻;2异源表达; 甲烷减排; 产甲烷菌; 甲烷氧化菌; 菌群丰度
稻田是最重要的农业甲烷排放源之一[1], 估计每年向大气中排放甲烷达50~60 Tg[2]。稻田甲烷排放包括甲烷产生、氧化和释放3个环节[3-7]。水稻(L.)生长过程中根系分泌的有机酸连同根脱落物在土壤微生物的作用下转变成为可被产甲烷菌利用的底物, 如CO2、H2、乙酸盐等简单分子。在厌氧条件下, 产甲烷菌利用这些小分子物质产生甲烷。所产生的甲烷大部分被根际土壤中的甲烷氧化菌氧化消耗, 剩余的少部分甲烷通过淹水层或者水稻植株通气组织排放到大气中去。通过这一系列复杂而又微妙的过程构成整个稻田甲烷的循环。
在稻田甲烷的循环系统中, 水稻品种、产甲烷菌和甲烷氧化菌是决定稻田甲烷产生和排放的关键因素。连接这3个因素的是土壤中的碳(碳库), 水稻品种通过根将光合同化碳以有机酸形式分泌到土壤中, 产甲烷菌以土壤中的碳为生存和繁殖的基质代谢产生甲烷, 甲烷氧化菌以甲烷为底物氧化消耗甲烷, 未被消耗的甲烷通过植株通道排放到大气中。因此, 水稻品种遗传背景所决定的根系结构和生理以及光合代谢特征不仅影响了甲烷底物的供给和甲烷排放的通道特性, 也影响了甲烷相关菌的丰度、结构及活性[8-10], 最终叠加作用于甲烷排放。由品种因素造成的甲烷排放差异可达1.5~3.5倍[11-12]。因此如果改变水稻根系的生物量和生理特征、茎的形态结构或者水稻光合产物分配, 就有可能相应地改变土壤中的碳含量, 继而降低稻田甲烷排放。基于此, 我们设想通过调控水稻光合产物的分配, 即增加光合产物在地上部的分配, 减少光合产物地下部的分配有可能减少稻田甲烷的排放。前期研究我们将一个来源于大麦(L.)的转录因子2构建在淀粉合成基因特异启动子的下游, 利用启动子在籽粒中优势表达的特性, 增加水稻种子淀粉含量, 调控水稻植株体内碳水化合物分配, 降低稻田甲烷排放[13]。初步证实了我们的设想。由于前期研究所用的材料为水稻模式品种‘日本晴’(subsp.), 该品种属于粳稻亚种, 与目前南方广大稻区种植的籼稻亚种(subsp.)遗传背景有较大差异。本研究我们将该基因导入籼稻, 研究该基因表达对籼稻稻田土壤甲烷排放的影响, 进一步理解2基因在不同遗传背景下对稻田甲烷减排的效果, 同时通过对水稻生长期产甲烷菌和甲烷氧化菌的种群丰度变化的检测, 探讨2水稻甲烷减排的机制, 探索通过生物技术实现稻田甲烷减排的新途径。
1 材料与方法
1.1 试验材料
水稻品种: 转2基因水稻纯合株系‘86R10-1’和‘86R27-3’及其野生型籼稻‘MH86’。
质粒: 含有产甲烷菌或甲烷氧化菌16S rRNA的pMD-18T载体转化的大肠杆菌, 由福建省农业遗传工程重点实验室构建并保存。
1.2 试验方法
1.2.1 水稻种植
试验设3个处理(株系), 每个处理30株, 3次重复, 随机区组试验。试验地点为福建省农业科学院福州寿山试验站(26°00¢N, 119°18¢E)。试验时间为2016年4月28日—9月23日。4月28日浸种, 2日后37 ℃温箱催芽, 5月7日育秧, 6月8日移栽。单本插, 株行距35 cm×35 cm。试验地四周栽种4行水稻作为保护行。田间水肥管理按当地管理方式, 仅施无机肥。整个生长季稻田保持水层5 cm淹水状态。为避免采集气体时扰动土壤造成采气不准确, 在试验田中安装两座木桥, 方便搬运器材及采气; 同时, 为避免采气过程中搬动静态箱而扰动植株, 在水稻移栽1周后, 在待测植株上安装特制的铁质基座, 保护水稻及根部土壤。
1.2.2 气体采集及检测
插秧后3周开始第1次气体采集, 之后每周采集1 d, 每个采集日上午和下午各1次, 直到水稻成熟, 如遇台风、大雨等恶劣天气, 则采气工作顺延至下一个晴天。以1株为一个采集单位。每个重复采集2份样品, 3次重复, 即每个株系共6份样品。从罩上集气装置开始计时, 在10 min、20 min、30 min时各采集30 mL气体, 每个单位植株的3个时段样品(共90 mL)混装在同一铝箔复合膜密闭气袋中, 采集后的样品放置4 ℃保存待测。采集时间为2016年6—9月, 上午采气时间为10:00—12:30, 下午为14:30—17:00, 过程中记录静态箱内温度、水稻生长发育阶段。采集的气体用Agilent 7890B气相色谱仪检测甲烷, 以纯甲烷作为标准气体, 检测器为前检测器FID; 载气(He)流量1 mL×min-1, 尾吹气(N2)流量25 mL×min-1, 燃气(H2)流量30 mL×min-1, 空气流量400 mL×min-1; 柱箱温度70 ℃,检测温度250 ℃;出峰时间2.9 min。甲烷通量按以下公式计算:
273(273) (1)
式中:表示甲烷气体通量(mg·m-2·h-1),表示标准状态下甲烷的气体密度,为静态箱的箱高(),表示静态箱内甲烷气体浓度变化率(%),表示采集过程中静态箱内的平均温度(℃)。采用最小二乘法计算生长季累计排放量, 按下列公式计算生长季减排率: 生长季减排率=(对照累计排放量-处理累计排放量)/对照累计排放量×100%
1.2.3 水稻根土采集及土壤总DNA提取
选与气体样品采集植株生长一致的相邻植株为采样株, 用土壤采样器等量采集水稻植株4个不同方向土壤, 采样点距离水稻根部水平距离5 cm、垂直距离地表5 cm, 4份土壤样品均匀地混合为1组, 3组重复。采集时间为气体采集日当天14:00—14:30, 采集后的土壤放入干净的自封袋, 4 ℃放置。带回实验室后-20 ℃暂时储藏, 1周内提取土壤总DNA。
土壤DNA提取: 准确称取1.0 g土壤样品2份, 一份装入2 mL EP管烘干测定土壤水分, 另一份装入试剂盒提供的Lysing Matrix ETube, 用于提取土壤总DNA。土壤总DNA提取采用Fast DNA™ SPIN Kit for Soil试剂盒提供的方法。用核酸定量仪检测土壤总DNA浓度, 将所提土壤总DNA稀释至100 ng∙μL-1, 并放置于-20 ℃备用。
质粒标准样品制备, 分别提取不同菌种的标准质粒, 用核酸定量仪检测质粒浓度, 加入ddH2O将样品调整至1×1010copy×L-1作为原液。
1.2.4 根土产甲烷菌及甲烷氧化菌定量分析
根据文献, 选稻田中常见的6类产甲烷菌群和2类甲烷氧化优势菌群以及土壤总细菌为检测目标。6对引物对应的产甲烷菌分别是: 甲烷古菌域Archaea (ARC)、产甲烷菌Methanogens (MET)、甲烷微菌目Methanomicrobiales (MMb)、甲烷杆菌目Methanobacteriales (MBT)、甲烷八叠球菌科Methanosarcinaceae(Msc)和甲烷鬃菌科Methanosaetaceae (Mst), 其中的ARC和MET与所测定的其他产甲烷菌群有部分重叠。2对甲烷氧化菌引物对应的甲烷氧化菌为: Ⅰ型甲烷氧化菌Methytobacter/Met-hylosarcina (MBAC)和Ⅱ型甲烷氧化菌Methylosinus (TYPEII); 土壤总细菌用土壤细菌通用引物扩增。引物设计参考文献[14-17], 详见表1。
采用荧光定量PCR法分别在水稻分蘖期、幼穗分化期、开花期、灌浆期和成熟期等关键时期测定稻田土壤菌群丰度。PCR反应体系如下: SYBR 10 µL、浓度为0.2 µmol×L-1引物各1 µL、浓度为100 ng×μL-1DNA 1 µL、加水补至20 µL。扩增程序见表2。每份DNA均做3次平行重复。上述不同菌标准样品质粒梯度稀释, 制备及扩增102~108copy×L-1的梯度浓度标准品, 建立各菌对应基因的标准曲线, 根据标准曲线和扩增所得CT值计算单位样品中的基因拷贝数, 再根据土壤水分比换算得到单位干土中菌的含量[×106copies×g-1(DWS)]。
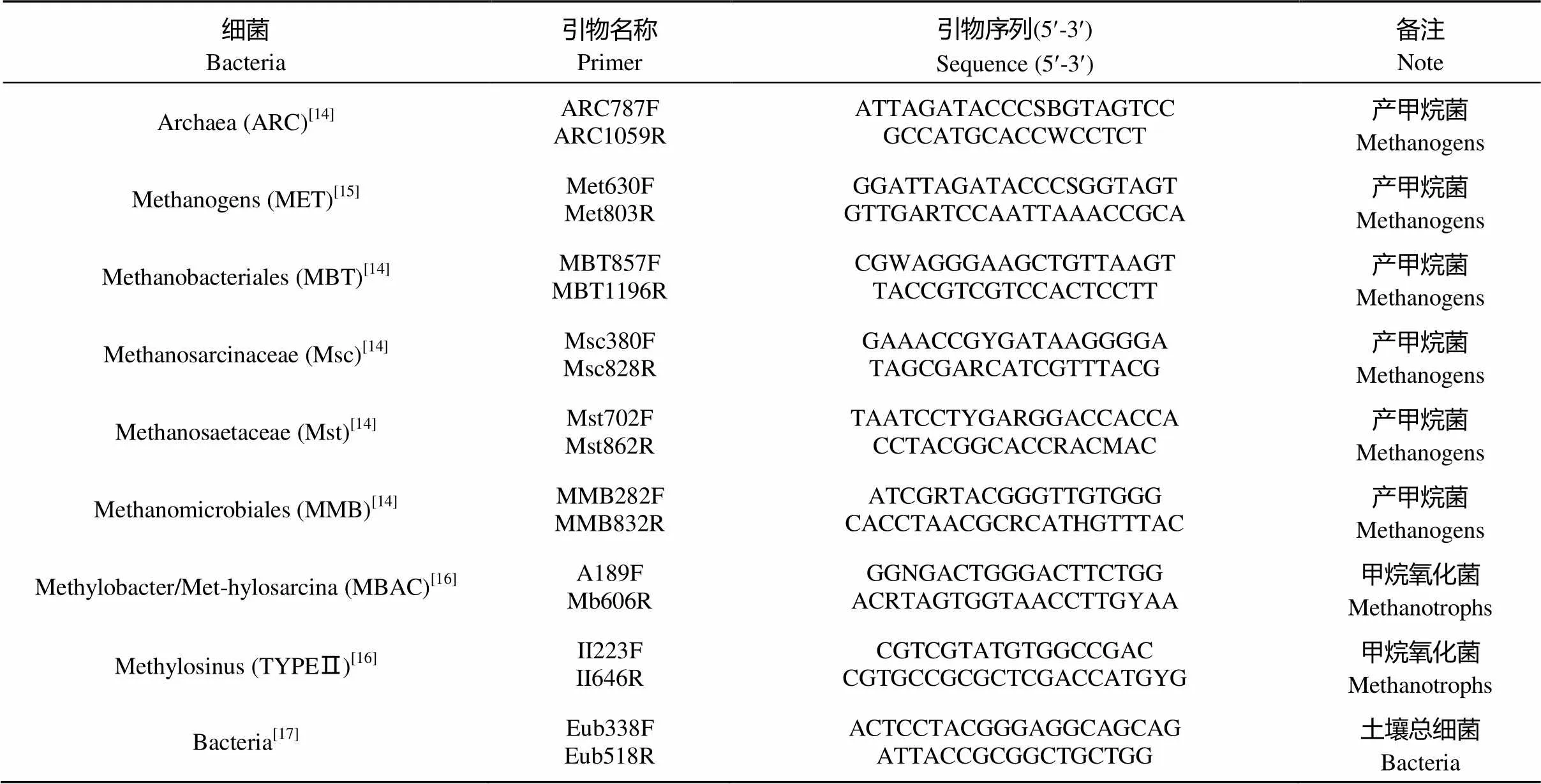
表1 产甲烷菌(群)和甲烷氧化菌(群)16S rRNA荧光定量PCR引物
表2 产甲烷菌(群)和甲烷氧化菌(群)荧光定量PCR扩增程序
Table 2 qPCR programs for 6 methanogens and 2 methanotrophs
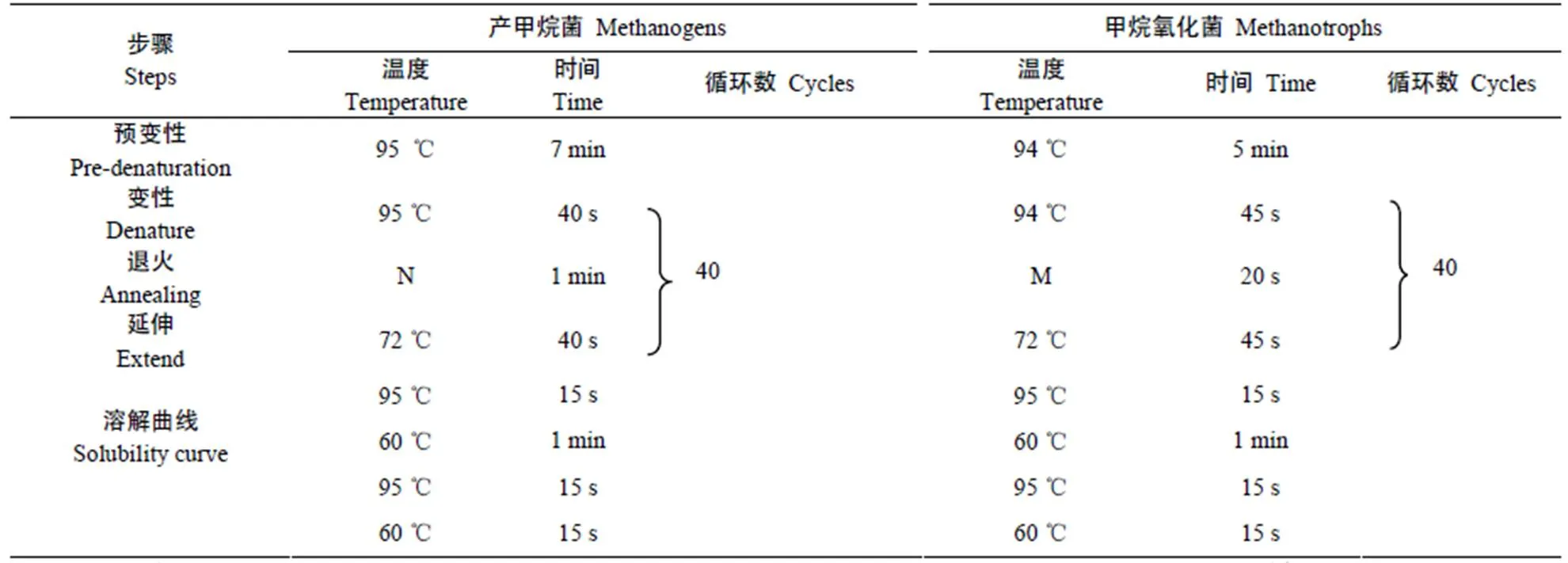
N: 产甲烷菌退火温度, ARC为60 ℃, Msc为60 ℃, Mst为61 ℃, MMb为66 ℃, MBT为58 ℃, Met为60 ℃。M: 甲烷氧化菌退火温度, MBAC为58 ℃, TYPEⅡ为65 ℃。N and M: PCR annealing temperature for methanogens andmethanotrophs, for ARC it is 60 ℃, for Msc it is 60 ℃, for Mst it is 61 ℃, for MMb it is 66 ℃, for MBT it is 58 ℃, for Met it is 60 ℃, for MBAC it is 58 ℃, for TYPEⅡ it is 65 ℃.
1.2.5 数据处理及作图
所有数据均用Microsoft Excel进行初步计算, 用SPSS(version 19.0; IBM)进行显著性分析, Excel制图。
2 结果分析
2.1 Hvsusiba2籼稻稻田甲烷排放特征分析
稻田甲烷排放特征见图1。2个表达2籼稻株系全生育期稻田甲烷排放趋势基本与对照相似。表现为随着水稻生长, 甲烷排放趋势呈波动状态上升。上午时段2个高峰期分别出现在幼穗分化期(播种后72 d)和开花灌浆期(播种后98 d )(图1A)。下午时段的排放趋势与上午时段略有不同, 对照‘MH86’有3个明显的高峰期分别出现在分蘖期(播种后59 d)、幼穗分化期(播种后78 d)和开花灌浆期(播种后98 d);2株系的甲烷排放趋势较缓和, 峰值较低(图1B)。不论是上午时段或下午时段,2水稻全生育期稻田甲烷排放量都明显低于对照‘MH86’, 在分蘖期、抽穗期、开花期以及灌浆期等生长旺盛期显著(<0.05)或极显著(<0.01)低于对照(图1A, B)。‘86R10-1’株系全生育期甲烷减排率幅度为54.7%~3.8%, 减排率最高的时期为幼穗分化期(播种后78 d)和成熟期(播种后118 d), 减排率分别达到54.7%和52.1%。‘86R27-3’全生育期甲烷减排率幅度范围和最高减排时期与‘86R10-1’株系相似。
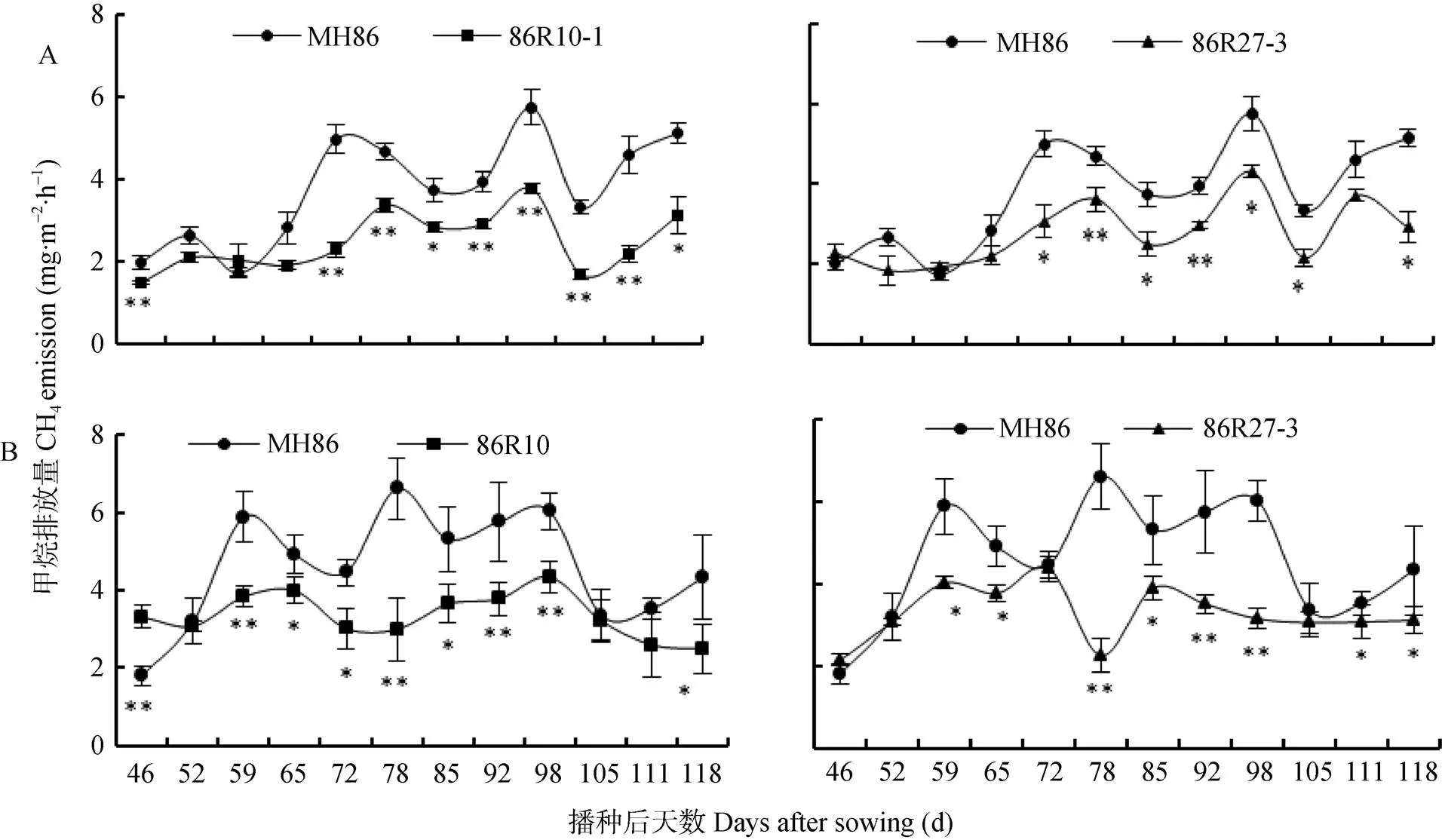
图1 播种后不同时间Hvsusiba2籼稻‘86R10-1’和‘86R27-3’稻田不同时段甲烷排放特征
A:10:00—12:30采集的样品; B: 14:30—17:00采集的样品。每个值是6个生物学重复的平均值。*和**分别表示2籼稻和对照‘MH86’的差异显著性达5%和1%水平。Figures A are samples collected at 10:00-12:30. Figures B are samples collected at 14:30-17:00. Each value is the mean of 6 biological replicates. * and ** indicate significance differences between2 indica rice and wild rice ‘MH86’ at 5% and 1% levels, respectively.
生长季累计排放量按第一次采气到最后一次采气的间隔时间73 d天计算, ‘86R10-1’生长季累计排放量为5 057.16 mg×m-2, ‘86R27-3’为5 250.24 mg×m-2, 对照‘MH86’累计排放量为7 249.68 mg×m-2, 2个2籼稻折算减排率分别为30.20%和27.60%。
2.2 表达Hvsusiba2籼稻全生育期根土总细菌的丰度变化
水稻根系分泌的有机酸及根脱落物等大分子有机物, 需要一系列细菌降解消化成H2、CO2、乙酸、甲醇等小分子物质, 才能进一步被甲烷菌所利用。这些细菌参与了包括甲烷前体形成的水解、发酵、纤维素降解、硫酸盐还原、互营、产酸产气等生化过程, 是甲烷产生过程中重要细菌[17]。甲烷是这一系列生化反应的终产物, 研究这些细菌的丰度和结构是评价甲烷排放的重要参数。2籼稻土壤中总细菌的丰度达到1011数量级, 高于甲烷菌109和甲烷氧化菌1010数量级。在水稻全生育期的变化趋势为分蘖期(播种后59 d)最高, 之后迅速下降。2籼稻土壤细菌在分蘖期(播种后52 d, 播种后59 d)、幼穗分化期(播种后78 d)以及开花期(播种后92 d)显著(<0.05)或极显著(<0.01)低于对照‘MH86’, 但灌浆期(播种后105 d)和成熟期(播种后111 d)与对照没有差异(图2)。2个籼稻株系全生育期土壤总细菌平均丰度分别为(31.82±3.70)×1010copies∙g-1(DWS)和(33.71±5.10)×1010copies∙g-1(DWS), 显著(<0.05)低于野生型‘MH86’的(42.59±4.38)×1010copies∙g-1(DWS)。
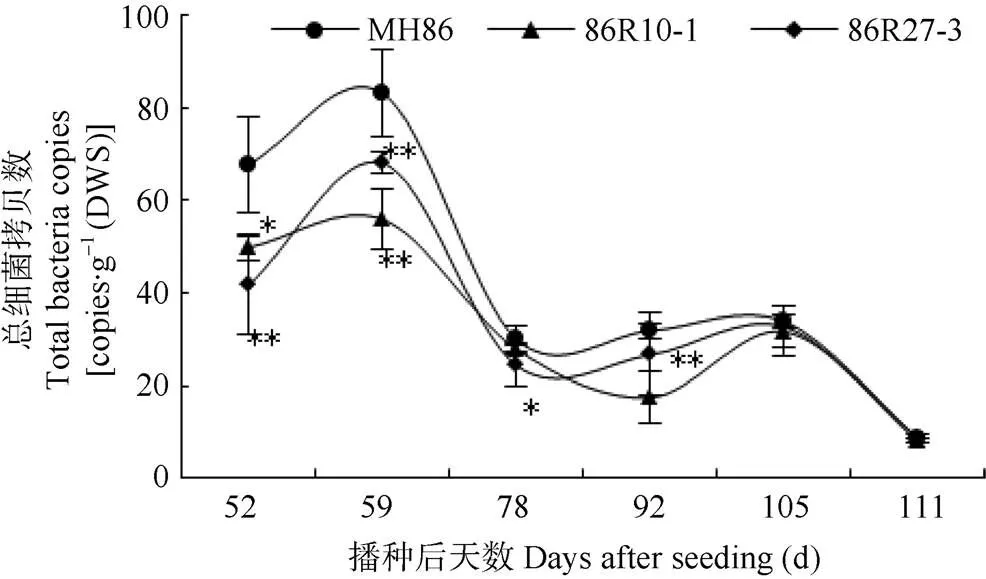
图2 Hvsusiba2籼稻‘86R10-1’和‘86R27-3’不同生育期根土总细菌丰度
每个值是3个生物学重复的平均值; *和**分别表示2籼稻和对照‘MH86’的差异达5%和1%水平。Each value is the mean of 3 biological replicates. * and ** mean significant differences between2 indica rice and wild rice ‘MH86’ at 5% and 1% levels, respectively.
2.3 Hvsusiba2籼稻全生育期根土6类产甲烷菌丰度动态变化
2个2水稻株系6类产甲烷菌丰度变化呈现出与水稻生长发育期密切相关的动态关系(图3)。总体是前期高、后期低, 但不同菌群表现不尽相同。其中ARCMst和MMb菌群丰度的高峰出现在分蘖期(播种后59 d), Msc出现在幼穗分化穗期(播种后78 d), MET和MBT几乎是分蘖期(播种后52 d)最高。随着生育期的推进, 所有菌群丰度逐渐下降, 除Msc在灌浆期(播种后105 d)最低外, 其余菌群在开花灌浆期(播种后92 d)丰度最低, 成熟期又略有上升。与对照‘MH86’相比, 除Mst在灌浆和成熟期与对照没有显著差异外, 2个2籼稻株系全生育期根土5类产甲烷菌丰度在水稻整个生育期的大部分时段均显著(<0.05)或极显著(<0.01)下降。
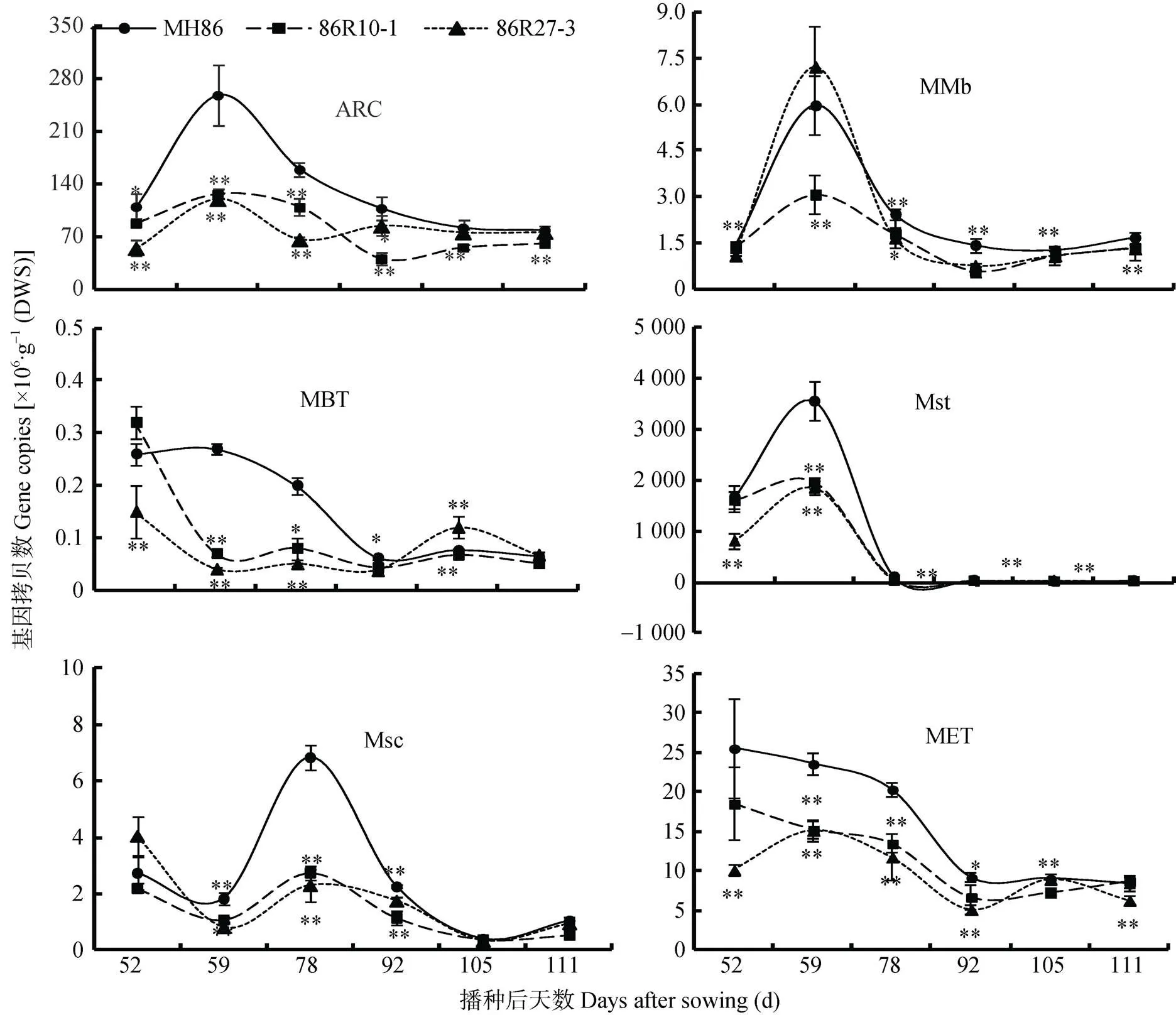
图3 表达Hvsusiba2籼稻‘86R10-1’和‘86R27-3’全生育期稻田根土6类产甲烷菌丰度变化
每个值是3个生物学重复的平均值; *和**分别表示2籼稻和对照‘MH86’的差异达5%和1%水平。Each value is the mean of 3 biological replicates. * and ** mean significant differences between2 indica rice and wild rice ‘MH86’ at 5% and 1% levels, respectively.
进一步分析表明, ‘86R10-1’根土产甲烷菌群量与对照相比, Mst、ARC和MBT最大减少量出现在分蘖期(播种后59 d), 分别为(1 599.00±282.45)×106copies∙g-1(DWS)、(131.80±14.75)×106copies∙g-1(DWS)和(0.12±0.01)×106copies∙g-1(DWS); ‘86R27-3’的Mst、ARC和MBT的减少量则为(1 707.40±257.60)×106copies∙g-1(DWS)、(138.85±34.42)×106copies∙g-1(DWS)和(0.15±0.01)×106copies∙g-1(DWS)。其余各生长期也都有不同程度减少; 2个株系Msc最大减少量则出现在幼穗分化期(播种后78 d), 分别为(4.09±0.24)×106copies∙g-1(DWS)和(4.54±0.28)×106copies∙g-1(DWS)。
将不同水稻株系同时期的土壤产甲烷菌总和与同期总细菌数进行比较(图4), 结果显示, 大部分测定期内2个2株系稻田土壤甲烷菌与总细菌的相对值比对照‘MH86’显著(<0.05)或极显著(<0.01)下降。虽然2水稻土壤产甲烷菌和总细菌丰度都显著下降(<0.05), 但2水稻土壤中产甲烷菌在总细菌中的占比下降, 说明2水稻对产甲烷菌的影响要大于对总细菌的影响。
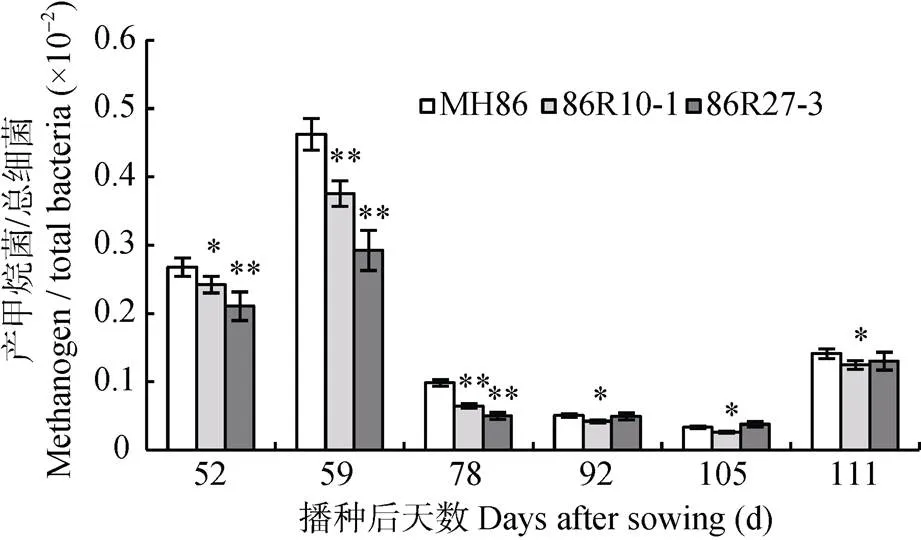
图4 Hvsusiba2籼稻‘86R10-1’和‘86R27-3’全生育期稻田土壤产甲烷菌的相对丰度(产甲烷菌/总细菌)
每个值是3个生物学重复的平均值; *和**分别表示2籼稻和对照‘MH86’的差异达5%和1%水平。Each value is the mean of 3 biological replicates. * and ** mean significant differences between2 indica rice and wild rice ‘MH86’ at 5% and 1% levels, respectively.
2.4 表达Hvsusiba2籼稻全生育期根土2类甲烷氧化菌丰度动态变化
2籼稻根土中2类甲烷氧化菌群MBAC和TYPEⅡ动态变化趋势如图5所示。2个2株系全生育期的MBAC丰度变化趋势与对照‘MH86’不一致, ‘86R10-1’和‘86R27-3’在分蘖期(播种后52 d)最高, 随着水稻生长进程, 逐渐下降, 在开花灌浆期(播种后92 d)最低, 后期略有提升; 而对照‘MH86’在分蘖期(播种后59 d)达到高峰, 之后逐渐下降, 在开花灌浆期(播种后92 d)降到最低, 之后又上升, 在灌浆期(播种后105 d)再次出现一个峰。‘86R10-1’和‘86R27-3’株系生育期内TYPEⅡ菌群的变化趋势与对照类似, 分蘖期和幼穗分化期较高。总体而言, 在大部分测试时段内2籼稻的2类甲烷氧化菌群丰度比对照有显著(<0.05)或极显著下降(<0.01)。
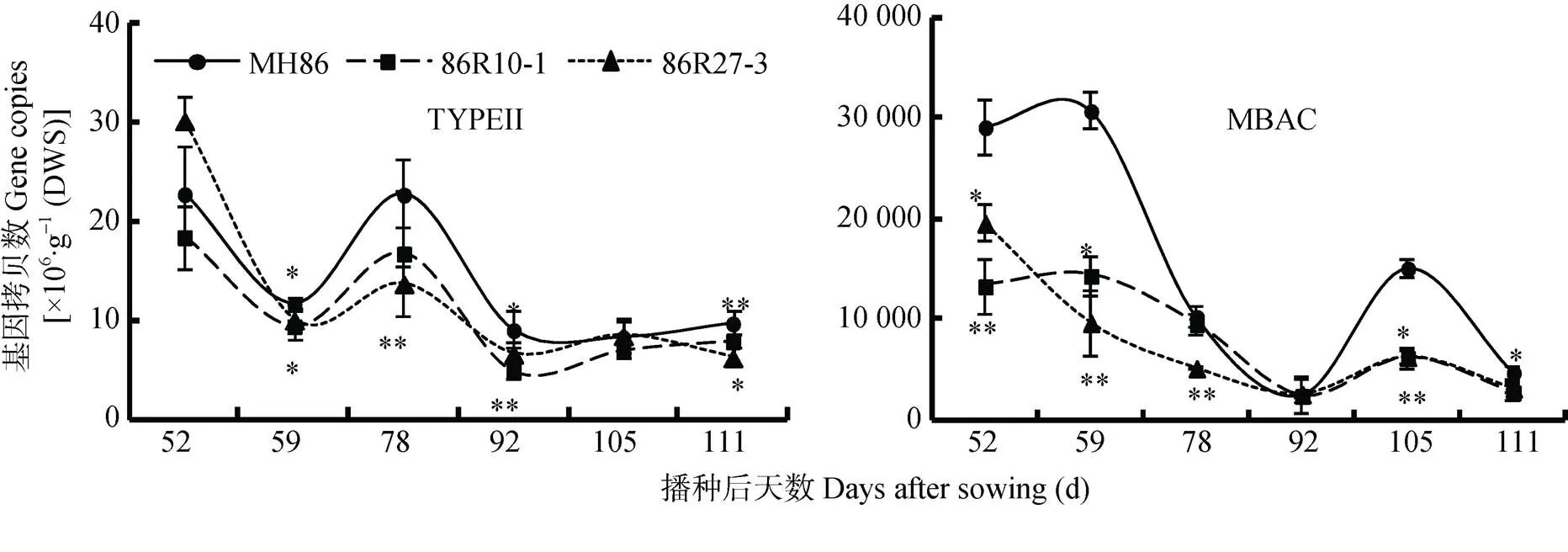
图5 Hvsusiba2籼稻‘86R10-1’和‘86R27-3’稻田土壤2类甲烷氧化菌丰度变化
每个值是3个生物学重复的平均值; *和**分别表示2籼稻和对照‘MH86’的差异达5%和1%水平。Each value is the mean of 3 biological replicates. * and ** mean significant differences between2 indica rice and wild rice ‘MH86’ at 5% and 1% levels, respectively.
‘86R10-1’和‘86R27-3’水稻根土中MBAC最大减少量出现在分蘖期(播种后59 d), 分别为(16 381.00± 2 092.10)×106copies∙g-1(DWS)和(21 038.00±2 092.10)× 106copies∙g-1(DWS)。‘86R10-1’和‘86R27-3’水稻根土中TYPEⅡ在根土中的最大减少量出现在幼穗分化期(播种后78 d), 分别为(6.00±3.63)×106copies∙g-1(DWS)和(9.06±2.00)×106copies∙g-1(DWS)。
从甲烷菌/甲烷氧化菌相对丰度看,2水稻甲烷菌/甲烷氧化菌比值显著提高(图6), 说明2水稻土壤甲烷氧化菌的降幅比产甲烷菌降幅大。甲烷氧化菌是以甲烷为惟一底物的土壤细菌, 甲烷氧化菌丰度减少, 表明土壤中可利用的甲烷浓度降低, 限制了甲烷氧化菌种群的繁殖。说明2水稻田甲烷减排的原因是甲烷产量降低了, 而非甲烷氧化消耗增加所致。
2.5 Hvsusiba2籼稻根土6类产甲烷菌和2类甲烷氧化菌结构
产甲烷菌是一个庞大的微生物种群, 目前已经鉴定了34个属的甲烷菌, 分属于7个目[18]。对6类稻田土壤常见产甲烷菌的检测显示: 整个生育期内, 水稻根土甲烷菌群平均丰度依次为甲烷鬃菌科(Mst)>古细菌域甲烷菌(ARC)>普通甲烷菌(MET)>甲烷微菌目(MMb)≥甲烷八叠球菌科(Msc)>甲烷杆菌目(MBT) (表3)。其中甲烷鬃菌为优势菌群, 在分蘖期可达109拷贝数, 甲烷古菌位于其次达到108拷贝数, 普通甲烷菌为107拷贝数, 丰度最低的是甲烷杆菌目仅105拷贝数。2个2株系菌群的群落结构与对照相比没有差异。
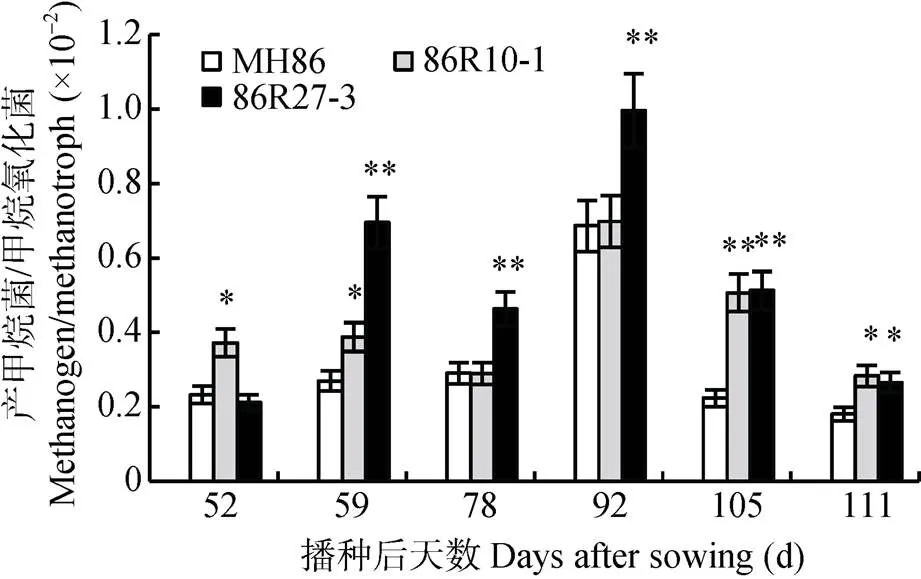
图6 Hvsusiba2籼稻‘86R10-1’和‘86R27-3’全生育期稻田土壤产甲烷菌相对丰度(产甲烷菌/甲烷氧化菌)
每个值是3个生物学重复的平均值; *和**分别表示2籼稻和对照‘MH86’的差异达5%和1%水平。Each value is the mean of 3 biological replicates. * and ** mean significant differences between2 indica rice and wild rice ‘MH86’ at 5% and 1% levels, respectively.
甲烷氧化菌是以甲烷为惟一碳源的功能微生物。主要有2类, 分别是MBAC和TypeⅡ。2籼稻根土中MBAC的丰度要极显著大于TYPEⅡ(表4), 达109~1010拷贝数, 而TYPEII仅为106~107拷贝数。表明2籼稻稻田中主导甲烷氧化菌群是MBAC, 占据压倒性优势。
此外, 结果还提示: ‘86R10-1’和‘86R27-3’株系与‘MH86’相比在生育期内各个阶段根土6类产甲烷菌和2类甲烷氧化菌丰度的位次顺序没有差异。
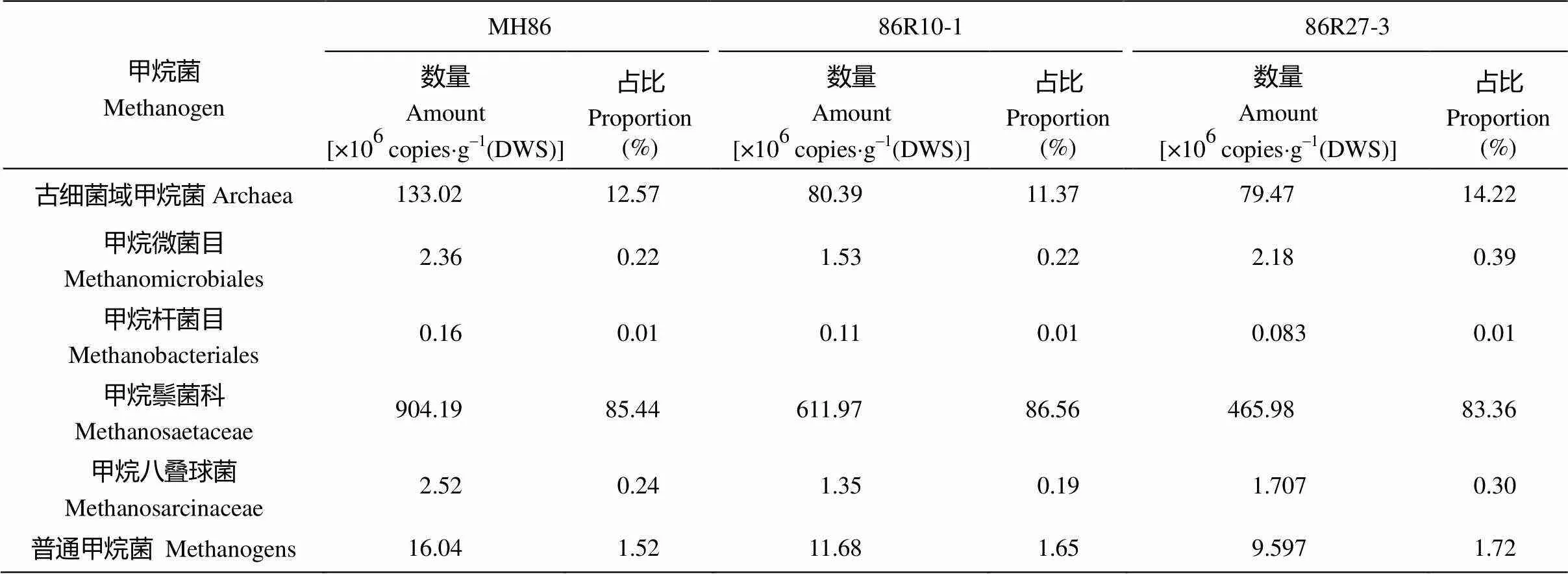
表3 6类甲烷菌在Hvsusiba2籼稻田土壤总甲烷菌中的比例
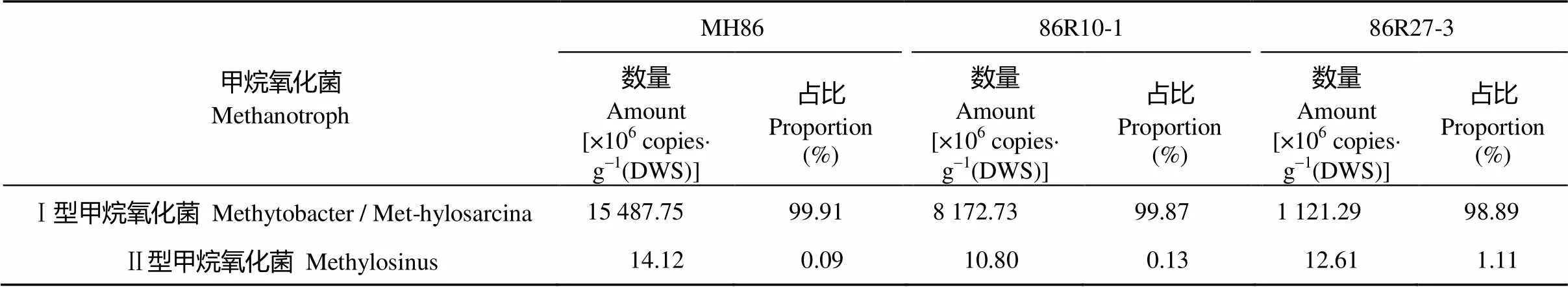
表4 2类甲烷氧化菌在Hvsusiba2籼稻田土壤中的比例
3 讨论和结论
水稻植株在稻田甲烷排放过程中起着十分重要的作用, 它既是稻田甲烷产生的基质供应者, 又是甲烷排放的主要通道[5]。因此不同品种植株形态和光合生理差异对稻田甲烷的排放有显著影响。由品种因素造成的甲烷排放差异可达1.5~3.5倍[11-12]。现代分子生物学表明: 通过基因操作是可以改变植株形态或光合生理的, 因此理论上通过基因操作降低甲烷排放是可能的。基于此, 我们将2基因导入水稻, 探讨生物技术降低稻田甲烷排放的可行性。2是一个糖信号诱导转录因子(sugar signaling in barley,2), 能够被蔗糖信号诱导激活, 激活后的2通过绑定大麦异淀粉酶基因(,ISO1)启动子区的SURE元件和序列, 调控异淀粉酶基因的转录水平, 促进淀粉合成[19-20]。我们将2构建在淀粉分支酶基因(,SBE)启动子p下游, 利用p启动子胚乳优势表达特性, 驱动2在胚乳表达, 促进胚乳淀粉累积, 目的是促进碳水化合物向籽粒运送, 减少向地下运送, 达到甲烷减排的效果。从甲烷排放趋势看, 全生育期稻田甲烷排放量都显著低于对照品种。结合2对粳稻‘日本晴’稻田甲烷减排的结果[13], 进一步证实了2降低水稻田甲烷排放量是可行的, 并且在不同的水稻遗传背景中的作用是稳定的。
稻田甲烷排放是产甲烷菌、甲烷氧化菌以及其他相关细菌共同作用后的结果。产甲烷菌和甲烷氧化菌位于土壤碳循环的末端, 产甲烷菌只能以CO2、H2、乙酸、甲酸等简单分子为底物[21], 土壤有机质必须降解成简单分子后才能被产甲烷菌利用。有机质的降解过程需要包括发酵、降解、酸化等众多微生物协同作用。稻田土壤总细菌丰度下降意味着这些功能菌群数量下降, 其结果是有机物的降解能力下降, 产甲烷底物供给量减少, 影响甲烷的生成。土壤总细菌下降也暗示土壤中供给微生物繁殖所需的有机物含量减少。稻田土壤有机质一般由两部分组成, 一部分是前茬作物及土壤原有有机质构成的本底, 另一部分是当季水稻生长过程中向土壤输入的新增有机质, 在水稻生长过程中如果没有外加有机质, 稻田土壤有机物质增量主要来源于水稻根分泌物和脱落物[22]。2水稻稻田土壤总细菌丰度下降, 表明当季水稻向土壤输入的有机碳减少。
稻田土壤甲烷菌群落丰度变化也一定程度上反映了水稻根系向土壤输入有机物的能力。2水稻各个生长期内产甲烷菌丰度低于野生型对照, 暗示2水稻在整个生长期根系向土壤输入有机物量都要低于野生型对照。该结果与2具有调控水稻光合产物分配、增加茎和胚乳总淀粉含量、减少水稻地下部生物量[13]的功能相符。2稻田土壤总细菌、甲烷菌和甲烷氧化菌丰度都显著下降, 但并不是等比例下降, 其中产甲烷菌丰度降低幅度大于总细菌降低幅度, 甲烷氧化菌丰度降低幅度大于甲烷菌降低幅度, 菌群的丰度下降幅度随甲烷代谢链下移而增大。我们分析这是由于土壤中含碳有机物减少引发的作用首先影响的是与碳代谢有关的菌群的繁殖, 产甲烷菌是有机物厌氧降解的末端功能菌群[23], 甲烷氧化菌是以甲烷为惟一底物的氧化菌[24], 两者都是土壤碳代谢下游的菌群, 土壤有机物减少对细菌的影响作用传导到甲烷代谢链末端菌的作用是累积的。
根据以上分析我们推断: 导致2水稻甲烷减排的主要原因是稻田土壤甲烷菌群丰度下降, 而造成甲烷菌群丰度下降的因素是土壤中产甲烷基质减少, 结合前期对2水稻光合生理以及基因表达分析[13], 我们认为2基因异源表达降低水稻甲烷排放的机制是光合产物的分配发生变化, 光合产物向地下部的运送减少, 导致甲烷相关菌群丰度下降, 甲烷产生量减少, 相应地达到甲烷减排的效果。当然, 这些研究只是提供了间接的证据, 进一步的证据还有待于对水稻根系分泌物以及稻田土壤输入性碳进行跟踪分析。
虽然来自田间测定数据显示2水稻甲烷排放显著下降、甲烷菌群丰度也显著下降, 但二者在对应的时间上没有显著相关性(数据未列出)。所测定的6类主要产甲烷菌中, 5类峰值出现在水稻生长分蘖期, 而甲烷排放的高峰期则在分蘖期至灌浆期之间。甲烷排放高峰期明显滞后于产甲烷菌的旺盛生长期。究其原因我们认为是稻田甲烷排放除与土壤中甲烷相关菌和基质有关外, 还受土壤中氧化还原电位、土壤温度、pH等诸多因素的影响, 因素与因素之间相互交织互相影响, 任何一个因素的变化, 都会导致其他所有因素发生变化, 最终导致系统性变化。王明星[25]研究也发现: 水稻生长季甲烷菌的数量不是甲烷产量变化的原因。此外, 我们检测甲烷菌丰度是基于16S rRNA定量法, 所获得的是菌群拷贝数数据, 而非菌群的活性数据, 而甲烷产量与甲烷菌群的活性关系更为密切。
稻田土壤中甲烷相关菌的多样性也受温度、有机质含量、土壤中电子受体含量等因素影响。任何一个因素的变化, 都会导致其他因素发生变化, 最终会导致系统性变化。例如温度的变化可以影响所有的生化过程, 包括甲烷相关菌的代谢以及为产甲烷菌提供底物的微生物的代谢活性; 同时温度也会通过影响土壤的理化性质影响甲烷菌。因此这个过程是复杂的、交织和变化的。本研究仅从水稻生理角度讨论光合产物分配对稻田甲烷排放及土壤甲烷相关菌的影响, 阐明通过遗传修饰改变水稻光合同化物分配可以达到稻田甲烷减排的预期, 为今后低甲烷排放水稻品种的培育提供了一个研究思路。
[1] YVON-DUROCHER G, ALLEN A P, BASTVIKEN D, et al. Methane fluxes show consistent temperature dependence across microbial to ecosystem scales[J]. Nature, 2014, 507(7493): 488–491
[2] LIU Y C, WHITMAN W B. Metabolic, phylogenetic, and ecological diversity of the methanogenic archaea[J]. Annals of the New York Academy of Sciences, 2008, 1125(1): 171–189
[3] 魏海苹, 孙文娟, 黄耀. 中国稻田甲烷排放及其影响因素的统计分析[J]. 中国农业科学, 2012, 45(17): 3531–3540 WEI H P, SUN W J, HUANG Y. Statistical analysis of methane emission from rice fields in China and the driving factors[J]. Scientia Agricultura Sinica, 2012, 45(17): 3531–3540
[4] 陈槐, 周舜, 吴宁, 等. 湿地甲烷的产生、氧化及排放通量研究进展[J]. 应用与环境生物学报, 2006, 12(5): 726–733 CHEN H, ZHOU S, WU N, et al. Advance in studies on production, oxidation and emission flux of methane from wetlands[J].Chinese Journal of Applied & Environmental Biology, 2006, 12(5): 726–733
[5] 贾仲君, 蔡祖聪. 水稻植株对稻田甲烷排放的影响[J]. 应用生态学报, 2003, 14(11): 2049–2053 JIA Z J, CAI Z C. Effects of rice plants on methane emission from paddy fields[J]. Chinese Journal of Applied Ecology, 2003, 14(11): 2049–2053
[6] MA K, QIU Q F, LU Y H. Microbial mechanism for rice variety control on methane emission from rice field soil[J]. Global Change Biology, 2010, 16(11): 3085–3095
[7] CAI Z C, TSURUTA H, RONG X M, et al. CH4emissions from rice paddies managed according to farmer’s practice in Hunan, China[J]. Biogeochemistry, 2001, 56(1): 75–91
[8] YAO H, YAGI K, NOUCHI I. Importance of physical plant properties on methane transport through several rice cultivars[J]. Plant and Soil, 2000, 222(1/2): 83–93
[9] LU Y, WASSMANN R, NEUE H U, et al. Impact of phosphorus supply on root exudation, aerenchyma formation and methane emission of rice plants[J]. Biogeochemistry, 1999, 47(2): 203–218
[10] AULAKH M S, BODENBENDER J, WASSMANN R, et al. Methane transport capacity of rice plants Ⅱ. Variations among different rice cultivars and relationship with morphological characteristics[J]. Nutrient Cycling in Agroecosystems, 2000, 58(1/3): 367–375
[11] 王增远, 徐雨昌, 李震, 等. 水稻品种对稻田甲烷排放的影响[J].作物学报, 1999, 25(4): 441–446 WANG Z Y, XU Y C, LI Z, et al. Effect of rice cultivars on methane emissions from rice field[J]. Acta Agronomica Sinica, 1999, 25(4): 441–446
[12] 傅志强, 黄璜, 朱华武, 等. 水稻CH4和 N2O的排放及其与植株特性的相关性[J]. 湖南农业大学学报: 自然科学版, 2011, 37(4): 356–360FU Z Q, HUANG H, ZHU H W, et al. Relativity between CH4and N2O emission and rice plant characteristics[J]. Journal of Hunan Agricultural University: Natural Sciences, 2011, 37(4): 356–360
[13] SU J, HU C, YAN X, et al. Expression of barley SUSIBA2 transcription factor yields high-starch low-methane rice[J]. Nature, 2015, 523(7562): 602–606
[14] YU Y, LEE C, KIM J, et al. Group-specific primer and probe sets to detect methanogenic communities using quantitative real-time polymerase chain reaction[J]. Biotechnology and Bioengineering, 2005, 89(6): 670–679
[15] HOOK S E, NORTHWOOD K S, WRIGHT A D G, et al. Long-term monensin supplementation does not significantly affect the quantity or diversity of methanogens in the rumen of the lactating dairy cow[J]. Applied and Environmental Microbiology, 2009, 75(2): 374–380
[16] KOLB S, KNIEF C, STUBNER S, et al. Quantitative detection of methanotrophs in soil by novelA-targeted real-time PCR assays[J]. Applied and Environmental Microbiology, 2003, 69(5): 2423–2429
[17] DUBEY S K, SINGH A, SINGH R S, et al. Changes in methanogenic population size and CH4production potential in response to crop phenology in tropical rice field[J]. Soil Biology and Biochemistry, 2013, 57: 972–978
[18] NARIHIRO T, SEKIGUCHI Y. Oligonucleotide primers, probes and molecular methods for the environmental monitoring of methanogenic archaea[J]. Microbial Biotechnology, 2011, 4(5): 585–602
[19] SUN C X, HÖGLUND A S, OLSSON H, et al. Antisense oligodeoxynucleotide inhibition as a potent strategy in plant biology: Identification of SUSIBA2 as a transcriptional activator in plant sugar signalling[J]. The Plant Journal, 2005, 44(1): 128–138
[20] SUN C X, PALMQVIST S, OLSSON H, et al. A novel WRKY transcription factor, SUSIBA2, participates in sugar signaling in barley by binding to the sugar-responsive elements of the iso1 promoter[J]. The Plant Cell, 2003, 15(9): 2076–2092
[21] 张广斌, 马静, 徐华, 等. 稻田甲烷产生途径研究进展[J]. 土壤, 2011, 43(1): 6–11 ZHANG G B, MA J, XU H, et al. Advances on methanogenic pathways in rice fields[J]. Soils, 2011, 43(1): 6–11
[22] LU Y H, WATANABE A, KIMURA M. Contribution of plant- derived carbon to soil microbial biomass dynamics in a paddy rice microcosm[J]. Biology and Fertility of Soils, 2002, 36(2): 136–142
[23] 方晓瑜, 李家宝, 芮俊鹏, 等. 产甲烷生化代谢途径研究进展[J]. 应用与环境生物学报, 2015, 21(1): 1–9 FANG X Y, LI J B, RUI J P, et al. Research progress in biochemical pathways of methanogenesis[J]. Chinese Journal of Applied and Environmental Biology, 2015, 21(1): 1–9
[24] 邓永翠, 车荣晓, 吴伊波, 等. 好氧甲烷氧化菌生理生态特征及其在自然湿地中的群落多样性研究进展[J]. 生态学报, 2015, 35(14): 4579–4591 DENG Y C, CHE R X, WU Y B, et al. A review of the physiological and ecological characteristics of methanotrophs and methanotrophic community diversity in the natural wetlands[J]. Acta Ecologica Sinica, 2015, 35(14): 4579–4591
[25] 王明星. 中国稻田甲烷排放[M]. 北京: 科学出版社, 2001: 192 WANG M X. Methane Emission from Rice Field in China[M]. Beijing: Science Press, 2001: 192
Effects of heterogous expression of2 rice on methane mitigation and related micro-organism abundance in paddy fields*
SU Jun, SHAN Zhen, CHEN Zaijie
(Biotechnology Institute of Fujian Academy of Agricultural Sciences / Fujian Provincial Key Laboratory of Genetic Engineering for Agriculture, Fuzhou 350003, China)
A field experiment was conducted to explore the effects of genetically modified rice with2 gene on paddy field methane mitigation.2 gene is a transcription factor that acts on the upstream of starch synthesis pathway and is recognized as a key regulator for barley starch accumulation and assimilation distribution. Previous studies have shown that japonica rice (L. subsp.) integrated with2 gene significantly reduces methane emission in paddy fields and increases content of seed starch. To further understand gene effects on cutting down of methane emissions under different rice genetic conditions, we introduced2 into indica rice (L.subsp.) and then investigated methane emissions from2 rice field as well as the population size of bacteria associated with methane emissions in paddy fields during the growing season from April to September 2016. The results showed that the range of methane mitigation for the whole season was 54.7%–3.8%, compared with the control (wild rice). The highest mitigation rate was during booting period, reaching 54.7%. Total methane emissions of the two lines of2 rice were respectively 5 060.16 mg∙m-2and 5 250.60 mg∙m-2, while that under wild rice was 7 249.68 mg∙m-2for the period from the first measurement to harvest. Methane reduction rates of the two lines were 30.30% and 27.58%, respectively. The abundance of 6 orders or families of methanogens and 2 groups of methanotrophs in2 rice fields showed significant (< 0.05,< 0.01) decreases almost throughout the entire growing season when2 rice was compared with wild rice. In addition, total bacteria populations during rice tillering, heading and flowering periods were significantly (< 0.05,< 0.01) lower in2 rice than in wild rice. Population size of 6 methanogens were in the order of: Methanosaetaceae (Mst) > Archaea (ARC) > methanogens (MET) > Methanomicrobiales (MMb) > Methanosarcinaceae (Msc) > Methanobacteriales (MBT). Among these, Methanosaetaceae had the largest community, followed by Archaea. Of the 2 groups of methanotrophs, the abundance of MBAC was much larger than that of TYPE Ⅱ. After comparison of our experimental data with other studies, we concluded that2 rice mechanism for reducing methane emission more likely regulated carbohydrate flow to ground parts of the plant, reduced assimilates transported to soil and lowered methane-related bacteria abundance, which ultimately reduced methane emissions.
Rice; Heterogous expression of2; Methanon mitigation; Methanogen; Methanotroph; Bacteria abundance
, SU Jun, E-mail: sj@fjage.org
Jan. 23, 2018;
May 15, 2018
10.13930/j.cnki.cjea.180109
Q89; Q78
A
1671-3990(2018)09-1333-10
2018-01-23
2018-05-15
* This study was supported by the National Natural Science Foundation of China (31670416), Fujian Science and Technology Project (2015N0037), Fujian Public Scientific Research Institution Foundation (2018R1019-1) and the Science and Technology Project of Fujian Academy of Agricultural Sciences (A2015-02).
* 国家自然科学基金项目(31670416)、福建省科技项目(2015N0037)、福建省属公益类项目(2018R1019-1)和福建省农业科学院科技项目(A2015-02)资助
苏军, 主要从事水稻分子研究。E-mail: sj@fjage.org
苏军, 单贞, 陈在杰. 异源表达2水稻对稻田甲烷排放及土壤相关菌群的影响[J]. 中国生态农业学报, 2018, 26(9): 1333-1342
SU J, SHAN Z, CHEN Z J. Effects of heterogous expression of2 rice on methane mitigation and related micro-organism abundance in paddy fields[J]. Chinese Journal of Eco-Agriculture, 2018, 26(9): 1333-1342

