生物医学大数据发展的新挑战与趋势
张国庆 李亦学 王泽峰 赵国屏
1 中国科学院计算生物学重点实验室生物医学大数据中心,中国科学院-马普学会计算生物学伙伴研究所,中国科学院上海生命科学研究院(上海营养与健康研究院),中国科学院大学 上海 200031 2 上海生物信息技术研究中心 上海 201203
人类基因组计划启动以来,以新一代测序技术和质谱技术为代表的各类组学技术的飞速发展,推动了基因组、转录组、表观遗传组、蛋白质组、代谢组等海量生命科学组学数据的指数级的增长[1,2]。一方面,机器学习和人工智能技术极大提升了医学影像和分子影像技术的分析能力,正在改变以影像组、放射组为代表的医学影像数据的应用方式。高通量实验技术的突破,直接把生物医学数据从以基因组为代表的 PB 量级时代推升到多组学融合的 EB 量级时代。另一方面,人群队列研究、分子流行病学研究产生了大量长时间、广空间的数据,表型组从分子、细胞、组织、器官、个体等多层面描述了高维数据,真实世界数据(real world data)回顾性地汇总分析海量的临床信息数据[3,4],这些数据构成了复杂的高维度生物医学大数据。
我们已经进入了具备相当深度和广度的生物医学大数据时代。生物医学临床数据呈现数量巨大、增长迅速、质量控制困难、来源广泛繁杂、难以标准化与结构化等特点,生物医学研究数据呈现种类繁多、内部结构高维复杂、内涵丰富、数据相对分散、难以高维度多层次交汇共享等特点,生物医学数据总体表现为数据零散分布、难以有效整合分析,从而导致难以挖掘生物医学大数据的潜在高价值。对我国生物医学而言,数据无汇交机制,导致存储碎片化、管理分散、流失损耗严重;数据无安全保障,无国际交流窗口,被迫持续成为世界最大组学数据输出国;数据无共享平台,标准化管理混乱,质量参差不齐,开放共享受国际、国内的政策与技术的双重限制。
生物医学研究正在发生面向数据密集型的第四科学范式的深刻变革,如何实现从“组学”到临床与健康人群数据的生物医学大数据的交汇、综合管理、利用和共享,将多层次临床与研究数据进行深度挖掘和高维度、全方位的有机整合,将大数据迅速转化为新知识,成为我们所面临的挑战,其中研究建设下一代生物医学大数据存储、共享和转化中心的关键要素(图 1)。
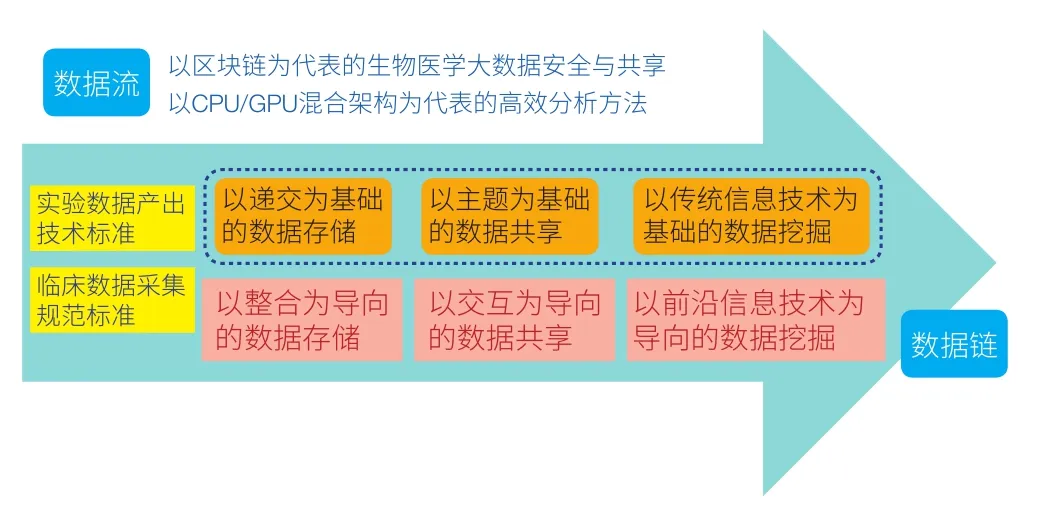
图 1 生物医学大数据平台的技术关键
1 以递交为基础、以整合为导向的数据存储
早在 20 世纪 80—90 年代,美国、欧洲和日本即已分别建立世界三大生物数据中心,即美国国家生物技术信息中心(NCBI)、欧洲生物信息研究所(EBI)和日本DNA 数据库(DDBJ)。这三大数据中心经过近 3 0年的建设,已经形成了完备的数据汇交技术体系,在基因组、转录组、蛋白质组等领域发挥着重要影响力[5-11]。国内机构也已经开始按照数据类型建设了 GSA[12]、iPROX①http://www.iprox.org/.、WDCM[13]等基因组、蛋白质组、微生物资源等组学数据中心。我国健康医疗大数据中心的“1+5+X”规划已经落地,即国家数据中心与江苏、福建、山东、安徽、贵州的东、南、西、北、中 5 个健康医疗大数据区域中心已经形成,将容纳全体公民健康医疗大数据。
各类已建、在建的生命科学和健康医疗数据中心,极大地丰富了生命科学、临床医疗等生物医学大数据的采集能力。但是随着数据规模的增加,如何更加有效地利用数据成了挑战。传统的数据模型和数据组织方式,已经无法满足海量数据的结构、数量快速增长以及数据结构不断变化的管理需求,难以按照实际情况动态调整。对于已有或者将要建设的综合性生物医学大数据平台而言,有必要突破传统的严格按照一类数据建设一个数据库的模式,采用新的仓储式的数据仓库模式,在底层数据结构上以整合为导向,按照样本、宿主、环境等信息,以及时间、空间信息,预留不同类型的数据之间的联系,形成弹性的数据结构,支持数据结构动态调整,为后期数据集成与整合工作奠定坚实的基础。
2 以主题为基础、以交互为导向的数据共享
NCBI 和 EBI 等机构通过数据递交服务汇聚了大量的数据资源,并通过网络提供数据共享。截至 2018 年 7月,NCBI 和 EBI 提供的生物序列、分子结构、遗传信息、表型信息等可以共享的数据接近资源都已经超过 60 项[7],这些数据资源极大地促进了生命科学与生物医学研究。除了共享第三方递交的数据资源外,以美国国家癌症研究院(NCI)建立的 TCGA(The Cancer Genome Atlas)数据库[14]、英国的国家队列 UK Biobank(UKB)②http://www.ukbiobank.ac.uk.等,采用的是另外一种模式,即依托大型科研项目产出的数据,提供分级共享,满足不同类型的科研需求。介于这两者之间,中小型研究团队利用自身的数据采集能力和整合能力,建立了大量的种类繁多、规模悬殊、质量参差不齐的数据库和知识库,提供数据查询、浏览、下载服务,部分数据库还提供在线分析服务。Nucleic Acids Research 每年第 1 期都出版数据库专刊,到目前为止,已经发表了 1 737 篇数据库相关论文[15],其已经成为生物医学数据库领域最有影响力的专刊。
这些按照数据类型(如基因组、转录组、蛋白质组等)、物种(如人类、人类以外、脊椎动物、无脊椎动物、微生物等)、研究目的(如遗传变异、转录因子、调控网络)等方式建设的数据库,在推进数据共享方面发挥了巨大的作用。但是随着数据类型和规模的日益扩大,如何存储、组织、访问存放在不同平台上的不同类型的生物医学数据成为新的挑战。为此,研究者提出 FAIR 原则,即可发现(findable)、可访问(accessible)、互操作(interoperable)和重用(reusable)[16]。基于 FAIR 原则,BD2K[17]、OmicsDI[18]等平台采用搜索引擎等技术突破传统的以主题为基础建设的数据库的局限性,对 EBI、NCBI 等数据中心的数据资源提供统一检索服务,实现以搜索引擎为核心的数据跨库整合,更好地满足用户一站式的数据共享需求。
除了搜索技术外,数据可视化、在线分析也是用户利用数据的重要手段。新的可视化技术,包括 HTML5、JavaScript 等 Web 展示技术在数据平台中的应用越来越广泛,用于大分子展示、分子影像、基因组浏览器等[19-21]。此外,依托数据库的分子序列、分子结构、调控及相互作用网络等数据,数据库根据自身特点,集成了序列比对、多序列比对、结构相似性比较、网络结构分析等在线分析的工具,也极大地加强了数据的可交互性。
在建设生物医学大数据平台时,TB 量级的数据下载需求对数据下载、单库检索等数据共享手段提出了严峻的挑战。因此在延续按照主题(数据类型、物种、研究领域)组织数据的基础上,引入跨库搜索引擎、可视化、在线分析等在线交互技术,通过更加准确地返回用户数据访问结果的方式,提高数据共享效率。
3 以传统信息技术为基础、以前沿信息技术为导向的数据挖掘
从分析的角度来看,生物医学大数据包括生命科学研究数据,以及临床医学数据。在生物信息学、计算生物学、系统生物学等计算学科的支持下,以基因组、转录组、蛋白质组、代谢组等组学数据为代表的生命科学研究数据的分析方法已经日趋成熟,分析流程日益普及,正在逐步成为传统的信息技术。临床医学数据在数据统计、数据建模、机器学习等技术的支持下,SAS、MATLAB、R 语言等分析工具也得到了广泛应用。
数据挖掘能力,尤其是组学数据挖掘能力,越来越难以满足飞速增长的数据产出。其面临的主要挑战在于:数据量越来越大,需要速度更快的数据压缩、传输、分析方法[22,23];数据维度越来越高,需要更加准确的降维方法[24]。基于 GPU(图形处理器)、FPGA(现场可编程门阵列)等硬件技术,对传统的生物信息分析方法的限速步骤进行算法优化,在序列比对、分子对接得到越来越多的应用[25,26]。而以深度神经网络为代表的人工智能技术,在医学影像处理、高维数据降维等方面的应用呈现爆发式的增长,包括致盲性视网膜疾病与肺炎、阿尔茨海默病、皮肤癌、脑膜瘤等医学影像辅助诊断等[27-30]。此外,区块链技术由于其去中心的特性,也开始在生物医学数据共享方面得到应用[31,32]。
前沿信息技术在生物医学大数据中的应用,将涵盖数据预处理、数据传输、数据分析、数据共享等范围,提升数据挖掘能力。
4 数据标准与质量控制
生物医学大数据的数据标准包括术语集、数据标准、综合标准等。典型的术语集包括基因本体 GO[33]、人类表型本体 HPO[34]等,序列最简描述信息标准集包括 MIxS 与 MIGS[35-37]以及 ICD10③http://apps.who.int/classi fi cations/icd10/browse/2016/en.、SNOMED-CT④https://www.snomed.org/snomed-ct.等医学数据标准。生命科学领域的数据标准大多由有国际影响力的机构或协会率先提出,伴随配套的数据解析或分析软件,逐步得到学术界的认可。例如:由国际核酸序列数据库协会(INSDC)定义的“The DDBJ/ENA/GenBank Feature Table Definition”[8]是 NCBI、EBI 等数据中心最早的核酸序列数据标准,以及基因组拼接数据标准;EBI 和 NCBI 等定义的基因芯片实验数据标准 MIAME[38]、GEO[39],FGED 定义的二代测序数据标准 MINSEQE⑤http://fged.org/site_media/pdf/MINSEQE_1.0.pdf.,以及拼接文件格式 BAM、变异文件格式 VCF、遗传特征描述格式 GFF3⑥http://gmod.org/wiki/GFF3.等,医学领域得到最为广泛认可的数据标准是医学影像标准 DICOM⑦https://www.dicomstandard.org/.。医学领域的标准比生命科学领域的数据标准要复杂得多,规范化程度也更高。医学领域的标准大多需要经过立项、草案、发布等阶段,得到了更为广泛的认可,如国际标准化组织健康信息学标准化技术委员会的 ISO/TC 215 系列标准⑧https://www.iso.org/committee/54960.html.、HL7(卫生信息用户层,ISO 定义的信息交换7层协议规范中的第七层)⑨http://www.hl7.org.、临床数据交换标准协会 CDISC⑩https://www.cdisc.org.等;标准的范围也远比生命科学领域的数据标准复杂,包括词汇术语、数据描述、技术操作、应用服务和医疗管理等。
生命科学的标准主要集中在术语集和数据标准,不同的标准之间相对独立,对数据产出过程、分析过程的规范性表述较少。医学的数据标准更强调互操作、互联互通等,不同的标准自成体系,但是对支撑科研的数据标准的描述反而较少。因此,生物医学大数据亟待加强临床科研的数据标准体系的建设,以及数据分析过程的操作相关的标准的建设。
数据质量控制受到数据产出、数据分析的影响,不同的数据质控有所差别。芯片、基因组数以美国食品药品监督管理局(FDA)主导的 MAQC、MAQC-II、MAQC-III 等[40-44],由于独立于技术系统之前,得到了较为广泛的认可。蛋白质组的数据质控,缺少与 MAQC 相匹配的大项目,而是主要通过 PRIDE、iPROX 等数据汇交平台的质控工具[45,46]来体现。数据质量控制需要提供参考数据集作为基准,包括实验方法产出的原始数据与参考数据集的吻合情况,以及数据分析形成的分析结果与参考数据集的吻合情况。因此,针对有广泛用途或者重要用途的数据类型,建设参考数据集、参考数据分析流程,是数据质量控制的关键环节,也是生物医学大数据平台的重要建设内容。
5 实践与思考
我们正在建设以组学数据百科全书——NODE⑪为代表的开放式基础性平台,并达到了一定的数据规模。其中,在整合存储方面,数据平台与数据库包括以微生物组大数据平台为代表的领域示范平台,以骆驼基因组变异数据库、可翻译转录组 RNA 数据库等为代表的专题数据库。在交互共享方面,正在向 NODE 系统集成全基因组、外显子组、转录组等常规组学数据分析流程,微生物 16S RNA、宏基因组、微生物功能注释等领域组学数据分析流程。在前沿信息技术方面,利用 GPU 技术对转录组、宏基因组等组学数据拼接、映射等高资源消耗的环节进行优化。在标准质控方面,开展了包括描述信息和原始数据在内的质量控制,并建立了自动化的质控流程,将实现数据汇交时就自动完成质控评估的功能。
面对生物医学大数据的挑战,建立全面支撑生命科学研究数据与健康医学大数据的汇交、管理、共享与挖掘的技术与资源体系,形成以递交为基础、以整合为导向的数据存储中心,以主题为基础、以交互为导向的数据共享中心,以及以传统信息技术为基础、以前沿信息技术为导向的下一代生命科学数据转化中心,将有效地支撑生物医学、健康医疗等领域的基础研究、应用研究和产业示范。

