基于SNP芯片分析的蓝塘猪遗传群体结构
王 晨,马 宁,郭春和,袁仁强,曾检华,宋德清,张惠文,陈瑶生,刘小红
(1.中山大学生命科学学院/有害生物控制与资源利用国家重点实验室,广东 广州 510006;2.紫金东瑞农牧发展有限公司,广东 河源 517447;3.广东壹号食品股份有限公司,广东 广州 510620)
蓝塘猪(Sus scrofa)属于典型的华南型品种,中心产区位于河源市紫金县蓝塘镇,个体毛色以躯体水平上下黑白各占一半为特征,具有体型较小、早熟易肥、性情温顺和耐高温高湿等特点。由于地理环境和历史条件等原因,采用“父老子继、母死女代、代代相传”的单传法,使得蓝塘猪长期处于闭锁选育和高度近交繁殖状态,从而具有耐近交的特性,遗传稳定性非常高。蓝塘猪几乎不含有欧洲猪种的渗入,并且被认为是美洲猪种中亚洲血缘的主要贡献品种[1]。蓝塘猪还经常被用作研究肌肉与脂肪发育的实验动物,为基础科学实验提供了重要素材[2-4]。
20世纪大量西方瘦肉型猪种被引进我国,使得我国大部分地方猪群体数量下降,一些品种处于濒危状态甚至灭绝。我国地方猪种遗传资源保护起步较晚,技术力量有限,缺乏系统的数据管理,出现遗传多样性降低和种质退化等现象。从DNA水平分析地方猪的种质特性和遗传基础,将有利于种猪遗传资源的保护和利用[5]。
SNP作为第三代分子标记,在基因组中具有分布广、数量多和遗传稳定等特点,被广泛应用于分子鉴定和关联分析等研究。随着二代测序成本的不断下降和商业化SNP芯片的普及,全基因组SNP逐渐被推广和应用到农业动植物的育种实践工作中。猪的商业化SNP芯片已经有多种,其中应用最为广泛的是Illunima公司的PorcineSNP60芯片和GeneSeek公司的PorcineSNP80芯片。然而,这些芯片并不适合中国地方猪种。目前,猪的SNP芯片主要应用在全基因组关联分析和全基因组选择领域,特别是应用在肉质[6]、繁殖[7]和生长[8]等性状,而对地方猪品种的群体结构和保护的研究还比较少。本研究利用Illumina CAUPorince 50K SNP芯片,以一个蓝塘猪种猪群体作为研究对象,分析蓝塘猪的群体结构并构建家系,为地方猪种的保种和利用实践提供分子水平的参考。
1 材料与方法
1.1 试验材料
供试的蓝塘猪样品均采自紫金东瑞农牧发展有限公司,共139头种猪,其中16头公猪、123头母猪。121头种猪具有出生日期和部分系谱记录,另外18头种猪(13头公猪)无任何记录。采集的样品均为耳组织,置于75%酒精中,-20℃低温保存。
1.2 试验方法
1.2.1 SNP芯片分型 DNA提取采用酚氯仿抽提,经Nanodrop 2000仪器和0.8%琼脂糖凝胶电泳检测DNA质量,DNA浓度调整至50 ng/μL。所有合格的DNA样品采用 Illumina CAUPorince 50K SNP芯片进行基因型分型,然后按照以下标准进行质控:先去除性染色体上的位点,再选择SNP检出率大于0.9和最小等位基因频率大于0.01的位点。
1.2.2 种群亲缘关系分析 利用Plink1.9b软件[9]对质控后的数据进行主成分分析(principal component analysis,PCA),采用R软件绘制第一主成分与第二主成分的二维图,分析品种内的群体结构。采用Gmatix(v2)软件构建群体G矩阵,并绘制热图展现种猪之间的遗传亲缘关系。
1.2.3 遗传距离分析 在质控后的基础上,进一步去除强连锁(r2>0.15)位点。使用Plink1.9b软件计算个体间的遗传距离(cluster-distance-matrix),构建状态同源(identity-bystate,IBS)矩阵。通过 MEGA7.0 软件[10]构建邻接法进化发育树(Neighbour-joining tree,NJ tree)。另外,还对16头种公猪Y染色体上的SNP进行单倍型分型。
1.2.4 ROH分析 使用Plink1.9b软件进行基因组上长纯合片段(runs of homozygosity,ROH)分析,具体参数如下:20个SNP的滑动窗口沿染色体滑动,每个滑动窗口的杂合子个数不超过1个。其中涉及的4个重要ROH定义参数:最小长度>10 kb,SNP个数/ROH>20,最小SNP密度>1 000 kb/SNP。
2 结果与分析
2.1 基因型质量控制
供试的Illumina CAUPorince 50K SNP芯片共含有43 832个SNP位点。139个个体的基因型检出率在0.9488~0.9843、平均值为0.9824,表明该款芯片适合分析地方猪种。对139个样本进行质控后,样本全部合格,剩下28 676个有效SNP进行后续分析。当除去强连锁(r2>0.15)位点后,剩余1 962个位点。
2.2 种群亲缘关系分析
为了研究蓝塘猪个体间的亲缘关系,将139个样本的28 676个SNP进行主成分分析。从PCA结果(图1)可以看出,整个蓝塘猪个体间的亲缘关系不太紧密,表明蓝塘猪的遗传多样性较高。但在第一主成分中,左侧的个体之间亲缘关系比较紧密,其中公猪之间以及部分母猪与公猪聚集一起,表明这些蓝塘猪的亲缘关系较近,可能存在近交的情况。
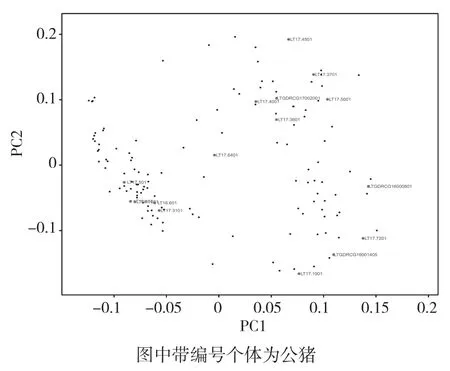
图1 蓝塘猪种群的主成分分析结果
为了进一步地分析139头蓝塘猪的亲缘关系,构建了G矩阵。从G矩阵的热图(图2,封二)来看,结果与PCA相似,大部分个体间的亲缘关系呈中等程度,而部分种猪之间亲缘关系较近,表明这些个体之间存在近交趋势。

图2 蓝塘猪种群的G矩阵热图
本研究还比较了139头蓝塘猪之间的遗传距离,IBS值在0.1566~0.4218之间,平均遗传距离值为0.3326±0.0339,表明蓝塘猪个体之间的遗传距离较远,且差异较大。16头公猪之间的遗传距离在0.2801~0.3933之间,平均遗传距离为0.3392±0.0275,表明种公猪之间的遗传距离均较远。
2.3 蓝塘猪家系构建
由于公猪在建立家系和整个保种群体中均具有极高的重要性,因此本研究选择16头公猪的1 962个位点构建NJ树。结果(图3)表明,公猪群体主要分成4支,同时结合G矩阵(>0.1)和遗传距离(<0.33)结果,最终从上而下分别命名为家系A、家系B、家系C、家系D,分别有6、4、2、4头公猪。

图3 16个蓝塘猪种公猪的NJ树
将16头公猪Y染色体上的6个SNP位点进行单倍型分析,共发现两个单倍型,分别命名为单倍型A和单倍型B(表1)。这两个单倍型之间存在3个SNP,均属于A>G转换突变。家系A和家系B的所有个体都属于单倍型A,而家系C和家系D的所有个体则属于单倍型B。

表1 蓝塘猪种公猪Y染色体单倍型分布
根据139头种猪的NJ树结果、G矩阵以及遗传距离,发现大部分母猪与4个家系的公猪聚集一起,形成4个家系(A、B、C和D)。但是,其中11头母猪与这些家系个体亲缘关系较远,则合并构建为家系E。
2.4 近交程度分析
在139头蓝塘猪中共检测到1 680个ROH片段,其中10~20 Mb长度的ROH数量最多、占44.52%(图4)。最短的ROH长度为5.72 Mb,含有100个SNP,位于第8号染色体上;最长的ROH长度为181.42 Mb,含有2 209个SNP,位于13号染色体上。在猪的18条常染色体上,13号染色体上的ROH数量最多、为189个,18号染色体最少、仅18个。ROH的数量随着染色体的长度增加而增多(图5)。每个蓝塘猪个体含有的ROH数量为2~28个,平均个数为12.09±5.90,其中含有7~8个ROH的个体数最多(图6);每个个体的ROH总长度在19.98~902.89 Mb,平均长度为246.90(±170.93)Mb,个体ROH长度在400 Mb内的个体数最多、占87.05%(图7)。
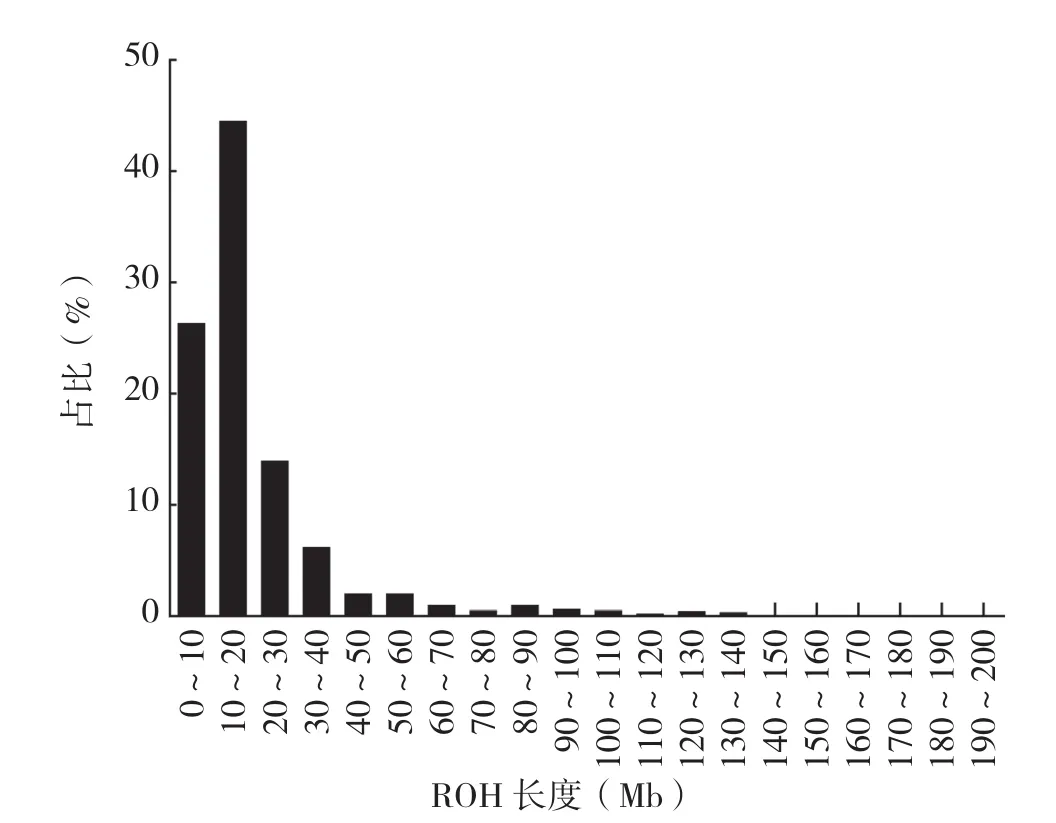
图4 蓝塘猪种群的ROH长度分布
3 结论与讨论
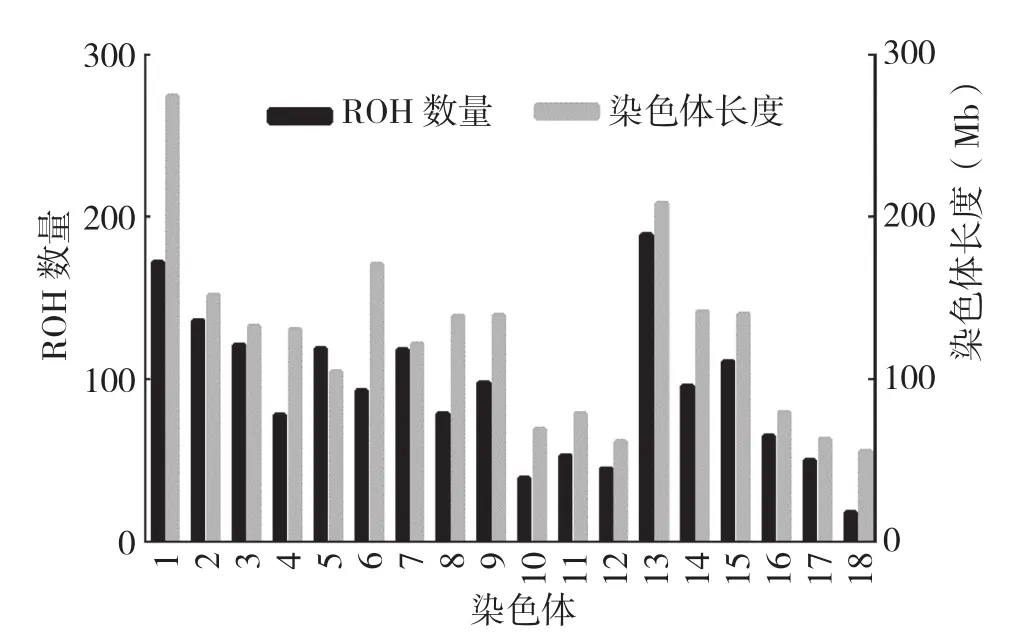
图5 蓝塘猪染色体上ROH数量分布
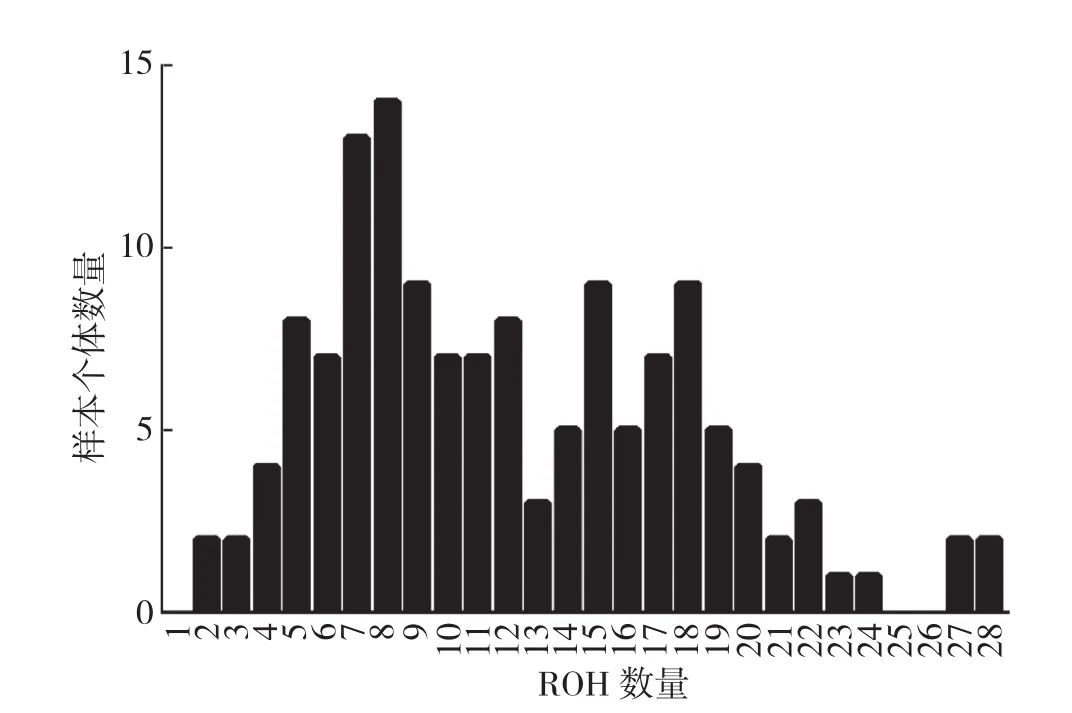
图6 蓝塘猪个体ROH数量的样本数分布
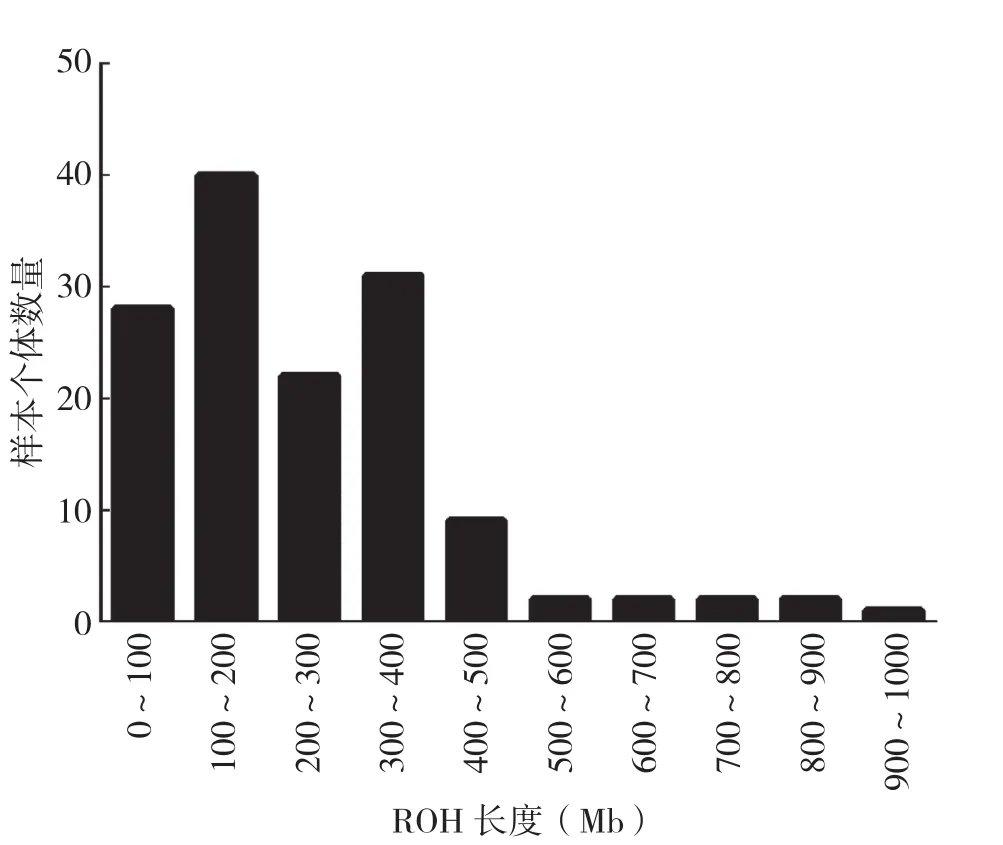
图7 蓝塘猪个体ROH长度的样本数分布
利用全基因组SNP标记可以对遗传多样性和群体结构进行科学评估,从分子水平上客观反映群体内或群体间的遗传差异。目前,市场上最常用的是猪60K SNP芯片,而这款芯片在染色体上分布的均匀性不高,大量位点在基因组上的位置不详,并且对中国地方猪中的基因型分型效率较低。因此,盛中华等[11]采用简化基因组方法分析上海白猪种群,发现上海白猪的遗传多样性比西方猪种和太湖猪种都要高,并且与西方猪种的遗传距离较太湖猪种更接近,印证了上海白猪是一个独立的品种。由于SNP芯片成本比简化基因组低,因此本研究利用由中国农业大学开发的Illumina CAUPorince 50K SNP芯片,此芯片在中外多个猪种多组学数据的基础上,加入了我国地方猪种特有的SNP以及重要经济功能基因候选位点。本次蓝塘猪样本的基因型分型率较高,表明该款SNP芯片非常适合我国地方猪种,为我国地方猪种选育和新品种培育提供了有效途径。
在畜禽的保种和育种过程中,关键在于生产记录和系谱的管理。在实际生产操作中,由于配种、产仔、数据录入以及人为因素等,通常会造成系谱错误,从而使得品种保护与利用效果降低。研究表明,世界范围内的奶牛系谱错误率约为 11%[12]。张哲等[13]利用 SNP芯片对一个杜洛克猪群体进行亲子鉴定,发现其系谱的错误率高达6%。由于条件有限,我国很多地方猪品种的系谱通常记录不完全甚至无系谱。在本研究的蓝塘猪猪场内,公猪仅有3头具有系谱来源;母猪的完整性相对较好,56%的个体具有其父母本和血统来源的记录。地方猪的保种来源主要从不同农户搜集而来,难以得到准确的个体信息。利用SNP标记鉴定二代和三代以内猪的血缘关系与系谱记录可以达到完全一致[14]。本研究通过SNP芯片分析,139头蓝塘猪种群可以分为5个家系,其中4个家系均含有种公猪,共有2个父系起源。根据当前的群体结构分析结果,不同家系的个体数量和近交程度不均衡。在一定的保种成本下,需要合理安排各个家系种猪的配种和数量。保种场内的存栏公猪的正常使用率往往较低,可以通过SNP芯片进行基因型分型,鉴定亲缘关系后,构建种公猪的冻精库,从而降低遗传资源的保种费用。
动物保种和育种的主要目标在于维持种群遗传多样性的最大化,从而减少近交导致的有害基因积累的效应,保证群体生存适应能力,同时具有足够多的目标性状变异可供选择,为今后的育种提供遗传素材。当蓝塘猪的平均近交系数达到26.7%水平时,对品种特征、繁殖力、仔猪出生重以及成活率均无不良影响[15]。本研究中的蓝塘猪群体,从PCA和G矩阵结果发现,群体中一部分种猪个体之间亲缘关系非常近,可能存在近交的可能。牛荣等[16]利用微卫星标记,发现近交系版纳小耳猪近交系的遗传距离在0~0.2766之间,本研究蓝塘猪群体的遗传距离比该近交系高,表明蓝塘猪的近交程度相对较低。但是,这两个品种均有较小的遗传距离,也存在较大的基因组成差异。李凯等[17]研究发现五指山猪近交系的基因纯合度随着近交世代数增加而递增,达到18个世代时,仍有约43%的基因座呈现较高水平的多样性,推测与这些基因座紧密连锁的功能基因在高度近交水平下具有维持生存能力的关键作用。
ROH是个体基因组内纯合基因型的连续片段,其长度和频率可以反映群体历史,片段越长近交的可能性越高。Bosse等[18]对欧亚家猪和野猪基因组中的ROH进行比较分析,发现亚洲猪种比欧洲的ROH数量少且总长度短。另外,还发现用Illumina porcine 60K SNP芯片能够检测到大于5 Mb的ROH,并且认为分析5 Mb以上的ROH时,60K SNP芯片与全基因组重测序结果一样准确。本试验中,蓝塘猪的ROH最短长度为5.72 Mb,表明本结果得到的ROH 具有较高的可靠性。蓝塘猪中ROH的数量与染色体的长度呈正相关,但是研究表明ROH的数量和大小与染色体的位置也有关,端粒区域由于重组率较高而含有较少的ROH,在染色体中部却更容易产生长片段的ROH[19-20]。全基因组重测序比较濒危猪种Sus cebifrons和商业猪种皮特兰,发现在较小群体中ROH片段的增加更容易引起有害等位基因表达的风险[21]。利用猪的60K SNP芯片对华南地方猪进行分析,发现蓝塘猪处于较高的连锁不平衡状态,相邻SNP间的r2为0.285,有效群体大小从1 092世代前的1 476头下降到2世代前的14头,表明受到了较强的人工选择[22]。本研究中的蓝塘猪群体只有2个父系起源,每个家系种群个体数量较少,且小部分个体的ROH片段总长度达到了800~900 Mb以上。因此,在蓝塘猪的保种过程中,需要对种群中一些个体进行合理选配,以保证蓝塘猪遗传资源的多样性。

