家禽屠宰场环境和屠宰器械表面微生物菌群结构和耐药基因分析
王佩佩,杨 华,戴贤君,桂国弘,肖英平,*
(1.中国计量大学 生命科学学院,浙江 杭州 310018; 2.浙江省农业科学院 农产品质量标准研究所,浙江 杭州 310021; 3.浙江省植物有害生物防控省部共建国家重点实验室培育基地,浙江 杭州 310021)
鸡肉作为世界上最主要的肉食消费品之一,具有高蛋白、低胆固醇、低脂肪的营养特点,受到人们的喜爱。我国为全球第二大肉鸡消费国,仅次于美国[1-3]。为保障公共卫生安全,目前我国多城市已禁止活禽交易,实行家禽定点屠宰[4]。然而肉鸡在屠宰加工过程易受到微生物的污染,如各屠宰区域的水、屠宰设备中微生物的交叉污染而增加鸡肉的微生物污染水平[5]。屠宰场环境中存在的腐败菌可影响鸡肉品质和货架寿命,一些特定病原菌也对人类健康造成威胁[6]。肉鸡在屠宰过程中产生的废弃物含有血、毛、粪便等直接污染屠宰场区域的水体和地面,其含有一定量的病原菌,有些病原菌甚至携带耐药基因等,可能会污染周边水体和土壤。因此,家禽屠宰场的微生物分布状况不仅直接影响鸡肉的质量安全,也关系到生态环境和人类健康。目前,国内对肉鸡的屠宰加工过程中特定食源性致病菌的研究较多,如James等[7]发现,肉鸡屠宰加工生产链中存在单核细胞增生李斯特菌、弯曲杆菌、沙门氏菌、粪便梭菌等病原微生物,但对易造成鸡肉微生物交叉污染的家禽屠宰场区域水体、地面以及屠宰器械表面微生物结构和耐药基因研究较少。本研究采用基于16S rRNAV3-V4的Illumina 高通量测序方法分析家禽屠宰场不同区域水体、地面和屠宰器械表面微生物结构特征,以及通过PCR技术检测微生物所携带的磺胺类、喹诺酮类、四环素类、大环内酯类和β-内酰胺类基因等9大类24种耐药基因,为全面而准确地认识家禽屠宰场环境微生物多样性、耐药基因传播扩散与环境风险之间的关系提供科学依据。
1 材料与方法
1.1 采样方法
1.1.1 屠宰区域水体微生物样品采样
选择浙江省某屠宰场,屠宰区域主要包括挂禽间、宰杀沥血间、浸烫间、脱毛间、净膛间(包括净膛和清洗),分别用5 mL无菌枪头吸取30 mL的地面水或器械水到无菌离心管中,4 ℃保存。样品编号为宰杀沥血间地表水(WSl)、浸烫间浸烫水(WScM)、浸烫间地表水(WSc)、脱毛间地表水(WD)、清洗间地表水(WC)、净膛间地表水(WE)、净膛后清洗水(WCE)、干净屠宰用水(WU)。
1.1.2 地面和屠宰器械表面微生物样品采样
将7.5 cm×7.5 cm的纱布在无菌生理盐水中沾湿,均匀擦拭屠宰场各个加工区域中地面以及与鸡肉直接接触挂钩、操作台或传送带,最后放置到盛有生理盐水的无菌离心管中,4 ℃保存。样品编号为挂禽间地表面纱布(SFH)、宰杀沥血间挂钩表面纱布(SHSl)、宰杀沥血间地表面纱布(SFSl)、浸烫机器面纱布(SScM)、浸烫间地表面纱布(SFSc)、脱毛机器表面纱布(SDM)、脱毛间地表面纱布(SFD)、清洗间传送带表面纱布(SBc)、净膛间挂钩表面纱布(SHE)、净膛间桌面纱布(STE)。
1.2 样品处理和细菌基因组DNA提取
将屠宰区域水体、地面和屠宰器械表面微生物样品按照ZR Fungal/Bacterial DNA MiniPrepTM(Zymo Research)提取总细菌DNA。具体操作:将纱布擦拭微生物样品置于摇床振荡30 min,然后离心机离心10 min,去除上清液;采集水样直接离心将沉淀重悬于200 μL无菌生理盐水后,加入ZR BashingBead Lysis Tube,按照试剂盒说明书提取DNA,进行电泳检测。
1.3 耐药基因的PCR扩增
本实验从9类抗生素耐药基因中共选取了24种耐药基因,包括2种磺胺类基因(sulⅠ、sulⅡ),5种喹诺酮类基因(qnrA、qnrD、qnrS、qepA和oqxB),4种四环素类基因(tetC、tetB、tetK和tetA),3种氯霉素类基因(cmlA,floR和cfr),3种大环内酯类基因(ereA、ermB和mef),2种β-内酰胺类基因(blaTEM和blaCTX-M),2种青霉素类基因(mecA和mecC),1种氨基糖苷类基因(aadA1),2种多粘菌素类基因(pmrA和pmrB)。所用引物见表1。PCR扩增反应程序为95 ℃ 5 min预变性;然后94 ℃ 30 s,58 ℃ 30 s;72 ℃ 1 min,共32个循环;最后16 ℃保存。扩增反应后进行琼脂糖凝胶电泳并成像。
1.4 高通量测序及数据分析
以提取的DNA为模板,扩增环境细菌16S rRNA基因V3-V4区,引物为338F(5′-ACTCCTACGGGAGGCAGCA-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)。测序由北京诺禾致源生物信息科技有限公司完成。通过QIIME 软件对获取的序列进行质控和过滤,采用ucluster方法根据相似性≥97%原则将通过质控的有效序列聚类成为操作分类单元(operational taxonomic unit,OTU[24])。通过绘制稀释曲线(rarefaction curve)评价所测序量是否覆盖全部类群。采用QIIME默认参数计算各样品的alpha多样性指数(ChaoⅠ、ACE、Simpson、Shannon和Coverage指数),以及Sliva和RPD数据库对所有OTU的代表性序列进行物种匹配,分别在门和属的水平上对样品的细菌组成进行菌群统计,并绘制柱状图。根据各个样品OTU的种类及其丰度,计算样品的加权UniFrac距离,再对18个样品进行主坐标分析(PCoA)。

表1 PCR反应引物
2 结果与分析
2.1 样品微生物物种丰富度及多样性
经过质控、过滤、拼接等处理,本实验最终获得902 029条有效序列,其中屠宰区域水体样品(W)有392 394条有效序列,屠宰器械表面微生物样品(S)有509 635条有效序列。所获得的有效序列根据97%相似性水平进行OTU聚类分析,各样品最终获得517~810个OTU,各样品的覆盖度指数(goods coverage)均为0.994~1.000,表明样本中序列没有被测出的概率极低,因此本次测序结果能够代表样本的真实多样性组成。各样品的稀释曲线均趋于平缓(图1),说明测序已趋于饱和,测序深度已基本覆盖样品中所有的物种。进一步使用ChaoⅠ指数、ACE指数、Shannon指数及Simpson指数对样本内细菌群落的丰富度和多样性进行评估(表2)。

表2 样品测序概况
WSl,宰杀沥血间地表水;WSc,浸烫间地表水;WD,脱毛间地表水;WC,清洗间地表水;WE,净膛间地表水;WScM,浸烫水;WCE,净膛后清洗水;WU,干净屠宰用水;SFH,挂禽间地表面纱布;SFSl,宰杀沥血间地表面纱布;SFSc,浸烫间地表面纱布;SFD,脱毛间地表面纱布;SHSl,宰杀沥血间挂钩表面纱布;SHE,净膛间挂钩表面纱布;SScM,浸烫机器表面纱布;SDM,脱毛机器表面纱布;SBC,清洗间传送带表面纱布;STE,净膛桌面表面纱布。下同。
WSl, water from slitting and blood draining district; WSc, water from scalding district; WD, water from dehairng district; WC, water from cleaning district ; WE, water form eviscerating district; WScM, water from scalding machine; WCE, water after cleaning the carcass in evisceration district; WU, water used in this slaughter house; SFH, surface swabs of floor in hanging district; SFSl, surface swabs of floor in slitting and blood draining district; SFSc, surface swabs of floor in scalding district; SFD, surface swabs of floor in dehairng district; SHSl, surface swabs of hanger in scalding district; SHE, surface swabs of hanger in eviscerating district; SScM, surface swabs of scalding machine; SDM, surface swabs of dehair machine; SBC, surface swabs of belt in clean district; STE, surface swabs of table for evisceration. The same as below.

图1 各样品稀释曲线Fig.1 Rarefaction curves of each sample
2.2 屠宰区域水体、地面和屠宰器械表面细菌菌群结构
将18个屠宰区域水体、地面与屠宰器械表面微生物样品用于细菌结构测序分析,获得的OTU分别在门和属水平上进行物种注释,具体结果见图2。可以看出在门的水平上,屠宰区域水体、地面和屠宰器械表面微生物主要分属于9个门,其中变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)和放线菌门(Actinobacteria)的相对丰度分别为27.55%~88.99%、3.45%~59.78%和4.35%~31.78%,三者共同构成了屠宰场区域水体、地面和屠宰器械表面细菌菌群的主要结构。
从在属的水平上分析,不动杆菌属(Acinetobacter)、嗜冷杆菌属(Psychrobacter)、栖热菌属(Thermus)、假单胞菌属(Pseudomonas)、希瓦氏菌属(Shewanella)为主要菌属,约占总菌群的46%以上;还有少量的考克氏菌属(Kocuria)、副球菌属(Paracoccus),(Methylotenera)、厌氧芽孢杆菌属(Anoxybacillus)、弓形杆菌属(Arcobacter)。其中不动杆菌属(Acinetobacter)在屠宰区域水体、地面和屠宰器械表面的相对丰度分别为4.71%~24.27%,4.71%~24.27%,6.99%~42.67%。各样品丰度较高的前35种菌属见图3。
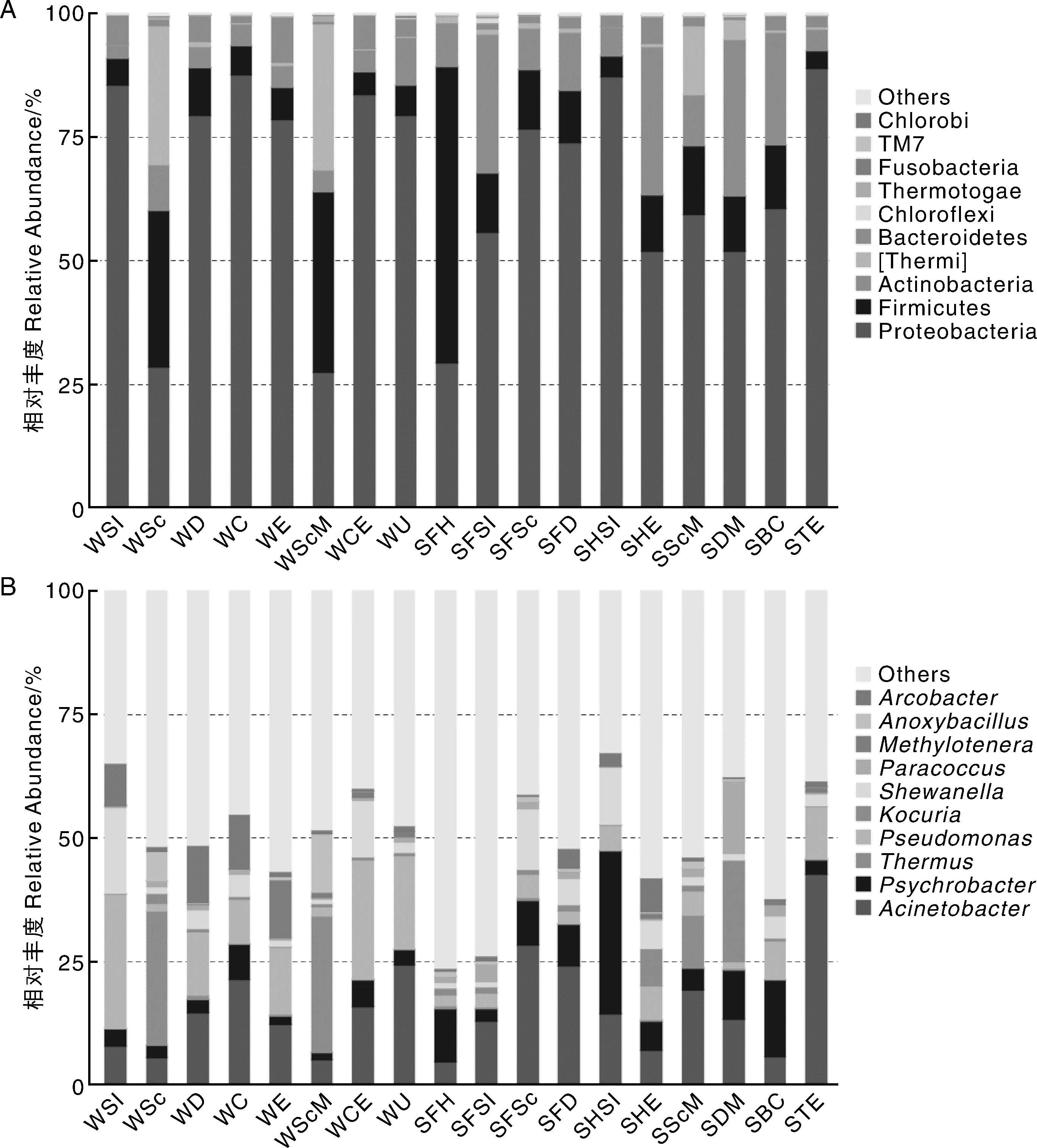
图2 各样品在门(A)和属(B)水平上丰度较高的前10菌群结构Fig.2 The top 10 phyla (A) and genera (B) of each sample
假单胞菌属是典型的腐败菌,因其营养要求低,黏附力强,极易在低温潮湿的环境中繁殖,其生长代谢能加速禽肉的腐败[25]。在临床上假单胞菌可感染人体皮肤、呼吸道、泌尿道、胃肠道等,是医院感染常见的病原菌。此外不动杆菌属、嗜冷杆菌属和希瓦氏菌属在低温潮湿环境下也能快速繁殖,是禽肉及水产品低温保存过程中的常见腐败菌[26-28]。不动杆菌属具有一定的致病性,属于条件致病菌,可引起肺部、伤口、皮肤和泌尿生殖系统感染,甚至会引起菌血症、脑膜炎等临床疾病[30]。假单胞菌属在净膛后清洗水微生物中的相对丰度较高,达到24%之多。有相关研究表明假单菌属能耐受清洗水中的氯浓度而生存下来,其他菌属会因为抑菌物质的存在而相对减少[31]。这些条件致病菌和腐败菌广泛存在于屠宰场区域水体、地面和屠宰器械表面中,可与屠宰加工的鸡肉构成交叉污染,致鸡肉加速腐败,还可能引起食源性疾病的发生。
2.3 样品的聚类与主坐标分析
根据各样品OTU的种类及其丰度,计算样品间的加权UniFrac距离,对18个屠宰区域水体与屠宰器械表面微生物样品进行了主坐标分析(principal coordinate analysis,PCoA)(图4)。结果表明,同种样品如屠宰区域水体微生物之间、屠宰场地面和屠宰器械表面微生物之间显示出明显聚集,而屠宰区域水体跟屠宰场地面和屠宰器械表面微生物之间相距较远,与图3的菌群丰度热点图相类似。

图3 家禽屠宰场不同区域菌属热点图和聚类分析Fig.3 Heatmaps and dendrograms of the most abundant bacterial genera in different area of poultry slaughterhouse

图4 各样品细菌菌落结构的主坐标分析Fig.4 Principal coordiante analysis (PCoA) of the dissimilarity between the bacterial samples
2.4 屠宰区域水体、地面和屠宰器械表面微生物样品的耐药基因检测结果
通过对18个屠宰区域水体、地面和屠宰器械表面微生物样品中的9大类抗生素24种耐药基因的PCR扩增发现,21种耐药基因分布在这些样品中。由此可见屠宰场的耐药基因检出率很高,其中sulⅠ、sulⅡ和qnrS的检出率为100%,blaTEM、tetB和aadA1的检出率为94.4%,floR和mecC的检出率为88.9%(表3和表4)。但是在干净屠宰用水(WU)中只检测出sulⅠ、sulⅡ、qnrS和tetA这4种耐药基因,表明屠宰场区域水体、地面和屠宰器械表面微生物中的大多数耐药基因不是来源于干净的屠宰用水。
畜牧业中滥用抗生素会使有害菌种产生耐药性及畜产品药物残留,通过食物链进入到人体中,对人类健康造成巨大的威胁[31-32]。环境中耐药微生物促进了耐药致病菌的产生和扩散,抗生素耐药基因作为一种环境中难以去除的新型污染物,具有可遗传性和可转移性[33-34]。家禽屠宰场环境中存在21种耐药基因之多,屠宰场的的水体、地面和屠宰器械表面存在的耐药基因有些许差异,tetK和pmrB基因只分布在屠宰场器械表面,cmlA、mef和tetC基因只存在于屠宰场区域水体中。一旦肉鸡在屠宰加工生产链中受到处理水、屠宰器械的微生物交叉污染,这些耐药基因极有可能会移转到鸡肉上。人们通过消费食用这些受耐药基因污染的鸡肉,耐药基因随之进入人体中。此外含有耐药基因的屠宰场废弃物若不经过处理直接排放,还有可能会污染地下水和增加土壤的微生物耐药性。本文在定性水平上展示了耐药基因在屠宰场的分布状况,屠宰场对耐药基因的传播起到了推进作用,对人类健康和生态环境存在着一定的危害。

表3 屠宰场不同区域水体中微生物耐药基因PCR检测结果
+,检出;-,未检出。下同。
+, Detected; -, Undetected. The same as below.

表4 屠宰场地面和器械表面微生物耐药基因PCR检测结果
3 讨论
James等[7]发现肉鸡屠宰加工生产链中存在单核细胞增生李斯特菌、弯曲杆菌、沙门氏菌、粪便梭菌等病原微生物;樊静等[6]发现屠宰场环境中存在的腐败菌可影响鸡肉品质和货架寿命,但对易造成鸡肉微生物交叉污染的家禽屠宰场区域水体、地面以及屠宰器械表面微生物结构和耐药基因研究较少。本文通过Illumina高通量测序技术对家禽屠宰场区域水体、地面和屠宰器械表面微生物的菌群结构进行分析,结果显示,在门的水平上以变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)和放线菌门(Actinobacteria)为主;在属的水平上主要为假单胞菌属(Pseudomonas)、不动杆菌属(Acinetobacter)、栖热菌属(Thermus)、嗜冷杆菌属(Psychrobacter)和希瓦氏菌属(Shewanella)。从定性水平上展示耐药基因在屠宰场的分布状况来看,屠宰场环境中存在21种耐药基因,其中8种耐药基因的检出率为88.9%以上。由此反映出屠宰场不同区域的水体、地面和屠宰器械表面存在种类较多的耐药基因,且存在一些条件性致病菌和腐败菌,但耐药基因各来自何种菌种需要进一步的验证。

