桃小食心虫成虫GOBPs与PBPs的基因克隆及 表达谱分析
田志强, 孙丽娜, 李艳艳, 张怀江, 闫文涛, 岳 强, 仇贵生
(中国农业科学院果树研究所, 兴城 125100)
桃小食心虫CarposinasasakiiMatsumura隶属鳞翅目Lepidoptera,蛀果蛾科Carposinidae,为害多种果树,包括苹果、桃、山楂、梨、枣等[1]。由于其具有分布广、隐蔽性强、世代重叠、自然天敌少等特点,一度被美国、智利、南非、俄罗斯等多个国家列为检疫性害虫[2]。桃小食心虫钻蛀时间短,幼虫蛀果为害时间长,使得在化学防治时容易错过最佳防治时间而常常需要多次重复喷药,致使事倍功半,同时还对环境造成不良影响。果实套袋虽可以起到较好的防治效果,但是枣、山楂等不便于套袋,并且随着劳动力费用日益增高和“无袋化栽培技术”政策的提出,桃小食心虫的为害有逐年加重的趋势[3]。近年来,引诱剂与驱避剂的开发为害虫防治提供了新的思路与策略。自1997年以来,研究发现桃小食心虫性信息素主要成分是顺-7-二十烯-11-酮和顺-7-十九烯-11-酮,但不同地区使用两种性信息素分子的比例存在显著差异,分别为150∶1,90∶10,20∶1等[4-5]。另外,在田间诱芯使用中两者发挥最佳效果的比例也不尽相同[6]。因此加强对桃小食心虫嗅觉系统的研究将会为桃小食心虫的防治与监控奠定理论基础。
研究表明,昆虫之所以具有寻找寄主、繁殖等能力,主要依靠其触角淋巴液中的气味结合蛋白(odorant binding protein,OBPs)[7-9]。通常昆虫脑部触角叶由两个平行亚系统组成,一个主要负责处理寄主植物气味分子,而另一个特化为专门处理种内或相似种之间的性信息素分子[10]。因此,OBP根据其功能主要分为普通气味结合蛋白(general odorant binding proteins,GOBPs)和性信息素结合蛋白(pheromone binding proteins,PBPs)[11]。PBPs主要存在于触角的毛形感器,并能够专一性地识别性信息素分子[12-13];GOBPs中GOBP1存在于毛形感器和锥形感器中,而GOBP2仅存在于锥形感器中,两者主要识别普通气味分子[14-15]。在功能领域,近年的研究表明,有些昆虫的GOBPs也可以与性信息素分子发生结合,甚至结合力高于PBPs[16];另外,PBPs也可以与植物挥发物等气味分子发生不同程度的结合[17-18]。
本研究在桃小食心虫触角转录组的基础上,利用RACE技术克隆GOBPs和PBPs基因,使用生物信息学方法对序列进行分析,并通过qPCR方法分别检测5个基因在雌雄成虫不同组织中的表达情况。本试验为进一步研究其在桃小食心虫气味识别的分子机制中的功能奠定了基础。
1 材料与方法
1.1 供试昆虫
于2015年7月在果树研究所(40.61°N,120.73°E)苹果园采集带有泪滴为害状的‘金冠’苹果。随后以未成熟的‘金冠’苹果在温度为(25±1)℃、相对湿度70%±5%、光周期为L∥D=15 h∥9 h的养虫室饲养获得桃小食心虫种群[19]。
1.2 总RNA提取,GOBPs和PBPs全长基因克隆
分别取桃小食心虫成虫的头、胸、腹、足、翅和触角组织各50 mg,放入盛有1 mL RNAiso Reagent试剂且无RNA酶的1.5 mL离心管中,利用组织破碎机匀浆后参照TRIzol试剂盒说明书提取总RNA。最后用RNase-free水溶解,在检测其浓度与纯度后,采用Prime ScriptTMRT Reagent Kit with gDNA Eraser试剂盒的方法将RNA反转录合成cDNA第一链,于-20℃保存备用。
GOBPs与PBPs cDNA全长序列扩增:根据桃小食心虫触角转录组数据(GFQL00000000)与NCBI库中的基因序列的BLAST结果,利用生物信息学分析,获得桃小食心虫2个GOBP和3个PBP的基因序列片段。根据5个基因片段序列并兼顾3′-RACE和5′-RACE至少重复200 bp的条件下,使用Premier 5.0分别设计3′-RACE和5′-RACE的Outer Primer和Inner Primer引物(表1)。利用Outer Primer和Inner Primer配对进行套式PCR反应,按照TaKaRa RACE试剂盒进行反转录和PCR扩增该基因3′和5′-末端cDNA序列。RACE Mix体系:PCR-Grade H2O 15.5 μL,2×Ex Seq Amp Buffer 25 μL,Seq Amp DNA Polymerase 1 μL;RACE反应体系:UMP(Universal Primer Mix)2 μL,cDNA 1 μL,引物各0.5 μL,Mix 16.6 μL。反应条件:98℃1 min;94℃ 30 s,62℃ 30 s,72℃ 1 min,35个循环;72℃ 10 min。将获得的目的片段切胶纯化,与pMD19-T载体连接后转化至DH5α感受态细胞,筛选阳性克隆测序后进行拼接可获得目的基因全长cDNA序列。
1.3 桃小食心虫成虫GOBPs与PBPs基因的生物信息学分析
将克隆得到的5个基因的全长序列进行生物信息学分析,利用在线软件ORF Finder (https:∥www.ncbi.nlm.nih.gov/orffinder/)确定其开放阅读框(open reading frame,ORF),并翻译为氨基酸序列,运用ExPASy服务器中的Compute pI/MW程序对蛋白质分子量与等电点进行预测。采用在线软件Signa1P 4.1(http:∥www.cbs.dtu.dk/services/SignalP/)进行信号肽预测。通过NCBI中的BLASTx程序搜索鳞翅目其他昆虫的OBP家族序列,分析它们的序列一致性;最后使用MEGA6.0软件进行CsasGOBPs和CsasPBPs与其他昆虫OBPs的系统发育进化树的构建,方法采用邻接法(neighbor-joining),并将结果进行1 000次bootstrap重复抽样分析。
1.4 桃小食心虫成虫GOBPs与PBPs基因的组织表达谱
以桃小食心虫触角转录组中的β-actin为内参基因,采用Primer Premier 5.0软件设计各目的基因与内参基因的定量引物(表1),使用TaKaRa公司的荧光定量试剂盒定量分析GOBPs与PBPs在桃小食心虫雌雄成虫不同组织间的表达水平。以1.2节中获得的cDNA为模板,无菌水为阴性对照,在定量PCR仪(CFX96 Real-Time Syste,BioRad)中进行荧光定量PCR。每个样品3次重复,反应体系为25 μL:2×SYBR Premix ExTaqⅡ(Tli RNaseH Plus)12.5 μL,10 μmol/L正反向引物各0.4 μL,200 ng/μL cDNA 1 μL,ddH2O 10.7 μL。反应程序:95℃ 30 s;95℃ 5 s,60℃ 30 s,72℃ 20 s,40个循环。反应结束后,根据各目的基因及内参基因Ct值,利用2-ΔΔCt相对定量法统计CsasGOBPs和CsasPBPs基因在成虫不同组织的表达量差异。
表1基因克隆与荧光定量所用引物
Table1PrimersforgenecloningandqRT-PCR

名称Name序列Sequence (5'-3')用途Purpose3'F-GOBP1-OGAAGCAGGTGTCGTTAGTTTTGGG3'R-GOBP1-IGCTGTCGCTACTGGTCCCACTAA5'F-GOBP1-OAGTGGTCCTCCTCAGCCTCGTTCT5'R-GOBP1TGAACTGTTGTCAGCATTATCGTGG3'F-GOBP2-OCAGTGAAAGGTGATGCGGTCCAGA3'R-GOBP2-ICTGCTTGCTTCAAACGAGATGCTAA5'F-GOBP2ACTTCTCCATCACCGCCTCAATCA5'R-GOBP2TCACTACTCGGTCGCAGTCATCAAC3'F-PBP1GCTGCTGCTTTGGATAAGTGTA3'R-PBP1AGGAATTTGGGTGCGTGGTTAT5'F-PBP1CTCAATCAGTGTCTCCATGTTCGG5'R-PBP1CGCATTCGTGGATCATGGTCACTA3'F-PBP2AGTGTCGGTGGTGGTTGCTCTA3'R-PBP2TTCTGGAAGGAGGGGTATGAG5'F-PBP2CAGCCAATATCTCTCCGATGACCA5'R-PBP2GTTAGGGATGGAGTTTTCACAGCC3'F-PBP3TAAACGCTGTCTCCTTGTCGGT3'R-PBP3CTTCTGGAAAGAGGAGTACGAGC5'F-PBP3CTTCAGTGACAAGCACGTCCATCT5'R-PBP3GAGACACTGGTCCTCCAATGCTTC扩增全长Primersfor genecloneF-GOBP1CGACACTTCAACCTGCTCACGGR-GOBP1CAGGACTCGGCGACAACAAACTCF-GOBP2GATGCGGTCCAGAGTGAGAGR-GOBP2CACCATCTTTGGGGACAGCF-PBP1TTGGGTGCGTGGTTATGTGCR-PBP1TCAATCAGTGTCTCCATGTTCGF-PBP2TCTGGAAGGAGGGGTATGAGCR-PBP2GACGCCATCTTTGAAGCAGCF-PBP3GTAAGCAAGGAGGTCGGATGR-PBP3CCATCTTCGGTGTCCAGTCGF-ActinGTGTTATGGTGTCTAGTTGGTR-ActinGATGTTCAGTTCTGTCTGGTAT荧光定量PrimersforqRT-PCR
2 结果与分析
2.1 GOBPs与PBPs基因的克隆与序列分析
本研究获得了桃小食心虫GOBP1、GOBP2、PBP1、PBP2和PBP3的cDNA全长序列,提交至NCBI数据库,登录号分别为:MG256559、MG256560、MG256556、MG256557和MG256558。5个基因的cDNA全长序列分别为617、811、755、795和561 bp,开放阅读框长度分别为504、492、507、492和498 bp,各基因去除信号肽后所编码的氨基酸序列特征如表2所示。桃小食心虫GOBP1与GOBP2的3′端非编码区长均为40 bp左右,5′端非编码区分别为74 bp和279 bp;在PBPs中,PBP3的5′端非编码区仅有14 bp,3′端非编码区长49 bp;而PBP1和PBP 2的5′端与3′端非编码区序列相对较长,5′端序列长度分别为73 bp和51 bp,3′端序列长度分别为340 bp和252 bp。桃小食心虫GOBPs和PBPs的特点均符合昆虫OBP基因家族的序列特征,通过结构模型“C1-X15-39-C2-X3-C3-X21-44-C4-X7-12-C5-X8-C6”(X代表相邻两个半胱氨酸之间的氨基酸残基数目)鉴定发现桃小食心虫5个气味结合蛋白均具有保守的6个半胱氨酸[20-21]。
2.2 CsasGOBPs和CsasPBPs的进化树构建与多重序列分析
桃小食心虫的CsasGOBPs和CsasPBPs的系统进化关系分析结果发现桃小食心虫与其他鳞翅目昆虫的GOBPs和PBPs这几个亚家族分成5大类,分别为GOBP1、GOBP2、PBP1、PBP2和PBP3。从进化树中可以看出PBP2和PBP3两个亚族的亲缘关系较近,它们所在的分支与PBP1亚家族所构的单系群平行;然而,相较于PBP亚家族,GOBP1和GOBP2虽然也与其他鳞翅目昆虫的GOBPs形成不同的分支,但是与它们的亲缘关系较远,这与它们之间所具有序列一致性较低的结果相符。
将GOBPs与PBPs序列在NCBI中采用BLASTx进行比对发现,桃小食心虫的GOBPs与小卷叶蛾科的GOBPs一致性较高,其中CsasGOBP1与大豆食心虫的LglyGOBP1一致性最高(72%),而CsasGOBP2与苹果蠹蛾CpomGOBP2的一致性最高(78%);在PBPs的BLASTN结果中CsasPBP1与烟芽夜蛾的HvirPBP1一致性最高(85%),CsasPBP2与麦蛾的ScerPBP一致性最高(71%),CsasPBP3与甜菜夜蛾的SexiPBP3一致性最高(69%)。将用于构建进化树的氨基酸序列进行多重序列比对发现,它们除了具有保守的6个半胱氨酸外,在其他位置的氨基酸也具有高度的一致性,例如甲硫氨酸(M)、苯丙氨酸(F)、亮氨酸(L)、缬氨酸(V)等一些疏水性氨基酸(图2)。
表2CsasGOBPs与CsasPBPs的序列分析1)
Table2SequenceanalysisofCsasGOBPsandCsasPBPs

基因Gene全长/bpFull length开放阅读框/bpORF氨基酸长度AA length信号肽(AA)SP蛋白分子量/KDaMW等电点PIGOBP16175041682416.604.85GOBP28114921642116.344.92PBP17555071692616.505.41PBP27954921642115.764.91PBP35614981661816.664.93
1) ORF:开放阅读框; SP: 信号肽; MW: 相对分子量; PI: 理论等电点。
ORF: Open reading frame;SP: Signal peptide; MW: Molecular weight; pI: Isoelectric point.

图1 桃小食心虫GOBPs和PBPs与其他鳞翅目昆虫相关蛋白的系统发育树Fig.1 Phylogenetic tree of the GOBPs and PBPs from Carposina sasakii and other lepidopteran insects

图2 CsasGOBPs和CsasPBPs与其他鳞翅昆虫相关基因的氨基酸序列相似性比较Fig.2 Alignment of amino acid sequences of CsasGOBPs and CsasPBPs with those from other insects
2.3 桃小食心虫成虫GOBPs与PBPs在不同部位的表达谱分析
本研究以桃小食心虫的β-actin为内参基因,采用qPCR技术调查了CsasGOBPs和CsasPBPs基因在桃小食心虫雌雄成虫头、胸、腹、足、翅和触角中的相对表达量。结果表明这5个基因都在雄虫触角中显著表达;在PBPs中尤其PBP2在雄虫触角中表达量更高,约为雌虫触角的600倍;另外,结果还显示这5个基因在雌雄成虫的翅和腹部有不同程度的表达(图3)。
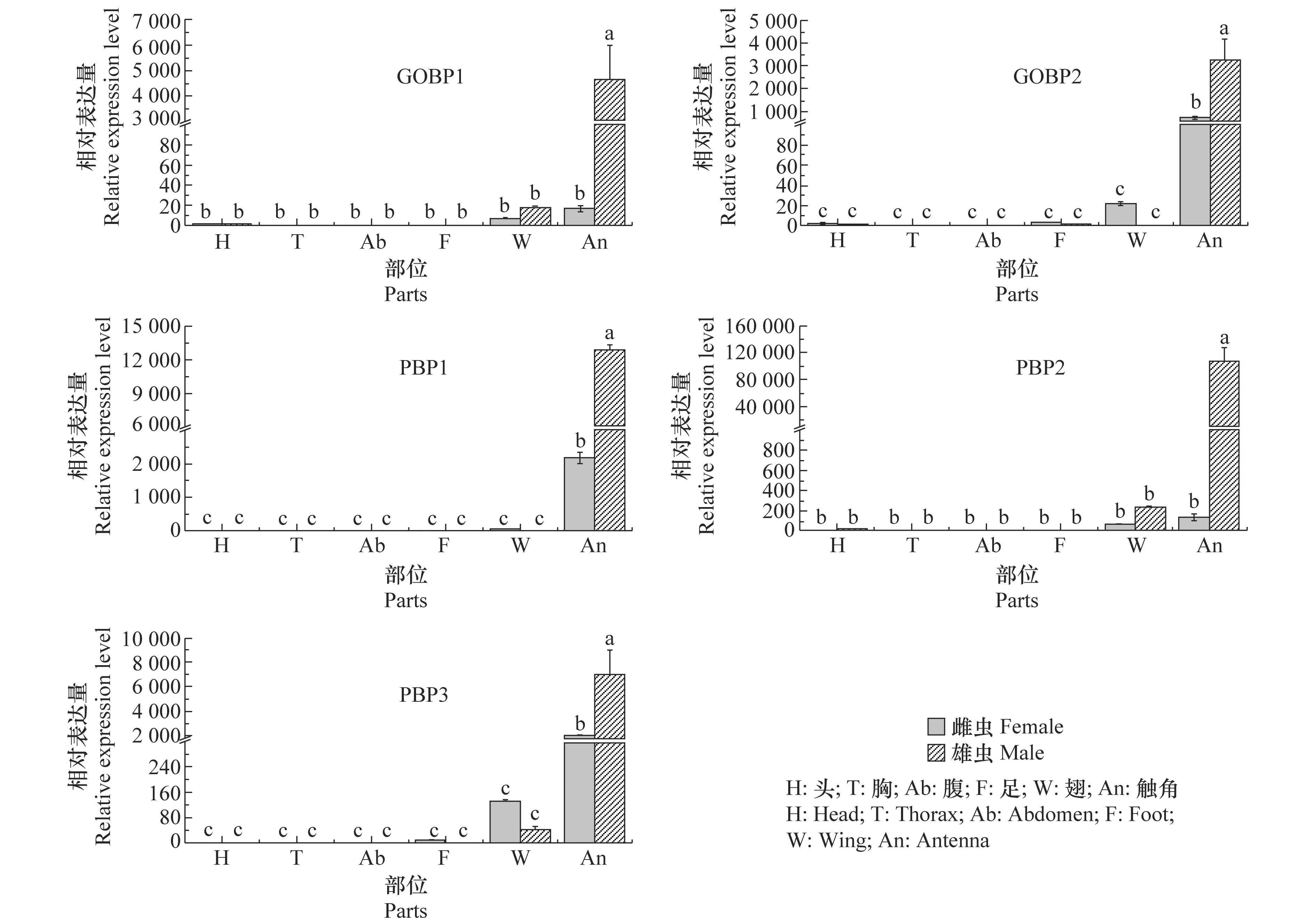
图3 CsasGOBPs和CsasPBPs在桃小食心虫雌雄成虫不同部位的表达谱Fig.3 Relative expressions of CsasGOBPs and CsasPBPs in different parts of female and male Carposina sasakii
3 讨论
OBPs在昆虫识别并结合寄主及自身所释放的气味分子的嗅觉感受过程发挥着关键作用;一般认为是PBPs识别并结合性信息素分子后,将其运输至嗅觉受体,指导昆虫的求偶行为。本研究以实验室已有的桃小食心虫触角转录组测序数据为基础,利用基因功能注释等方法筛选出这5个基因的序列片段,并通过RACE技术克隆得到5个基因的全长序列。利用生物信息学对这5个基因的信号肽、等电点和蛋白分子量等的分析表明这5个基因均具有OBPs家族的典型特征,即由120~160个氨基酸组成的酸性、水溶性的多肽分子,N端具有20个氨基酸构成信号肽序列。在对这5个基因蛋白序列的亲脂性分析中发现它们都有较为明显的疏水区域,同时,序列多重比对结果也表明不同种昆虫之间的PBPs与GOBPs在多个疏水性氨基酸位点高度保守,而气味分子多数为疏水性分子,这可能意味着它们在与配体分子结合的过程中发挥着关键作用,因此可能为结合的关键位点[22]。从已鉴定PBPs基因的昆虫中发现,不同昆虫中所具有的PBPs数目不同,例如家蚕只存在1种PBP,小地老虎存在3种PBPs,而亚洲玉米螟有PBP4和PBP5的存在[23-25];而不同昆虫中的性信息素分子的组成数也不尽相同,这可能表明不同的PBPs用于识别不同的性信息素分子。
现在越来越多的研究表明不仅PBPs可以与性信息素分子发生特异性结合,类似的GOBPs也可以与之相结合,甚至结合力更强于PBPs;例如在甜菜夜蛾的研究中发现SexiGOBP2相较于SexiPBP1对性信息素分子的结合能力更强[18]。同时系统进化树构建结果表明,GOBPs中GOBP1与PBPs所形成的单系群的亲缘关系更近,而与GOBP2所构成的分支距离较远;另外,在与其他鳞翅目昆虫的GOBPs与PBPs进行多重序列比对时发现它们之间的序列一致性能达到50%以上,有研究表明家蚕的BmorPBPs与BmorGOBPs亚家族基因簇中的基因很相似,因此,他们很有可能来自同一祖先基因的复制,后来在环境变化时由自然选择压而分化成不同的基因型[26-27]。
起初学者们认为PBPs在雄性昆虫的触角中特异性表达,但后来的研究在雌虫触角中也检测到了PBPs表达[13,22],本试验结果发现,CsasGOBPs与CsasPBPs在雌虫触角中也有表达;另有研究表明雌虫触角中的嗅觉感器存在大量PBPs,因此推断雌虫可能通过检测自身所释放的性信息素浓度,以此来辨别附近是否有同种同性个体的存在,进而避免同种之间的竞争;而有趣的是雄虫也可以释放信息素来增加雌雄昆虫的交配成功率,当雄蛾接近雌蛾后,便开始释放雄性信息素来抑制雌蛾释放雌性信息素,从而避免其他同种雄蛾求偶行为的发生[25]。此外,越来越多的研究证明GOBPs也可以与性信息素分子结合,在本研究中发现CsasGOBPs在桃小食心虫雄虫触角中显著高于雌虫触角中,而昆虫基因的表达模式在一定程度上可以反映其功能,而这种性别差异性表达可能表明它们在桃小食心虫性信息素分子的识别中也存在差异。
触角在昆虫的嗅觉识别机制中发挥着极为关键的作用,而多数嗅觉相关蛋白基因也在触角中特异性或高量表达。在小地老虎PBP1-3研究中发现,小地老虎的3个PBPs除了在触角中显著性表达外,在一些非嗅觉器官中也有表达,例如喙、唇须、头、胸、腹等部位也有低量的表达[13]。在本试验的组织表达谱中同样也检测到这5个基因在足、翅等组织中表达,这种现象可能与雌蛾寻找寄主产卵场所有关。当雌蛾产卵时其性信息素也随之分泌,而当同种雌虫在同一寄主的同一部位产卵时,能敏锐地接收到此性信息素信号并转移至新的寄主,最终避免后代之间的食物竞争以有利于后代的繁衍[28]。
综上所述,本研究获得了桃小食心虫CsasGOBPs和CsasPBPs,为进一步研究桃小食心虫GOBPs和PBPs与性信息素分子及寄主挥发物气味分子之间的结合机制及研发驱避或者引诱技术奠定基础。

