马氏珠母贝F8代4种壳色选育群体的EST-SSR遗传多样性分析
谭 杰,孙宗红,陶雅晋,姚高友,刘付少梅,刘锦上,刘志刚
( 1.广东海洋大学 水产学院,广东省南海经济无脊椎动物健康养殖工程技术研究中心, 广东 湛江 524088; 2.湛江银浪海洋生物技术有限公司,广东 湛江 524000; 3.雷州市源源至水产种苗繁育有限公司,广东 湛江 524200 )
马氏珠母贝(Pinctadamartensii)又称合浦珠母贝(P.fucata),在中国主要分布在南海、广东、广西、海南及台湾南部沿海海域[1-2]。该贝是培育海水珍珠的重要经济贝类,其孕育的珍珠称为“南珠”,在国际珍珠市场占据重要地位,具有极高的经济价值[3]。
1965年中国首次成功突破马氏珠母贝人工育苗技术,随后南珠产业迅猛发展,至20世纪90年代,该产业已成为广东、广西和海南等地区经济发展的重要支柱[4]。然而快速发展的同时,问题也日益突显。长期的累代养殖、不科学的育苗方式、频繁的病害侵扰和恶化的生存环境,使得马氏珠母贝种质退化严重,具体表现为生长速度缓慢、存活率降低、珍珠质量和产量下降等[5-6]。目前许多养殖户因为养殖经济效益低而处于停产观望状态。改良旧品种,培育新品种,克服种质退化,促进国内马氏珠母贝产业健康可持续发展已成为业界的共同呼声。为此,科研工作者开展了马氏珠母贝品种改良研究,并取得了一定成效。如杜晓东等[7]采用群体和家系选育的方法,获得了马氏珠母贝“海选1号”新品种,该品种与未经选育的马氏珠母贝相比,2龄贝平均壳宽和平均壳长分别提高21.2%和20.8%,留核率与珍珠珠层厚度分别提高22.3%和22.2%;何毛贤等[8]经6代群体选育,培育出了壳型肥厚、优质珍珠率高的“南科1号”新品种;喻达辉等[9]通过家系基因聚合为核心的选择育种技术,选育出了生长快、育珠性能优良的“南珍1号”新品种。
马氏珠母贝的壳色具有多态性,且不同壳色个体在生长速度、育珠性能、存活率等方面存在差异[10]。本课题组以壳色为辅助标记,以生长快、存活率高为选育目标,采用群体继代选育的方法开展了马氏珠母贝红、黑、黄、白4种壳色新品种培育研究。迄今为止,该贝4种壳色群体已经历8代选育且取得了较理想的选育效果,壳色趋于纯化、各经济性状均优于普通群体。但在分子层面上,经过8代群体继代选育后的4种壳色群体遗传分化程度如何,遗传差异达到什么水平,尚未查明。EST-SSR标记来源于基因的编码区,具有多态性高、数量丰富及共显性遗传等特点,能较准确地反映物种的遗传多样性[11-15]。笔者采用10对EST-SSR 引物对马氏珠母贝4种壳色群体的遗传多样性进行研究,以期从分子层面上揭示该贝4种壳色选育系F8的遗传差异和分化水平,为选育工作提供分子科学理论依据。
1 材料与方法
1.1 材料来源
课题组在广东省湛江市雷州源源至公司经8年群体继代选育获得马氏珠母贝红、黑、白、黄4种壳色群体,每种壳色群体随机挑选40个健康、有活力的个体,清洗干净后活体运回广东南海经济无脊椎动物健康养殖工程技术研究中心实验室,暂养3 d后,取闭壳肌组织置于无水乙醇中-20 ℃保存,用于后续DNA提取。
1.2 DNA提取及引物筛选
取无水乙醇保存的闭壳肌组织,解冻、剪碎,采用上海生工生物公司Ezup柱式提取试剂盒提取DNA,用1.0%的琼脂糖凝胶电泳检测DNA的质量,用分光光度计测定DNA的质量浓度。检测合格后用无菌水稀释至同一质量浓度,分装-20 ℃保存备用。选取10对扩增效果好的EST-SSR引物用于4种壳色马氏珠母贝群体遗传多样性分析,引物参考文献[16],序列信息及退火温度见表1。EST-SSR引物由上海生工生物公司技术部合成。
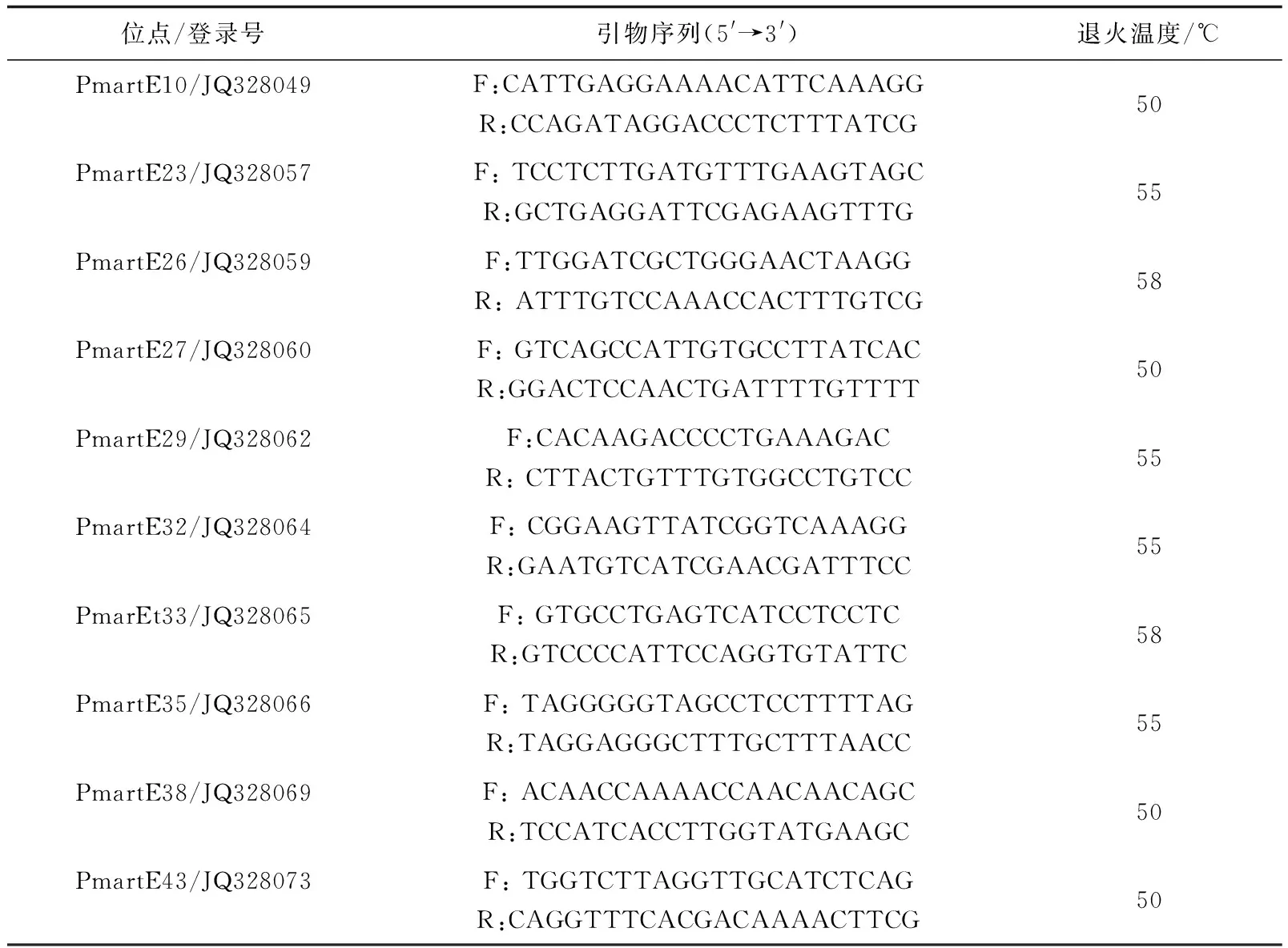
表1 EST-SSR引物序列及退火温度
1.3 PCR扩增及产物检测
10 μL反应体系:TaKaRa Taq TM酶(5 U/μL)0.1 μL,10×buffer 1.0 μL,MgCl2(2 mmol/L)0.6 μL, dNTPs(25 mmol/L)0.8 μL,正向引物和反向引物各0.8 μL,DNA模板1 μL,ddH2O 4.9 μL。
PCR反应程序:94 ℃预变性5 min;30次循环(94 ℃变性30 s,50~58 ℃退火30 s,72 ℃延伸1 min);72 ℃延伸10 min。
取2 μL PCR扩增产物,用1.0%琼脂糖凝胶电泳检测,若有清晰扩增条带则使用DYY-6C型电泳仪进行8.0%非变性聚丙烯酰胺凝胶电泳(恒定电压150 V,电泳3.5 h),扩增产物分离后,固定、银染、显色、观察、拍照。
1.4 数据统计与分析
使用Gel-Pro analyzer软件对聚丙烯酰胺凝胶图上的等位基因条带进行定位,确定等位基因的大小和基因型;利用Popgene32软件计算群体的等位基因数、有效等位基因数、期望杂合度、观测杂合度、遗传相似系数以及遗传距离等,同时进行Hardy-Weinberg平衡检验[17]。使用PIC-CALC软件计算多态信息含量;用MEGA 4.1构建UPGMA树[18]。
2 结 果
2.1 EST-SSR位点的多态性
10个EST-SSR位点的多态性参数见表2。10个位点共检测到46个等位基因,各位点的等位基因数为2~7个,平均等位基因数为4.6个,其中位点PmartE27的等位基因数最少(2个),位点PmartE32和PmartE43的等位基因数最多(7个);有效等位基因数为1.0880~5.1939个,平均有效等位基因数为3.1132个;期望杂合度为0.0812~0.8108,平均期望杂合度为0.6040;观测杂合度为0.0833~0.9917,平均观测杂合度为0.5595;多态信息含量以位点PmartE32为最高,达到0.7482,位点PmartE29最低,为0.0796,平均多态信息含量为0.5517,说明10个位点的遗传多样性丰富,其中有7个高度多态位点(多态信息含量>0.5),2个中度多态位点(0.25<多态信息含量<0.5)和1个低度多态位点(多态信息含量<0.25)。
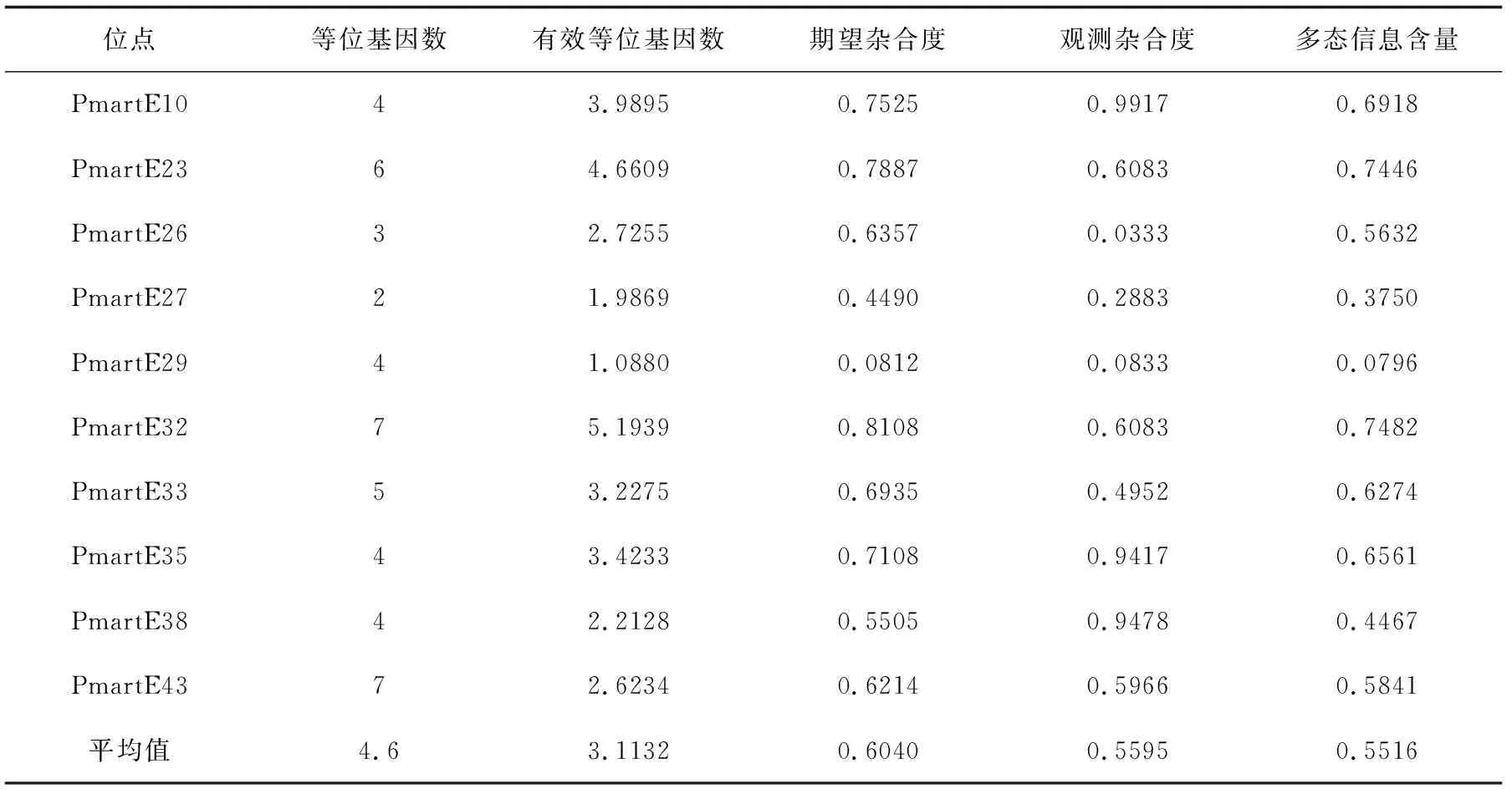
表2 10个EST-SSR位点的遗传多态性参数
2.2 4种壳色马氏珠母贝群体的遗传多样性
4种壳色马氏珠母贝群体的平均等位基因数为3.5~4.0,平均有效等位基因数为2.288~2.730;平均观测杂合度为0.445~0.627,平均期望杂合度为0.479~0.580,除黄壳色群体外,其余3种壳色群体的观测杂合度均大于期望杂合度,说明不存在杂合子缺失的情况;Shannon多样性指数为0.850~1.042;平均多态信息含量为0.423~0.509,每种壳色群体多态信息含量低于0.5的位点为3~6个,低于0.25的位点为1~2个,这与选择作用有关,4种壳色群体的多态性信息含量处于中低等水平。黄壳色群体中位点PmartE33以及白壳色群体中位点PmartE29的平均观测杂合度、平均期望杂合度和多态信息含量值均为0,说明这两个位点在这两个群体中杂合子缺失严重,基本达到纯合。黄壳色群体中各遗传参数值最小,而红壳色群体中除有效等位基因数外,其他参数值等位基因数、观测杂合度、期望杂合度、多态信息含量和Shannon多样性指数均最大,说明红壳色群体的遗传多样性最高,黄壳色群体的遗传多样性最低,4种壳色群体总的遗传多样性维持在较高水平。
2.3 Hardy-Weinberg平衡检验
Hardy-Weinberg平衡检验结果表明,40个群体—位点组合中有31个组合显著偏离平衡(P<0.05),占总位点的77.5%。4种壳色群体中均偏离的位点有5个(PmartE10、PmartE23、PmartE26、PmartE35、PmartE38),在白、黑、黄3种壳色群体中显著偏离的位点有1个(PmartE32)。红壳色群体中有6个位点(60%)显著偏离平衡,白壳色群体中有8个位点(80%)显著偏离平衡,黑壳色群体有8个位点(80%)显著偏离平衡,黄壳色群体中有9个位点(90%)显著偏离平衡。说明人工选择压力下,群体基因频率发生了改变。

表3 4种壳色马氏珠母贝群体在10个EST-SSR位点的遗传多样性参数
注:Na:等位基因数;Ne:有效等位基因数;Ho:观测杂合度;He:期望杂合度;I:Shannon多样性指数;PIC:多态信息含量;P:哈温平衡检验;*:显著偏离哈温平衡(P<0.05).
2.4 4种壳色马氏珠母贝群体的遗传分化
10个EST-SSR位点在4种壳色群体中的遗传分化指数为0.0101~0.3025,平均为0.1206(0.05<遗传分化指数<0.15)(表4),说明4种壳色群体间的遗传分化处于中等水平,这与课题组8年群体继代选育有关。各位点的基因流值0.5764~24.5841,除位点PmartE38的基因流值较大外,其位点的基因流值均较小,平均为1.8225,说明4种壳色马氏珠母贝群体间未发生基因流动。
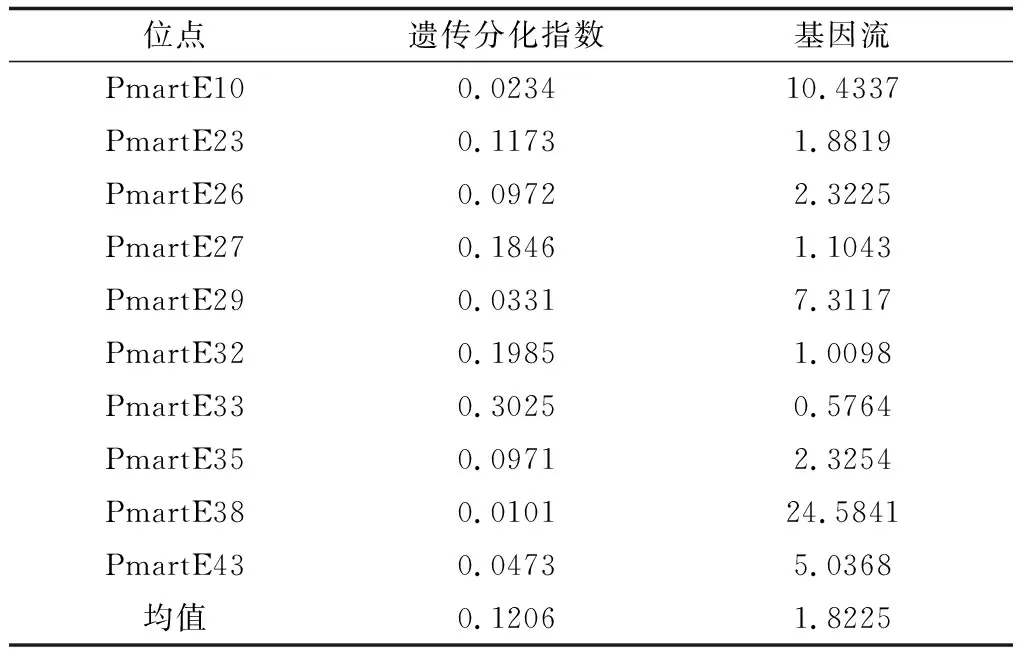
表4 4种壳色群体的遗传分化指数和基因流值
2.5 4种壳色马氏珠母贝群体间的遗传距离和遗传相似性
4种壳色马氏珠母贝群体间的遗传相似系数为0.7107~0.8418,遗传距离为0.1742~0.3415,说明4种壳色马氏珠母贝群体间存在一定的遗传差异,其中红壳色群体和黑壳色群体的遗传相似度最高(0.8418),遗传距离最小(0.1722),白壳色群体和黄壳色群体之间的遗传相似度最低(0.7107),遗传距离最大(0.3415)。基于Nei′s遗传距离,采用UPGMA法,对种壳色马氏珠母贝群体进行聚类分析(图1),结果显示4种壳色群体共分为3类,其中红壳色群体和黑壳色群体最先聚为一类,然后和白壳色群体聚集成一类,最后与黄壳色群体聚为一类。

图1 4种壳色马氏珠母贝群体的聚类图

群体红壳色白壳色黑壳色黄壳色红壳色—0.83170.84180.7458白壳色0.1842—0.79490.7107黑壳色0.17220.2296—0.8282黄壳色0.29330.34150.1885—
3 讨 论
3.1 4种壳色马氏珠母贝群体间的遗传分化
遗传分化指数和遗传距离是衡量群体间遗传分化程度的两个重要指标,在良种选育过程中,由于人为施加的选择压力,基因频率常常发生改变,群体间的遗传分化水平也随之改变。水产动物中,颉晓勇等[19]采用微卫星分子标记技术分析了吉富品系尼罗罗非鱼(OreochromisniloticusGIFT)9代选育群体的遗传结构,得到F0与F6、F7、F8和F9代之间的遗传分化指数分别为0.0377、0.0644、0.0609、0.0759,说明随着选育代数的增加,吉富品系尼罗罗非鱼的遗传分化程度逐渐变大。陈静等[10]以RAPD分子标记研究了马氏珠母贝黑、白、红、黄4种壳色选育系F3的遗传变异,发现各壳色选育系间的遗传分化系数为0.2143~0.3186,遗传距离为0.1135~0.1968,大于王爱民等[20]利用RAPD分子标记分析的马氏珠母贝3个不同地理野生群体间的遗传距离,说明本课题组的马氏珠母贝F3代壳色选育系的遗传结构已发生改变,这与王学颖等[3]报道的马氏珠母贝金黄壳色系F3和基础群体间的遗传分化系数一致。白成等[21]利用微卫星分子标记,分析了马氏珠母贝F5代白、黑、黄、红4种壳色家系的遗传结构,结果显示,4个家系间的遗传分化系数值为0.113~0.793,与普通养殖群体及壳色选育系F5相比,4种壳色家系间的遗传距离变大;朱晓闻等[1]报道马氏珠母贝F5代4种壳色选育系群体在10个微卫星位点处的平均遗传分化系数为0.0445,遗传分化程度较小,遗传距离为0.0629~0.1729;本研究中,红、白、黑、黄4种壳色群体间的遗传分化系数为0.0101~0.3025,平均值为0.1206,遗传距离为0.1722~0.3415,与选育系F5代相比,F8代4种壳色群体间的遗传距离和遗传分化程度增大,遗传变异提高,群体遗传结构改变明显,这是多年定向选育的结果。
3.2 人工选育对马氏珠母贝4种壳色群体遗传多样性的影响
遗传多样性是良种选育的基础和前提,因此,进行遗传选育改良时要尽可能的维持选育群体的遗传多样性,预防经济性状衰退,以获得高品质的新品种。本研究中,4种壳色马氏珠母贝群体的平均等位基因数为4.5,其中红、黑、白、黄4种壳色群体的等位基因数分别为4.0、3.7、3.6、3.5,其值均小于平均水平,说明每种壳色群体均存在等位基因丢失的现象且黄壳色群体丢失最严重,这与本课题组多年定向选育有关。因人工选育导致等位基因丢失的现象在鳜鱼(Sinipercachuatsi)[22]、日本囊对虾(Marsupenaeusjaponicus)[23]、长牡蛎(Crassostreagigas)[24]、大珠母贝(Pinctadamaxima)[25]和仿刺参(Apostichopusjaponicus)[26]等水产动物群体中均有报道。有文献资料显示,马氏珠母贝普通养殖群体的观测杂合度为0.15~0.56,期望杂合度为0.38~0.75。朱晓闻等[1]报道壳色选育系F5代的期望杂合度为0.662~0.685,多态信息含量为0.6025~0.6230。本研究中,4个群体的观测杂合度和期望杂合度分别为0.445~0.627和0.479~0.580,除黄壳色群体外,其余3种壳色群体的观测杂合度均大于期望杂合度,说明这3个群体受人工选育的影响小,遗传多样性较丰富。与马氏珠母贝普通养殖群体相比,4种壳色群体的观测杂合度和期望杂合度处于中等水平,两者之间存在一定的差异,出现差异一方面与人工施加的选择压力有关,另一方面与所选取位点的变异程度不同有关。与选育系F5相比,F8的期望杂合度和多态信息含量均降低,说明经过8代选育4种壳色马氏珠母贝群体的遗传多样性降低,但总体仍处于较高水平。这与选育时选取的方法有关,本研究采用群体继代选育的方式确保了有效繁育群体的数量,减少了近交衰退几率。这与聂鸿涛等[27]报道的菲律宾蛤仔(Ruditapesphilippinarum)人工选育群体及田野等[28]报道的泥蚶(Tegillarcagranosa)人工选育群体遗传多样性变化的趋势极相似。因此,在今后的选育工作中,应注重亲本群体的数量,尽可能避免亲缘近交,实现种质资源的健康可持续发展。

