温度影响下DNA分子欧拉临界力模拟研究
辜凌云,张敏,周勋
(1.贵州师范大学 物理与电子科学学院,贵州贵阳 550025;2.贵州大学 大数据与信息工程学院,贵州贵阳 550025)
0 引言
DNA力学性质一直以来都是研究的重点,原因之一是其在生物学行为中发挥着重要作用,如DNA的复制、转录、重组等。长期以来关于DNA性质的研究大多集中于碱基序列的生物化学性质,对物理性质研究较少,导致其力学相关性质认识有所不足。
随着单分子操纵技术的发展,实验上通常利用原子力显微镜(AFM)、磁镊子(MT)、光镊子(OP)等技术[1-4]对单个DNA分子施加力的作用,测量其物理性质。而相对于实验,利用计算机从理论上对DNA分子进行模拟,也是表述其性质的有效手段。
DNA分子拥有高曲率是其生物活性的基础,DNA发生弯曲在许多生理过程中是不可或缺的一部分,DNA欧拉临界力作为DNA分子开始发生弯曲时所需的力,对其进行探究十分有意义。因为DNA分子临界力与弯折刚度有关,而实验表明弯折刚度会受温度影响,当温度范围在3℃到53℃时,弯折刚度与温度之间的关系可以近似表示为g=(3.19×10-19-T·4.14×10-22)erg·cm[5],所以十分有必要探究温度对DNA欧拉临界力的影响。此外根据弹性理论[14]可知,DNA分子欧拉临界力还与DNA链长有关,所以同样有必要对不同长度下的DNA欧拉临界力进行研究,但是想要通过实验直接测量DNA分子弯折力十分困难,而采用计算模拟的方法则能很好的解决这一问题。
通过对理论和实验结果的分析[6],我们基于蠕虫链模型(Worm-Like Chain Model)[7]和胡克弹簧模型(Hooke spring model),建立可表征DNA微力学性质的力学模型(图1),此基础上考虑DNA分子的弯折和拉伸,利用Monte Carlo方法进行模拟。最终通过模拟,给出不同长度、温度下的DNA分子力以及临界力。
1 理论模型及计算方法
1.1 DNA微力学模型
研究通过结合目前的单分子实验,建立如图1所示的DNA微力学模型[8],主要考虑DNA分子弯折能、DNA分子片段弹性能以及弹簧的弹性能,体系总能量表示为:

Et为DNA分子弯折能,利用蠕虫链模型[9]表征。DNA分子每个片段取向矢量为t^i,相邻片段取向间的夹角为θ,弯折刚度为g,则DNA分子弯折能可以表示为:

对于DNA受温度影响,这里主要考虑温度对DNA分子驻留长度的影响,弯折刚度g与DNA驻留长度lp存在关系lp=g/kBTkB为玻尔兹曼常数,T为温度)[10][11],代入关系g=(3.19×10-19-T·4.14×10-22)erg·cm[5],则驻留长度可以表示为,又根据g=lp,b为DNA分子的片段长度,对于长度小于500个碱基对的DNA分子,b约为1nm,即g=lp,可以得到:

b为DNA分子片段的总弹性能,每个片段原长度为bnm,改变分子构象后,片段的长度变为binm,DNA分子弹性能可表示为:

k1为双链DNA分子片段的弹性模量[12],约为1200pN/nm。
El为连接在DNA分子首末两端的弹簧所具有的的弹性能,如图1所示,弹簧原长度为l0,〈R〉k1为DNA分子构象的首末端距,弹簧的弹性能可表示为:

k2为限制弹簧的弹性模量[12],约为1200pN/nm。

图1 DNA微力学模型示意图
图2 构象变化方法
1.2 Monte Carlo模拟[13]及Metropolis判据
在模拟过程中,产生DNA分子新构象的方法如图2所示,图2中为DNA分子链中的部分片段,模拟中随机选取DNA链上的点(M或N)沿着xyz方向进行扰动产生新的DNA分子构象,扰动的幅度由模拟产生的随机数进行控制。
抽样过程采用了Metropolis判据,将原构象能量Eold与新构象能量Enew进行对比。若新构象能量小于原构象能量(Enew<Eold),则新构象完全被接受(p=1);若新构象能量大于原构象能量(Enew>Eold),则将计算其概率p=exp[(Eold-Enew)/kBT],并与随机数χ(0~1)进行比较,若p<χ则被接受,反之不接受,其目的是为了使模拟能够遍历所有可能情况。
2 结果与讨论
2.1 DNA分子力随链长变化
常温下,DNA驻留长度lp为50nm,根据弹性理论[14],可以由公式fcr=π2E/L2计算其理论临界力大小,其中E为弹性模量,I为转动惯量,L为长度。对DNA分子链,可以将其视为弹性杆[15],即EI=lpkBT,理论上DNA分子链的欧拉临界力可以表示为:

模拟得到的DNA链的分子力则可以通过如图1所示,连接在其两端的弹簧所受力来表征。模拟过程中不断改变弹簧的长度,当弹簧长度接近DNA分子长度,即弹簧长度与DNA链长比值接近1时,力会出现转变,转变点的力即为该长度DNA分子的欧拉临界力,弹簧力可以表示为:

〈R〉为DNA分子链的首末端距,k2为弹簧的弹性模量,l0为弹簧的原长度。
基于上述理论,我们分别对长度为5 nm、9 nm、12 nm、15 nm、20 nm的DNA进行模拟,通过改变弹簧的长度,得到对应的欧拉临界力。将临界力的理论值与模拟值比较,得到表1的结果,其模拟值与理论值非常接近,说明模拟工作是可靠的。

表1 不同长度DNA欧拉临界力理论值与模拟值对比
2.2 温度对临界力的影响
对于DNA受温度的影响,主要由式(3)中弯折刚度与温度的关系体现。如图4,是DNA长度为9nm时,连接在其两端的弹簧所受力随温度的变化情况,曲线从上至下分别为10℃、20℃、30℃、40℃、50℃。从图中可以看出,温度越高,DNA分子受力越小,其转折处对应的欧拉临界力也越小。
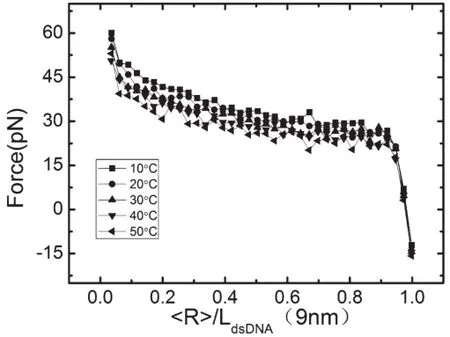
图4 温度对9 nmDNA分子力的影响

图5 不同长度下温度对DNA欧拉临界力的影响
图5为不同长度的DNA,其欧拉临界力随温度的变化情况,从上至下其长度依次对应9 nm、15 nm、20 nm。结果显示随着温度升高,欧拉临界力都呈现出逐渐减小趋势,并且DNA链长度越长,临界力波动越小,折线更加平滑,说明长度越长DNA分子欧拉临界力受温度影响越小。
3 结论
模拟结果显示,基于蠕虫链模型和胡克弹簧模型搭建的DNA微力学模型能够有效表征DNA的力学性质,得到的模拟值与理论值符合良好,说明DNA分子具备与弹性杆相类似的力学性质,可以用弹性理论进行解释。通过对不同温度下的DNA分子力进行模拟,结果显示随着温度升高,DNA分子力下降,不同长度下的所对应的DNA欧拉临界力均呈现减小的趋势,并且长度越短受温度影响越明显。
[1]Binnig G, Quate C F, Gerber C. Atomic force microscope[J]. Physical review letters, 1986, 56(9): 930.
[2]Gosse C, Croquette V. Magnetic tweezers: micromanipulation and force measurement at the molecular level[J]. Biophysical journal, 2002,82(6): 3314-3329.
[3]Wang M D, Yin H, Landick R, et al. Stretching DNA with optical tweezers[J]. Biophysical journal, 1997, 72(3): 1335-1346.
[4]Neuman K C, Nagy A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy[J]. Nature methods, 2008, 5(6): 491.
[5]Geggier S, Kotlyar A, Vologodskii A. Temperature dependence of DNA persistence length[J]. Nucleic acids research, 2010, 39(4): 1419-1426.
[6]Shroff H, Reinhard B M, Siu M, et al. Biocompatible force sensor with optical readout and dimensions of 6 nm3[J]. Nano letters, 2005, 5(7):1509-1514.
[7]Flory P J, Volkenstein M. Statistical mechanics of chain molecules[J].Physics Today, 1970, 23(5):71-71.
[8]刘艳辉. 微环 DNA 微力学, 拓扑性质及 DNA 凝聚机制研究[D].贵州大学, 2009.
[9]Colby R H, Rubinstein M. Polymer physics[J]. New-York: Oxford University, 2003: 274-281.
[9]De Groot S R, De Groot S R. Thermodynamics of irreversible processes[M]. Amsterdam: North-Holland, 1951.
[10]Schellman J A. Flexibility of DNA[J]. Biopolymers, 1974, 13(1): 217-226.
[11]Cocco S, Marko J F, Monasson R. Theoretical models for singlemolecule DNA and RNA experiments: from elasticity to unzipping[J].Comptes Rendus Physique, 2002, 3(5): 569-584.
[12]刘艳辉, 胡林. DNA 微环拓扑性质的 Monte Carlo 模拟[J]. 计算物理, 2009, 26(1): 152-158.
[13]Timoshenko S. Theory of Elastic Stability[M]. Tata McGraw-Hill Education, 1970.
[14]Hinner B, Tempel M, Sackmann E, et al. Entanglement, elasticity, and viscous relaxation of actin solutions[J]. Physical Review Letters, 1998,81(12): 2614.

