合作网络视角下的基因编辑领域科研团队识别及产出绩效测度
王菲菲,陈晓璇,杨辰毓妍
(1.北京工业大学 经济与管理学院,北京 100124;2.中国医学科学院 医学信息研究所,北京 100020)
基因编辑是指对基因组进行定点修饰的一项新技术,其中被业内誉为“基因剪刀”的CRISPR基因组编辑技术曾入选《科学》杂志公布的十大科学突破。利用该技术,可以精确地定位到基因组的某一位点上,在这位点上剪断靶标DNA 片段并插入新的基因片段。[1]现如今,基因编辑已经投入基因功能研究,基因治疗,构建模式动物以及改造和培育新品种等方面,取得了良好的成绩。在未来的发展中,基因编辑技术必将成为生命科学和生物医学等领域研究与应用的重要工具,《科学》杂志称其“将会给许多领域带来持久的兴奋和乐观”,“势必对研究产生革命性影响”。
在大数据背景下,科研工作的合作不断增强,科研人员的合作关系深刻地影响了科学的发展,合作成了研究中的普遍现象,对于新兴的基因编辑领域来说更是如此。因此,通过对合作规律的研究和分析,我们能更全方位地把握某一领域发展轨迹,进一步探索现有结构,预测未来发展方向。有的科研团队是作为一个研究实体而存在的,但是更多的科研合作从外部难以识别,科学研究中的合作往往是通过产出论文的共同署名来体现的。[2]因此,在合作网络视角下,通过对合著论文进行计量分析,利用社会网络分析法和可视化工具发现团队,研究团队合作模式成为一种常用手段。[3]而如何衡量合作模式的优劣则通常利用绩效测度的方法,即研究怎样改变与利用影响团队表现的因素来提高团队绩效,大大丰富了科研团队的研究内容,为提出可行建议提供依据。
本文将借鉴前人经验,对基因编辑这一热点领域中科研人员的合作模式进行研究,对团队绩效进行测度,以期为该领域人员选择合作对象和合作模式提供一定参考,也有助于进一步认识基因编辑领域当前研究脉络,把握未来的发展方向。
一、数据来源与研究方法
(一)数据来源
本研究的数据样本来源于“Web of Science”数据库,按“gene edit* or crispr”为主题进行检索,检索时间段为1980年至2015年,检索时间为2015年12月25日。共检索文章10503篇,截取article,proceedings paper,review,book review四种文献共9020篇为样本进行分析研究。
(二)研究方法与工具
本文运用社会网络分析法进行合著网络中科研团队的发现,探索团队的网络结构,确立其产出绩效因素,提出对科研团队建设的有效建议。社会网络分析法是对社会网络中各种关系结构及其属性加以分析的一套理论和方法,它主要分析的是行动者之间的关系模式。[4]该方法已经被证实可以应用于作者合作关系分析以及网络结构阐释。[5-6]本研究使用SPSS和Ucinet作为数据分析和可视化工具。
(三)研究对象界定
发文量是衡量一个研究者科研水平和学术能力的重要指标,一个领域的核心作者自然是在该领域产出较多,取得较高成就的带头人,故而在确定数据样本之后,将文献的所有作者提取出来。在此过程中笔者发现在基因编辑领域发文量较大的作者中中国研究者占很大比例,另外考虑到国外作者在署名时姓名缩写的问题,故而为了保证样本数据的准确性,对发文量大于等于10篇的作者共210人进行全名-机构人工比对消歧的方法,最终确定该领域173位核心作者如表1所示。
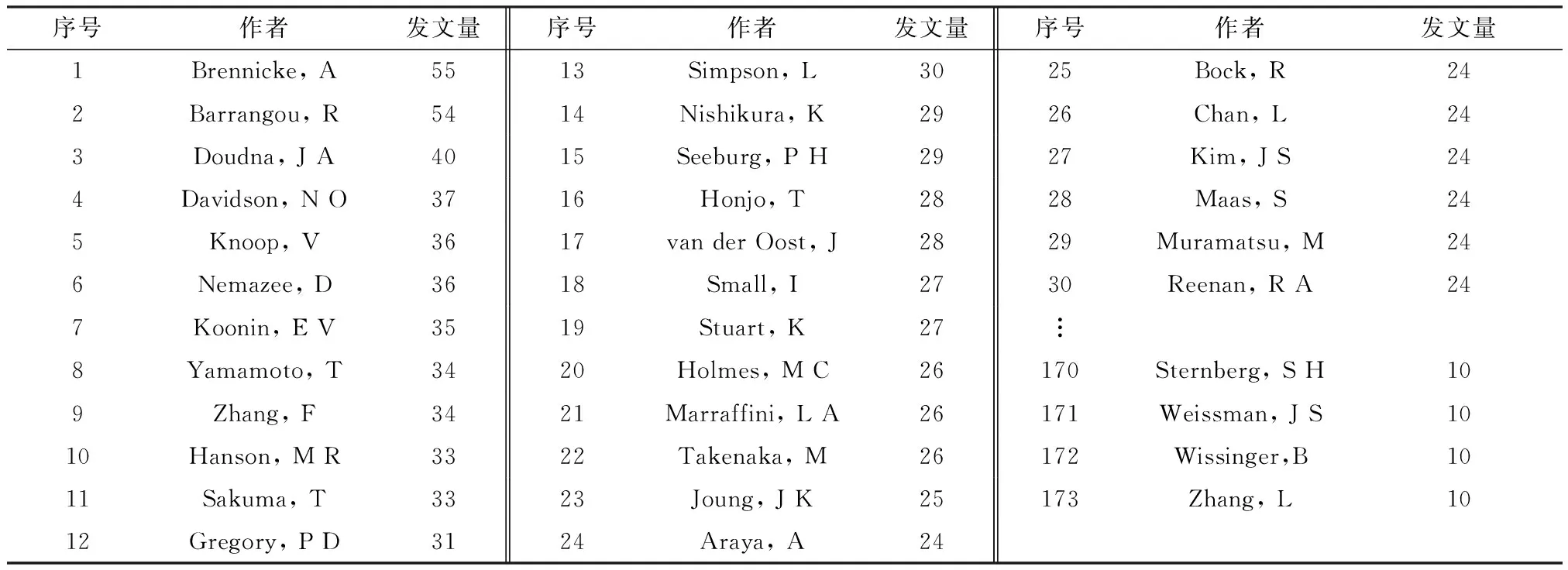
表1 发表10篇以上的核心作者群
二、基因编辑领域科研团队识别
本文根据社会网络分析方法对基因编辑领域作者合作网络进行如下几个步骤的分析:选定有173位作者构成的核心作者群作为研究样本;利用VBA编程自动构建核心作者间的合作矩阵;使用NetDraw绘制合作网络图;根据凝聚子群分析结合网络图确定核心作者团队;进行网络特征分析;对每个团队进行绩效测度指标计算及分析。
(一)构建作者合作关系网络
由于本文主要研究团队识别及其绩效测度,故已经对图进行了进一步处理,排除了孤立点,使得图像更加清晰,但是为了保证研究准确性,进一步使用了凝聚子群的方法对团队识别进行进一步排查。
该网络由基因编辑领域核心作者共同发文而形成的一个个子网组成,虽然有较大的桥梁型结构网络以及一个网架型网络,但是整体而言连通性不够好。网络中每一个顶点代表一个该领域研究人员,顶点的大小和作者的发表论文数量成正比,圆点越大,该作者产出越多;顶点之间的连线表示作者之间的合作发表文章的关系,线条的粗细与研究人员合作发表文章的频次成正比,线条越粗,研究人员合作发表论文频次越高。[7]大型桥梁型网络和网架型网络属于高产作者聚集较多和合作频次较高的网络,但是该大型桥梁型网络中桥梁节点较多,故采取凝聚子群法对网络进行进一步划分与合作关系网络图进行对比分析。
(二)凝聚子群分析及团队识别
为了确保团队识别的准确性,以及排除在构建可视化网络时作图的误差,本文还将凝聚子群法中N-clique法运用于团队发现的研究中。N-clique即n-派系,是指如果在一个团队中,任何两位作者之间在整体交往中的距离最大不超过n,就称为n-派系。[8]将当n值为2,最小节点数为3时得到的多个团体的作者进行合并处理,可进一步归结为12个小团体,这也与NetDraw绘制的社群图一致,分别为:
团队1网架型:Gregory,PDHolmes,MCUrnov,FD Rebar,E J Miller,J C Wang,J B Doyon,Y Zhang,L
团队2三角型:Harris,R S Neuberger,M S Petersen-Mahrt,S K
团队3桥梁型:Araya,A Maeder,M L Reyon,D Cathomen,T Blanc,V Joung,J K Davidson,N O Navaratnam,N Voytas,D F Anant,S Carlson,D F
团队4桥梁型:Stuart,K Salavati,R Panigrahi,A K Read,L K Lukes,J Benne,R Sloof,P
团队5网架型:Marusawa,H Chiba,T Kinoshita,K Honjo,T Muramatsu,M Nagaoka,H
团队6网架型:Jantsch,M F Levanon,E Y Eisenberg,E Rechavi,G Amariglio,N
团队7网架型:Higuchi,M Maas,S Seeburg,P H Sprengel,R
团队8三角型:Gallo,A Keegan,L P O’Connell,M A
团队9三角型:Gualberto,J M Bonnard,G Grienenberger,J M
团队10三角型:Vartanian,J P Wain-Hobson,S Henry,M
团队11桥梁型:Weissman,J SDoudna,J A Staals,R H J Jore,M M Wiedenheft,B Severinov,K Westra,E R Fineran,P C Semenova,E Brouns,S J J van der Oost,J Makarova,K S Wolf,Y I Gasiunas,G Charpentier,E Horvath,P Banfielf,J F Siksnys,V Fremaux,C Barrangou,R Moineau,S Koonin,E V Zhang,F Terns,R M Li,H Terns,M P
团队12桥梁型:Prangishvili,D She,Q X Shah,S A Garrett,R A Backofen,R Small,I Randau,L Okuda,K Nakamura,T Sugita,M Sugiura,M Kossel,H Maier,R M Marchfelder,A Knoop,V Brennicke,A Wissinger,B Schuster,W Zehrmann A Verbitskiy,D Takenaka,M
12个核心科研团队,人数为3—26人,差别较大。第11和12号团队之间联系也较密切,也可视为一整个比较庞大的桥梁型团队,但是根据凝聚子群分析结果,以及11号团队中多名成员贡献和其他团队成员比较,把其分为由26人组成的团队11和21人组成的团队12。需要说明的是,根据Ucinet带有的凝聚子群分析算法得出结果,有部分团体并未在合作图中显示,笔者认为这是因为为了使合作图清晰,在作图时使用阈值进行筛选,有的合作关系实际强度不是非常显著,故而被排除。并且本研究利用合作发文量作为合作指标,实际科研活动中,产出并不仅仅是文章的发表,也会有专利等其他类型产出。
(三)团队网络模式分析
根据上文团队识别结果,确定基因编辑领域存在上述12个核心作者合作团队。根据团队成员的数量以及成员间的关系特点,可以用三种团队类型来描述,分别是三角型、网架型以及桥梁型。[9-10]三角型团队是由三个作者相互合作形成的子网络,虽然也可实现某种程度上不同学科不同知识背景的研究人员合作,但是团队规模太小,不利于资源的传递和新知识的传播。网架型团队由多作者合著关系构成,成员之间合作非常密切,信息共享程度较高,但是缺少成员的流动,难以实现新知识的引入和融合。桥梁型团队也是由多作者的合著关系所构成,但是实际上可以看作是多个团队构成,但是有的团队成员起着桥梁的连接作用,这类网络结构有利于不同学科不同领域研究者的交流,实现跨领域运用技术,知识融合。

表2 各类型团队分布情况
如表2所示,三种团队类型所占比例一样,但是明显有两个桥梁型团队较为庞大,分别是团队11和团队12。虽然桥梁型团队具有跨学科合作的优点,有利于小团体之间优势互补,使得知识跨领域扩散融合,产生创新的动力,但是和其他团队不同的庞大的规模使得有必要通过数据指标对所有团队进行绩效测度分析。
三、基因编辑领域科研团队绩效测度
进行科研团队合作模式的研究,最大的目的是研究哪些具体的因素影响到团队的表现,怎样改变这些因素才最有利于团队进行工作,提高其产出,这也是许多研究者从不同角度探讨的重要问题。本研究试图发现网络的结构特性对团队效率的影响,所以采用网络的主要特征作为输入对象,如密度,聚类系数和距离[11]。团队的输出采用了5个指标,分别是团队总发文,人均发文,总被引频次,人均被引频次以及文章均被引频次,其中团队总发文与总被引频次属于绝对指标,而人均发文、人均被引频次和文章均被引频次属于相对指标(见表3)。[12]

表3 输入输出指标
(一)输入指标测度

(2)聚类系数:衡量网络的聚集性,在合著网络中表现为作者的所有合作对象里也存在实际合作关系的概率。网络的聚类系数为所有作者的聚类系数的平均值。假设网络中的一个节点i有ki条边将它和其他节点相连,这ki个节点就称为节点i的邻居。显然,在这 个节点之间最多可能有ki(ki-1)/2条边。而这ki个节点之间实际存在的边数Ei和总的可能的边数之比ki(ki-1)/2就定义为节点i的聚类系数Ci,即
Ci=2Ei/(ki(ki-1))
整个网络的聚类系数C就是所有节点i的聚类系数Ci的平均值。
(3)距离:两点之间的距离指的是连接这两点的最短路径长度。我们把关联图中最长的距离叫作图的距离。网络中两个节点i和j之间的距离dij定义为连接这两个节点的最短路径上的边数。网络中任意两个节点之间的距离的最大值称为网络的直径,记为D,即
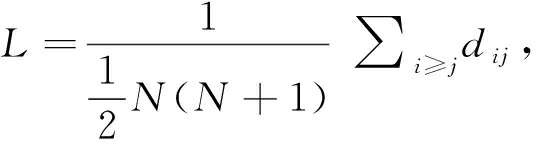
分别对12个团队的密度,聚类系数和距离进行计算,结果如表4所示。
(二)输出指标测度
本文采取不区分作者排名的方法计算发文量。所使用的有作者发文量,团队总发文,人均发文,总被引频次,人均被引频次以及文章均被引频次五个产出指标,结果见表5所示。

表4 三项网络特性输入指标计算

表5 五项团队产出指标计算
(三)输入-输出指标关联测度
在研究输入指标与输出指标的关系之前,先对输入指标做一个相关性研究分析,然后对输出指标也做一个类似的相关性分析,最后对团队产出的影响因素进行挖掘。
(1)输入指标分析

表6 输入指标Pearson相关分析
注:**表示显著性水平达到0.01;*表示显著性水平达到 0.05。
网络特性的输入指标分析主要讨论指标之间的相关性,结果见表6。。密度和聚类系数:正相关系数达到0.910,随着密度的增加,网络的聚类系数成急剧上升趋势,密度侧重于网络中各个点之间关联的紧密程度,定义为图中实际拥有的连线数与最多可能拥有的线数之比,而聚类系数衡量的是网络的聚类性质。密度和距离:负相关系数达到0.684,随着团队中作者合作强度的增加,网络平均路径长度快速下降。聚类系数和距离:负相关系数0.449,随着网络聚类程度的提高,网络的平均路径长度下降,但下降速度不是很高。
(2)输出指标分析

表7 输出指标Pearson相关分析
注:**表示显著性水平达到0.01。
五个相关输出指标团队总发文量,人均发文量,团队总被引频次,人均被引频次,文章均被引频次中,团队总发文量,团队总被引频次以及文章均被引频次衡量了团队整体绩效,而人均发文量,人均被引频次则衡量了团队中个人的研究绩效,分析结果见表7。从研究来看,团队总发文和团队总被引频次,还有人均被引频次和文章均被引频次间存在较强的正相关关系。团队总发文量和文章均被频次间存在较强的负相关关系。另外通过研究我们也发现,其他指标之间的相关性并不十分明显,比如总发文量和人均被引频次间的正相关系数为0.022,几乎没有相关性,还有总被引频次和文章均被引频次间相关性也较弱,这也证明了引入人均被引频次和文章被引频次两个指标的重要性。
(3)网络特性与团队产出的影响因素挖掘

表8 网络特性与团队产出指标Pearson相关分析
注:**表示显著性水平达到0.01;*表示显著性水平达到 0.05。
本文将先从单个的网络特性指标来挖掘其与团队产出之间的关系,分析结果见表8。
①密度:一般而言,会认为团队成员联系越密切越有利于科研活动顺利开展并取得良好成绩,但从得到的分析数据来看,密度与团队总发文量,人均发文量之间的负相关程度较为显著,这说明科研团队的密度越大反而越不利于团队产出,密度与被引频次之间达0.652的负相关系数也说明密度的增加也不利于团队产出的质量。而密度与人均被引频次及文章均被引频次之间的关系并不明显,这说明密度很大程度上与均被引频次无关。
②聚类系数:网络按组群分布,组群内的节点之间有密集的联系,而组群与组群之间的联系则较少,这种特性类似于关系网络中“物以类聚、人以群分”的特性,这种属性称为网络的聚类特性,聚类系数可以用来定量化网络的这种属性。聚类系数与团队人均发文的负相关系数达到了0.718,随着团队聚类程度的提升,团队人均发文显著下降。与传统认知不同,聚类程度越明显,团队成员的合作程度越高,反而影响了团队的产出,只有对人均被引频次和文章均被引频次影响不大。
③距离:距离与总发文量、人均发文量和团队被引频次都是正相关关系,尤其与总发文量,正相关系数达到0.800。随着网络距离的增大,团队的产出反而迅速增长,而人均被引频次和文章均被引频次与距离几乎没有太大关系,这也与上文关于密度这一维度的分析相互佐证。
四、结论
本文将社会网络分析方法运用于科研团队的识别及绩效测度研究中,从网络的角度为科研团队的研究提供了新的思路,研究结果显示,与传统思路不同,在基因编辑领域内,团队联系的密切还有高频的合作反而使团队产出效率降低,即连通度高的网架型团队结构不再适应基因编辑这一新兴领域的需要,过于紧密的联系降低了团队成员的自由度,使人员流动程度减弱,新的知识较难传播。通过数据推测,桥梁型这一把众多小团体结合起来的合作模式使不同专长的团体结合在一起,有利于激发团队成员的创造性,团队产出绩效较高。
基因编辑领域起源于20世纪80年代,兴盛于最近十年,把这样的新热点领域作为样本得出的研究具有较高的参考价值,得出的不同于传统的高效合作模式也可被其他领域研究人员参考。
针对本研究对影响科研的绩效因素分析,笔者认为在现今高速发展的信息社会背景下,可以从以下三个方面提高科研团队绩效:
(1)大力推进外延式团队合作,促进不同学科方向的团队之间的合作,提高知识的交流程度,推广跨领域技术的运用。通过开展国内外学术会议的方式为研究者合作提供机会,设立专项基金对跨国家跨领域跨机构的团队进行奖励以及提供保障,也可建设多样化学术交流平台,以期激发研究者知识的融合和创新。
(2)在继续保障团队合作的基础上,适度给予团队成员一定的自由度,不能让团队成为一个封闭的组织,鼓励团队内部成员多对外进行交流,学习新的知识,引入更多技术和思路。
(3)建立更为完备的监督体系,如建立新的私密平台供研究者备份研究思路和进度,对研究成果的审核严格要求。以往团队之间交流较少,团队内部抱团较严重的成因包括部分学术道德低下的研究者会利用交流的平台偷取其他研究者思路和成果,建立更为完备的监督体系为团队成员毫无顾忌对外交流起到保驾护航的作用,也为建设优良研究风气起着推动作用。
在后续研究中,可深入研究团队合作动机,细化团队划分,使研究对象更加准确;也可更加细化绩效指标,如考虑计算发文量时引入赋予不同权值的方法;还可以引入更多绩效测度指标,如论文影响因子,更加全面、准确地测度团队的整体及平均绩效;此外,还能引入更多维度来对团队研究内容进行更深一步的探讨,丰富该新兴领域合作网络的研究,以期对该领域研究者和合作模式研究者提供更多参考。
参考文献:
[1]刘蓓,尉玮,王丽华.基因编辑新技术研究进展[J].亚热带农业研究,2013,9(4):262-269
[2]温芳芳.基于社会网络分析的中外图书馆学合作模式比较研究[J].国家图书馆学刊,2014(1):76-83.
[3]李纲,李春雅,李翔.基于社会网络分析的科研团队发现研究[J].图书情报工作,2014,58(7):63-70,82.
[4]李远明,谭世明.高校科研团队的识别与网络分析研究[J].科技进步与对策,2012,29(11):147-150.
[5] Otte E,Rousseau R.Social network analysis:a powerful strategy,also for the information sciences[J].Journal of Information Science,2016,28(6):441-453.
[6] Kretschmer H,Aguillo I F.Visibility of collaboration on the Web[J].Scientometrics,2004,61(3):405-426.
[7]吴菲菲,段国辉,黄鲁成.基于团队识别的3D打印技术发展前景分析[J].情报杂志,2013,32(8):62-67.
[8]邱均平,王菲菲.基于SNA的国内竞争情报领域作者合作关系研究[J].图书馆论坛 2010,30(6):34-40.
[9]汤建民.学术研究团队的可视化识别及评估方法研究:以科学学研究领域为例[J].情报学报,2010,29(2):323-326.
[10]刘蓓,袁毅,Boutin Eric. 社会网络分析法在论文合作网中的应用研究[J].情报学报,2008,27(3):409-411.
[11]刘军.整体网分析——UCINET软件使用指南[M].2版.上海:上海人民出版社,2014:126.
[12] 刘璇,朱庆华,段宇锋.社会网络分析法运用于科研团队发现和评价的实证研究[J].信息资源管理学报,2011(3):32-37.

