由上呼吸道感染诱发的急性点滴型银屑病皮损生态微生物研究
张宝兰,王晓萌,常贵珍,张理涛
银屑病是皮肤科常见的自身免疫性疾病,病因尚不明确,但国际公认感染为诱发点滴型银屑病的主要原因。既往研究证实,点滴型银屑病与上呼吸道感染链球菌相关[1-2]。银屑病患者体内存在对链球菌反应特异的T淋巴细胞[3],可促进细胞因子如白细胞介素(IL)-2、IL-6[4]和γ干扰素(interferon-γ,IFN-γ)等产生,进一步诱导角质形成细胞过度增生,但并不直接影响该细胞DNA的合成[5]。这些研究表明微生物菌群的变化可能与银屑病发病存在因果关系。皮肤在体表微生物与非特异性免疫的共同作用下建立了适应性免疫[6],除其本身的物理屏障外[7],还会分泌抗菌物质如β-防御素、抗菌肽、S100蛋白等[8]来自我保护。本研究采用16SrDNA二代测序技术明确由上呼吸道感染诱发的急性点滴型银屑病患者皮损处的微生物状态,并与正常健康体检者体表微生物比较,以明确2种人群的差异及其组间的物种差异。
1 对象及方法
1.1 研究对象 选取2017年5月—9月于天津市中医药研究院附属医院门诊确诊的急性点滴型银屑病患者(银屑病组)11例,男3例,女8例,平均年龄(30.18±8.03)岁,病程多为2~4周。同期健康体检者11例为对照组,男3例,女8例,平均年龄(30.27±8.45)岁。研究对象的一般情况统计见表1。纳入标准:2个月内未口服、注射或静脉滴注任何抗生素及免疫抑制剂等药物,1周内未使用外用型抗生素制剂,至少24 h内未洗澡,无其他任何疾病、皮肤创伤或其他相关感染。本研究经本院伦理委员会批准且患者均签署知情同意书。
1.2 样本收集 使用无菌棉球经无菌水处理后在靶点部位加压擦拭取材,靶部位包括四肢伸侧、腰骶部、腹部,顺时针方向擦拭,每个部位至少30 s,收集完毕立即置于-80℃冰箱备用。
1.3 主要试剂及器材 DNA抽提试剂盒购自赛默飞,Phusion®的高保真酶购自新英格兰生物公司,胶回收试剂盒,TruSeq®DNA PCR-Free样本制备建库试剂盒购自凯杰公司,生物样本均质器、超声波DNA破碎仪、Illumina测序平台来自天津诺禾致源生物信息科技有限公司。
1.4 方法
1.4.1 样本DNA抽提与质检 样本经液氮冷冻后进行研磨,使用DNA抽提试剂盒按说明书抽提DNA。取1 g琼脂糖加入100 mL 1×TAE[TAE是由三羟甲基氨基甲烷(Tris base)、乙酸(acetic acid)和乙二胺四乙酸(EDTA)组成的缓冲液]电泳缓冲液配置琼脂糖凝胶,加入EB染色,取5 μL的DNA抽提液及1 μL的Loading Buffer于加样孔,电压100 V、40 min后在紫外灯下观察。
1.4.2 PCR扩增与文库构建 取抽提液1 μL,对16SrDNA(16SrRNA为核糖体RNA的一个亚基,16SrDNA就是编码该亚基的基因,这里单指这个基因)的V4区测序,特异引物:515 F 5′-GTGCCAGCMGCCGCGGTAA-3′;806 R 5′-GGACTACHVGGGTWTCTAAT-3′。5 μL 10×Buffer、4 μL 2.5 mmol/L dNTP、1.6 μL Taq 酶、4 μL Primers、37 μL ddH2O,配成50 μL的PCR反应体系。样本DNA抽提液中最低体积为1 μL。反应条件:94℃预变性2 min;94℃变性40 s,55℃退火40 s,72℃延伸130 s,循环35次;72℃延伸10 min。将PCR产物利用磁珠纯化两次后,产物置于2 mL的离心管中。使用Qubit 2.0仪器检测纯化后的PCR质量,质检通过后委托天津诺禾致源生物信息科技有限公司测序。
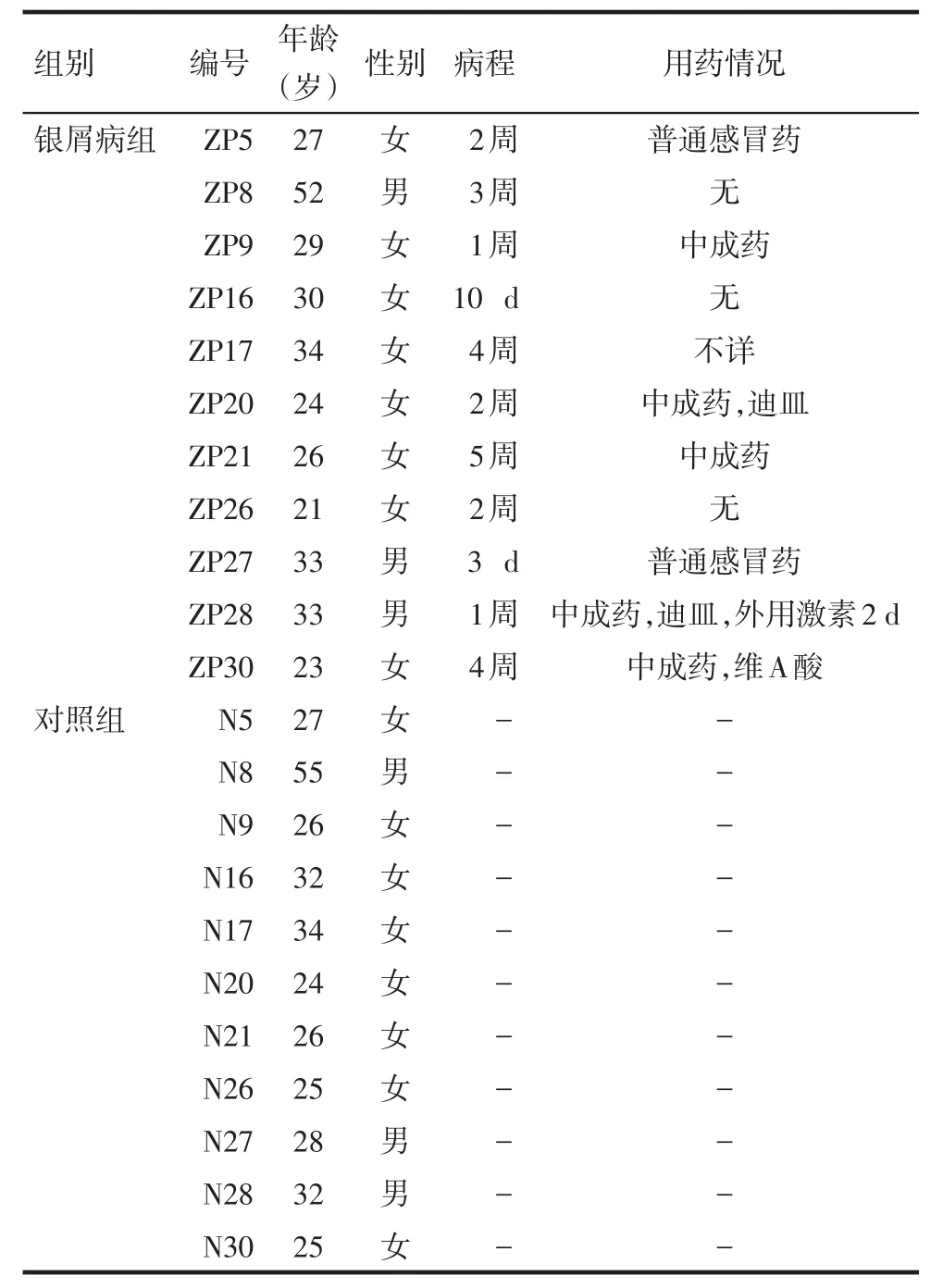
Tab.1 Comparison of the general situation of two groups of patients表1 银屑病患者与健康者的一般情况比较
1.5 统计学方法 (1)物种分布情况。利用Uparse软件(v7.0.1001)对样本有效序列进行聚类,并使Mothur方法与SILVA的SSUrRNA数据库[9-10]进行物种注释分析,了解物种分布情况。(2)样本复杂度分析。QIIME(Version 1.9.1)计算α多样性指数[Observed_species、香农-威纳指数(Shannonwiener diversity index)、辛普森多样指数(Simpson diversity index)、chao1丰富度估计量(Chao1 richness estimator)、基于丰度的覆盖估计(Abundance-based Coverage Estimator)],使用SPSS 24.0进行统计分析2组样本的物种丰富性与分布情况。(3)微生物结构β多样性分析。使用R软件(Version 2.15.3)绘制稀释性曲线、物种积累箱形图,主坐标分析(principal co-ordinates analysis,PCoA)图,观察2组间在物种多样性与微生物群落结构即微生物丰度是否存在差异。构建 UPGMA(unweighted pair-group method with arithmetic mean)样品聚类树。PCoA与UPGMA直观地描述了样本间的差异,本研究目的为银屑病组与对照组间的差异物种,因此选择非加权距离(unweighted unifrac)进行分析。(4)组间差异物种。用LEfSe软件进行分析寻找组间差异显著的物种[11],为寻找相对丰度较大,起主要作用的物种,把LED值设为4。寻找2组间差异显著的物种,主要观察属水平的差异,利用相对丰度(某一物种丰度与全部微生物物种丰度的比值)进行比较。P<0.05为差异有统计学意义。
2 结果
2.1 物种分布情况 按97%的相似性进行聚类,共得到3 427条OTU序列,对OTU序列进行生物注释,门、纲、目、科、属、种水平分别发现40、88、157、306、784、566个种类。按丰度排名前10位的物种绘制累加图,发现各样本组成相似,主要为变形菌门(proteobacteria)、放线菌门(actinobacteria)、厚壁菌门(firmicutes),见图1。银屑病组与对照组中上述3种菌门的相对丰度(某一菌门丰度/所有菌门丰度)分别为42.38%和34.34%、23.95%和31.44%、25.51%和27.35%。
全部样本中编号为ZP16的样本蓝藻菌门(cynaobacteria)未分类的叶绿体属相对丰度水平较其他银屑病患者高,查看一般情况记录发现提供此样本的银屑病患者为孕妇。为保证结果客观真实,将处于妊娠期的患者样本ZP16(很明显较其他样本多出许多橙色的条带,为蓝藻菌门)以及离散度较大的样本N5(在β多样性分析中,可看到该样本距离较其他样本十分远,且在距离矩阵的数值较其他离散)剔除,银屑病组与对照组各剩10例样本,主要物种未发生明显变化,见图2。
2.2 样本复杂度分析 根据此20例样本绘制的物种积累箱型图显示,箱形图最后趋于平缓,表明样本量足够,测序数据合理(图3)。银屑病组与对照组的稀释性曲线显示,2组在物种多样性上差异无统计学意义(图4)。同时2组α多样性指数差异均无统计学意义,见表2。
2.3 微生物结构β多样性分析 为了分析皮肤表面菌群的完整性对样本的分类影响,使用PCoA非加权算法计算样本分群情况(图5)。如果不考虑预试验与正式试验的差别,银屑病组与对照组之间存在明显分群现象,差异较为明显,患者组样本聚集在一起(红色点),对照组样本聚集在一起(蓝色点),整体事件解释率为29.1%(一般整体解释率大于20%,即可说明样本间关系)。这说明在皮肤菌群完整性上,银屑病组与对照组在皮肤菌相存在不同之处。对整体样本构建进化树(图6),可见除个别样本可能因为个体特异性聚类分散外,银屑病组与对照组大部分是分别聚集在一起,样本聚类情况理想,说明银屑病皮损与正常皮肤表面微生物存在一定程度的差异。结合样本一般情况,与聚类树结果对比发现,ZP8与其他银屑病患者距离较远,说明微生物分类与年龄有关,ZP26、ZP27与其他患者样本分开,说明在男女样本中年龄相对较小,病程短的患者微生物分布接近对照组。
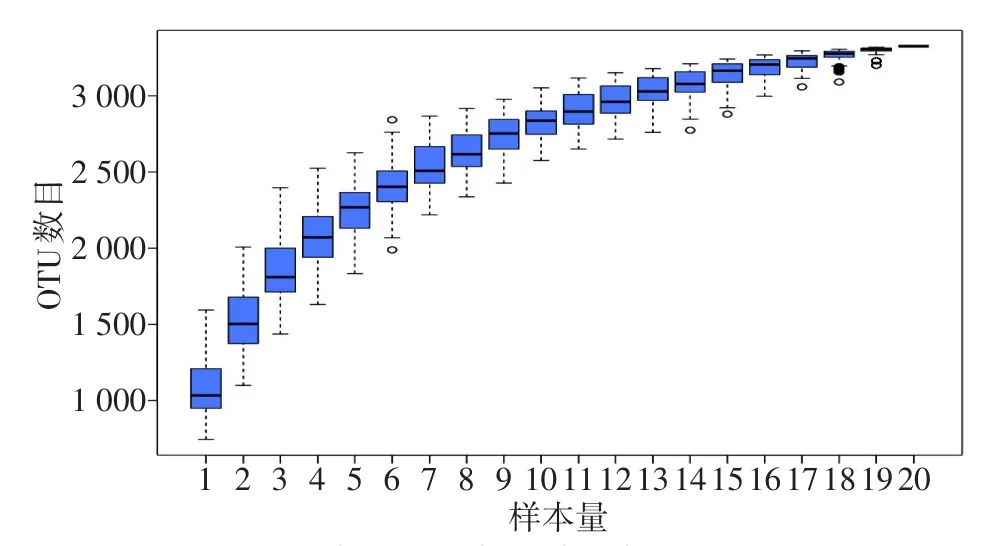
Fig.3 The accumulation boxplot of species图3 物种积累箱形图

Fig.4 The sample rarefaction curves in the two groups图4 2组样本稀释性曲线
Tab.2 Comparison of alpha diversity index between the two groups表2 2组α多样性指数的比较 (n=10,±s)

Tab.2 Comparison of alpha diversity index between the two groups表2 2组α多样性指数的比较 (n=10,±s)
*P<0.05;Shannon为香农-威纳指数,simpson为辛普森多样指数,chao1为chao1丰富度估计量,ACE为基于丰度的覆盖估计
组别银屑病组对照组t Observed_species 1 088.10±233.57 1 034.00±161.98 0.602 Shannon 5.81±0.89 5.56±1.18 0.547 simpson 0.92±0.05 0.89±0.13 0.725 chao1 1 340.33±240.27 1 295.93±295.19 0.369 ACE 1 373.83±239.27 1 333.86±285.88 0.339
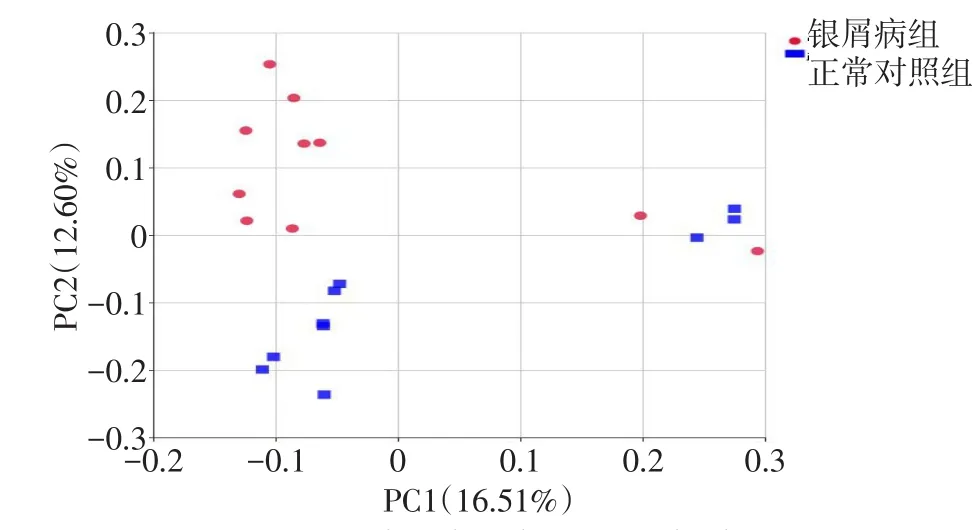
Fig.5 PCoA analysis based on Unweighted Unifrac distance for each sample图5 各样本基于Unweighted Unifrac距离的PCoA分析
2.4 组间差异物种 银屑病组与对照组在属水平主要物种的相对丰度分别为葡萄球菌属13.02%、15.10%,棒状杆菌属9.66%、15.97%,栖水菌属8.65%、4.89%,不动杆菌属9.82%、2.89%,未知的叶绿体0.27%、2.04%,链球菌属3.38%、2.70%,副球菌属2.34%、2.53%,丙酸杆菌属3.16%、5.81%。LEfSe分析(LED阈值为4)在银屑病组与对照组间筛选出具有显著性差异的物种为不动杆菌属(图7)。2组不动杆菌属和未分类的叶绿体这2种物种组间差异均有统计学意义,见表3。

Fig.7 The LEfSe analysis cladogram and the LDA value distribution histogram图7 LEfSe分析进化分支图和LDA值分布柱状图

Tab.3 Comparison of significant different species in acinetobacter and unidentified chloroplast between two groups表3 银屑病组与正常对照组具有显著差异物种不动杆菌属与未分类叶绿体的相对丰度比较 [n=10,M(P25,P75)]
3 讨论
目前,有关寻常型银屑病皮损微生物的研究尚无定论。有研究显示,银屑病与正常健康对照者存在链球菌、丙酸杆菌的显著差异[12]。另有研究显示,棒状杆菌、丙酸杆菌、葡萄球菌、链球菌在银屑病组与对照组间存在相对丰度的差异,但仅限于描述性分析[13-14]。以上研究的银屑病对象可能因人种、病程不同或在前期治疗中使用抗生素制剂而使表皮菌群重新分布有关,但都表明银屑病发病的确与皮损微生物的变化相关。
本实验运用16SrDNA测序技术对样本分析,所得数据进行α多样性分析发现银屑病组与对照组差异无统计学意义。β多样性分析中,对样本聚类分析示,PCoA与UPGMA聚类树结果多数样本都表现出银屑病组与对照组在微生物群落结构上有所不同。LEfSe分析与秩和检验结果发现不动杆菌在2组中有差异。不动杆菌属革兰阴性杆菌,黏附力极强,对生长环境要求较低,广泛分布于自然界、医院环境及健康人的皮肤表面[15]。其黏附在皮肤上成为潜在的致病菌原因之一是可以产生神经氨酸酶,后者在后续细菌黏附宿主的作用中发挥作用,可形成细菌生物膜,促进细菌微生物的定植[16]。有研究证实,皮肤表面微生物菌种的变化无论个体差异多大,均受到不动杆菌等相对丰度变化的影响,如金黄色葡萄球菌(SA)丰度随不动杆菌丰度升高而升高,丙酸杆菌则与之为负相关[17]。本研究检测到银屑病组不动杆菌相对丰度升高即符合这一现象。笔者推测在上呼吸道感染时,机体免疫力降低可导致不动杆菌感染,或因链球菌的作用致使体表微生物尤其不动杆菌相对丰度发生了改变。由于本研究病例均为初发或复发的患者,病程较短,体表生态微生物一般正在发展到另一个相对稳定的状态中。研究显示,由于微生物间的相互作用持久,而且这种微生物间相互作用可正可负,不一定处在同一种稳定状态[18];这种稳态的改变就像陆地森林生态系统一样,由一个物种的变化即可导致其他物种的生长或消亡[19],这种变化过程需要一定时间,且相互作用会持续存在。因此,短期病程内物种菌群还未凸显差异,皮损区与健康皮肤微生物在α多样性分析中没有太大差异,但β多样性分析图显示2组间微生物菌相有所差异。
具有显著差异的不动杆菌在微生物菌群相对稳态变化时可起到枢纽作用。不动杆菌介导致病菌如SA形成新的生物膜,而SA的毒力作用使IL-17等炎症因子释放增加[20];IL-17在T细胞炎症轴线及角质形成、细胞过度增生的过程中起到重要作用,可导致银屑病加重[21]。另外,不动杆菌本身具有致病性,可在上呼吸道感染期接收信号,作用皮肤,激活T淋巴细胞,产生细胞因子,并且刺激皮肤产生大量抗菌肽、防御素、S100蛋白等杀菌物质,促使银屑病的发生,此点也证实了微生物稳态对宿主的健康十分重要。
前期物种数据比对结果发现,银屑病组中有1例蓝藻菌门,叶绿体丰度较高,此患者在确诊银屑病后检查发现已妊娠,因此急性点滴型银屑病妊娠患者与其他患者皮损微生物物种存在差异还是偶发情况有待考证。然而,剔除已妊娠患者的样本后进行数据分析发现,该物种在健康对照组中较银屑病组相对丰度较高,蓝藻释放毒素对皮肤具有刺激性,却在健康对照组中有较高的丰度,该结果具有什么意义,目前还不明了,需要进一步研究论证。
本次实验突出的最主要问题是测序。笔者将预试验样本的测序结果与后期样本的测序结果进行统一分析,发现因测序批次的不同,样本聚类可能会有所偏差,如PCoA分析图,预试验与正式试验样本分离,但仅正式试验部分表示2组间是存在差异的。当下,人们关注自身卫生的同时存在“过度清洁”,滥用抗生素的现象,有可能导致自身免疫性疾病的发生。若发生此类与自身免疫性相关炎症性疾病,可考虑应用益生菌制剂来稳定微生物稳态,阻断由微生物菌群变化导致皮肤免疫反应的发生,以控制炎症的发展。
(图1、2、6见插页)

Fig.1 The schematic diagram of mechanical loading for a mouse图1 小鼠机械加载图示
Fig.2 The schematic diagram of whole body composition analyzer for a mouse图2 小鼠体成分分析图示
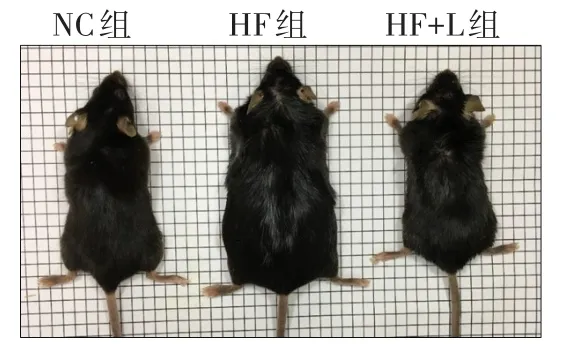
Fig.3 The body sizes of mice before being sacrificed图3 小鼠处死前体型
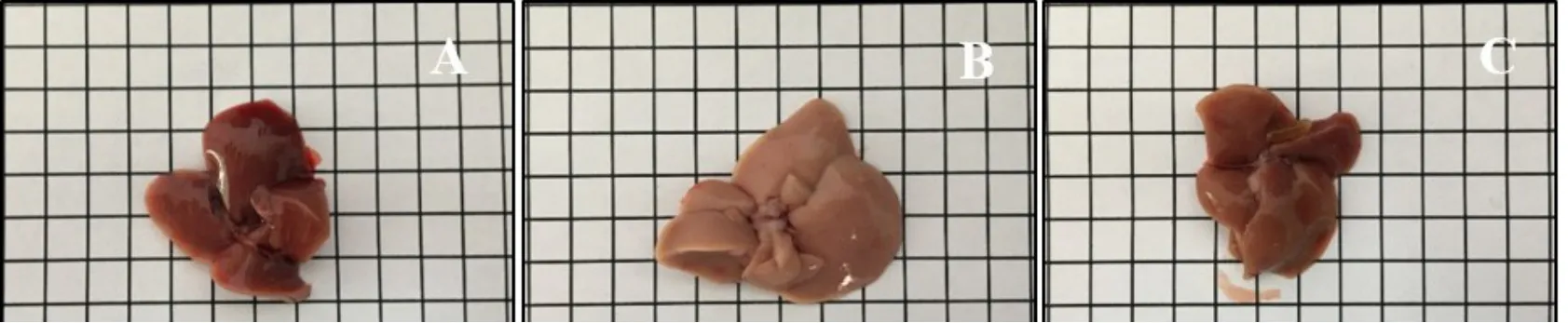
Fig.4 General observation of liver tissues in three groups of mice图4 小鼠肝脏组织肉眼观察
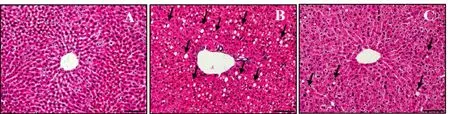
Fig.5 The changes of hepatic adipocytes under light microscopy in three groups(HE staining,×200)图5 各组小鼠肝脏脂肪细胞光镜下观察(HE染色,×200)
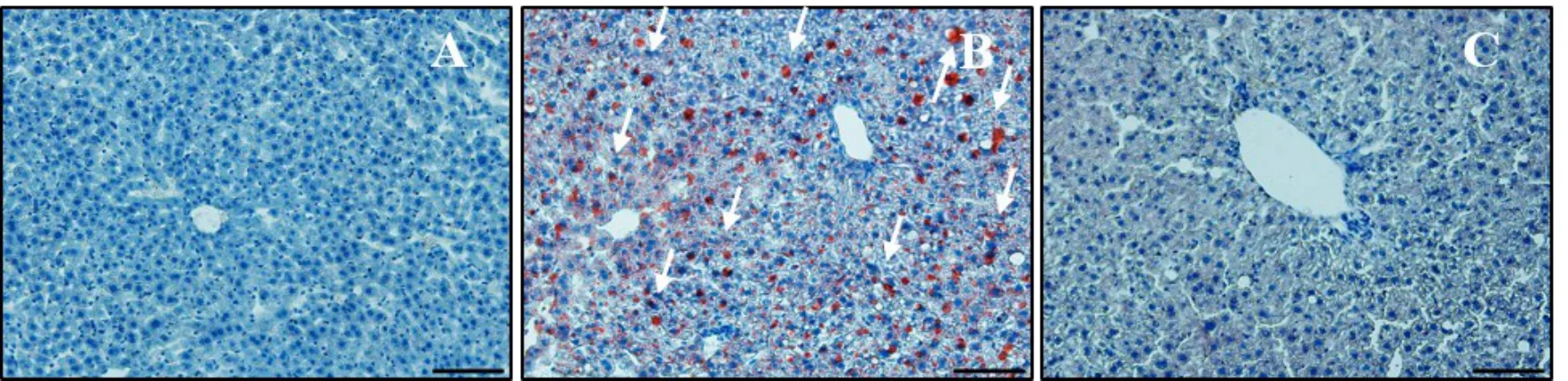
Fig.6 The changes of liver tissues under light microscopy in three groups(Oil red O staining,×200)图6 各组小鼠肝脏组织光镜下观察(油红O染色,×200)
[1]Telfer NR,Chalmers RJ,Whale K,et al.The role of streptococcal infection in the initiation of guttate psoriasis[J].Arch Dermatol,1992,128(1):39-42.
[2]Talanin NY.American academy of dermatology 1998 awards for young investigators in dermatology detection of streptococcal antigens in psoriasis[J].J Am Acad Dermatol,1998,39(2 Pt 1):270-271.
[3]张理涛,陈学荣,殷金珠.β溶血型链球菌诱发点滴型银屑病发病机制的研究[J].中华皮肤科杂志,1999,32(2):29-30.Zhang LT,Chen XR,Yin JZ.Study on pathogenesis of guttate type psoriasis induced by group a β-haemolyticstreptococcal infection[J].Chin J Dermatol,1999,32(2):29-30.
[4]张理涛,高兴华,陈洪铎,等.链球菌抗原刺激的银屑病患者外周血单一核细胞培养上清液中TNF-α和IL-6的检测[J].中华皮肤科杂志,2000,33(5):58.Zhang LT,Gao XH,Chen HD,et al.Detection of TNF-α and IL-6 in the culture supernatant of peripheral blood mononuclear cells from patients with psoriasis stimulated by streptococcus antigen[J].Chin J Dermatol,2000,33(5):58.
[5]张理涛,阎学贞,夏青,等.链球菌抗原和几种细胞因子对人类角质形成细胞株DNA合成的影响[J].中国皮肤性病学杂志,2000,14(6):383-384.Zhang LT,Yan XZ,Xia Q,et al.Effect of streptococcus antigen and several cytokines on the compose of human keratinocyte strain DNA[J].Chin J Derm Venereol,2000,14(6):383-384.
[6]Yang JJ,Chang TW,Jiang Y,et al.Commensal staphylococcus aureus provokes immunity to protect against skin infection of methicillin-resistant staphylococcus aureus[J].Int J Mol Sci,2018,19(5).pii:E1290.doi:10.3390/ijms19051290.[Epub ahead of pirint].
[7]Yuki T,Yoshida H,Akazawa Y,et al.Activation of TLR2 enhances tight junction barrier in epidermal keratinocytes[J].J Immunol,2011,187(6):3230-3237.doi:10.4049/jimmunol.1100058.
[8]Gallo RL,Nakatsuji T.Microbial symbiosis with the innate immune defense system of the skin[J].J Invest Dermatol,2011,131(10):1974-1980.doi:10.1038/jid.2011.182.
[9]Wang,Q,Garrity GM,Tiedje JM,et al.Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Appl Environ Microbiol,2007,73(16):5261-5267.doi:10.1128/AEM.00062-07.
[10]Quast C,Pruesse E,Yilmaz P,et al.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucl Acids Res,2013,41(Database issue):D590-D596.doi:10.1093/nar/gks1219.
[11]Segata N,Izard J,Waldron L,et al.Metagenomic biomarker discovery and explanation[J].Genome Biol,2011,12(6):R60.doi:10.1186/gb-2011-12-6-r60.
[12]Gao Z,Tseng CH,Strober BE,et al.Substantial alterations of the cutaneous bacterial biota in psoriatic lesions[J].PLoS One,2008,3(7):e2719.doi:10.1371/journal.pone.0002719.
[13]Alekseyenko AV,Perez-Perez GI,De Souza A,et al.Community differentiation of the cutaneous microbiota in psoriasis[J].Microbiome,2013,1(1):31.doi:10.1186/2049-2618-1-31.
[14]Fahlen A,Engstrand L,Baker BS,et al.Comparison of bacterial microbiota in skin biopsies from normal and psoriatic skin[J].Arch Dermatol Res,2012,304(1):15-22.doi:10.1007/s00403-011-1189-x.
[15]Ramalingam K,Lee VA.Antibiofilm activity of an EDTA-containing nanoemulsion on multidrug-resistant Acinetobacter baumannii[J].Artif Cells Nanomed B,2018,May 2:1-7.doi:10.1080/21691401.2018.1468771.[Epub ahead of print]
[16]Prakasam G,RohitA,PadmasiniE,etal.Production of neuraminidase in relation with biofilm formation among clinical and healthy skin isolates of Acinetobacter species[J].Indian J Med Microbiol,2014,32(4):459-460.doi:10.4103/0255-0857.142235.
[17]Leung MHY,Tong X,Wilkins D,et al.Individual and household attributes influence the dynamics of the personal skin microbiota and its association network[J].Microbiome,2018,6(1):26.doi:10.1186/s40168-018-0412-9.
[18]Blaser MJ,Kirschner D.The equilibria that allow bacterial persistence in human hosts[J].Nature,2007,449(7164):843-849.doi:10.1038/nature06198.
[19]Jackson CR.Changes in community properties during microbial succession[J].Oikos,2003,101(2):444-448.
[20]Howell MD,Boguniewicz M,Pastore S,et al.Mechanism of HBD-3 deficiency in atopic dermatitis[J].Clin Immunol,2006,121(3):332-338.doi:10.1016/j.clim.2006.08.008.
[21]Hawkes JE,Chan TC,Krueger JG.Psoriasis pathogenesis and the development of novel targeted immune therapies[J].J Allergy Clin Immunol,2017,140(3):645-653.doi:10.1016/j.jaci.2017.07.004.

