流式荧光点阵技术在乙型肝炎病毒基因分型中的应用
朱 俊,朱宇清,朱岭峰,蒋玲丽,徐 翀
(上海市临床检验中心,上海 200126)
乙型肝炎病毒(hepatitis B virus, HBV)感染是导致肝硬化和肝癌的首要原因,全球大约有2.4亿HBV感染者[1],我国的乙型肝炎患者达1.2亿[2]。由于HBV双链DNA的复制过程中RNA等中间产物和逆转录酶的作用,以及DNA聚合酶缺乏校正功能而使HBV基因组具有较大的变异性[3]。迄今为止,根据HBV全基因组序列分析将HBV分为10种(A~J)基因型和多种亚型,不同基因型之间的核酸序列异质性≥8%,不同亚型之间序列差异为4%~7%[2,4-6]。有研究结果表明,HBV基因型与乙型肝炎临床表现的异质性、对抗病毒治疗应答及预后有关[7-8]。目前,国内外对于HBV基因分型多采用测序方法,但测序方法存在繁琐耗时、成本较高的不足。我们研发了一种基于流式荧光点阵技术(又称液态芯片法)的HBV基因型B型和C型的检测方法,具有特异性强、操作简便灵活并易自动化的特点。
1 材料和方法
1.1 研究对象
收集2015年4月上海中医药大学附属曙光医院检验科采用测序法进行HBV基因分型的血清样本67份,其中B型24份、C型43份。
1.2 仪器与试剂
1.2.1 仪器 (1)TC-96/G/H(b)A Life Pro梯度基因扩增仪(杭州博日科技有限公司)。(2)EPS100核酸电泳仪(上海天能科技有限公司)。(3)FR-200A全自动紫外与可见分析装置(上海复日科技有限公司)。(4)Luminex 200多功能流式点阵(液相芯片)仪(美国Luminex公司)。
1.2.2 试剂 (1)DNA提取试剂盒(德国Qiagen公司)。(2)BestarTaq DNA Polymerase DNA聚合酶(德国DBI Bioscience公司)。(3)引物和探针由英潍捷基(上海)贸易有限公司合成。测序引物 HBV B型扩增引物:HBV-BF 5'-GTCCTCTAATTCCAGGAT-3',其中5'端第5个T用生物素标记,HBV-BR 5'-CGTAGGTTTTGTACAGCAACGT-3'。HBV C型扩增引物:HBV-CF 5'-ACAACATCTTGAGTCCCTTCTT-3',其中5'端第5个T用生物素标记,HBV-CR 5'-CCATGAAGTTAAGGGAGTTGC-3'。探针(5'起第9个碱基T用氨基修饰):HBV B型探针ACTCCTGCTCAAGGAACCTCTA;HBV C型探针TCTTTGGGTATACATTTAAACCCTAA。(4)链霉亲和素-藻红蛋白(美国Invitrogen公司)。(5)荧光编号的(表面羧基修饰)聚苯乙烯微球(美国Luminex公司)。
1.3 检测技术原理
1.3.1 核酸探针与微球偶联 从探针5' 起第9个碱基T氨基化,聚苯乙烯微球表面用羧基修饰。探针通过修饰的氨基偶联至修饰羧基的微球表面。
1.3.2 多重聚合酶链反应(polymerase chain reaction,PCR)产物与探针杂交 PCR引物5'端第5个T用生物素标记,PCR扩增产物如与微球表面探针互补,则生物素连接至微球表面,加入链霉亲和素-藻红蛋白后即可通过链霉亲和素与生物素的结合而使藻红蛋白荧光标记至微球。微球表面的藻红蛋白荧光在激光激发下发光。见图1。
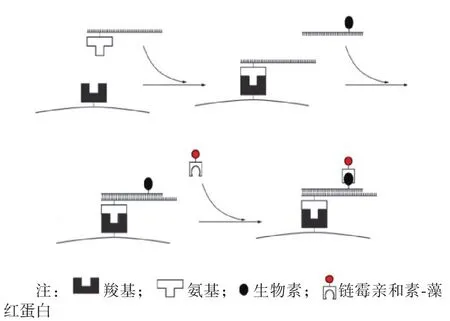
图1 荧光点阵技术原理
1.4 方法
1.4.1 多重PCR扩增 (1)DNA提取:根据DNA提取试剂盒的说明书进行DNA提取。(2)反应体系组成:模板DNA2 μL,上、下游引物2 μL,PCR预混液(含热启动TaqDNA酶、MgCl2和 dNTP Mix)10 μL,双蒸水6 μL,至总体积20 μL。(3)反应条件:95 ℃ 15 min;94 ℃ 30 s,55 ℃ 90 s,72 ℃ 90 s,35个循环;72 ℃ 10 min。反应产物于4 ℃保存。
1.4.2 杂交检测 (1)探针与微球偶联:用蒸馏水将2种寡核苷酸探针溶解,调节浓度为1 mM/L。分别从2个荧光编码的微球储存悬液中取2.5×106个微球到2个离心管中。以蒸馏水将1 mM/L浓度探针进一步稀释10倍,各取2 mL至混匀的相应的微球里混合并避光孵育30 min,探针通过修饰的氨基偶联至修饰羧基的微球表面。偶联后的微球经洗涤后用pH值8.0的三羟甲基氨基甲烷-乙二胺四乙酸溶液将偶联微球重悬浮。将分别偶联B型和C型探针的2种微球等比例混合,各种微球的终浓度为1 500个/μL,2~8 ℃避光保存。(2)PCR产物与探针杂交:用杂交液将偶联微球浓度稀释至150个/μL作为微球工作液。取33 μL微球工作液,与多重PCR扩增产物和TE溶液(共17 μL)混合,在95~100 ℃孵育5 min以去除寡核苷酸的二级结构。在杂交温度48 ℃下孵育反应60 min,加入10 μg/μL的链霉亲和素-藻红蛋白溶液75 μL后继续孵育15 min完成杂交和荧光标记。(3)上机检测:在Luminex 200液相芯片仪上,保持杂交温度,对各反应孔进行检测分析,测定平均荧光强度(mean fluorescence intensity, MFI)值。
1.4.3 结果分析 检测时以不含有HBV DNA序列的质粒作为阴性对照,以MFI值>150作为阳性的判断标准。阳性阈值是以10个阴性标本MFI的平均值加上2s确定。
2 结果
2.1 特异性试验
将多重PCR扩增产物与结合B型和C型探针的2种微球在同一反应体系中杂交,Luminex 200液态芯片仪检测结果显示,人DNA、金黄色葡萄球菌(Staphylococcus aureus, Sau)DNA和人乳头瘤病毒(human papillomavirus, HPV)DNA样本的MFI均<150,显示为阴性结果。18、19和20号样本与C型探针杂交呈阳性反应,与B型探针不发生反应;33和34号样本与B型探针杂交呈阳性反应,与C型探针不发生反应。这2个组杂交结果均与测序结果相符。见表1。
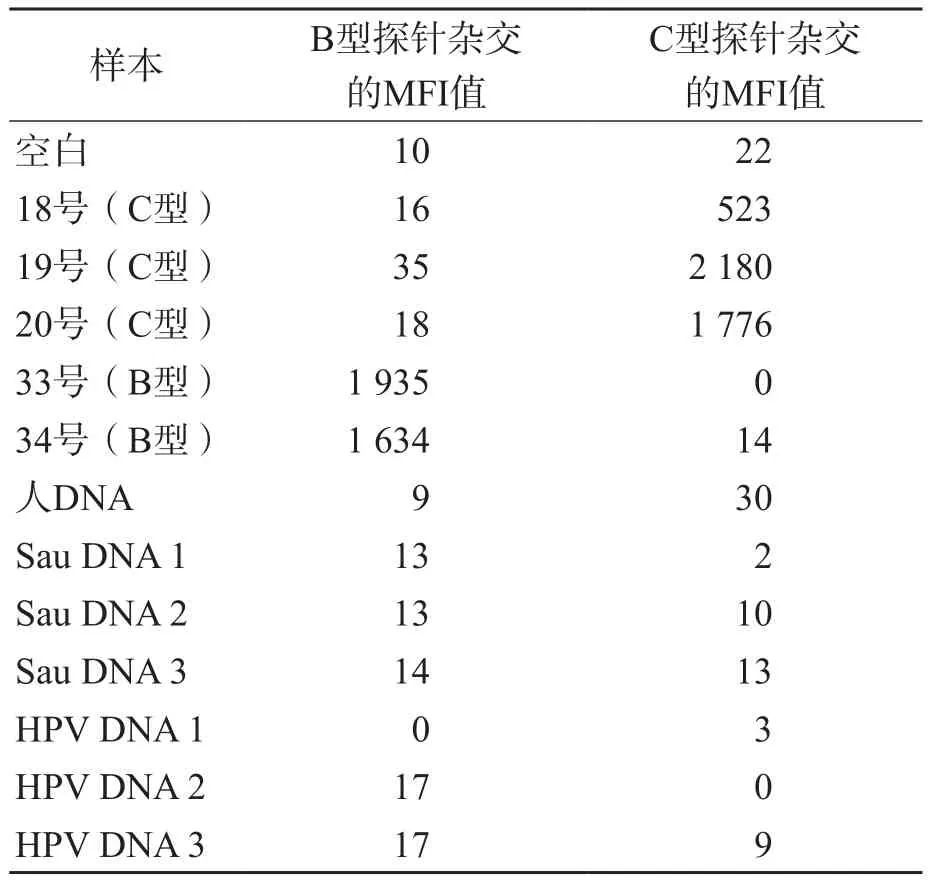
表1 B型和C型2种探针的特异性试验
2.2 液态芯片法与测序法结果比较
经测序法基因分型确定为B型的24份样本中,23份经液态芯片法检测获得与测序同样的结果,但有1份样本同时显示与B型和C型探针均有阳性杂交信号;经测序法确定为C型的43份样本中,有40份样本在液态芯片法的检测中获得与测序同样结果,但有3份样本同时与B型和C型探针有阳性杂交信号。见表2。
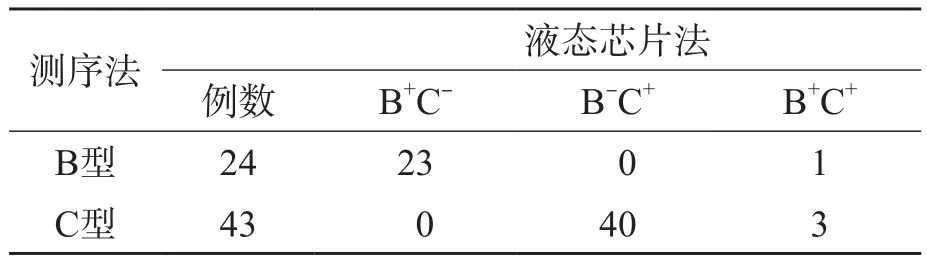
表2 液态芯片法与测序法结果的比较
3 讨论
HBV属于嗜肝DNA病毒科家族,呈全球性分布,是导致人类严重慢性肝病(如慢性肝炎、肝硬化和肝细胞癌)的最主要原因[9]。自1965年BLUMBERG等[10]发现HBV后,HBV的第1种疫苗于5年后问世[11]。从1982年起,由于 >10亿支HBV疫苗的使用,使全球感染HBV而导致的死亡率显著下降[12]。但尽管如此,数亿患者已经成为HBV慢性感染的事实使得根除乙型肝炎的任务异常艰巨。诺贝尔奖获得者Blumberg教授认为可以通过3种重要的方式来根除HBV[11]:有效的全球疫苗接种、准确的诊断分析技术和对于HBV携带者的有效治疗。而缺乏对HBV遗传特性的了解,则难以达到此目的。
自从1988年OKAMOTO等[13]首次建立了HBV基因型的分型标准并将HBV分为A、B、C和D 4种基因型以后,经过NORDER等[14]、STUYVER等[15]、WU等[16]、YU等[17]和TATEMATSU等[18]的补充和发展,根据HBV全基因系列不同基因型间核酸序列差异≥8%、亚型之间差异为4%~7%的原则,至今可将HBV分为A~J共10种基因型,并可进一步分为诸多亚型(以数字表示)。HBV基因分型与之前报道的根据HBV表面抗原2对互相排斥的亚型决定簇(d/y和w/r)而建立的血清分型部分相关[6],2种分型方式并不能建立完全的对应关系,比如血清型adw并不只存在于1种基因型中[19]。目前认为以基因型来反映HBV的变异更为准确。HBV基因型的分布具有显著的民族和地理区域差异[2],并且与乙型肝炎的临床表现、病程、抗病毒治疗应答以及预后有关[7-8]。因此,准确的HBV基因分型对于乙型肝炎的预防、诊断、治疗以及预后判断均有重要意义。
迄今为止,通过与美国国家生物技术信息中心(the National Center for Biotechnology Information, NCBI)网站上的BLAST软件的比对,对HBV进行全基因组序列分析是区分HBV基因型和亚型的“金标准”。其他用于HBV基因分型的分子生物学方法还包括:PCR-限制性片段长度法、型特异性引物PCR、寡核苷酸基因芯片法、限制性片段质谱多态性技术、质谱分析、实时荧光PCR、线形探针反向杂交技术以及反向点杂交等方法。但这些方法的缺点是不能准确分型、容易受基因组碱基突变而影响引物和DNA或探针和DNA的配对而导致分型结果难以归类,检测成本高、需要特殊仪器等[20]。测序法因其可以直接分析HBV全基因组的序列,所以分型结果最为可靠。因此,虽然有多种HBV分型方法,基因测序仍然是HBV分型的“金标准”。但是,测序方法检测成本较高、技术相对复杂且需要测序仪器,故不适合临床广泛应用[20]。
本研究以亚洲主要流行的B型和C型HBV为研究目标,研发了一种利用多重PCR技术结合液态芯片技术对HBV进行基因分型的方法,目前市场上尚无相同产品。引物和探针的设计参考了世界卫生组织等机构推荐的病毒株序列和引物序列,与NCBI的HBV核酸数据库进行序列比对,以确保所设计引物和探针序列的特异性、稳定性和代表性,并同时兼顾我国地区高发毒株的情况。本研究检测了经测序确认的B型和C型HBV患者样本各20份,结果显示自建的液态芯片法与测序法结果的一致性高达 94%, 达到了预期的目的。值得指出的是,在24份测序结果为B型患者中有1份样本检测出B型和C型双阳性探针信号;43份样本测序法结果为C型的患者中有3份样本检测出B和C型双阳性探针信号,我们认为这4份样本的患者可能是HBV B型和C型混合感染。KAO等[21]于2002年即报道了在日本献血员中发现有HBV的B型和C型混合型感染的存在。对于HBV混合型感染的鉴定,采用型特异性引物扩增的方法优于测序方法,因为如果只对PCR扩增产物进行直接测序,通常只能检测到优势毒株的基因型,载量低的毒株序列大多难以测出,除非我们对PCR产物先进行克隆,再选取多个克隆进行测序,这样才有可能检测到弱势基因型的毒株,但采取这样的技术需要更长的检测周期,在临床常规检测中并不可行。本研究设计的液态芯片法初步显示能够更敏感和特异地检测出混合型感染,但还需对这4份样本作进一步验证,以确定本技术在鉴定HBV混合感染中的优势。
本研究建立的HBV液态芯片法有广泛的应用前景。实验所用的微球如用不同强度的二维荧光编码,可以做到在同一个体系中同时检测到100多个不同荧光编码的微球群体,通过对每个微球群体标记不同的核酸探针,就可以在同一个体系中同时检测100多个分析物。因此,本方法开发成功的意义在于不仅能有效地检测HBV和鉴定其亚型,为其他各种病原体的确定及其亚型鉴定、病原体多种耐药基因分析等提供多重、简便、快速检测的可能,甚至还能为一些复杂性疾病、遗传性疾病多基因多位点实现多重平行检测提供可行的方法。
[1]OTT J J, STEVENS G A, GROEGER J, et al.Global epidemiology of hepatitis B virus infection:new estimates of age-specific HBsAg seroprevalence and endemicity[J]. Vaccine, 2012, 30(12):2212-2219.
[2]LI H M, WANG J Q, WANG R, et al. Hepatitis B virus genotypes and genome characteristics in China[J]. World J Gastroenterol, 2015, 21(21): 6684-6697.
[3]ZEHENDER G, EBRANATI E, GABANELLI E, et al. Enigmatic origin of hepatitis B virus: an ancient travelling companion or a recent encounter?[J]. World J Gastroenterol, 2014, 20(24):7622-7634.
[4]ARAUZ-RUIZ P, NORDER H, ROBERTSON B H, et al. Genotype H: a new Amerindian genotype of hepatitis B virus revealed in Central America[J]. J Gen Virol, 2002, 83(Pt 8): 2059-2073.
[5]MAGNIUS L O, NORDER H. Subtypes,genotypes and molecular epidemiology of the hepatitis B virus as reflected by sequence variability of the S-gene[J]. Intervirology, 1995, 38(1-2): 24-34.
[6]NORDER H, COUROUCÉ A M, COURSAGET P, et al. Genetic diversity of hepatitis B virus strains derived worldwide: genotypes, subgenotypes,and HBsAg subtypes[J]. Intervirology, 2004, 47(6): 289-309.
[7]ALEXOPOULOU A, DOURAKIS S P. Genetic heterogeneity of hepatitis viruses and its clinical significance [J]. Curr Drug Targets Inflamm Allergy,2005, 4(1): 47-55.
[8]AKUTA N, KUMADA H. Influence of hepatitis B virus genotypes on the response to antiviral therapies[J]. J Antimicrob Chemother, 2005, 55(2):139-142.
[9]IOANNOU G N. Chronic hepatitis B infection:a global disease requiring global strategies[J].Hepatology, 2013, 58(3): 839-843.
[10]BLUMBERG B S, GERSTLEY B J,HUNGERFORD D A, et al. A serum antigen(Australia antigen) in Down's syndrome,leukemia, and hepatitis[J]. Ann Intern Med,1967, 66(5): 924-931.
[11]BLUMBERG B S. Primary and secondary prevention of liver cancer caused by HBV[J]. Front Biosci(Schol Ed) 2010, 2: 756-763.
[12]VAN DAMME P, ZANETTI A R, SHOUVAL D,et al. Strategies for global prevention of hepatitis B virus infection[J]. Adv Exp Med Biol, 2010, 659:175-188.
[13]OKAMOTO H, TSUDA F, SAKUGAWA H, et al.Typing hepatitis B virus by homology in nucleotide sequence: comparison of surface antigen subtypes[J].J Gen Virol, 1988, 69(Pt 10): 2575-2583.
[14]NORDER H, COUROUCÉ A M, MAGNIUS L O. Complete genomes, phylogenetic relatedness,and structural proteins of six strains of the hepatitis B virus, four of which represent two new genotypes[J].Virology, 1994, 198(2): 489-503.
[15]STUYVER L, DE GENDT S, VAN GEYT C,et al. A new genotype of hepatitis B virus: complete genome and phylogenetic relatedness[J]. J Gen Virol, 2000, 81(Pt 1): 67-74.
[16]WU F, WU M J, ZHUGE X L, et al. Correlation of the occurrence of YMDD mutations with HBV genotypes, HBV-DNA levels, and HBeAg status in Chinese patients with chronic hepatitis B during lamivudine treatment[J]. Hepatobiliary Pancreat Dis Int, 2012, 11(2): 172-176.
[17]YU H, YUAN Q, GE S X, et al. Molecular and phylogenetic analyses suggest an additional hepatitis B virus genotype “Ⅰ”[J]. PLoS One, 2010, 5(2): e9297.
[18]TATEMATSU K, TANAKA Y, KURBANOV F,et al. A genetic variant of hepatitis B virus divergent from know human and ape genotypes isolated from a Japanese patient and provisionally assigned to new genotype J[J]. J Virol, 2009, 83(20): 10538-10547.
[19]OHBA K, MIZOKAMI M, OHNO T, et al.Relationships between serotypes and genotypes of hepatitis B virus: genetic classification of HBV by use of surface genes[J]. Virus Res, 1995, 39(1): 25-34.
[20]POURKARIM M R, AMINI-BAVIL-OLYAEE S,KURBANOV F, et al. Molecular identification of hepatitis B virus genotypes/subgenotypes: revised classification hurdles and updated resolutions[J].World J Gastroenterol, 2014, 20(23): 7152-7168.
[21]KAO J H, CHEN P J, LAI M Y, et al. Clinical and virological aspects of blood donors infected with hepatitis B virus genotypes B and C[J]. J Clin Microbiol, 2002, 40(1): 22-25.

