圈养及野生藏酋猴子代遗传多样性差异分析
牛丽红, 周亮, 杨帆, 吴爱明, 付国强, 候琳琳, 李霄, 窦科峰, 陈辉, 张新尚*
(1.四川省医学科学院·四川省人民医院实验动物研究所,成都610212; 2.中国人民解放军第四军医大学西京医院肝胆外科,西安710032)
藏酋猴Macacathibetana又名四川短尾猴、藏猴,隶属于灵长目Primates猴科Cercopithecidae猕猴属Macaca,是我国特有的非人灵长类物种,主要分布于中亚热带-北亚热带的四川、贵州、安徽、福建和浙江等地(蒋学龙等,1996)。藏酋猴为国家Ⅱ级重点保护野生动物,被《濒危野生动植物国际贸易公约(CITES)》列入附录Ⅱ。
藏酋猴在组织结构、生理代谢及遗传等方面与人类接近,是理想的非人灵长类实验动物,已被广泛应用于生命科学和医学研究中(王红星等,2009;杨峰等,2010)。藏酋猴体型较大,接近甚至超过异种器官移植中常用的东非狒狒Papioanubis,其器官形态、大小与小型猪Susscrofa相似,且免疫系统、凝血系统等与人类近似,已被成功应用于异种器官移植研究中(陶开山等,2017)。藏酋猴性情温顺,易驯化,可在清醒状态下进行眼压测定,其眼压昼夜节律波动及对药物(苏为坦、唆吗洛尔)的眼压反应与其他灵长类相似,表明藏酋猴是一种非常理想的眼睛疾病模型(Yietal. ,2012)。
为保护我国野生藏酋猴资源,分别于2004年和2016年从四川省马边彝族自治县和金阳县引入野生藏酋猴进行人工驯养繁殖。由于人工定向养殖会增加动物近交程度而在不同程度上造成遗传多样性的降低,同时有可能因为人工选择的作用而发生遗传分化(Notter,1999),因此,在实验动物标准化研究中,遗传学控制十分重要。圈养条件下,应避免因近亲繁殖导致的种群遗传多样性降低,遗传多样性降低会使物种环境适应能力和抗病性下降,对圈养藏酋猴种群生存产生威胁。相较于野生种群的随机交配,藏酋猴在人工繁殖时通常采用一雄配多雌的策略,但目前还没有对圈养种群子代的遗传背景及遗传多样性信息进行系统评估的研究,在一定程度上制约着藏酋猴封闭种群的建立和更广泛的应用。线粒体DNA控制区(mtDNA D-loop)作为mtDNA上最重要的非编码区,富含A、T,碱基替换率比mtDNA其他区域高5~10倍,是研究亲缘关系较近群体遗传分化的理想分子标记,被广泛应用于不同动物类群的遗传多样性和系统进化研究(Zhuetal. ,1994;谢振宇等,2006;黄雪贞等,2012)。本研究利用mtDNA D-loop全长序列对自然交配与人工选择2种不同繁殖模式下的子一代进行遗传多样性评估,为制定科学有效的繁殖计划,实现标准化遗传控制,增加圈养藏酋猴的遗传多样性和扩大种群数量提供理论指导依据。
1 材料与方法
1.1 材料
共选用102只藏酋猴个体,所有亲本及金阳地区子一代均为野外捕捉个体,包括27只金阳地区亲本个体(10岁以上)、27只金阳地区野生子一代(5岁以下)、24只马边地区亲本个体和24只马边地区亲本个体圈养繁殖子一代。采用EDTA抗凝管采集藏酋猴小隐静脉血2 mL,4 ℃保存。
1.2 方法
1.2.1基因组DNA的制备藏酋猴血液基因组DNA提取采用血液基因组DNA提取试剂盒(天根生化科技有限公司),经1.2%琼脂糖凝胶电泳和紫外分光光度法检测基因组DNA的纯度和浓度,合格样本作为PCR扩增的模板。
1.2.2mtDNAD-loop序列引物设计、合成根据藏酋猴mtDNA序列信息(GenBank登录号:EU294187),在D-loop上下游保守区Cytb基因和12S rRNA区域设计特异性引物,引物序列为:F:5’-CACTAGTCTCCCTAATCGAAAACAACCTACTC-3’,R:5’-GTGCGTGCTTGATGCTTGCTCCTCTTG-3’;引物由生工生物工程(上海)股份有限公司合成。
1.2.3PCR扩增及测序PCR反应体系为50 μL,包括50 ng基因组DNA,2×Premix LA Taq聚合酶25 μL,上下游引物各2.5 μL(10 μM),加灭菌双蒸水至50 μL。PCR扩增程序为:94 ℃预变性5 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1.5 min,30个循环;72 ℃延伸8 min,最后4 ℃保存。扩增产物用1.5%琼脂糖凝胶电泳检测片段大小及纯度。将确定为目的片段的PCR扩增产物送生工生物工程(上海)股份有限公司进行正、反向测序,测序引物与PCR扩增引物一致。
1.2.4数据分析利用Vector NTI对正、反向序列进行重叠区拼接,除去多余的碱基片段,得到D-loop全长序列。通过BLAST与GenBank中藏酋猴mtDNA序列进行比对,确认PCR扩增得到的序列是藏酋猴mtDNA D-loop序列。用DnaSP 5统计多态位点数(S)、单倍型数(H)、单倍型多样度(Hd)、核苷酸多样度(Pi)和平均核苷酸变异数(K)等参数。采用Arlequin 2.0计算群体遗传分化指数(FST)。用Kimura 2-parameter(K2P)模型计算遗传距离,并使用MEGA6构建邻接(NJ)树。
2 结果
2.1 藏酋猴mtDNA D-loop序列的碱基组成
通过对测序结果分析,去掉Cytb和12S rRNA区域序列,最终获得了D-loop全长序列1 091 bp,其中,T、C、A、G的平均含量分别为25.8%、31.4%、30.2%、12.6%,A+T和C+G的平均含量分别为56.0%和44.0%(表1)。
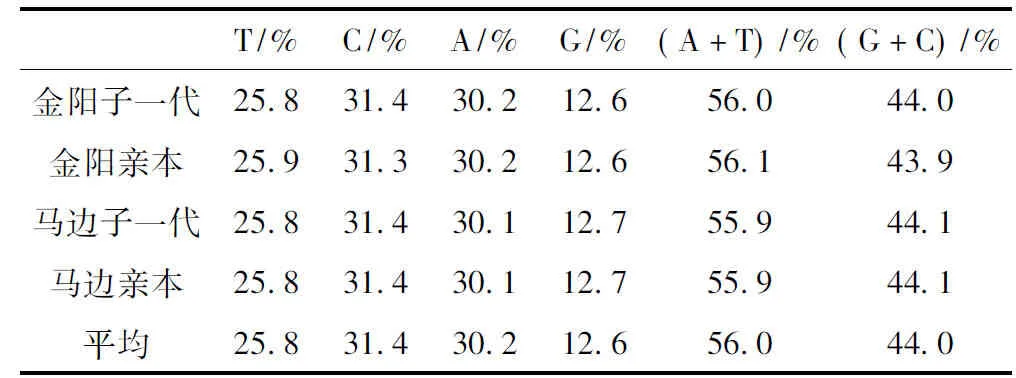
表1 不同群体藏酋猴mtDNA D-loop全长序列的碱基组成Table 1 Base composition of mtDNA D-loop indifferent groups of Macacca thibetana
2.2 藏酋猴不同群体及子代的遗传结构
102个序列共检测出115个变异位点,其中,转换96次、颠换19次,转换/颠换为5.1,插入或缺失位点2个(表2)。
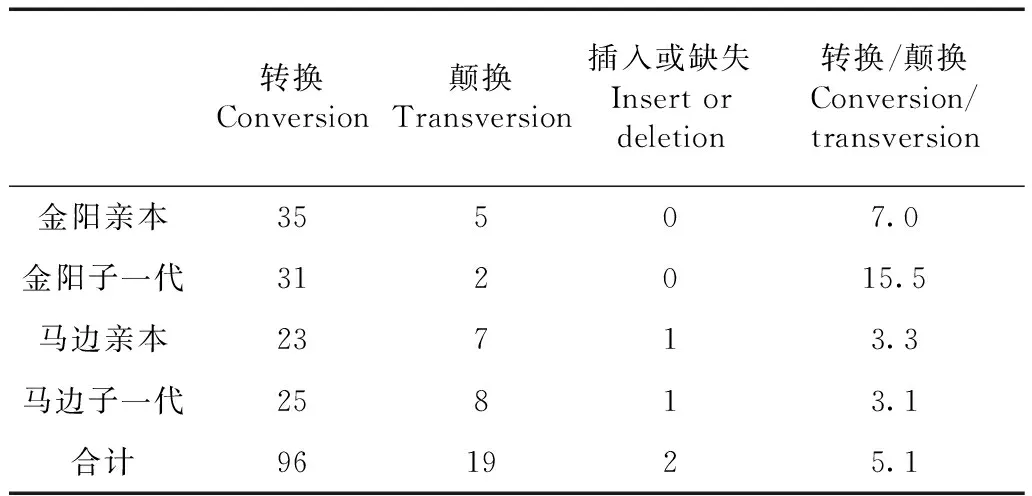
表2 不同群体藏酋猴mtDNA D-loop序列变异情况Table 2 Variation of mtDNA D-loop in different groupsof Macacca thibetana
102只个体共检测出66个单倍型,其中,金阳种群亲本20个,子一代16个,二者共享1个单倍型;马边种群亲本18个,子一代15个,二者共享 2个单倍型;马边种群与金阳种群无共享单倍型。2个种群亲本及其子一代的Hd为0.812~0.975(表3)。在不同世代之间,金阳子一代的Pi略低于亲本,而马边子一代的Pi略高于亲本,但差异无统计学意义,均处于较低水平。而对于藏酋猴不同地理种群而言,马边地区的Pi高于金阳地区。

表3 不同群体藏酋猴的遗传多样性参数Table 3 Genetic diversity parameters of D-loop indifferent groups of Macacca thibetana
藏酋猴2个种群亲本及其子一代D-loop序列的遗传距离见表4,金阳子一代与亲本之间的遗传距离为0.002 55,马边子一代与亲本之间的遗传距离为0.024 09,均未见明显的遗传分化。而马边地区和金阳地区之间的遗传距离均大于0.05,说明2个地区的藏酋猴群体发生了中度遗传分化。
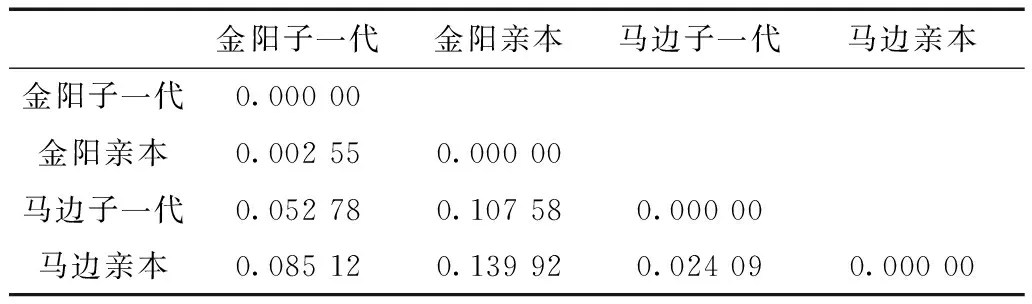
表4 藏酋猴2个种群间的遗传分化指数Table 4 Genetic differentiation between thetwo Macacca thibetana populations
2.3 藏酋猴种群的系统进化树
根据双参数法,结点的置信水平为1 000,计算了102只个体的遗传距离,基于该距离建立了藏酋猴2个地理种群亲本及其子一代的NJ树(图1),金阳子一代(001~027)与亲本(028~054)之间或马边子一代(055~078)与亲本(079~102)之间均未形成明显的分支,在进化树上互相交叉;而金阳地区(001~054)和马边地区(055~102)的个体出现了明显的分支。
3 讨论
藏酋猴在我国的地理分布范围比较广,仅次于猕猴Macacamulatta,但随着近现代人为活动的加剧,生境碎片化导致藏酋猴种群数量下降(李生强等,2017)。为保护我国野生藏酋猴资源,开始圈养藏酋猴。但圈养种群人为设定藏酋猴的繁殖单元(一雄配多雌),如果不通过遗传控制管理,容易造成近亲繁殖,影响种群的遗传多样性水平。

图1 基于mtDNA D-loop序列构建的藏酋猴群体间的邻接树Fig. 1 Neighbor-Joining tree based on the mtDNA D-loop sequences of Macacca thibetana
001~027. 金阳子一代, 028~054. 金阳亲本, 055~078. 马边子一代, 079~102. 马边亲本
001-027. F1of Jinyang population, 028-054. parents in Jinyang population, 055-078. F1of Mabian population, 079-102. parents in Mabian population
3.1 藏酋猴不同地理种群的遗传多样性
本研究基于mtDNA D-loop全长序列,选取S、H、Hd、Pi和K等参数研究了马边、金阳2个地理种群亲本及其子一代的遗传多样性。Hd和Pi是mtDNA中用来衡量种群遗传变异及遗传多样性的重要指标,由于Pi考虑了各单倍型在群体中所占的比例,因而较Hd可靠(Zhouetal. ,2006)。分析结果表明,金阳和马边地区藏酋猴种群均处于单倍型多态性较高而核苷酸多态性较低的水平,这与贵州和云南地区藏酋猴种群遗传多样性的分析结果一致(Zhongetal. ,2013),推测藏酋猴种群可能正从更小的有效种群经历种群扩张,所以它们拥有足够的时间通过突变来积累单倍型多态性,但却不足以增加核苷酸多态性。
3.2 野生与圈养条件下子一代的遗传多样性比较
目前,关于藏酋猴遗传多样性的报道还十分有限。Li等(2008)利用mtDNA D-loop的部分序列(476 bp)对四川和安徽黄山的藏酋猴进行了遗传多样性分析,认为这2个种群出现了明显遗传分化,是2个独立的种群,并指出是由地理隔离导致的。姚永芳等(2013)利用mtDNA D-loop的部分序列(505 bp)对四川地区(峨眉、马边)和安徽黄山藏酋猴种群的遗传多样性进行分析,表明不同地区的藏酋猴种群均存在不同程度的遗传分化,且种群间基因交流贫乏。同样的现象在云南地区和贵州地区的藏酋猴种群中也有发现(Zhongetal. ,2013)。由于野外采样的不确定性,目前还没有对同一种群来源的不同世代间的藏酋猴群体遗传多样性分析研究的报道。本研究首次分析了藏酋猴不同世代之间的群体遗传变异情况,同时采用mtDNA D-loop全长序列对藏酋猴不同群体进行分析,使结果更为可靠。金阳地区藏酋猴子一代为野生繁殖后代,而马边地区子一代为圈养条件下人工繁殖后代,通过这2个群体与亲本群体的比较分析,有利于对比自然条件下和人工繁殖条件下子代群体遗传多样性的变化。相较于自然条件下的随机交配,藏酋猴在人工繁殖的过程中多采用一雄配多雌的策略,且有多个繁殖单元,在遗传背景信息不全的情况下,可能出现近交机率增加,有效种群数目减少,导致遗传多样性降低,遗传效应减弱,造成遗传瓶颈。本研究发现,金阳地区藏酋猴子一代Pi与亲本间并无显著变化,马边地区子一代Pi也无显著降低,金阳或马边地区不同世代的藏酋猴个体在NJ树上互相交叉,各世代未形成独有的进化支,说明无论是自然繁殖还是人工繁殖,藏酋猴的子一代依然能够保持与亲本相当的遗传多样性水平。虽然在目前繁殖方式下,藏酋猴子代的遗传多样性得到了保存,但藏酋猴整体的核苷酸多样性还较低,在以后的繁育过程中,可利用更多的分子标记对藏酋猴进行遗传多样性及家系的遗传管理,进一步提高种群的遗传多样性水平。
本研究利用藏酋猴mtDNA D-loop全长序列分析了野外繁殖的亲本与子一代、人工繁殖的亲本与子一代以及子一代间的遗传差异,建立了藏酋猴实验动物标准化遗传控制体系,为科学有效地制定繁殖计划、增加圈养藏酋猴种群的遗传多样性和扩大种群数量提供理论指导依据。
:
黄雪贞, 钱国英, 李彩燕. 2012. 中华鳖3个地理群体线粒体基因D-loop区遗传多样性分析[J]. 水产学报, 36(1): 18-24.
蒋学龙, 王应祥, 王歧山. 1996. 藏酋猴的分类与分布[J]. 动物学研究, 17(4): 361-369.
李生强, 汪国海, 施泽攀, 等. 2017. 广西藏酋猴种群数量、分布及威胁因素的分析[J]. 广西师范大学学报(自然科学版), 35(2): 126-132.
陶开山, 李霄, 窦科峰. 2017. 异种肝移植的历史与发展[J]. 器官移植, 8(2): 86-88.
王红星, 姚方, 周亮. 2009. 藏酋猴人工繁殖研究概况[J]. 中国科技成果, 23: 38-40.
谢振宇, 杜继曾, 陈学群, 等. 2006. 线粒体控制区在鱼类种内遗传分化中的意义[J]. 遗传, 28(3): 362-368.
杨锋, 王红星, 周亮, 等. 2010. 藏酋猴外周血细胞及血清生化指标测定与分析[J]. 四川动物, 29(6): 1000-1001.
姚永芳, 钟丽菁, 刘家斌, 等. 2013. 峨眉山藏酋猴mtDNA控制区序列变异及种群遗传多样性[J]. 四川动物, 32(6): 801-807.
Li D, Fan L, Ran J,etal. 2008. Genetic diversity analysis ofMacacathibetanabased on mitochondrial DNA control region sequences[J]. Mitochondrial DNA, 19(5): 446-452.
Notter DR. 1999. The importance of genetic diversity in livestock populations of the future[J]. Journal of Animal Science, 77(1): 61-69.
Yi Y, Zeng T, Zhou L,etal. 2012. Expreimental Tibetan monkey domestication and its application for intraocular pressure measurement[J]. International Journal of Ophthalmology, 5(3): 277-280.
Zhong LJ, Zhang MW, Yao YF,etal. 2013. Genetic diversity of two Tibetan macaque (Macacathibetana) populations from Guizhou and Yunnan in China based on mitochondrial DNA D-loop sequences[J]. Genes & Genomics, 35(2): 205-214.
Zhou H, Li DQ, Zhang YG,etal. 2006. Study on mitochondrial DNA genetic diversity of Tibetan antelope[J]. Heredias, 28(3): 299-305.
Zhu D, Jamikson BG, Hugall A,etal. 1994. Sequence evolution and phylogenetic signal in control-region and cytochrome b sequences of rainbow fishes (Melanotaeniidae)[J]. Molecular Biology & Evolution, 11(4): 672-683.

